2. 国家民委青藏高原动物疫病防控创新团队, 成都 610041
2. Innovation Team for Animal Epidemic Diseases Prevention and Control on Qinghai-Tibet Plateau, State Ethnic Affairs Commission, Chengdu 610041, China
Newbury-1 virus目前是杯状病毒科(Caliciviridae)纽布病毒属(Nebovirus)的唯一成员(https://talk.ictvonline.org/),常用Nebovirus(NeV)表述。NeV为无囊膜的单股正链RNA病毒,是致犊牛腹泻的重要病毒[1]。不像杯状病毒科的诺如病毒属(Norovirus)和兔病毒属(Lagovirus)[2-3],NeV尚未建立基于RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase, RdRp)和主要衣壳蛋白(major capsid protein, VP1)的双重命名系统。因此,仍然分别基于部分RdRp或完整VP1序列描述NeV的分子特征。根据RdRp的核苷酸序列可将NeV分为3种基因型(NB-like、NA1-like和SC-Yak)[4];根据完整VP1氨基酸序列可将NeV分为3种基因型(基因1、2和3型),其中基因1型又可进一步分为4个谱系(谱系1~4)[4-5]。尽管目前尚无NeV的分离培养体系,但用排除了其他病毒的含有NeV的腹泻粪便除菌处理后接种,可引起剖腹产且隔离饲养的小牛严重腹泻[6-7]。2018年,作者实验室首次证实NeV在我国的存在[8],随后我们又证实该病毒在我国奶牛和牦牛中广泛流行[4, 9-10]。目前,NeV已经在中国、英国、美国、韩国、意大利等13个国家检出[11-20],其危害也受到越来越广泛的重视。本文就NeV的生物学特性、流行概况、所致疾病的临床症状与病理变化、检测方法和防控措施等做一综述。
1 NeV的生物学特性 1.1 病原分类Nebovirus是杯状病毒科(Caliciviridae)纽布病毒属(Nebovirus)成员。杯状病毒科其他成员包括诺如病毒属(Norovirus)、札幌病毒属(Sapovirus)、水疱疹病毒属(Vesivirus)、兔病毒属(Lagovirus)等(https://talk.ictvonline.org/)。所有杯状病毒科的成员之间均有相似的基因组结构,但是不同病毒属之间的遗传进化和抗原性显著不同[21]。NeV与兔病毒属成员的亲缘关系相对较近,如兔出血病病毒(rabbit hemorrhagic disease virus)和欧洲野兔综合征病毒(European brown hare syndrome virus)[21-22]。
1.2 基因组结构及蛋白NeV的线性RNA基因组为7 453~7 460 bp,有2个开放阅读框(ORF1和ORF2)、两个非编码区(5′-TUR和3′-TUR)和一个多聚腺苷酸尾(polyA)(图 1)[4, 10, 21]。ORF1长度为2 210~2 211个氨基酸(amino acid, aa),依次编码NH2-terminal(P34)、2C-解旋酶(NTPase)、3A(P30)、3B(VPg)、3C-蛋白酶(3CLpro)、RdRp和VP1;ORF2编码225个aa,编码次要衣壳蛋白(minor capsid protein, VP2)。ORF1和ORF2之间有1个核苷酸间隔。5′-TUR有74~80 bp,3′-TUR有66~67 bp[4, 10, 22]。RdRp和VP1常用于NeV的基因分型,但NeV其他蛋白的结构与功能鲜见报道。
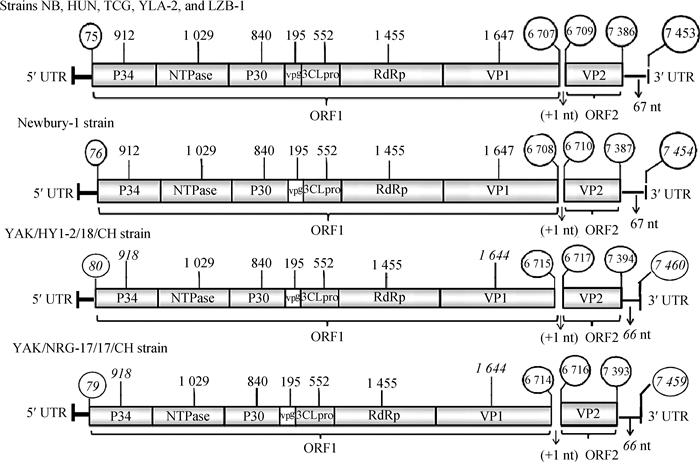
|
NeV毒株之间的差异用斜体数字表示 The difference among all NeV strains were showed with italic numbers 图 1 NeV的基因组结构示意 Fig. 1 Diagram of NeV genome structure |
目前,关于NeV RdRp的结构与功能的研究尚未见报道。对诺如病毒、札幌病毒和兔病毒等其他杯状病毒的研究表明,RdRp在杯状病毒的复制过程中起着关键作用[23-25]。NeV完整的RdRp基因长度均为1 455 bp,编码485 aa。已有的8个完整RdRp序列之间的核苷酸相似性和氨基酸相似性分别为66.1%~90.0%和74.2%~99.0%。像其他杯状病毒一样,NeV的RdRp 3′端核苷酸序列常用于遗传进化分析[14-15, 19, 26-27]。尽管目前缺乏足够的NeV序列来计算NeV基因型分类的遗传距离截止值,但基于已有的158株NeV毒株的RdRp 3′端序列(≥396 bp)遗传进化分析表明,NeV可以进一步分为NB-like、NA1-like和SC-Yak 3种基因型(图 2)[4]。当前我国NeV的流行毒株有NB-like和SC-Yak两个基因型,其中NB-like为主要基因型[4, 9]。有趣的是,我国奶牛NeV毒株和NB-like基因型的牦牛NeV毒株共同聚为一个独立的大支,区别于其他已知的NB-like基因型毒株[4];而SC-Yak基因型仅在牦牛中检出[4]。序列分析显示,NA1-like基因型之间的核苷酸相似性为78.5%~ 99.7%,NB-like基因型之间的核苷酸相似性为82.5%~100.0%,SC-Yak基因型之间的核苷酸相似性为87.1%~100.0%;而不相同基因型之间的核苷酸相似性为61.8%~80.8%。前期研究表明,与非大流行和较少检测到的毒株相比,引起大流行的诺如病毒毒株(GII.4)RdRp显示出较低的复制保真度,这导致更高的突变率和病毒进化速率[28]。因此,不同的NeV RdRp基因型流行情况值得进一步关注。

|
“YAK”表示我国69株牦牛源NeV毒株,黑色三角形和黑色方框分别表示我国牦牛源和奶牛源的NeV毒株。这69株牦牛源NeV毒株的GenBank登录号为MK452017~MK452020、MK452022~MK452026、MK452028~MK452070和MK452075~MK452095 "YAK"represents 69 NeVs strains from yak in China, and black triangle and black box represents NeVs strains from yak and dairy cows in China, respectively. These 69 NeVs partial RdRp sequence were deposited in the GenBank database under accession No. MK452017-MK452020, MK452022-MK452026, MK452028-MK452070, and MK452075-MK452095 图 2 部分RdRp基因片段(396 bp)的最大似然法进化树 Fig. 2 The maximum-likelihood phylogenetic tree based on partial RdRp gene fragment (396 bp) |
VP1是杯状病毒的主要结构蛋白,参与病毒与宿主细胞受体识别、宿主特异性、抗原性和免疫原性[29-31]。最近研究表明,NeV MA415-KOR毒株(VP1基因1型谱系3)衣壳蛋白的VLPs能与HBGAs结合,证实NeV衣壳蛋白参与受体识别[1]。值得注意的是,该研究显示NeV的VLPs具有广谱的HBGAs,并可与人的部分HBGAs结合,可能具有感染人的潜力[1]。目前,GenBank中登录了62条完整的VP1序列,其大小为1 647或1 644 bp,编码549或548 aa。尽管缺乏足够的VP1序列来计算基因型分类的遗传距离截止值,但基于已有的62条NeV完整VP1序列的遗传进化分析表明,VP1可以进一步分为基因1型、基因2型和基因3型,其中基因1型又分为4个VP1谱系(谱系1~4)(图 3)。目前,在GenBank中有30条国内NEV VP1序列,其中27株为VP1基因1型(21株为谱系1、4株为谱系3、2株为谱系4)、1株为基因2型、2株为基因3型,表明我国NeV VP1具有丰富的遗传多样性。基因1型之间的核苷酸和氨基酸相似性分别为83.0%~100.0%和88.4%~100.0%,基因2型之间的核苷酸相似性和氨基酸相似性分别为87.9%和96.5%,基因3型之间的核苷酸相似性和氨基酸相似性分别为91.2%和96.4%;而不相同基因型之间的核苷酸相似性和氨基酸相似性分别为68.0%~72.3%和69.5%~78.5%。

|
国内NeV毒株用方框表示 NeV strains from China were showed with box 图 3 完整VP1蛋白氨基酸序列的最大似然法进化树 Fig. 3 The maximum-likelihood phylogenetic tree based on complete VP1 protein amino scid sequence |
目前,GenBank数据库中有8个NeV基因组,其中英国、美国、日本和匈牙利各登录了1个(登录号为DQ013304.1、AY082891.1、AB117797.2和JX018212.1),作者实验室登录了4个(登录号为MG599036.1、MH718886.1、MK452012.1和MK452013.1),毒株具体的背景信息见图 4。8个NeV基因组之间的核苷酸相似性为68.1%~90.2%。其中,2株牦牛源NeV基因组之间的核苷酸相似性为90.2%,与其余6个NeV基因组之间的核苷酸相似性仅为68.1%~69.3%;其余6株奶牛源和肉牛源NeV毒株基因组之间的核苷酸相似性为82.0%~88.7%。基因组的遗传进化树显示,2株牦牛源NeV毒株共同聚为一个独立大支,与其他NeV毒株(包括国内奶牛NeV毒株)遗传关系较远(图 4);此外,基于P34、NTPase、P30、VPg、3CLpr、RdRp、VP1和VP2的遗传进化树均显示,2株牦牛源NeV毒株均共同聚为一个独立大支[4],表明牦牛源NeV基因组与其他NeV基因组相比有较大的异质性,有必要进一步研究这种牦牛源NeV的起源和进化。
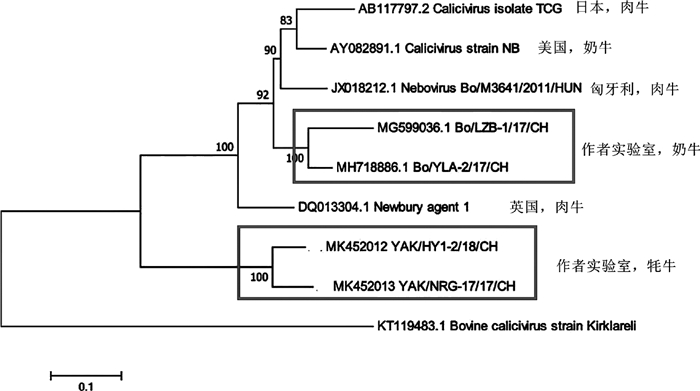
|
国内NeV毒株用方框表示, 小牛中鉴定的Kirklareli virus(Kirklareli毒株)被认为与NeV相关[32] NeV strains from China were showed with box. Kirklareli virus (Kirklareli strain) was found in calves and may be related to the genus Nebovirus[32] 图 4 NeV和Kirklareli病毒的遗传进化树 Fig. 4 Phylogenetic tree based on the complete genomic sequence of NeV and Kirklareli virus |
NeV的进化有几种表现:①氨基酸突变,特别是VP1易于发生。②VP1和RdRp进化不一致:如国内的Bo/LZB-1/CH/17毒株和Bo/YLA-2/17/CH毒株,这2个毒株的RdRp均为NB-like基因型,而Bo/LZB-1/CH/17毒株的VP1为基因2型,Bo/YLA-2/17/CH毒株的VP1为基因1型谱系3。③基因重组,目前GenBank登陆的NeV序列中,有27毒株被鉴定为重组毒株[9-10, 12],其中26株重组毒株(包括8株国内NeV毒株)的重组位点均位于VP1[9, 12],仅有1株来自国内NeV的重组毒株的重组位点位于RdRp 3′端[10]。重组的形式主要表现为单断点重组和区域重组。氨基酸突变的累积和基因重组使得杯状病毒具有丰富的遗传多样性,甚至进化出新型变异株从而引起流行[33-37]。关于诺如病毒的研究表明,重组可以交换病毒VP1而保留参与病毒基因组复制的RdRp来逃避群体免疫[27, 37-38]。
1.4 抗原性和其他杯状病毒一样[39-40],NeV VP1是感染过程中诱导抗体产生的重要蛋白[41],但目前关于NeV抗原性研究的报道还很少。根据完整VP1的氨基酸序列,NeV可以分为3种基因型(基因1、2和3型)[4],不同基因型之间氨基酸的差异较大(特别是P2结构域),因此有必要进一步研究不同基因型的抗原性差异。
1.5 培养特性目前,除了猪札幌病毒参考毒株Cowden[42]、鼠诺如病毒[43]和人诺如病毒[44]能在体外分离培养,其他肠道杯状病毒(包括NeV)尚没有体外分离培养体系。杯状病毒难以分离的主要原因有以下两点:首先是合适的细胞系;例如,人诺如病毒(HuNoVs)体外培养的早期研究都集中在肠道上皮细胞[45],但都未成功。最近研究证实,HuNoVs能在B细胞系[46]和干细胞衍生的人肠道类器官[47]中进行体外感染和复制。其次,病毒培养所需的辅助因子对病毒分离的影响也很大。例如,在HuNoVs感染B细胞系时,需要游离H抗原或者表达HBGA肠菌群的参与[48]。在HuNoVs感染干细胞人肠道上皮培养物,GII.10基因型的复制需要用胆酸处理肠状单层细胞,然而GII.4基因型的复制不需要用胆酸处理[49]。
1.6 理化特性由于尚无NeV的分离培养体系,因此NeV的理化性质并不清楚。来自杯状病毒科其他成员的研究表明,杯状病毒在环境中稳定,耐酸和耐热,对醚和氯仿具有抵抗力。
2 流行概况病牛和带毒牛均是传染源,其粪便中含有大量病毒。NeV的传播途径是粪-口途径。目前,NeV的易感动物只有牛,包括肉牛、奶牛和牦牛[4, 7-8]。主要感染3月龄以内犊牛,无明显的季节性和地域性[4, 6-7]。
2.1 国外流行概况迄今为止,NeV已经在美国、英国、巴西、土耳其等13个国家检出,地域涵盖北美洲、南美洲、欧洲、亚洲、非洲;国外的检出率为3.0%~25.2%[6, 11-16]。目前,RdRp的NB-like基因型NeV为世界流行的基因型,在中国、美国、英国、韩国、日本、法国、摩洛哥、意大利、匈牙利、突尼斯、土耳其和伊朗均有检出[6, 9, 13-15, 18];NA1-like毒株在英国、巴西、伊朗和法国检出[11, 14, 19, 22];而SC-Yak基因型仅在中国牦牛中检出[4]。另外,VP1的基因1型为世界流行的主要基因型;基因1.1型(基因1型谱系1)已经在美国、匈牙利、英国、法国、意大利和中国检出[6, 9, 12, 14, 20]; 基因1.2型在美国和日本检出[6]; 基因1.3型在中国和韩国检出[9, 18]; 基因1.4型在英国、法国、意大利、土耳其和中国检出[12, 14, 22];基因2型在中国和法国检出[9, 14];基因3型仅在中国牦牛中检出[4]。
2.2 国内流行概况2018年,作者实验室首次证实NeV在中国的存在[8]。进一步调查表明,NeV在新疆维吾尔自治区、四川省、辽宁省、河南省、山东省和陕西省的奶牛中广泛流行;腹泻样本中检出率为48.1%,显著高于健康粪便样本的检出率(5.7%,P < 0.001),表明该病毒与我国奶犊牛腹泻密切相关[9]。混合感染调查结果显示,91.2%的NeV阳性腹泻样本与牛轮状病毒(bovine rotavirus, BRV)、牛冠状病毒(bovine coronavirus, BCoV)和/或牛病毒性腹泻病毒(bovine viral diarrhea virus, BVDV)混合感染,可能增加临床表现的严重程度,给犊牛腹泻的诊断带来困难。目前,NB-like基因型为国内流行的RdRp基因型,其他基因型均未检出[9];而国内奶牛NeV的VP1具有丰富的遗传多样性,包括基因1.1型、基因1.3型、基因1.4型和基因2型,其中主要基因型为基因1.1型[9]。
2019年,作者实验室首次证实NeV在牦牛中存在,是牦牛腹泻的新发病毒[4]。NeV在西藏自治区、四川省和云南省的牦牛腹泻粪便样本中均检出,平均阳性率为22.0%,场阳性率为69.1%[4];87.2%的NeV阳性腹泻样本与BRV、BCoV和/或BVDV混合感染,与国内奶牛NeV混和感染结果相似。目前,牦牛NeV具有2种RdRp基因型,其中NB-like基因型为主要基因型,还有新RdRp基因型“SC-Yak”[4]。在四川省的红原县和诺尔盖县的牦牛腹泻样本中鉴定了一种具有RdRp SC-Yak基因型和VP1基因3型的新型NeV毒株,并且这种新型NeV毒株已经在当地牦牛群中流行[4]。
3 所致疾病的临床症状与病理变化NA1-like和NB-like基因型是最早确定的RdRp基因型,其原型毒株分别为Bo/Newbury1/76/UK(NA1)和Bo/Nebraska/80/US(NB)。排除了其他病毒的含NA1或NB毒株的腹泻粪便样本经无菌处理后,口服或静脉接种1~42日龄的剖腹产且隔离饲养的小牛,1~3 d后均出现精神沉郁、厌食、木糖吸收不良、腹泻,粪便颜色从正常浓稠的棕绿色糊状物先变成稀薄的棕褐色糊状物,之后变为具有絮状碎片的黄色液体[6-7]。病理变化均局限于小肠,其中十二指肠和空肠最严重;组织病理学表现为肠绒毛萎缩、上皮细胞空泡变性和嗜碱性粒细胞增多[6-7]。感染NA1和NB毒株的小牛临床表现和病理变化相似[6-7]。
4 NeV的检测 4.1 病原检测由于缺乏NeV的分离培养体系,该病毒的病原检测主要依靠RT-PCR和Real-time RT-PCR方法。目前有5篇关于检测NeV的RT-PCR方法的报道和2篇关于检测NeV的Real-time RT-PCR方法的报道[4, 8, 16, 50-53],其中6篇报道的检测引物均靶向RdRp的3′端,1篇报道的检测引物靶向RdRp的3′端和VP1的5′端。与Real-time RT-PCR方法相比,RT-PCR方法的引物所扩增片段较长,既能用于检测又能用于遗传进化分析;但是RT-PCR方法的灵敏度低于Real-time RT-PCR方法,且不能用于定量检测。
值得注意的是,国外报道的RT-PCR和Real-time RT-PCR方法对国内NeV毒株的检出效果并不理想[53],其可能的原因是国内NeV毒株在国外检测引物的扩增位点上具有不同程度的点突变[53],同时还有新基因型的存在[4]。因此,我们重新设计引物并建立了RT-PCR和Real-time RT-PCR方法[4, 53],大大提高了国内NeV的检出率。
4.2 基于酶联免疫吸附试验的抗体检测技术目前,关于NeV抗体检测技术仅见1篇文献报道,即Thomas等[41]利用昆虫细胞-杆状病毒表达系统构建了NeV VP1的VLPs,通过CsCl梯度超速离心纯化VLPs后将其作为特异性抗原建立的检测抗体的间接ELISA方法,并对1999—2001年期间美国不同地区的小牛进行了血清流行病学调查,发现不同地区牛群的NeV感染率为78%~100%,平均感染率为92% (263/287)。
5 防控措施目前,尚无针对NeV的疫苗,也没有特异性的治疗抗体。由于病牛和带毒牛的粪便是主要的传染源,并且该病毒在环境中比较稳定。因此,隔离病牛和带毒牛,做好环境消毒(粪便的清洁和圈舍消毒等)是预防该病的重要措施。常见的含氯消毒药对该病毒都是敏感的。对于病牛和脱水严重的牛要及时补液,调节酸碱平衡和电解质平衡。
6 问题与展望作为一个致犊牛腹泻的病毒,有关NeV的基础研究和应用研究还很薄弱。首先,病毒的分离鉴定是病毒生物学特性和疫苗研究的基础,而目前还没有NeV分离成功的报道。因此,NeV的分离培养成为该病毒研究最迫切需要解决的问题。第二,杯状病毒VP1参与病毒与宿主细胞受体识别、抗原性和免疫原性。NeV VP1具有不同的基因型,不同基因型诱导中和抗体能力及其交叉保护性有待于进一步研究。第三,流行病学是疫病防控的基础,应进一步加强NeV的分子流行病学调查。第四,在没有NeV成功分离培养的情况下,亚单位疫苗的研制可能是一个重要的方向。
7 结论NeV是国内新发现的致犊牛腹泻病毒,已在我国奶牛和牦牛中广泛流行,具有独特的进化趋势,且有新基因型毒株的出现。因此,进一步加强对国内NeV的病原生物学、流行病学、诊断和防控技术等研究对于犊牛腹泻的防控具有重要意义。
| [1] | CHO E H, SOLIMAN M, ALFAJARO M M, et al. Bovine nebovirus interacts with a wide spectrum of histo-blood group antigens[J]. J Virol, 2018, 92(9): e02160–17. |
| [2] | KRONEMAN A, VEGA E, VENNEMA H, et al. Proposal for a unified norovirus nomenclature and genotyping[J]. Arch Virol, 2013, 159(10): 2059–2068. |
| [3] | LE PENDU J, ABRANTES J, BERTAGNOLI S, et al. Proposal for a unified classification system and nomenclature of lagoviruses[J]. J Gen Virol, 2017, 98(7): 1658–1666. DOI: 10.1099/jgv.0.000840 |
| [4] | GUO Z J, HE Q F, ZHANG B, et al. First detection of neboviruses in Yak (Bos grunniens) and identification of a novel neboviruses based on complete genome[J]. Vet Microbiol, 2019, 236: 108388. DOI: 10.1016/j.vetmic.2019.108388 |
| [5] | D'MELLO F, JERVIS S M, EDWARDS P M, et al. Heterogeneity in the capsid protein of bovine enteric caliciviruses belonging to a new genus[J]. Virology, 2009, 387(1): 109–116. DOI: 10.1016/j.virol.2009.01.035 |
| [6] | SMILEY J R, CHANG K O, HAYES J, et al. Characterization of an enteropathogenic bovine calicivirus representing a potentially new calicivirus genus[J]. J Virol, 2002, 76(20): 10089–10098. DOI: 10.1128/JVI.76.20.10089-10098.2002 |
| [7] | BRIDGER J C, HALL G A, BROWN J F. Characterization of a calici-like virus (Newbury agent) found in association with astrovirus in bovine diarrhea[J]. Infect Immun, 1984, 43(1): 133–138. DOI: 10.1128/IAI.43.1.133-138.1984 |
| [8] | GUO Z J, HE Q F, YUE H, et al. First detection of nebovirus and norovirus from cattle in China[J]. Arch Virol, 2018, 163(2): 475–478. DOI: 10.1007/s00705-017-3616-6 |
| [9] | GUO Z J, HE Q F, ZHANG B, et al. Detection and molecular characteristics of neboviruses in dairy cows in China[J]. J Gen Virol, 2019, 100(1): 35–45. |
| [10] | GUO Z J, HE Q F, YUE H, et al. Genomic characterization of a RdRp-recombinat nebovirus strain with a novel VP1 genotype[J]. Virus Res, 2018, 251: 6–13. DOI: 10.1016/j.virusres.2018.04.016 |
| [11] | CANDIDO M, ALENCAR A L F, ALMEIDA-QUEIROZ S R, et al. First detection and molecular characterization of Nebovirus in Brazil[J]. Epidemiol Infect, 2016, 144(9): 1876–1878. DOI: 10.1017/S0950268816000029 |
| [12] | DI MARTINO B, DI PROFIO F, MARTELLA V, et al. Evidence for recombination in neboviruses[J]. Vet Microbiol, 2011, 153(3-4): 367–372. DOI: 10.1016/j.vetmic.2011.05.034 |
| [13] | HASSINE-ZAAFRANE M, KAPLON J, SDIRI-LOULIZI K, et al. Molecular prevalence of bovine noroviruses and neboviruses detected in central-eastern Tunisia[J]. Arch Virol, 2012, 157(8): 1599–1604. DOI: 10.1007/s00705-012-1344-5 |
| [14] | KAPLON J, GUENAU E, ASDRUBAL P, et al. Possible novel nebovirus genotype in cattle, France[J]. Emerg Infect Dis, 2011, 17(6): 1120–1123. DOI: 10.3201/eid/1706.100038 |
| [15] | KARAYEL-HACIOGLU I, ALKAN F. Molecular characterization of bovine noroviruses and neboviruses in Turkey:detection of recombinant strains[J]. Arch Virol, 2019, 164(5): 1411–1417. DOI: 10.1007/s00705-019-04186-0 |
| [16] | TURAN T, IŞIDAN H, ATASOY M O, et al. Detection and molecular analysis of bovine enteric norovirus and nebovirus in Turkey[J]. J Vet Res, 2018, 62(2): 129–135. DOI: 10.2478/jvetres-2018-0021 |
| [17] | CHO Y I, HAN J I, WANG C, et al. Case-control study of microbiological etiology associated with calf diarrhea[J]. Vet Microbiol, 2013, 166(3-4): 375–385. DOI: 10.1016/j.vetmic.2013.07.001 |
| [18] | PARK S I, JEONG C, PARK S J, et al. Molecular detection and characterization of unclassified bovine enteric caliciviruses in South Korea[J]. Vet Microbiol, 2008, 130(3-4): 371–379. DOI: 10.1016/j.vetmic.2008.01.017 |
| [19] | POURASGARI F, KAPLON J, SANCHOOLI A, et al. Molecular prevalence of bovine noroviruses and neboviruses in newborn calves in Iran[J]. Arch Virol, 2018, 163(5): 1271–1277. DOI: 10.1007/s00705-018-3716-y |
| [20] | REUTER G, KÁTAI A, KÁLMÁN M, et al. First detection of human calicivirus infection in Hungary[J]. Orv Hetil, 2000, 141(38): 2071–2074. |
| [21] | DESSELBERGER U. Caliciviridae other than noroviruses[J]. Viruses, 2019, 11(3): E286. DOI: 10.3390/v11030286 |
| [22] | OLIVER S L, ASOBAYIRE E, DASTJERDI A M, et al. Genomic characterization of the unclassified bovine enteric virus Newbury agent-1 (Newbury1) endorses a new genus in the family Caliciviridae[J]. Virology, 2006, 350(1): 240–250. DOI: 10.1016/j.virol.2006.02.027 |
| [23] | SMERTINA E, URAKOVA N, STRIVE T, et al. Calicivirus RNA-Dependent RNA Polymerases: evolution, structure, protein dynamics, and function[J]. Front Microbiol, 2019, 10: 1280. DOI: 10.3389/fmicb.2019.01280 |
| [24] | DEVAL J, JIN Z N, CHUANG Y C, et al. Structure(s), function(s), and inhibition of the RNA-dependent RNA polymerase of noroviruses[J]. Virus Res, 2017, 234: 21–33. DOI: 10.1016/j.virusres.2016.12.018 |
| [25] | LEE J H, CHUNG M S, KIMM K H. Structure and function of Caliciviral RNA polymerases[J]. Viruses, 2017, 9(11): E329. DOI: 10.3390/v9110329 |
| [26] | OKA T, WANG Q H, KATAYAMA K, et al. Comprehensive review of human sapoviruses[J]. Clin Microbiol Rev, 2015, 28(1): 32–53. DOI: 10.1128/CMR.00011-14 |
| [27] | DI FELICE E, MAUROY A, DAL POZZO F, et al. Bovine noroviruses: a missing component of calf diarrhoea diagnosis[J]. Vet J, 2016, 207: 53–62. DOI: 10.1016/j.tvjl.2015.10.026 |
| [28] | BULL R A, EDEN J S, RAWLINSON W D, et al. Rapid evolution of pandemic noroviruses of the GII. 4 lineage[J]. PLoS Pathog, 2010, 6(3): e1000831. DOI: 10.1371/journal.ppat.1000831 |
| [29] | SMITH H Q, SMITH T J. The dynamic capsid structures of the Noroviruses[J]. Viruses, 2019, 11(3): 235. DOI: 10.3390/v11030235 |
| [30] | HANSMAN G S, NATORI K, SHIRATO-HORIKOSHI H, et al. Genetic and antigenic diversity among noroviruses[J]. J Gen Virol, 2006, 87(4): 909–919. DOI: 10.1099/vir.0.81532-0 |
| [31] | ZHANG G B, WANG J, LIU J J, et al. The surface-exposed loop region of norovirus GII. 3 VP1 plays an essential role in binding histo-blood group antigens[J]. Arch Virol, 2019, 164(6): 1629–1638. DOI: 10.1007/s00705-019-04256-3 |
| [32] | ALKAN F, KARAYEL I ·, CATELLA C, et al. Identification of a bovine enteric calicivirus, klrklareli virus, distantly related to Neboviruses, in calves with enteritis in Turkey[J]. J Clin Microbiol, 2015, 53(11): 3614–3617. DOI: 10.1128/JCM.01736-15 |
| [33] | MATHIJS E, DE OLIVEIRA-FILHO E F, DAL POZZO F, et al. Infectivity of a recombinant murine norovirus (RecMNV) in Balb/cByJ mice[J]. Vet Microbiol, 2016, 192: 118–122. DOI: 10.1016/j.vetmic.2016.07.012 |
| [34] | LUDWIG-BEGALL L F, MAUROY A, THIRY E. Norovirus recombinants: recurrent in the field, recalcitrant in the lab-a scoping review of recombination and recombinant types of noroviruses[J]. J Gen Virol, 2018, 99(8): 970–988. DOI: 10.1099/jgv.0.001103 |
| [35] | SILVÉRIO D, LOPES A M, MELO-FERREIRA J, et al. Insights into the evolution of the new variant rabbit haemorrhagic disease virus (GI. 2) and the identification of novel recombinant strains[J]. Transbound Emerg Dis, 2018, 65(4): 983–992. DOI: 10.1111/tbed.12830 |
| [36] | KHAMRIN P, KUMTHIP K, SUPADEJ K, et al. Noroviruses and sapoviruses associated with acute gastroenteritis in pediatric patients in Thailand: increased detection of recombinant norovirus GII. P16/GII. 13 strains[J]. Arch Virol, 2017, 162(11): 3371–3380. DOI: 10.1007/s00705-017-3501-3 |
| [37] | WHITE P A. Evolution of norovirus[J]. Clin Microbiol Infect, 2014, 20(8): 741–745. DOI: 10.1111/1469-0691.12746 |
| [38] | EDEN J S, TANAKA M M, BONI M F, et al. Recombination within the pandemic norovirus GII. 4 lineage[J]. J Virol, 2013, 87(11): 6270–6282. DOI: 10.1128/JVI.03464-12 |
| [39] | LUCERO Y, VIDAL R, O'RYAN G M. Norovirus vaccines under development[J]. Vaccine, 2018, 36(36): 5435–5441. DOI: 10.1016/j.vaccine.2017.06.043 |
| [40] | YANG B, YANG B, SHAN X N, et al. Short communication: Immune responses in sows induced by porcine sapovirus virus-like particles reduce viral shedding in suckled piglets[J]. Res Vet Sci, 2018, 117: 196–199. DOI: 10.1016/j.rvsc.2017.12.016 |
| [41] | THOMAS C, JUNG K, HAN M G, et al. Retrospective serosurveillance of bovine norovirus (GIII. 2) and nebovirus in cattle from selected feedlots and a veal calf farm in 1999 to 2001 in the United States[J]. Arch Virol,, 2014, 159(1): 83–90. DOI: 10.1007/s00705-013-1795-3 |
| [42] | LU Z Y, YOKOYAMA M, CHEN N, et al. Mechanism of cell culture adaptation of an enteric calicivirus, the porcine sapovirus Cowden Strain[J]. J Virol, 2015, 90(3): 1345–1358. |
| [43] | WOBUS C E, KARST S M, THACKRAY L B, et al. Replication of Norovirus in cell culture reveals a tropism for dendritic cells and macrophages[J]. PLoS Biol, 2004, 2(12): e432. DOI: 10.1371/journal.pbio.0020432 |
| [44] | ESTES M K, ETTAYEBI K, TENGE V R, et al. Human norovirus cultivation in nontransformed stem cell-derived human intestinal enteroid cultures:success and challenges[J]. Viruses, 2019, 11(7): 638. DOI: 10.3390/v11070638 |
| [45] | TAKANASHI S, SAIF L J, HUGHES J H, et al. Failure of propagation of human norovirus in intestinal epithelial cells with microvilli grown in three-dimensional cultures[J]. Arch Virol, 2014, 159(2): 257–266. DOI: 10.1007/s00705-013-1806-4 |
| [46] | JONES M K, WATANABE M, ZHU S, et al. Enteric bacteria promote human and mouse norovirus infection of B cells[J]. Science, 2014, 346(6210): 755–759. DOI: 10.1126/science.1257147 |
| [47] | ETTAYEBI K, CRAWFORD S E, MURAKAMI K, et al. Replication of human noroviruses in stem cell-derived human enteroids[J]. Science, 2016, 353(6306): 1387–1393. DOI: 10.1126/science.aaf5211 |
| [48] | JONES M K, GRAU K R, COSTANTINI V, et al. Human norovirus culture in B cells[J]. Nat Protoc, 2015, 10(12): 1939–1947. DOI: 10.1038/nprot.2015.121 |
| [49] | KILIC T, KOROMYSLOVA A, HANSMAN G S. Structural basis for human norovirus capsid binding to bile acids[J]. J Virol, 2019, 93(2): e01581–18. |
| [50] | PARK S I, PARK D H, SAIF L J, et al. Development of SYBR Green real-time RT-PCR for rapid detection, quantitation and diagnosis of unclassified bovine enteric calicivirus[J]. J Virol Methods, 2009, 159(1): 64–68. DOI: 10.1016/j.jviromet.2009.03.001 |
| [51] | SMILEY J R, HOET A E, TRÅVÉN M, et al. Reverse Transcription-PCR Assays for Detection of Bovine Enteric Caliciviruses (BEC) and analysis of the genetic relationships among BEC and human Caliciviruses[J]. J Clin Microbiol, 2003, 41(7): 3089–3099. DOI: 10.1128/JCM.41.7.3089-3099.2003 |
| [52] | PARK S J, JEONG C, YOON S S, et al. Detection and characterization of bovine coronaviruses in fecal specimens of adult cattle with diarrhea during the warmer seasons[J]. J Clin Microbiol, 2006, 44(9): 3178–3188. DOI: 10.1128/JCM.02667-05 |
| [53] |
郭紫晶, 何琪富, 汤承, 等. 检测纽布病毒的Real-time RT-PCR方法的建立与应用[J]. 畜牧兽医学报, 2019, 50(4): 893–900.
GUO Z J, HE Q F, TANG C, et al. Establishment and application of a Real-time RT-PCR assay for detecting bovine nebovirus[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(4): 893–900. (in Chinese) |



