2. 中国农业科学院北京畜牧兽医研究所, 北京 100193;
3. 湖南农业大学, 长沙 410128;
4. 云南农业大学, 昆明 650201
2. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China;
3. Hunan Agricultural University, Changsha 410128, China;
4. Yunnan Agricultural University, Kunming 650201, China
基因编辑是揭示基因功能、调控基因表达、制备转基因动植物等研究的重要技术手段,可实现人为主观地对目的基因进行修改、删除或插入。目前,主流的基因编辑技术有锌指核酸酶(zinc-finger nuclease, ZFN)、转录激活因子样效应物核酸酶(transcription activator-like effector nuclease, TALEN)和CRISPR/Cas9。其中,CRISPR/Cas9技术快速简单,成本低廉,能有效地靶向基因组任何位点,且多次被《Nature》和《Science》杂志评为年度十大科学突破成果。近些年,该技术被广泛地应用于动植物遗传改良领域,并且给动植物育种带来了突破性进展,极大地提高了品种的选育速度。猪既是人类最主要的动物蛋白来源,也是生物医学研究最理想的模型之一,但是,猪基因组中大部分基因和位点的功能及调控机理仍不清楚,CRISPR/Cas9技术的蓬勃发展将极大推动优良种猪品种培育以及转基因猪医学模型的构建。本文主要介绍了CRISPR/Cas9技术及其在猪基因组上的应用,并讨论了该技术在未来猪遗传育种和人类医学研究中的重要价值。
1 CRISPR/Cas的发现1987年,Ishino等[1]在分析大肠杆菌(Escherichia coli)基因序列时,偶然发现了一段聚集且规则间隔的短回文重复序列,并将其命名为CRISPR(clustered regularly interspaced short palindromic repeat)。在多数细菌及古细菌中,该区域是一段由众多保守的短重复序列区(repeat)以及细菌捕获的外源基因片段组成的间隔区(spacer)所构成的DNA重复序列[2]。
直至2005年,Pourcel等[3]进一步揭示了CRISPR的结构,并发现CRISPR/Cas是一种微生物体内可遗传的获得性免疫防御体系,用于抵抗外源核酸的入侵,这为基因位点的特异辨识提供了一种新方法。随后,研究人员发现,Cas基因家族可以编码不同功能的CRISPR相关蛋白,并与CRISPR一起形成CRISPR/Cas工作系统,从而识别外源基因特异序列,同时联合CRISPR/Cas系统启动子前导区转录生成的CRISPR RNAs(crRNA),以靶向剪切的方式破坏入侵的DNA[4]。Cas核酸酶可以通过接触反式激活crRNA(transactivating crRNA,tracrRNA)与crRNA前体形成的二聚体进一步成为复合体,协助细菌识别基因组内的特定基因序列进行有效切割,这是一种适应性的免疫防御机制。
基于上述原理,科学家们借助crRNA对目标序列特异性识别的特点,引导Cas核酸酶准确定位目标基因,并对单核酸位点或多核酸位点进行精确切割与编辑[5]。因此,CRISPR/Cas渐渐成为主流基因编辑工具。
2 CRISPR/Cas9的作用机理由于存在不同类型的Cas蛋白,所以CRISPR/Cas基因编辑系统分为Ⅰ型、Ⅱ型与Ⅲ型3大类。Ⅰ型与Ⅲ型均需要依赖复杂的蛋白质复合体进行基因编辑。Ⅰ型需多个Cas蛋白与crRNAs结合形成复合物,进而识别外源靶DNA的3′末端序列(protospacer adjacent motif, PAM)元件位点进行特异性结合,并介导Cas3蛋白对目标DNA进行剪辑;在Ⅲ型系统中,Cas6 /Cas10在不需要PAM的情况下,即可作用于crRNA前体并与之结合,从而识别并破坏外源性DNA[6]。Ⅱ型Cas蛋白仅存在于细菌中,包含有Cas9蛋白、crRNA及tracrRNA等组分,由于tracrRNA-crRNA-Cas9复合物可以靶向识别PAM元件并切割外源性DNA,因此,只需体外设计合成一条单链向导RNA(single guide RNA,sgRNA)即可代替tracrRNA-crRNA,从而引导Cas9蛋白完成DNA剪辑工作[7]。由此,根据CRISPR/CasⅡ型演变而成了精简高效的CRISPR/Cas9靶向切割系统,并开始广泛应用于各种基因编辑研究中。
CRISPR/Cas9系统工作原理主要分为以下4个步骤:第一阶段是外源DNA的俘获,CRISPR/Cas9系统可以将新的一小段双链外源DNA插入到间隔区内,并整合到细菌自身基因中;第二阶段是合成成熟的crRNA,通过CRISPR基因座转录形成的crRNA前体,在Cas9和核酸酶的作用下被剪切为成熟的crRNA,从而形成tracrRNA-crRNA-Cas9复合物;第三阶段是对外源基因的靶向切割,tracrRNA-crRNA-Cas9复合物中的crRNA通过PAM位点扫描识别外源DNA序列中匹配的位点并与之结合,从而进行对外源DNA双链的切割;第四个阶段是CRISPR/Cas9系统的应用,主要有两种:一是基因敲除(knock-out),即在体外构建一对含有目的基因PAM反义序列的gRNA,引导Cas9蛋白定点在打靶基因处切割形成DNA双链断裂(DNA double-strand breaks, DSBs),并通过非同源末端连接(non-homologous end joining,NHEJ)途径进行断链DNA的修复;二是基因敲入(knock-in),是在基因敲除的操作基础上,再构建一条包含目的基因在内的供体基因,利用电转法与上述原件同时转入目的细胞中,在Cas9蛋白的切割序列位置利用同源直接修复(homology directed repair,HDR)途径引入供体基因片段,完成供体基因对目的基因的替换[8]。CRISPR/Cas9系统的具体分子机制如图 1所示。
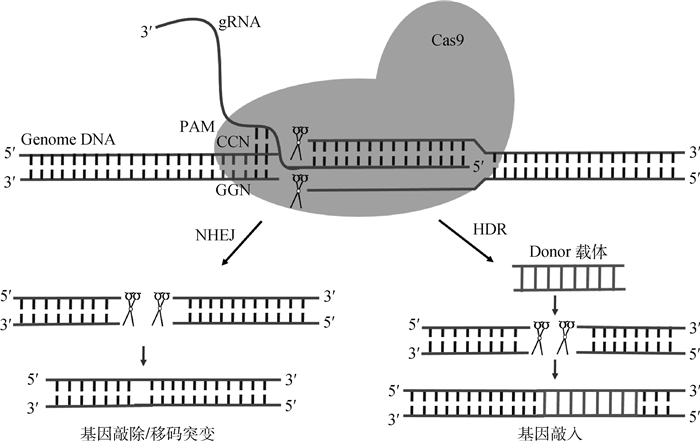
|
Genome DNA.基因组DNA;Cas9. Cas9蛋白; gRNA.向导RNA;PAM.向导RNA识别位点;NHEJ.非同源末端连接; HDR.同源直接修复; Donor载体.供体载体 Genome DNA. Genome DNA; Cas9. Cas proteim; gRNA. Guide RNA; PAM. Protospacer adjacent motif; NHEJ. Non-homologous end joining; HDR. Homology directed repair; Donor载体. Donor vector 图 1 CRISPR/Cas9系统在基因组中的工作示意图 Fig. 1 Genome editing using the CRISPR/Cas9 system |
CRISPR/Cas9系统已广泛应用于猪基因编辑及相关研究中,为优质高效猪品种的培育和基因编辑猪的创制带来了巨大的推动力。本文主要论述CRISPR/Cas9技术在猪遗传性状改良和生物医用模型猪中的应用现状。CRISPR/Cas9系统基于gRNA的协助,可精准定位并编辑猪基因组上的一段或多段序列,有助于将其他物种的优质基因、环境友好型基因以及功能性基因等定点转入猪基因组内并稳定表达。同时,该技术与全基因组测序技术的结合可高密度筛选猪基因组中与育种相关的关键位点和功能基因。
3.1 农用基因编辑猪的研究进展目前,利用CRISPR/Cas9技术结合体细胞核移植技术(somatic cell nuclear transfer,SCNT)已获得多种基因编辑猪。2015年,Ruan等[9]利用TALEN、CRISPR/Cas9n与CRISPR/Cas9三种基因编辑系统对猪基因组中H11位点进行打靶编辑,证明CRISPR/Cas9系统具有最高的编辑效率,并在猪基因组中插入了一段长达9.4 kb可稳定高效表达的外源DNA片段。为了提高猪的瘦肉率,Wang等[10]利用CRISPR/Cas9技术成功打靶编辑了猪的肌肉生长抑制素(myostatin, MSTN)基因,并制备了MSTN突变的基因编辑猪,显著提高了猪的瘦肉率。这些结果表明,CRISPR/Cas9基因编辑技术可以高效改良猪生长性状,提高经济效益。Xiang等[11]利用CRISPR/Cas9技术对胰岛素样生长因子2(insulin-like growth factor 2, IGF2)的三号内含子中一段序列进行精确编辑,培育出了产肉量优于野生型巴马猪的基因修饰巴马猪,这是首次通过基因编辑的手段证明猪的非编码区上具有能改善产肉量的位点。2019年,Liu等[12]对猪胚胎成纤维细胞(porcine embryonic fibroblasts, PEFs)进行了前期编辑,即利用CRISPR/Cas9基因敲除系统成功破坏了IGF2内含子3序列中与ZBED6结合的模体,进而通过SCNT制备了肌肉发育增强型的两广小花猪。由于猪的体内缺少一种名为解偶联蛋白1(uncoupling protein 1, UCP1)的线粒体内膜蛋白,从而降低了猪的产热能力,加速脂肪沉积。Zheng等[13]在2017年利用CRISPR/Cas9成功将UCP1基因插入猪的基因组中,制备出能够抵抗严寒且脂肪率低于野生型的转基因猪,并发现该基因的转入还能提高猪的瘦肉率。
猪繁殖与呼吸综合征(porcine reproductive and respiratory syndrome, PRRS)又称“蓝耳病”,是造成全世界养猪产业经济损失最严重的猪传染病之一,该病的病原是猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus, PRRSV)。一种被称作清道夫受体的CD163蛋白是PRRSV感染宿主的必需受体[14-15]。2014年,Whitworth等[16]成功制备了CD163双等位基因敲除猪,该基因编辑猪在进行攻毒试验后未表现出蓝耳病的临床症状。该研究表明,病毒受体基因敲除后能显著提高猪的抗病能力。2018年,Burkard等[17]利用CRISPR/Cas9对猪的受精卵进行基因编辑,将CD163基因第7外显子上富含半胱氨酸的结构域5(scavenger receptor cysteine-rich domain 5, SRCR5)全部敲除,成功制备了能抵抗PRRSV-1的抗病猪。Guo等[18]进一步在长白猪和两广小花猪中利用CRISPR/Cas9特异性删除了SRCR5结构域里一段长度仅为41个氨基酸的序列,并培育出可抵抗PRRSV-2的长白猪与两广小花猪抗病猪。进一步研究发现,只需对SRCR5进行单位点酶切便可制备能抵抗高致病性蓝耳病的杜洛克抗病猪[19]。以上编辑手段均能有效的抵抗蓝耳病,且不影响CD163的生物学功能。
Rosa26基因座在哺乳动物中具有优良的保守型和包容性,因此,大规模应用于基因编辑研究中[20-22]。研究人员利用CRISPR/Cas9系统在猪基因组中的Rosa26位点插入了一条抗经典猪瘟病毒(classical swine fever virus, CSFV)的“短发夹状RNA”(short hairpin RNA, shRNA),随后制备出可抵抗CSF的抗病猪,该抗病性状可稳定遗传给后代[23]。
3.2 生物医用基因编辑猪的研究进展由于猪的生理和解剖结构与人类高度类似,因此猪已成为一种理想的生物医学模型[24-25]。目前,免疫排斥反应尤其是慢性排斥反应是猪作为异种器官移植供体需要解决的首要问题。猪的α1, 3-半乳糖转移酶基因(α1, 3- galactosyltransferase, GGTA1)是一个关键基因,它可以编码Gal蛋白表位,从而导致超急性排斥反应。科研人员曾尝试通过ZFN、TALEN等技术制备敲除或突变猪GGTA1基因[26-27]。Sato等[28]利用CRISPR/Cas9技术先在猪成纤维细胞中敲除该基因,并获得了GGTA1双等位基因敲除的细胞,随后在猪的胚胎中也成功敲除了GGTA1基因。2017年,Chuang等[29]利用CRISPR/Cas9技术,在已敲除GGTA1基因的巴马小型猪上又敲除了β1, 4 N-乙酰半乳糖胺转移酶(β1, 4 N-acetylgalactosaminyl transferase, β4GalNT2)基因,进一步降低了异种移植的免疫排斥反应。此外,唐雨婷等[30]也成功制备了GGTA1/β4GalNT2双基因敲除猪。异种器官移植的成功除了要解决上述免疫排斥的问题外,还需去除猪组织中携带的内源性逆转录病毒(porcine endogenous retroviruses, PERV),以免感染移植手术的受者。2017年,Niu等[31]通过CRISPR/Cas9技术使猪基因组中62种PERV失活,阻止了物种间PERV的传播,推进了猪脏器成为异种移植器官的进程。
Zhou等[32]针对猪基因组中Tyrosinase、PARK2和PINK1三个基因的外显子区域设计了多个打靶位点,并通过多条gRNA的协助,成功在版纳小型猪和巴马小型猪的PEFs中敲除了以上目的基因,最终培育出两个不同品种的多基因敲除猪,并由此建立了人白化病和帕金森综合症两种医用猪模型。2018年3月,Yan等[33]再次通过CRISPR/Cas9与SCNT技术,成功制备出世界首例亨廷顿舞蹈病基因敲入猪,并首次准确地在猪模型中模拟出人神经退行性疾病。
此外,利用猪作为生物反应器,也是当下研究的一大热点[34]。2015年,Peng等[35]利用CRISPR/Cas9技术在猪的受精卵中插入人白蛋白基因,成功获得了可以生产人白蛋白的转基因猪,推进了大型动物生物反应器的制备进程。猪与人的胰岛素基因只差一个氨基酸,Yang等[36]利用该技术对猪胰岛素基因进行定点突变,制备了能够产生人胰岛素的转基因纯合猪。
黑素皮质素3受体(melanocortin 3 receptor, MC3R)可以调控机体的能量稳态,有研究报道该基因的突变将显著提高人体肥胖的风险[37-38]。为进一步阐述MC3R在哺乳动物体内的生物学功能,Yin等[39]通过CRSPR/Cas9系统并结合SCNT技术研制了MC3R敲除猪,MC3R敲除后的猪比野生型猪具有更多的脂肪沉积,为人类肥胖疾病的研究建立了良好的医学模型。其次,鉴于猪的某些疾病需借助非猪源细胞系建立最佳表型,并开展功能研究,CRISPR/Cas9在细胞系中的应用为建立猪的相关疾病模型奠定了前期研究基础[40-43]。
3.3 CRISPR/Cas9技术面临的问题与挑战CRISPR/Cas9系统的编辑效率根据生物材料的不同会有差异,该系统在受精卵和胚胎中的操作更简洁高效,且不损伤合子的生命活性,只需将gRNA和Cas9蛋白酶的mRNA显微注射入受精卵中即可[24, 44]。鉴于斑马鱼、小鼠和绵羊等模式生物都已有稳定的胚胎编辑材料,因此,CRISPR/Cas9系统在这类模式生物的基因编辑中会有更高的工作效率[45-47]。然而,由于猪的受精卵收集困难且成本高,目前,研究人员普遍采用先在体外编辑猪的成纤维细胞,再将已编辑成功的成纤维细胞通过SCNT技术制备基因编辑猪[32, 48]。这种方法既增加了基因编辑猪的制备步骤和复杂性,也降低了工作效率。另外,CRISPR/Cas9系统中的另一大难题是打靶效率低,从而导致阳性编辑细胞筛选率低、基因编辑系统不稳定以及人力和时间成本增加等问题的产生。目前,研究人员通过逐步优化CRISPR/Cas9系统、筛选打靶位点和增强gRNA在基因组中的特异性识别能力来提高打靶效率[49-52]。因此,如何进一步优化基因编辑手段,从而将基因组的精准编辑效率提高到100%,是未来猪基因组编辑的研究方向。
目前,很多学者利用多种方式检测了CRISPR/Cas9打靶效率,但检测手段都较为繁琐且昂贵[53-54]。经研究发现,现有的基因编辑手段仍受到有效编辑序列范围和PAM位点依赖等问题的限制,因此无法直接得知目的基因的编辑效率及其编辑后的分子生物学功能变化。尽管CRISPR/Cas9基因编辑技术在DNA与RNA编辑、动物免疫、动物疾病模型和转基因动物制备等领域的拓展应用中取得了多方面的突破性进展[55-56],基因编辑猪的器官异种移植到人体内的工作仍处于材料制备和动物测试阶段,临床试验亟须突破[57-59]。其次,尽管目前已成功制备了多种转基因猪,但暂未扩繁为大群体,转基因动物产业之路任重道远。
综上,CRISPR/Cas9技术为基因组编辑和研究基因功能调控等提供了一个简便、经济、高效且精准的手段,为猪品种改良和医学动物模型制备提供了新的方法,极大地促进了农业科学和生物医学等基础研究的发展[60-61]。其次,CRISPR/Cas9技术也广泛应用于基因治疗中,该技术为实现分子治疗、靶向治疗复杂遗传病及癌症等多种重大疾病提供了新的治疗途径。
4 结语目前,基因编辑技术蓬勃发展,其应用领域也在迅速扩大。CRISPR/Cas9的胞嘧啶单碱基基因编辑技术可对基因组中的任一段DNA序列进行单碱基编辑[62-63],该方法可结合分子生物学,高效鉴定猪基因组中单核苷酸多态性(single nucleotide polymorphisms, SNP)位点的生物学功能。随着生物信息学和高通量测序技术的不断更新,科学家们开发了一系列高通量筛选和设计动物全基因组内gRNA的软件,以供基因组编辑文库的构建和应用[64-66],进而,CRISPR/Cas9结合生物信息学分析将更有助于推进基因组编辑和基因功能研究的发展。随着基础研究领域与临床应用需求的不断扩大,CRISPR/Cas9基因编辑系统已逐步优化并应用于更多的农业分子育种和生命科学研究中,基因编辑过程中出现的特异性识别能力不强与脱靶率高等问题将逐渐解决,其操作性和编辑效率会逐步提高,从而使猪基因组编辑变得更加简便、高效和安全[67]。在未来,CRISPR/Cas9及其相关基因编辑技术将会在猪基因组编辑的研究中获得更多突破性进展。
| [1] | ISHINO Y, SHINAGAWA H, MAKINO K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J]. J Bacteriol, 1987, 169(12): 5429–5433. |
| [2] | JANSEN R, VAN EMBDEN J D A, GAASTRA W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43(6): 1565–1575. |
| [3] | POURCEL C, SALVIGNOL G, VERGNAUD G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies[J]. Microbiology, 2005, 151(3): 653–663. |
| [4] | BROUNS S J, JORE M M, LUNDGREN M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science, 2008, 321(5891): 960–964. |
| [5] | JIANG W Y, BIKARD D, COX D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nat Biotechnol, 2013, 31(3): 233–239. |
| [6] | SHAH S A, GARRETT R A. CRISPR/Cas and Cmr modules, mobility and evolution of adaptive immune systems[J]. Res Microbiol, 2011, 162(1): 27–38. |
| [7] | JINEK M, CHYLINSKI K, FONFARA I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816–821. |
| [8] | WHITWORTH K M, ROWLAND R R R, EWEN C L, et al. Gene-edited pigs are protected from porcine reproductive and respiratory syndrome virus[J]. Nat Biotechnol, 2016, 34(1): 20–22. |
| [9] | RUAN J X, LI H G, XU K, et al. Highly efficient CRISPR/Cas9-mediated transgene knockin at the H11 locus in pigs[J]. Sci Rep, 2015, 5: 14253. |
| [10] | WANG K K, OUYANG H S, XIE Z Z, et al. Efficient generation of myostatin mutations in pigs using the CRISPR/Cas9 system[J]. Sci Rep, 2015, 5: 16623. |
| [11] | XIANG G H, REN J L, HAI T, et al. Editing porcine IGF2 regulatory element improved meat production in Chinese Bama pigs[J]. Cell Mol Life Sci, 2018, 75(24): 4619–4628. |
| [12] | LIU X F, LIU H B, WANG M, et al. Disruption of the ZBED6 binding site in intron 3 of IGF2 by CRISPR/Cas9 leads to enhanced muscle development in Liang Guang Small Spotted pigs[J]. Transgenic Res, 2019, 28(1): 141–150. |
| [13] | ZHENG Q T, LIN J, HUANG J J, et al. Reconstitution of UCP1 using CRISPR/Cas9 in the white adipose tissue of pigs decreases fat deposition and improves thermogenic capacity[J]. Proc Natl Acad Sci U S A, 2017, 114(45): E9474–E9482. |
| [14] | VAN GORP H, VAN BREEDAM W, VAN DOORSSELAERE J, et al. Identification of the CD163 protein domains involved in infection of the porcine reproductive and respiratory syndrome virus[J]. J Virol, 2010, 84(6): 3101–3105. |
| [15] | WELLS K D, BARDOT R, WHITWORTH K M, et al. Replacement of porcine CD163 scavenger receptor cysteine-rich domain 5 with a CD163-like homolog confers resistance of pigs to genotype 1 but not genotype 2 porcine reproductive and respiratory syndrome virus[J]. J Virol, 2017, 91(2): e01521–e01516. |
| [16] | WHITWORTH K M, LEE K, BENNE J A, et al. Use of the CRISPR/Cas9 system to produce genetically engineered pigs from in vitro-derived oocytes and embryos[J]. Biol Reprod, 2014, 91(3): 78. |
| [17] | BURKARD C, OPRIESSNIG T, MILEHAM A J, et al. Pigs lacking the scavenger receptor cysteine-rich domain 5 of CD163 are resistant to porcine reproductive and respiratory syndrome virus 1 infection[J]. J Virol, 2018, 92(16): e00415–e00418. |
| [18] | GUO C H, WANG M, ZHU Z B, et al. Highly efficient generation of pigs harboring a partial deletion of the CD163 SRCR5 domain, which are fully resistant to porcine reproductive and respiratory syndrome virus 2 infection[J]. Front Immunol, 2019, 10: 1846. |
| [19] | YANG H Q, ZHANG J, ZHANG X W, et al. CD163 knockout pigs are fully resistant to highly pathogenic porcine reproductive and respiratory syndrome virus[J]. Antiviral Res, 2018, 151: 63–70. |
| [20] | YU M, SUN X S, TYLER S R, et al. Highly efficient transgenesis in ferrets using CRISPR/Cas9-mediated homology-independent insertion at the ROSA26 locus[J]. Sci Rep, 2019, 9(1): 1971. |
| [21] | WANG M, SUN Z L, ZOU Z Y, et al. Efficient targeted integration into the bovine ROSA26 locus using TALENs[J]. Sci Rep, 2018, 8(1): 10385. |
| [22] | WU Y M, LUNA M J, BONILLA L S, et al. Characterization of knockin mice at the Rosa26, Tac1 and Plekhg1 loci generated by homologous recombination in oocytes[J]. PLoS One, 2018, 13(2): e0193129. |
| [23] | XIE Z C, PANG D X, YUAN H M, et al. Genetically modified pigs are protected from classical swine fever virus[J]. PLoS Pathog, 2018, 14(12): e1007193. |
| [24] | HAI T, TENG F, GUO R F, et al. One-step generation of knockout pigs by zygote injection of CRISPR/Cas system[J]. Cell Res, 2014, 24(3): 372–375. |
| [25] | WANG J Z, LIU M L, ZHAO L H, et al. Disabling of nephrogenesis in porcine embryos via CRISPR/Cas9-mediated SIX1 and SIX4 gene targeting[J]. Xenotransplantation, 2019, 26(3): e12484. |
| [26] | LAI L X, KOLBER-SIMONDS D, PARK K W, et al. Production of α-1, 3-galactosyltransferase knockout pigs by nuclear transfer cloning[J]. Science, 2002, 295(5557): 1089–1092. |
| [27] | XIN J G, YANG H Q, FAN N N, et al. Highly efficient generation of GGTA1 biallelic knockout inbred mini-pigs with TALENs[J]. PLoS One, 2013, 8(12): e84250. |
| [28] | SATO M, MIYOSHI K, NAGAO Y, et al. The combinational use of CRISPR/Cas9-based gene editing and targeted toxin technology enables efficient biallelic knockout of the α-1, 3-galactosyltransferase gene in porcine embryonic fibroblasts[J]. Xenotransplantation, 2014, 21(3): 291–300. |
| [29] | CHUANG C K, CHEN C H, HUANG C L, et al. Generation of GGTA1 mutant pigs by direct pronuclear microinjection of CRISPR/Cas9 plasmid vectors[J]. Anim Biotechnol, 2017, 28(3): 174–181. |
| [30] |
唐雨婷, 高景波, 杜敏杰, 等. CRISPR/Cas9介导的β4GalNT2基因敲除猪制备[J]. 农业生物技术学报, 2017, 25(10): 1697–1705.
TANG Y T, GAO J B, DU M J, et al. Generation of β4GalNT2 gene knockout pigs (Sus scrofa) via CRISPR/Cas9[J]. Journal of Agricultural Biotechnology, 2017, 25(10): 1697–1705. (in Chinese) |
| [31] | NIU D, WEI H J, LIN L, et al. Inactivation of porcine endogenous retrovirus in pigs using CRISPR-Cas9[J]. Science, 2017, 357(6357): 1303–1307. |
| [32] | ZHOU X Q, XIN J G, FAN N N, et al. Generation of CRISPR/Cas9-mediated gene-targeted pigs via somatic cell nuclear transfer[J]. Cell Mol Life Sci, 2015, 72(6): 1175–1184. |
| [33] | YAN S, TU Z C, LIU Z M, et al. A huntingtin knockin pig model recapitulates features of selective neurodegeneration in huntington's disease[J]. Cell, 2018, 173(4): 989–1002. |
| [34] |
靳伟, 代敏敏, 李德娟, 等. CRISPR/Cas9介导的外源基因在猪PSP位点的定点整合[J]. 农业生物技术学报, 2019, 27(9): 1569–1581.
JIN W, DAI M M, LI D J, et al. CRISPR/Cas9-mediated site specific integration of foreign genes in pig (Sus scrofa) PSP locus[J]. Journal of Agricultural Biotechnology, 2019, 27(9): 1569–1581. (in Chinese) |
| [35] | PENG J, WANG Y, JIANG J Y, et al. Production of human albumin in pigs through CRISPR/Cas9-mediated knockin of human cDNA into swine albumin locus in the zygotes[J]. Sci Rep, 2015, 5: 16705. |
| [36] | YANG Y, WANG K P, WU H, et al. Genetically humanized pigs exclusively expressing human insulin are generated through custom endonuclease-mediated seamless engineering[J]. J Mol Cell Biol, 2016, 8(2): 174–177. |
| [37] | YANG Z, TAO Y X. Mutations in melanocortin-3 receptor gene and human obesity[J]. Prog Mol Biol Transl Sci, 2016, 140: 97–129. |
| [38] | EHTESHAM S, QASIM A, MEYRE D. Loss-of-function mutations in the melanocortin-3 receptor gene confer risk for human obesity:a systematic review and meta-analysis[J]. Obes Rev, 2019, 20(8): 1085–1092. |
| [39] | YIN Y J, HAO H Y, XU X B, et al. Generation of an MC3R knock-out pig by CRSPR/Cas9 combined with somatic cell nuclear transfer (SCNT) technology[J]. Lipids Health Dis, 2019, 18(1): 122. |
| [40] |
王晓朋, 徐奎, 魏迎辉, 等. CRISPR/Cas9介导的猪IPEC-J2细胞CD13基因敲除细胞系的建立[J]. 畜牧兽医学报, 2019, 50(7): 1319–1327.
WANG X P, XU K, WEI Y H, et al. Establishment of CD13 gene knockout IPEC-J2 cell lines mediated by CRISPR/Cas9 system[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(7): 1319–1327. (in Chinese) |
| [41] | PANG L, ZHANG N, XIA Y, et al. Serum APN/CD13 as a novel diagnostic and prognostic biomarker of pancreatic cancer[J]. Oncotarget, 2016, 7(47): 77854–77864. |
| [42] |
张东杰, 刘娣, 张旭, 等. 利用CRISPR-Cas9系统定点突变猪MSTN基因的研究[J]. 畜牧兽医学报, 2016, 47(1): 207–212.
ZHANG D J, LIU D, ZHANG X, et al. Study of pig MSTN gene point mutation based on the CRISPR-Cas9 system[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(1): 207–212. (in Chinese) |
| [43] |
李聪聪, 张永辉, 赵婉霞, 等. CRISPR/Cas9系统介导的miR-155基因敲除细胞的制备[J]. 生物技术通报, 2019, 35(11): 231–239.
LI C C, ZHANG Y H, ZHAO W X, et al. Preparation of miR-155 knockout cells mediated by CRISPR/Cas9 technology[J]. Biotechnology Bulletin, 2019, 35(11): 231–239. (in Chinese) |
| [44] | SATO M, KOSUKE M, KORIYAMA M, et al. Timing of CRISPR/Cas9-related mRNA microinjection after activation as an important factor affecting genome editing efficiency in porcine oocytes[J]. Theriogenology, 2018, 108: 29–38. |
| [45] | KIMURA Y, HISANO Y, KAWAHARA A. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering[J]. Sci Rep, 2014, 4: 6545. |
| [46] | ZUO E W, CAI Y J, LI K, et al. One-step generation of complete gene knockout mice and monkeys by CRISPR/Cas9-mediated gene editing with multiple sgRNAs[J]. Cell Res, 2017, 27(7): 933–945. |
| [47] | CRISPO M, MULET A P, TESSON L, et al. Efficient generation of myostatin knock-out sheep using CRISPR/Cas9 technology and microinjection into zygotes[J]. PLoS One, 2015, 10(8): e0136690. |
| [48] | ZHANG J P, ZHU Z S, YUE W, et al. Establishment of CRISPR/Cas9-mediated knock-in system for porcine cells with high efficiency[J]. Appl Biochem Biotechnol, 2019, 189(1): 26–36. |
| [49] | FU Y F, FODEN J A, KHAYTER C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells[J]. Nat Biotechnol, 2013, 31(9): 822–826. |
| [50] | RAN F A, HSU P D, LIN C Y. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1380–1389. |
| [51] | KIM E J, KOO T, PARK S W, et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni[J]. Nat Commun, 2017, 8: 14500. |
| [52] | RAN F A, CONG L, YAN W X, et al. In vivo genome editing using Staphylococcus aureus Cas9[J]. Nature, 2015, 520(7546): 186–191. |
| [53] |
吴金青, 梅瑰, 刘志国, 等. 应用SSA报告载体提高ZFN和CRISPR/Cas9对猪IGF2基因的打靶效率[J]. 遗传, 2015, 37(1): 55–62.
WU J Q, MEI G, LIU Z G, et al. Improving gene targeting efficiency on pig IGF2 mediated by ZFNs and CRISPR/Cas9 by using SSA reporter system[J]. Hereditas, 2015, 37(1): 55–62. (in Chinese) |
| [54] | ZUO E W, SUN Y D, WEI W, et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos[J]. Science, 2019, 364(6437): 289–292. |
| [55] | ABUDAYYEH O O, GOOTENBERG J S, FRANKLIN B, et al. A cytosine deaminase for programmable single-base RNA editing[J]. Science, 2019, 365(6451): 382–386. |
| [56] | CHEN Y C, ZHENG Y H, KANG Y, et al. Functional disruption of the dystrophin gene in rhesus monkey using CRISPR/Cas9[J]. Hum Mol Genet, 2015, 24(13): 3764–3774. |
| [57] | YANG L H, GÜELL M, NIU D, et al. Genome-wide inactivation of porcine endogenous retroviruses (PERVs)[J]. Science, 2015, 350(6264): 1101–1104. |
| [58] | YAN J J, YEOM H J, JEONG J C, et al. Beneficial effects of the transgenic expression of human sTNF-αR-Fc and HO-1 on pig-to-mouse islet xenograft survival[J]. Transpl Immunol, 2016, 34: 25–32. |
| [59] | CHAN J L, SINGH A K, CORCORAN P C, et al. Encouraging experience using multi-transgenic xenografts in a pig-to-baboon cardiac xenotransplantation model[J]. Xenotransplantation, 2017, 24(6): e12330. |
| [60] | BALIOU S, ADAMAKI M, KYRIAKOPOULOS A M, et al. CRISPR therapeutic tools for complex genetic disorders and cancer (Review)[J]. Int J Oncol, 2018, 53(2): 443–468. |
| [61] | KRUMINIS-KASZKIEL E, JURANEK J, MAKSYMOWICZ W, et al. CRISPR/Cas9 technology as an emerging tool for targeting amyotrophic lateral sclerosis (ALS)[J]. Int J Mol Sci, 2018, 19(3): 906. |
| [62] | GAUDELLI N M, KOMOR A C, REES H A, et al. Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage[J]. Nature, 2017, 551(7681): 464–471. |
| [63] | KOMOR A C, BADRAN A H, LIU D R. CRISPR-based technologies for the manipulation of eukaryotic genomes[J]. Cell, 2017, 168(1-2): 20–36. |
| [64] | XIE S S, SHEN B, ZHANG C B, et al. sgRNAcas9:a software package for designing CRISPR sgRNA and evaluating potential off-target cleavage sites[J]. PLoS One, 2014, 9(6): e100448. |
| [65] | ZHAO C Z, ZHENG X G, QU W B, et al. CRISPR-offinder:a CRISPR guide RNA design and off-target searching tool for user-defined protospacer adjacent motif[J]. Int J Biol Sci, 2017, 13(12): 1470–1478. |
| [66] |
吴海波, 边雪娇, 贾丽玲, 等. 二代靶向测序在CRISPR/Cas9靶区筛选中的应用性研究[J]. 畜牧兽医学报, 2019, 50(8): 1587–1595.
WU H B, BIAN X J, JIA L L, et al. Screening of CRISPR/Cas9 gene editing targets using next generation targeted sequence[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(8): 1587–1595. (in Chinese) |
| [67] | LINO C A, HARPER J C, CARNEY J P, et al. Delivering CRISPR:a review of the challenges and approaches[J]. Drug Deliv, 2018, 25(1): 1234–1257. |



