2. 中华人民共和国荣成海关, 荣成 264300
2. Rongcheng Customs of the People's Republic of China, Rongcheng 264300, China
动物肠道中的微生物由细菌、古生菌及真菌构成,微生物数量可达宿主细胞总量的1~10倍,其总基因数是宿主的100倍[1]。肠道正常菌群数量庞大、结构复杂,且彼此之间相互作用,相互制约,处于动态平衡状态。
肠道正常菌群对宿主健康有重要意义,肠道菌群成员可以执行碳水化合物、氨代谢和维生素的合成与降解等消化代谢作用,促进宿主的正常生长[2];肠道健康菌群可以与致病性肠道微生物竞争肠道上皮细胞黏附位点和营养物质,产生抗菌物质等构成菌群抗病原微生物的屏障作用[3-4];且肠道正常菌群可以促进宿主免疫系统的发育与成熟,维持宿主的健康状态[5]。在后基因组时代,优化肠道微生物群落的分类组成及其生化功能,促进动物健康生产是一个可实现的目标。健康动物个体或群体具有特征性、稳定的菌群结构,而对健康群体特征性、稳定性细菌菌群结构解析,是继续讨论微生物群落调节与干预的基础[6]。鉴于肠道微生物菌群作为生命体的重要组成部分,对宿主的生长发育、生产力提高有重要影响。高邮鸭是我国肉蛋兼用型地方良种,耐粗饲、适应力强。
本研究以健康高邮鸭为动物模型,通过16S rDNA V3-V4区,分析鸭肠道内容物中菌群区系特征,揭示肠道内容物中微生物丰度和多样性,同时分析拟杆菌种在鸭肠道内的分布特征,以期为鸭健康肠道菌群的调控与干预提供基础数据。
1 材料与方法 1.1 主要仪器与试剂ProFlex型PCR仪(美国ThermoFisher公司);琼脂糖电泳仪;数显恒温水浴锅;NanoDrop2000(美国ThermoFisher公司)。
Iso Plus Fragment Library Kit 48 rxns试剂盒(美国ThermoFisher公司);十六烷基三甲基溴化铵(CTAB,cetyltrimethylammonium bromide)、琼脂糖等(分析纯,国药产品)。
1.2 鸭肠道内容物取样健康成年高邮鸭20只,公母各半。正常饲养于江苏省家禽科学研究所水禽试验基地,试验鸭群饲粮配方及营养水平见表 1。70日龄时(无禁食),处死鸭之后,打开腹腔,分离出十二指肠、空肠、回肠和盲肠,分别取各肠段内容物放入5 mL无菌离心管,-80 ℃保存。
|
|
表 1 试验基础日粮组成及营养水平 Table 1 Diet composition and nutrient levels of experimental basal diet |
采用CTAB法提取肠道内容物中细菌基因组DNA。DNA浓度和纯度利用NanoDrop2000进行检测,1%琼脂糖凝胶电泳检测DNA提取质量。取适量的样品DNA于离心管中,使用无菌水稀释样品至1 ng·μL-1。
以稀释后的基因组DNA为模板,根据测序区域的选择,使用带Barcode的特异引物,New England Biolabs公司的PhusionⓇ High-Fidelity PCR Master Mix with GC Buffer和高效高保真酶进行PCR,确保扩增效率和准确性。
1.4 建库和测序根据Iso Plus Fragment Library Kit 48 rxns (Thermo Scientific)试剂盒构建文库,利用Qubit@2.0 Fluorometer (Thermo Scientific)进行文库质控,经检测合格后,利用IonS5TMXL平台进行测序,测序引物为343F: TACGGRAGGCAGCAG,798R: AGGGTATCTAATCCT。建库与测序工作由北京诺禾致源生物信息科技有限公司完成。
1.5 数据处理原始测序序列使用Cutadapt(V1.9.1, http://cutadapt.readthedocs.io/en/stable/)剪切低质量reads,再去除Barcode和引物序列,并进行初步质控获得原始数据(raw reads);原始数据序列通过(UCHIME Algorithm,http://www.drive5.com/usearch/manual/uchime_algo.html)[7]与物种注释数据库进行比对检测嵌合体序列,并最终去除其中的嵌合体序列[8],得到最终的有效数据(clean reads)。
使用Uparse v7.0.1001,http://drive5.com/uparse/,根据97%的相似度对序列进行操作单元(operational taxonomic unit, OTU)聚类;用Mothur方法与SILVA(http://www.arb-silva.de/)[9]的SSUrRNA数据库对OTUs代表序列进行物种注释分析,设置比对阈值为70%。以数据量最少的样品为标准均一化处理各样品的数据。
1.6 鸭肠道菌群多样性分析使用Qiime软件(Version 1.9.1)计算α多样性指数(observed-species,Chao1,Shannon,Simpson指数)和Unifrac距离,构建UPGMA样品聚类树;用R软件(Version 2.15.3)的ggplot2包进行PCoA分析。LEfSe分析使用LEfSe软件,默认设置LDA Score的筛选值为4。使用SAS9.0统计分析α多样性指数,单因素方差分析检验不同组别间差异,P < 0.05为差异有统计学意义。
2 结果 2.1 鸭不同肠段内容物多样性分析本研究共采集了80份肠道内容物样本,其中1份空肠内容物所提取细菌DNA不符合测序要求,共79份细菌DNA样本进行测序和后续分析。测序检测到10 331个OTUs,其中十二指肠、空肠、回肠和盲肠各有7 000、8 109、4 902、2 950个OTUs,不同肠段共有OTUs为2 057个,十二指肠、空肠、回肠和盲肠内容物中特有的OTUs分别为1 239、2 003、297、196个(图 1)。回肠和盲肠中总OTUs以及特有OTUs数量远少于十二指肠和空肠。
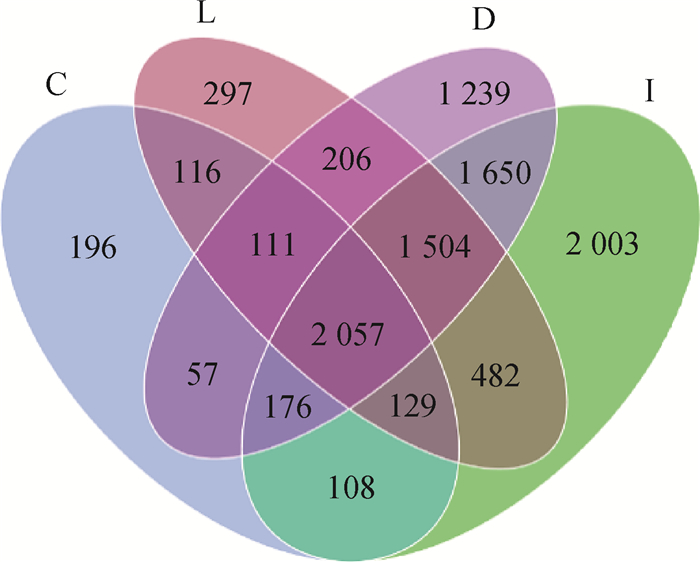
|
D.十二指肠;I.空肠;L.回肠;C.盲肠。下同 D. Duodenum; I. Jejunum; L. Ileum; C. Cecum. The same as below 图 1 十二指肠、空肠、回肠和盲肠内容物微生物OTUs花瓣图 Fig. 1 OTUs petaline graphs in duodenal, jejunal, ileal and caecal contents in duck |
样品复杂度分析α多样性结果显示(表 2),回肠和盲肠内容物中菌群丰度Chao1值和物种观测值(observed species)显著水平低于十二指肠和空肠(P < 0.05),前肠段(十二指肠和空肠)、后肠段(回肠和盲肠)内容物中菌群丰度差异不显著;而空肠内容物中菌群多样性指数Shannon值显著高于其他肠段(P < 0.05),回肠内容物菌群多样性指数Shannon和Simpson值则最低,显著低于空肠和盲肠(P < 0.05)。
|
|
表 2 鸭十二指肠、空肠、回肠和盲肠内容物菌群α多样性分析 Table 2 α diversity of microbiota in duodenal, jejunal, ileal and caecal contents in duck |
以加权UniFrac算法对菌群组成进行进化聚类分析,结果表明,不同肠段空间内肠道菌群结构有较大差异,十二指肠和空肠内容物菌群组成距离较近,而十二指肠和空肠与回肠、盲肠内容物中菌群组成距离较远(图 2A)。基于OTUs计算的加权Unifrac距离结果绘制的PCoA图(图 2B)所示结果与进化聚类分析结果类似,前肠段(十二指肠和空肠)内容物中群落结构差异较小,样本个体在PCoA图中重叠分布,但是十二指肠、空肠内容物与回肠和盲肠内容物的群落结构相似度较低,特别是盲肠内容物样本的微生物群落与其他肠段内容物样本有明显的区别分布。
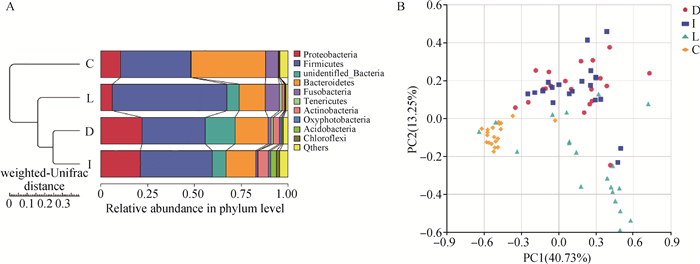
|
图 2 鸭不同肠段内容物菌群进化聚类分析(A)和加权Unifrac距离的PCoA分析(B) Fig. 2 Cluster analysis (A) and PCoA analysis (B) of microbiota from different intestinal segments in ducks based on weighted Unifrac distance |
鸭肠道内容物中细菌菌门相对丰度分析结果表明,在十二指肠、空肠、回肠和盲肠内容物中,优势菌门为拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和梭杆菌门(Fusobacteria),但优势菌门在不同肠段中表现出不同的相对分布,如表 3所示,厚壁菌门在回肠内容物中相对丰度最高(61.17%),其次为空肠(38.40%)、盲肠(37.52%)和十二指肠(33.81%);而回肠内容物中变形菌门相对丰度(6.19%)则小于十二指肠(22%)、空肠(21.16%)和盲肠(10.49%);盲肠内容物中拟杆菌门的相对丰度最高,达39.84%,远高于其他肠段(13.91%~17.70%),盲肠内容物中其他菌门如unidentified Bacteria、Actinobacteria、Acidobacteria、Chloroflexi相对丰度则小于其他肠段。
|
|
表 3 鸭十二指肠、空肠、回肠和盲肠内容物中主要菌门的相对丰度 Table 3 Relative abundance of top 10 phyla in duodenal, jejunal, ileal and caecal contents of duck |
主要细菌菌属在不同肠段内容物中也呈现出不同的丰度水平,相对丰度水平较高的10个优势菌属如表 4所示。和菌门水平结果一致,拟杆菌属在盲肠内容物中有最高相对丰度水平(29.12%),其在十二指肠、空肠和回肠内容物中的相对丰度水平比较一致(6.82%~7.74%);乳杆菌属(Lactobacillus)随着肠段的延伸,从十二指肠到盲肠,表现为相对丰度越来越小。螺杆菌属(Helicobacter)、肠球菌属(Enterococcus)在十二指肠、空肠和回肠内容物中有较高的相对丰度分布,盲肠内容物中多个主要菌属相对丰度小于其他肠段内容物。
|
|
表 4 鸭十二指肠、空肠、回肠和盲肠内容物中主要菌属的相对丰度 Table 4 Relative abundance of top 10 genus in duodenal, jejunal, ileal and caecal contents of duck |
LEfSe分析了在不同肠道内容物中显著差异的微生物物种。十二指肠、空肠、回肠和盲肠内容物中肠段环境特异的细菌菌门和菌科如图 3所示。十二指肠、空肠、回肠和盲肠内容物中差异菌门分别是变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)和拟杆菌门(Bacte- roidetes)。十二指肠中差异细菌科有螺杆菌科(Helicobacteraceae)、乳杆菌科(Lactobacillaceae)、肠杆菌科(Enterobacteriaceae)、Muribaculaceae、假单胞菌科(Pseudomonadaceae)、链球菌科(Streptococcaceae);空肠中差异细菌科有根瘤菌科(Rhizobiaceae)和伯克氏菌科(Burkholderiaceae);回肠中差异细菌科有Peptostreptococcaceae、不明梭菌科(unidentified Clostridiales)、肠球菌科(Enterococcaceae);盲肠中差异细菌科有拟杆菌科(Bacteroidaceae)、疣微菌科(Ruminococcaceae)、琥珀酸弧菌科(Succinivibrionaceae)、脱硫弧菌科(Desulfovibrionaceae)。
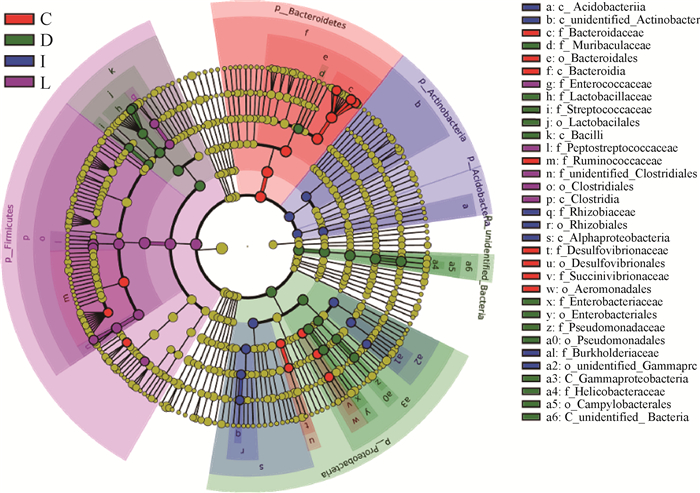
|
图 3 十二指肠、空肠、回肠和盲肠中差异物种进化分支图 Fig. 3 Evolutionary branching map of differential species in duodenal, jejunal, ileal and caecal contents of duck |
由鸭肠道内容物细菌门、属水平丰度分布结果发现,拟杆菌是健康鸭肠道内容物中的绝对优势菌,但是在不同肠段内容物中的相对丰度分布差异较大,在盲肠内容物中表现出最高的相对丰度。进一步在种(species)水平分析拟杆菌种,结果在健康鸭肠道内容物中共测序到28种拟杆菌种,具体拟杆菌种见表 5,相对丰度分布较高的为:B. caecigallinarum、B. plebeius和B. barnesiae,在盲肠内容物或者十二指肠、回肠内容物中的相对丰度大于1%,B. caecigallinarum在盲肠内容物中的相对丰度高达8.37%;其他有9种拟杆菌种如:B. acidifaciens、B. caccae、B. coprocola、B. coprophilus等在多个肠道中的相对丰度大于0.1%。
|
|
表 5 拟杆菌种在鸭肠道内容物中的平均相对丰度 Table 5 Relative abundance of Bacteroides in duodenal, jejunal, ileal and caecal contents of duck |
28种拟杆菌种在不同肠段内容物中相对丰度具有空间差异,其肠段相关性聚类结果如图 4所示。B2(B. acidifaciens)、B3(B. barnesiae)、B4(B. caccae)、B5(B. caecicola)、B8(B. coprocola)、B21(B. sp)和B14(B. luti)在盲肠内容物中显著聚类。
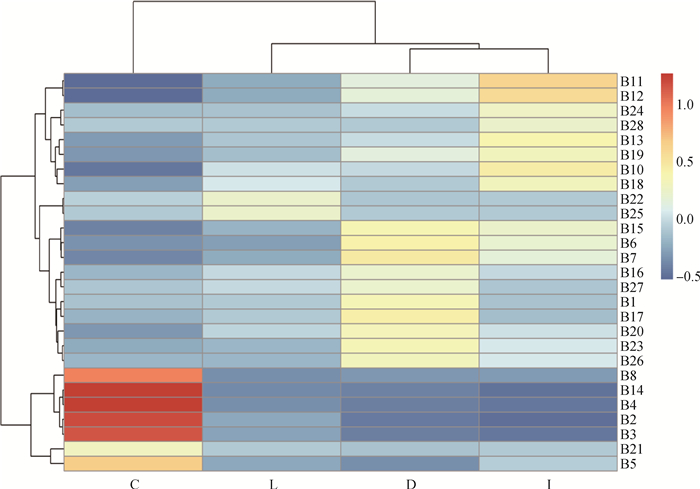
|
图 4 鸭肠道内容物中拟杆菌种聚类图 Fig. 4 Cluster diagram of Bacteroidetes species in duck intestinal contents |
本研究基于16S rRNA高通量测序方法分析了健康鸭肠道内容物中菌群组成多样性与菌群结构,结果发现,鸭肠道内容物中共发现10 331个OTUs,不同肠段内容物中OTUs数量不等,随着肠道结构深入,肠道内容物中的OTUs数量逐渐减少,且回肠和盲肠中特有的OTUs数量远少于十二指肠和空肠,初步表明,在健康鸭肠道内容物中肠段空间不同,其内容物中的微生物存在差异。赵远等[10]分析人工饲养恒河猴幼猴不同肠段微生物结构也发现,十二指肠内OTUs数目显著高于盲肠,与本研究结果类似。肠道内容物菌群复杂度分析和多样性比较分析结果表明,肠段空间差异显著影响菌群丰度,回肠和盲肠内容物中菌群丰度显著低于十二指肠和空肠(P < 0.05),而回肠内容物菌群的多样性最小。研究认为,动物消化道内菌群多样性从胃到粪便增加,前肠段,如胃、十二指肠内处于较低的酸性环境,不能保证细菌生长和维持更高的多样性[11]。李艳艳等[12]比较了野生树鼩和人工养殖树鼩各肠段菌群多样性,结果发现,野生树鼩α多样性结果与传统结论一致,后肠段盲肠菌群物种丰度最高,结肠次之;而人工养殖树鼩各肠段菌群多样性与之差别较大,空肠、回肠多样性较其他肠段高,十二指肠和盲肠次之,上述结论与本研究类似。这一结果可能与饲喂颗粒料的肠道环境有关,上述结果需要扩大鸭品种和数量进行验证,并设计试验进一步深入研究。
健康鸭肠道内容物中优势菌门为拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和梭杆菌门(Fusobacteria),这一结果和Best等[13]的报道一致,本研究中,上述4大菌门占十二指肠、空肠、回肠和盲肠内容物测序到菌门相对丰度总量的73.51%、75.48%、81.27%、87.85%。不同报道的主要菌门相对丰度值差异较大[14-16]。特别是随着细菌分类水平的降低,不同研究对细菌属、种相对丰度报道的相似性越低[17-19]。肠道内容物中菌群丰度结构受到宿主状态、环境、营养以及菌群结构本身等多种因素的影响,报道称营养结构的改变可以引起57%的肠道菌群发生改变,并且菌群的改变会从饮食变化的第2天开始且能持续维持,所以不同试验群体的菌群结构存在较大差异[20-21]。
健康鸭肠道内容物中的优势菌属为拟杆菌属(Bacteroides)、梭杆菌属(Fusobacterium)、乳杆菌属(Lactobacillus)、螺杆菌属(Helicobacter)、肠杆菌属(Enterococcus)等。就相对丰度分布分析结果可以看出,上述优势菌属的丰度分布也具有肠段空间差异,如耐酸性的兼性厌氧菌乳杆菌属(Lactobacillus)和肠球菌属(Enterococcus)在十二指肠、空肠和回肠内容物中相对丰度较高[22-23];而专性厌氧的拟杆菌属(Bacteroides)在健康鸭盲肠内容物中有最高的相对丰度,上述菌属丰度分布差异与不同肠段空间的内环境和肠段功能有关[24-25]。
随着肠道环境的改变,不同的共生菌会定植到不同的肠段中[26]。不同肠段空间中差异微生物物种分析结果与相对丰度分布结果一致,十二指肠中,其pH较小,呈酸性环境,与其他肠道内容物中差异显著的细菌菌门是变形菌门(Proteobacteria),以及螺杆菌、乳杆菌、肠杆菌、链球菌等兼性厌氧、且具一定耐酸能力的菌属种[27-28]。盲肠中差异细菌为拟杆菌科(Bacteroidaceae)、琥珀酸弧菌科(Succinivibrionaceae)、脱硫弧菌科(Desulfovibrionaceae)等。拟杆菌科和琥珀酸弧菌科中存在较多的多糖降解菌、淀粉降解菌和蛋白质降解菌[29-30],推测其在家禽盲肠特定的消化环境中发挥重要作用。
肠道微生态平衡的建立受宿主机体状态、生存环境、营养条件等多种因素的影响,肠道菌群微生态平衡建立的同时优势菌群的种类和数量就被固定下来。研究证实,拟杆菌在健康人的粪便中数量可达3.2×1010CFU[31],是健康人群粪便中的绝对优势菌;此外,拟杆菌在猪、牛、羊、兔、鸡、麻鸭、鲤鱼等众多物种肠道中也是优势菌群之一[32-34]。研究报道,拟杆菌在肠道中发挥重要功能,主要作用于类固醇、多糖和胆汁酸的代谢,有助于宿主对多糖的吸收以及蛋白质的合成[35];而且在宿主体内可以直接抑制其他有害菌群的黏附。研究发现,在菌群失调时重新引入拟杆菌,可以尽快使宿主肠道内微生态体系恢复平衡。禽类的盲肠是位于小肠和大肠之间的一对长形管道,在禽类中有着重要消化和免疫功能:细菌发酵、氮的再循坏、渗透压调节、营养的吸收、维生素的细菌合成和免疫反应等。随着肠道的深入,盲肠内严格厌氧,是拟杆菌适宜的生存环境,本研究结果显示,健康鸭盲肠内容物中拟杆菌属(Bacteroides)相对丰度显著高于其他肠段内容物[36-37]。
从十二指肠到盲肠的肠道内容物中,共测序到28种不同的拟杆菌种,但是这些拟杆菌种在不同的肠道内容物中相对丰度不同。对不同人群肠道中菌群宏基因组测序结果发现,人体肠道中丰度较高的拟杆菌种包括单形拟杆菌(B. uniformis)、P. merdae、粪拟杆菌(B.caccae)、多形拟杆菌(B. thetaiotaomicron)、普通拟杆菌(B.vulgatus)及吉氏副拟杆菌(P.distasonis),相对丰度均高于1%;相对丰度在1%以下的拟杆菌,如卵形拟杆菌(B.ovatus)、B.xylanisolvens、脆弱拟杆菌(B.fragilis)等属于相对低丰度的拟杆菌种[34]。鸭肠道内容物中的检测结果和其他物种如人粪便拟杆菌丰度分布有较大差异。鸭盲肠内容物中相对丰度最高的是B. caecigallinarum,高达8.37%,其次是B. plebeius和B. barnesiae,相对丰度分别为4.81%和4.01%。在十二指肠和回肠内容物中,B. caecigallinarum和B. plebeius相对丰度大于1%。有研究认为,日粮的纤维水平影响肠道菌群结构,食物中的膳食纤维,如抗性淀粉、果胶、菊粉等能显著上调拟杆菌门丰度[38-40]。中、高纤维日粮组健康力克斯兔盲肠中有益菌如双歧杆菌、乳酸杆菌、拟杆菌和消化球菌显著优于低纤维日粮组[41]。所以不同物种以及不同的营养结构可能是拟杆菌分类结构以及丰度分布差异的重要因素。
鸭盲肠中显著聚类的拟杆菌种有B. acidifaciens、B. barnesiae、B.caccae、B. caecicola、B. coprocola、B. sp和B. luti,其中仅粪拟杆菌B. caccae有少量文献报道[42-43],其他拟杆菌种的研究报道较少。禽类与其他脊椎动物相比,盲肠结构和功能有很大差异,禽类盲肠内容物中拟杆菌的生理生化功能是值得深入探究的一个重要方向。
4 结论本研究结果表明,鸭肠段空间显著影响肠道内容物中菌群丰度与多样性;健康鸭肠道内容物中优势菌门为拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria);优势菌属为拟杆菌属拟杆菌属(Bacteroides)、梭杆菌属(Fusobacterium);不同肠段内定植了差异的优势微生物物种,其相对丰度分布差异与其在肠道中功能相关,其中拟杆菌属(Bacteroides)在盲肠内容物中优势定植,推测拟杆菌属(Bacteroides)参与盲肠生理生化过程。本研究结果将为科学研究和生产中鸭肠道菌群的管理与调控提供基础数据。
| [1] | SENDER R, FUCHS S, MILO R. Revised estimates for the number of human and bacteria cells in the body[J]. PLoS Biol, 2016, 14(8): e1002533. DOI: 10.1371/journal.pbio.1002533 |
| [2] | RICKE S C, ROTHROCK JR M J. Gastrointestinal microbiomes of broilers and layer hens in alternative production systems[J]. Poult Sci, 2020, 99(2): 660–669. DOI: 10.1016/j.psj.2019.12.017 |
| [3] | RUBIO L A. Possibilities of early life programming in broiler chickens via intestinal microbiota modulation[J]. Poult Sci, 2019, 98(2): 695–706. DOI: 10.3382/ps/pey416 |
| [4] | CLAVIJO V, FLÓREZ M J V. The gastrointestinal microbiome and its association with the control of pathogens in broiler chicken production:A review[J]. Poult Sci, 2018, 97(3): 1006–1021. DOI: 10.3382/ps/pex359 |
| [5] | MCFALL-NGAI M, HADFIELD M G, BOSCH T C G, et al. Animals in a bacterial world, a new imperative for the life sciences[J]. Proc Natl Acad Sci U S A, 2013, 110(9): 3229–3236. DOI: 10.1073/pnas.1218525110 |
| [6] | SCHROEDER B O. Fight them or feed them:how the intestinal mucus layer manages the gut microbiota[J]. Gastroenterol Rep (Oxf), 2019, 7(1): 3–12. DOI: 10.1093/gastro/goy052 |
| [7] | EDGAR R C, HAAS B J, CLEMENTE J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194–2200. DOI: 10.1093/bioinformatics/btr381 |
| [8] | HAAS B J, GEVERS D, EARL A M, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Res, 2011, 21(3): 494–504. DOI: 10.1101/gr.112730.110 |
| [9] | WANG Q, GARRITY G M, TIEDJE J M, et al. Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Appl Environ Microbiol, 2007, 73(16): 5261–5267. DOI: 10.1128/AEM.00062-07 |
| [10] |
赵远, 杨凤梅, 禹文海, 等. 人工饲养恒河猴幼猴不同肠段微生物宏基因组学研究[J]. 中国微生态学杂志, 2017, 29(8): 869–873, 883.
ZHAO Y, YANG F M, YU W H, et al. Metagenomic analysis on microbiota in different parts of gut tracts of artificially reared baby Rhesus macaques[J]. Chinese Journal of Microecology, 2017, 29(8): 869–873, 883. (in Chinese) |
| [11] |
范慧宁, 瞿国强. 十二指肠球部和末端回肠黏膜菌群多样性分析[J]. 同济大学学报:医学版, 2020, 41(2): 228–234.
FAN H N, QU G Q. Mucosal microbiome diversity and composition in duodenal bulb and terminal ileum under normal and inflammatory conditions[J]. Journal of Tongji University:Medical Science, 2020, 41(2): 228–234. (in Chinese) |
| [12] |
李艳艳, 杨凤梅, 王俊斌, 等. 野生树鼩与人工饲养树鼩消化道不同部位微生物组成的比较研究[J]. 中国比较医学杂志, 2020, 30(4): 46–55.
LI Y Y, YANG F M, WANG J B. A comparative study of the microbial composition of different segments of the gastrointestinal tract in wild and captive tree shrews[J]. Chinese Journal of Comparative Medicine, 2020, 30(4): 46–55. (in Chinese) |
| [13] | BEST A A, PORTER A L, FRALEY S M, et al. Characterization of gut microbiome dynamics in developing Pekin Ducks and impact of management system[J]. Front Microbiol, 2017, 7: 2125. |
| [14] |
朱振, 徐琪, 赵文明, 等. 日粮添加微生态制剂对樱桃谷鸭肉品质、肠道菌群及其舍内NH3浓度的影响[J]. 中国家禽, 2012, 34(9): 29–32.
ZHU Z, XU Q, ZHAO W M, et al. Effect of probiotics on the meat quality, intestinal microflora of Cherry Valley and NH3 concentration in house[J]. China Poultry, 2012, 34(9): 29–32. (in Chinese) |
| [15] | WANG S M, CHEN L, HE M Z, et al. Different rearing conditions alter gut microbiota composition and host physiology in Shaoxing ducks[J]. Sci Rep, 2018, 8(1): 7387. DOI: 10.1038/s41598-018-25760-7 |
| [16] | ANGELAKIS E, RAOULT D. The increase of Lactobacillus species in the gut flora of newborn broiler chicks and ducks is associated with weight gain[J]. PLoS One, 2010, 5(5): e10463. DOI: 10.1371/journal.pone.0010463 |
| [17] |
梁恒之, 王波, 尹达菲, 等. 不同饲料转化率的北京鸭和枫叶鸭消化器官发育及盲肠微生物菌群的比较研究[J]. 中国畜牧杂志, 2013, 49(19): 42–46.
LIANG H Z, WANG B, YIN D F, et al. Comparisons of digestive organs development and cecal microflora between Pekin Duck and Maple Leaf Duck with different feed conversion efficiency[J]. Chinese Journal of Animal Science, 2013, 49(19): 42–46. (in Chinese) |
| [18] | ZHAO L L, YIN H C, LU T F, et al. Application of high-throughput sequencing for microbial diversity detection in feces of specific-pathogen-free ducks[J]. Poult Sci, 2018, 97(7): 2278–2286. DOI: 10.3382/ps/pex348 |
| [19] | DAI S J, ZHANG K Y, DING X M, et al. Effect of dietary non-phytate phosphorus levels on the diversity and structure of cecal microbiota in meat duck from 1 to 21 d of age[J]. Poult Sci, 2018, 97(7): 2441–2450. DOI: 10.3382/ps/pey090 |
| [20] | TURNBAUGH P J, RIDAURA V K, FAITH J J, et al. The effect of diet on the human gut microbiome:a metagenomic analysis in humanized gnotobiotic mice[J]. Sci Transl Med, 2009, 1(6): 6r. |
| [21] | ZHANG C H, ZHANG M H, WANG S Y, et al. Interactions between gut microbiota, host genetics and diet relevant to development of metabolic syndromes in mice[J]. ISME J, 2010, 4(2): 232–241. DOI: 10.1038/ismej.2009.112 |
| [22] |
阮忱.益生菌对无量山乌骨鸡雏鸡生产性能和肠道菌群的动态影响[D].昆明: 昆明理工大学, 2018.
RUAN C.The dynamic influence of probiotics on the growth performance and gut microbiota of Wuliang-shan Black-bone Chickens[D].Kunming: Kunming University of Science and Technology, 2018.(in Chinese) |
| [23] |
陶振阳, 张耀文, 曹忠洋, 等. 粪肠球菌对产蛋后期蛋鸡生产性能、蛋品质及肠道组织形态的影响[J]. 中国畜牧兽医, 2020, 47(5): 1413–1420.
TAO Z Y, ZHANG Y W, CAO Z Y, et al. Effects of Enterococcus faecalis on production performance, egg quality and intestinal morphology of laying hens during the late laying period[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(5): 1413–1420. (in Chinese) |
| [24] |
李娟娟, 王凤云, 唐旭东, 等. 肠道菌群失调与功能性消化不良的相关性研究[J]. 中国中西医结合消化杂志, 2019, 27(1): 77–81.
LI J J, WANG F Y, TANG X D, et al. The correlation between intestinal microbiota imbalance and functional dyspepsia[J]. Chinese Journal of Integrated Traditional and Western Medicine on Digestion, 2019, 27(1): 77–81. (in Chinese) |
| [25] |
张娟.丁酸梭菌对肉鸡生长性能、肠道生理及盲肠菌群的影响[D].杭州: 浙江大学, 2015.
ZHANG J.Effects of Clostridium butyricum on growth performance, intestinal physiology and cecal microflora in broiler chickens[D].Hangzhou: Zhejiang University, 2015.(in Chinese) |
| [26] | COSTEA P I, HILDEBRAND F, ARUMUGAM M, et al. Enterotypes in the landscape of gut microbial community composition[J]. Nat Microbiol, 2018, 3(1): 8–16. DOI: 10.1038/s41564-017-0072-8 |
| [27] |
赵东伟, 范建华, 赵宝华, 等. 益生菌和壳聚糖的不同组合对肉鸡生产和肠道内环境的影响[J]. 江苏农业科学, 2010(6): 352–354.
ZHAO D W, FANG J H, ZHAO B H, et al. Effects of different probiotics and chitosan combinations on production and intestinal enviroment of meat chickens[J]. Jiangsu Agricultural Sciences, 2010(6): 352–354. (in Chinese) |
| [28] |
钟航, 刘作兰, 王启贵, 等. 四川白鹅不同肠段菌群多样性比较研究[J]. 中国家禽, 2019, 41(8): 29–33.
ZHONG H, LIU Z L, WANG Q G, et al. Comparative study of bacteria diversity in different intestinal segments in Sichuan White goose[J]. China Poultry, 2019, 41(8): 29–33. (in Chinese) |
| [29] |
李子健, 李大彪, 高民, 等. 不同生理阶段荷斯坦奶牛瘤胃细菌多样性研究[J]. 动物营养学报, 2018, 30(8): 3017–3025.
LI Z J, LI D B, GAO M, et al. Rumen bacteria diversity in Holstein Dairy Cows at different physiological phases[J]. Chinese Journal of Animal Nutrition, 2018, 30(8): 3017–3025. (in Chinese) |
| [30] | PANASEVICH M R, MEERS G M, LINDEN M A, et al. High-fat, high-fructose, high-cholesterol feeding causes severe NASH and cecal microbiota dysbiosis in juvenile Ossabaw swine[J]. Am J Physiol Endocrinol Metab, 2018, 314(1): E78–E92. DOI: 10.1152/ajpendo.00015.2017 |
| [31] | ZITOMERSKY N L, COYNE M J, COMSTOCK L E. Longitudinal analysis of the prevalence, maintenance, and IgA response to species of the order Bacteroidales in the human gut[J]. Infect Immun, 2011, 79(5): 2012–2020. DOI: 10.1128/IAI.01348-10 |
| [32] | RYCHLIK I. Composition and function of chicken gut microbiota[J]. Animals (Basel), 2020, 10(1): 103. |
| [33] | CRESPO-PIAZUELO D, MIGURA-GARCIA L, ESTELLÉ J, et al. Association between the pig genome and its gut microbiota composition[J]. Sci Rep, 2019, 9(1): 8791. DOI: 10.1038/s41598-019-45066-6 |
| [34] | JOHNSON E L, HEAVER S L, WALTERS W A, et al. Microbiome and metabolic disease:revisiting the bacterial phylum Bacteroidetes[J]. J Mol Med, 2017, 95(1): 1–8. |
| [35] | MURPHY E F, COTTER P D, HEALY S, et al. Composition and energy harvesting capacity of the gut microbiota:relationship to diet, obesity and time in mouse models[J]. Gut, 2010, 59(12): 1635–1642. DOI: 10.1136/gut.2010.215665 |
| [36] |
王姝妹.不同养殖方式及添加益生菌对蛋鸭肠道微生物及其各项生理生化指标的影响[D].杭州: 浙江师范大学, 2018.
WANG S M.The effects of different rearing conditions and adding probiotics on gut microbiota and physiological and biochemical indexes in laying ducks[D].Hangzhou: Zhejiang Normal University, 2018.(in Chinese) |
| [37] |
谭惠子.健康人群肠道中低丰度拟杆菌的筛选及生理作用研究[D].无锡: 江南大学, 2019.
TAN H Z.Isolation and physiological function investigation of low-abundant Bacteroidales in healthy human intestine[D].Wuxi: Jiangnan University, 2019.(in Chinese) |
| [38] | MARTÍNEZ I, KIM J, DUFFY P R, et al. Resistant starches types 2 and 4 have differential effects on the composition of the fecal microbiota in human subjects[J]. PLoS One, 2010, 5(11): e15046. DOI: 10.1371/journal.pone.0015046 |
| [39] |
周海柱, 高云航, 徐博, 等. 日粮纤维水平对鹅盲肠细菌多样性的影响[J]. 畜牧兽医学报, 2018, 49(3): 565–571.
ZHOU H Z, GAO Y H, XU B, et al. Effects of different dietary fiber levels on bacteria diversity in cecum of goose[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(3): 565–571. (in Chinese) |
| [40] |
张玲, 李华, 赵瑶, 等. 短期饲喂两种类型纤维对生长猪后肠菌群结构和主要代谢产物的影响[J]. 四川农业大学学报, 2019, 37(1): 70–77.
ZHANG L, LI H, ZHAO Y, et al. The short-term feeding of two different dietary fibers affects the microbial community and main metabolites in the hindgut of growing pigs[J]. Journal of Sichuan Agricultural University, 2019, 37(1): 70–77. (in Chinese) |
| [41] |
谷子林, 李江, 张玉华, 等. 日粮粗纤维水平对断乳力克斯兔腹泻率、消化道pH和盲肠菌群的影响[J]. 中国农学通报, 2008, 24(2): 21–25.
GU Z L, LI J, ZHANG Y H, et al. Effect of crude fiber level on diarrhea, intestinal pH and caecal microbe of Rex rabbit[J]. Chinese Agricultural Science Bulletin, 2008, 24(2): 21–25. (in Chinese) |
| [42] | CHENG Z L, HUANG Y, WIE W, et al. Bloodstream infection caused by bacteroides caccae in a diabetic patient:a case report and review of the literature[J]. Clin Lab, 2019, 65(12): 2425–2427. |
| [43] | SIROTEK K, SLOVÁKOVÁ L, KOPEČNY J, et al. Fermentation of pectin and glucose, and activity of pectin-degrading enzymes in the rabbit caecal bacterium Bacteroides caccae[J]. Lett Appl Microbiol, 2004, 38(4): 327–332. DOI: 10.1111/j.1472-765X.2004.01492.x |



