2. 四川农业大学资源学院, 成都 611130
2. College of Resources, Sichuan Agricultural University, Chengdu 611130, China
动物肠道是动物机体中最重要、最复杂的微生态系统。肠道微生态系统主要由肠道微生物构成,大量研究表明肠道微生物与宿主的营养物代谢、能量获取、免疫系统发育以及炎症性肠病和肥胖等疾病密切相关[1-2]。作为肠道中的优势微生物,细菌对机体的健康和疾病发生有着重要影响[3]。肠道中真菌所占比例虽较小,但其可影响动物疾病的产生以及食草动物对纤维素的消化利用[4]。肠道微生物也是重要的耐药基因的储存库,由于肠道微生物之间关系密切,十分有利于耐药基因在不同细菌间水平传播,加速了耐药性的产生,增大了许多细菌性疾病的救治难度[5]。肠道寄生虫与微生物共享肠道栖息地,通过相互作用可影响宿主免疫系统的调节[3]。寄生虫病是严重危害动物健康、影响濒危动物保护的多发性疾病,寄生虫的主要危害表现为掠夺动物营养,使生长发育受阻、造成消化道病理损伤、产生代谢毒素、引发病原菌感染等[6-7]。目前,对大熊猫肠道寄生虫的报道不多,因此,研究大熊猫肠道内寄生虫的情况,对大熊猫寄生虫病的预防有着重要意义。
以往对大熊猫肠道微生物的研究,主要以大熊猫粪便为研究对象,以传统分离鉴定和宏基因组测序等方法为主[2-3, 8]。由于大量微生物不可培养,通过分离培养方法获得的微生物在种类、数量和功能上无法反映自然状态下微生物群落的真实情况。宏基因组学研究包括系统进化分子标记测序和宏基因组DNA测序,基于16S rRNA和ITS测序的微生物组分析由于快速、简单、样品制备和分析成本低、数据库容量大等特点,得到了广泛应用[9]。但该方法也存在一定的缺陷,如无法同时对细菌与真菌进行定量,不能将微生物与其功能联系起来。宏基因组DNA测序以特定环境下整个微生物群落作为研究对象,通过提取微生物总DNA进行研究,可同时获得细菌、真菌及病毒等物种组成和丰度信息,可进行微生物基因和功能层面的研究。但通过宏基因组学获得的微生物信息同时包括活的和死亡微生物,故不能如实反映微生物与其功能的关系。宏转录组兴起于宏基因组之后,相对于宏基因组,宏转录组是原位衡量宏基因组表达的一种方法,是以样品中的全部微生物RNA信息为分析对象,从整体水平上研究某一特定环境、特定时间点活性微生物种类,通过测定基因转录来描述基因的活性及分析它们的代谢路径和功能表达等[10-11]。
目前,宏转录组技术已应用于肠道、食品、土壤等微生物群落和功能研究[11-12],但尚无利用宏转录组针对大熊猫粪便中微生物的研究。鉴于此,本研究利用宏转录组学技术,对大熊猫粪便中微生物菌群、耐药基因和寄生虫组成情况进行研究,分析微生物菌群功能表达以及菌群与耐药基因的相关性,可为了解大熊猫肠道群菌功能、大熊猫疾病的合理用药及寄生虫病的防治提供依据。
1 材料与方法 1.1 样品采集在中国大熊猫保护研究中心神树坪基地,采集3只健康雄性大熊猫(编号M1~M3,分别为6、9、11岁)和3只健康雌性大熊猫(编号F1~F3,分别为6、8、9岁)的新鲜粪便样品。使用无菌一次性手套,在大熊猫饲养内圈中采集黏液明显的新鲜整块粪便,每只大熊猫采集3份重复样品,迅速冻于液氮中,于6 h内送回实验室,并在无菌条件下进行RNA提取。
1.2 总RNA提取用百泰克粪便总RNA快速提取试剂盒提取样品中的总RNA,具体操作步骤参考说明书。用每只大熊猫采集的3份重复样品混合后提取RNA。采用NanoDrop2000、Agilent 2100分析仪对总RNA样品的纯度、浓度进行检测。
1.3 宏转录组测序将检测合格的样品送至北京诺禾致源科技股份有限公司采用Illumina Hiseq 2000测序平台进行高通量测序。从总RNA中去除核糖体RNA,从而得到mRNA。随后在NEB Fragmentation Buffer中用二价阳离子将得到的mRNA随机打断,按照NEB普通建库方式或链特异性建库方式进行建库。文库构建完成后,先使用Qubit2.0 Fluorometer进行初步定量,稀释文库至1.5 ng·μL-1,随后使用Agilent 2100 bioanalyzer对文库的insert size进行检测,用qRT-PCR对文库有效浓度进行准确定量(文库有效浓度高于2 nmol·L-1)。库检合格后,把不同文库按照有效浓度(大于2 nmol·L-1)及目标下机数据量(5G)的需求pooling后进行Illumina测序。
有效浓度:大于2 nmol·L-1,测序数据量5G(clean data)。
1.4 数据分析针对样品经预处理得到的clean reads,使用TopHat v2.0.10软件将得到的clean reads与UCSC数据库中大熊猫参考基因组序列进行比对,去除相似性高的污染reads。使用NCBI的rRNA、tRNA以及SILVA数据库进行比对分离出来宏基因组中rRNA序列,剩下的mRNA序列则使用拼接软Trinity[13]分别进行从头组装,然后对所有样品的序列整合并使CD-HIT-EST去冗余(设定序列一致性阈值为0.95),得到unigenes集合。使用DIAMOND软件将unigenes与eggNOG、GO和KEGG数据库进行比对(blastp,Evalue≤0.000 01),获得各自对应的分类信息和注释。比对雄性(M)和雌性(F)两组间的差异表达unigenes情况,差异unigenes筛选条件为Padj < 0.05。筛选出的差异unigenes采用GOseq和KOBAS软件进行GO功能富集和KEGG通路富集分析。使用DIAMOND软件将unigenes与从NCBI的NR数据库中抽提出的序列进行比对(blastp,evalue≤0.000 01), 获得对应物种信息及在各个分类水平物种丰度情况。采用wormbase parasite数据库[3]和抗生素耐药基因数据库(the Comprehensive Antibiotic Research Database, CARD)[14]分别对寄生虫和耐药基因进行比对。使用R软件进行Spearman分析,研究物种与耐药基因的相关性。
2 结果 2.1 Unigenes功能分析2.1.1 Unigenes功能注释分析 经Trinity拼接后共得283 377个unigenes,共有194 368个unigenes被注释到24个已知COG功能分类中,其中,最多的三类为一般基因功能(general function prediction only,35 793个unigenes)、碳水化合物运输以及代谢(carbohydrate transport and metabolism,20 516个unigenes)以及氨基酸运输和代谢(amino acid transport and metabolism,15 295个unigenes)。
通过GO功能分类预测,发现生物过程(biological process)包含的分支和unigenes最多,共有25个分支和315 714个unigenes。细胞合成(cellular component)有20个分支和112 497个unigenes,分子功能(molecular function)有10个分支和160 200个unigenes。在生物过程中,功能注释为代谢过程(metabolic process)、细胞过程(cellular process)和单一组织过程(single-organism process)的unigenes最多,在细胞合成中细胞(cell)、细胞组分(cell part)和膜(membrane)的unigenes最多,在分子功能中催化活性(catalytic activity)、连接(binding)和转运活性(transporter activity)的unigenes最多。
KEGG分析显示,共有126 725个unigenes被注释到6个代谢通路大类40个亚类中。在6个大类中,涉及unigenes最多的为代谢(metabolism,83 747个unigenes),最少的为生物系统(organismal systems,3 177个unigenes),遗传信息处理(genetic information processing)、环境信息处理(environmental information processing)、疾病(diseases)和细胞过程(cellular processes)分别包含14 470、13 491、7 414和4 426个unigenes。在亚类代谢通路中,涉及基因最多的通路主要有碳水化合物代谢(carbohydrate metabolism,21 184个unigenes)、概述(overview,14 049个unigenes)、氨基酸代谢(amino acid metabolism,11 695个unigenes)。
2.1.2 差异unigenes富集分析 对雄性和雌性大熊猫中unigenes进行了差异表达分析,结果表明有45个显著性差异表达unigenes,在雄性大熊猫中上调和下调差异表达unigenes分别有20和25个。进一步对差异表达unigenes进行GO和KEGG富集分析,未发现差异unigenes被注释到GO功能中,但发现共有12个差异unigenes被注释到10个KEGG生物学通路中。由表 1可知,这些生物学通路主要被划分到氨基糖与核苷酸糖代谢(amino sugar and nucleotide sugar metabolism)、抗坏血酸与醛酸代谢(ascorbate and aldarate metabolism)和叶酸一碳单位循环代谢(one carbon pool by folate)等6个新陈代谢相关的通路中。分析发现,叶酸一碳单位循环代谢、果糖与甘露糖代谢(fructose and mannose metabolism)、原核生物碳固定途径(carbon fixation pathways in prokaryotes)和碳代谢(carbon metabolism)中注释到的4个差异unigenes在雄性大熊猫中表现为上调,其余通路中的差异unigenes表现为下调。
|
|
表 1 雄性与雌性大熊猫差异表达unigenes KEGG通路富集情况 Table 1 KEGG pathway enrichment of differentially expressed unigenes between male and female sub-adult giant pandas |
共有189 524个unigenes经物种注释获得结果,得3界41门82纲161目300科573属。其中在门水平由32个细菌、8个真菌和1个病毒组成,属水平由436个细菌、78个真菌和51个病毒组成。在门分类水平上,相对丰度前10的物种如图 1a所示,其相对丰度在雄性组(M1~M3)和雌性组(F1~F3)没有显著性差异(P>0.05)。
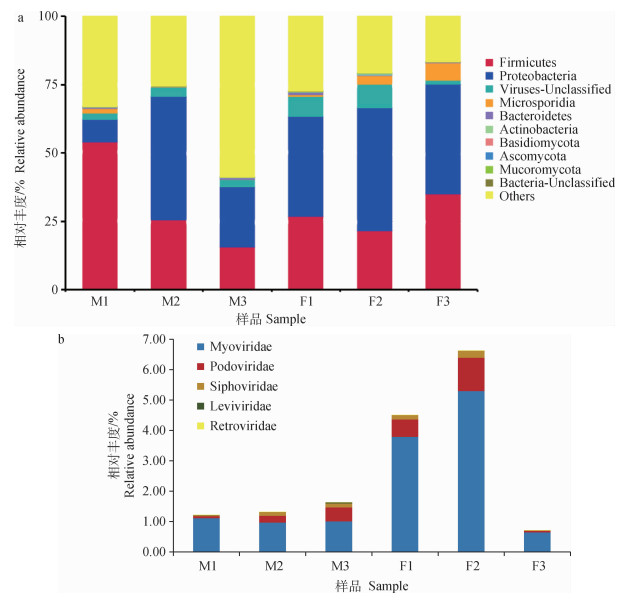
|
图 1 门(a)、科(b)水平大熊猫粪便内微生物相对丰度情况 Fig. 1 Relative abundance of microbial in the feces of giant pandas at phylum(a) and family (b) level |
在门水平,各样品中物种主要由细菌组成(41.12%~83.31%),厚壁菌门(15.67%~53.96%)和变形菌门(8.30%~45.30%)是成年大熊猫粪便中细菌菌群的两大主要门,其次为拟杆菌门(Bacteroidetes)(0.10%~0.83%)和放线菌门(Actinobacteria)(0.07%~0.35%)。圈养大熊猫粪便内均含有相对丰度含量较高的真菌为担子菌门(0.003%~0.07%)、子囊菌门(0.01%~0.05%)和毛霉亚门(0.001%~0.04%)。
在属分类水平,总相对丰度大于1%的物种见表 2,其相对丰度在雄性组(M1~M3)和雌性组(F1~F3)没有显著性差异(P>0.05),物种由6个细菌菌属组成。梭菌(Clostridium)和链球菌在样品M2、F1、F2和F3中所占比列较高,在样品M1和M3中梭菌和Intestinibacter所占比列较高。结果显示,细菌梭菌和链球菌为6只大熊猫粪便内的优势物种,平均相对丰度较高。
|
|
表 2 属水平大熊猫粪便微生物相对丰度情况 Table 2 Relative abundance of microbial in the feces of giant pandas at genus level |
由于病毒相对丰度较小(属水平)且获得注释较少,因此在科水平筛选所有样种品共有且总相对丰度大于0.01%的病毒进行分析。如图 1b所示,共有5个病毒科,其相对丰度在雄性组(M1~M3)和雌性组(F1~F3)没有显著性差异(P>0.05),肌尾噬菌体科(Myoviridae)在各样品中相对丰度最高(0.64%~5.30%),其次为短尾噬菌体科(Podoviridae)(0.06%~1.10%)和长尾噬菌体科(Siphoviridae)(0.01%~0.23%)。光滑噬菌体科(Leviviridae)和反录病毒科(Retroviridae)在各样品种相对丰度较低,其总相对丰度分别为0.04%和0.02%。
2.3 大熊猫粪便中耐药基因组成经CARD数据库比对,共发现304种抗生素耐药基因,其中有217种耐药基因为所有样品共有。分析发现大熊猫粪便内耐药基因的耐药类型多样,可分为25大类,耐药基因种类最多的为外排泵类(n=125)、糖肽类(n=38)、四环素类(n=23)、β-内酰胺类(n=18)和喹诺酮类(n=17)。相对丰度排名前10耐药基因大类如表 3所示,各样品均显示外排泵类耐药基因相对丰度最高(77 860~166 484 ppm),此外, 多肽类(10 233~34 651 ppm)、elfamycin类(9 848~29 535 ppm)、β-内酰胺类(6 888~27 915 ppm)和喹诺酮类(7 755~18 783 ppm)耐药基因样品中的相对丰度也较高。图 2所示为相对丰度前10的耐药基因,在这些耐药基因种有5个属于外排泵类(patA、macB、evgA、bcr-1和mdtF),1个elfamycin类(Streptomyces_cinnamoneus_EF-Tu),1个多肽类(pmrF),1个β-内酰胺类(nmcR),1个大环内酯类(chrB)和1个利福霉素类(rphB), 其中Streptomyces_cinnamoneus_EF-Tu(115 744 ppm)在各个样本中的相对丰度之和最高,其次为patA(78 557ppm)和nmcR(75 543 ppm)。
|
|
表 3 大熊猫粪便内抗生素耐药基因类型分布情况 Table 3 Distributions of antibiotic resistance gene types detected in the feces of giant pandas |
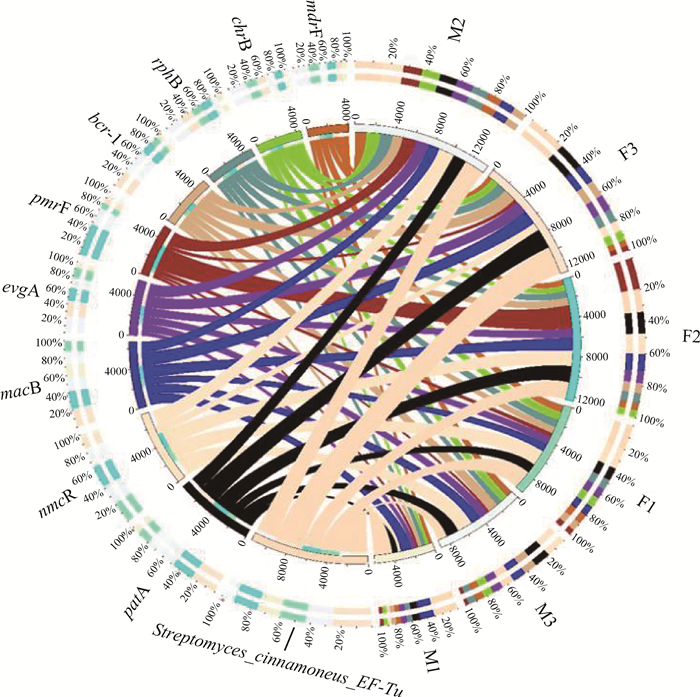
|
圈图分为两个部分,右侧为样品,左侧为耐药基因(ARGs)。内圈不同颜色表示不同的样品和ARGs,左侧为某ARG在各个样本中的相对丰度之和,右侧为各ARG在某样本中的相对丰度之和;外圈左侧为某ARG中各个样品的相对百分含量,外圈右侧为某样品中各ARG的相对百分含量 The different colors of the inner ring represent different samples and ARGs, is the sum of relative abundance in each sample of an ARG, and the right side is the sum of relative abundance of each ARG in a sample. The left side of the outer ring is the relative percentage of each sample in a certain ARG, and the right side of the outer ring is the relative percentage of each ARG in a certain sample 图 2 大熊猫粪便内主要抗生素耐药基因分布Circos图 Fig. 2 Circos representation of antibiotic resistance gene detected in the feces of giant pandas |
经Spearman相关性分析,发现部分细菌属水平优势物种与耐药基因具有显著相关性[Padj (P值经过多重校验校正后的值) < 0.001或 < 0.05]。属水平物种与耐药基因大类及优势耐药基因的相关性分别如图 3和图 4所示,总体来看,所有耐药基因大类和优势耐药基因与链球菌、埃希杆菌、肠球菌和Turicibacter属均呈正相关,与Intestinibacter均呈负相关。其中,与埃希杆菌呈显著正相关的耐药基因大类最多,包括外排泵、喹诺酮类、四环素类、糖肽类和磺胺类,埃希杆菌与基因patA呈显著正相关。此外,肠球菌与喹诺酮类和糖肽类耐药基因,链球菌与多肽类和磺胺类耐药基因具有显著正相关性。
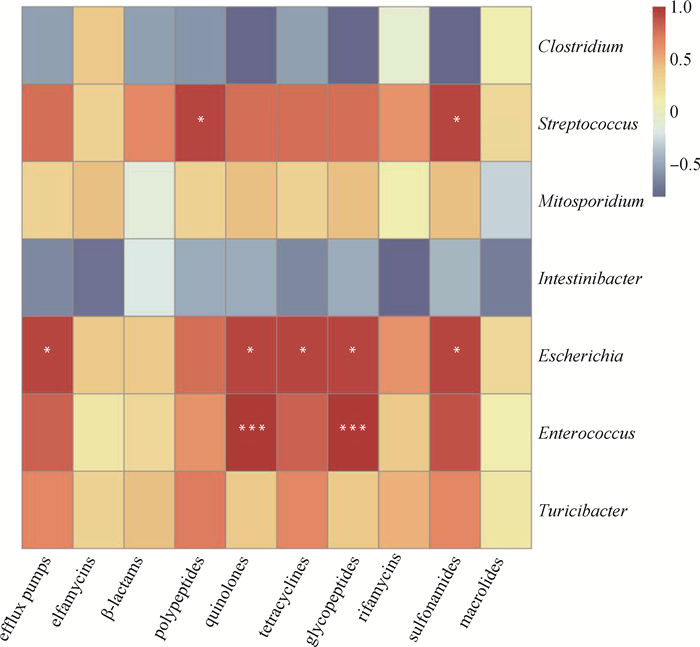
|
***.Padj < 0.001; *.Padj < 0.05 图 3 细菌(属水平)与耐药基因大类的相关性 Fig. 3 The correlations between bacteria at genus level and antibiotic resistance gene types |
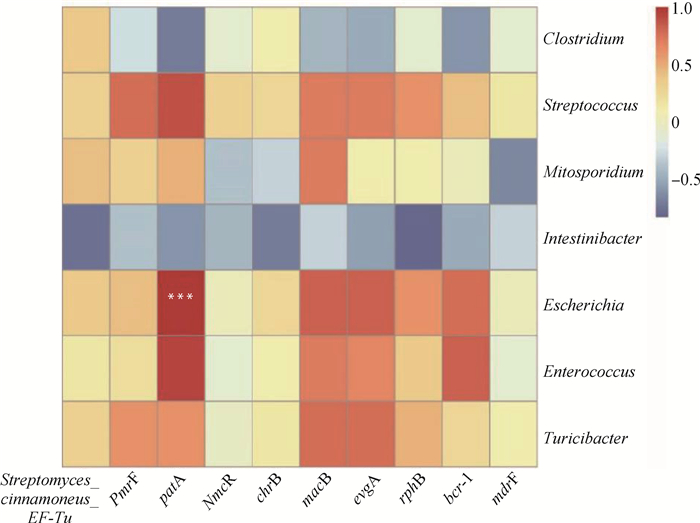
|
***. Padj < 0.001 图 4 细菌(属水平)与耐药基因的相关性 Fig. 4 The correlations between bacteria at genus level and antibiotic resistance genes |
通过对所有样品中氨基酸序列进行比对,共发现63属126种寄生虫。对主要寄生虫属(包含寄生虫种≥3)分析(表 4),共发现13属67种寄生虫,其中线虫种类最多,如北方根结线虫(Meloidogyne hapla)、蝼蛄斯氏线虫(Steinernema scapterisci)、猪蛔虫(Ascaris suum)等共51种。此外还包括绦虫猪带绦虫(Taenia solium)、牛带绦虫(T. saginata)和细粒刺球绦虫(Echinococcus granulosus)等10种以及吸虫曼氏血吸虫(Schistosoma mansoni)和埃及血吸虫(S. haematobium)等6种。在属分类水平上,毛线虫属(Trichinella)中检测到的寄生虫种最多,包括伪旋毛虫(T. pseudospiralis)、本地毛形线虫(T. nativ)和纳氏旋毛虫(T. nelsoni)等12种,其次为隐杆线虫属(Caenorhabditis),包括秀丽隐杆线虫(C. elegans)、双桅隐杆线虫(C. briggsae)和C. sp.34等11种。
|
|
表 4 大熊猫粪便中寄生虫组成 Table 4 Species of parasites in the feces of giant pandas |
由于大熊猫这一物种的特殊性,难以直接从肠道内获取样品,因此,常以大熊猫粪便为试验样品,对其肠道微生物进行分析[2-3, 8]。通过对宏转录组文库中的unigenes进行功能注释和富集分析,从基因层面分析了成年大熊猫粪便中微生物功能的整体表达特征。分析显示3个数据库结果基本一致,即成年大熊猫肠道微生物的功能主要涉及代谢过程,如COG分类中的碳水化合物运输、代谢与氨基酸运输和代谢,GO功能注释中的代谢过程,以及KEGG通路中的碳水化合物代谢和氨基酸代谢等,此结果提示成年大熊猫的代谢活动较强。相关报道也表明,动物肠道微生物能够通过生化修饰参与营养物质的分解与加工促进宿主营养代谢, 哺乳动物血液中10%的代谢产物来自肠道微生物[15-16],而碳水化合物代谢和蛋白代谢这类代谢活动的优势地位在仔猪肠道微生物转录组学研究中也被发现[17]。通过富集分析发现少量雄性和雌雄大熊猫间的差异unigenes被注释到KEGG通路的代谢相关通路中,表明雄性和雌雄大熊猫肠道微生物在代谢方面存在一定差异,这可能与雌雄大熊猫肠道菌群结构差异有关。此外,Baars等[18]研究发现雌雄生理过程受到不同的调节,不同性别小鼠的脂质代谢也受到肠道菌群的影响,其中雌雄胆汁酸水平的差异也可能影响导致脂质代谢的性别差异。
由于大熊猫粪便刚排出时,粪便表面一般有一层黏液包裹,可使松散的竹子残渣粘成一团[19],采集黏液明显的粪便可确保样品的新鲜,同时, 可在一定程度上避免粪便样品被环境污染。转录组结果显示,大熊猫肠道内微生物种类多样,包括了细菌、真菌和病毒,且以细菌为主。发现雌性和雄性大熊猫肠道微生物没有显著性差异(P>0.05),与相关报道一致[20],可见性别差异对大熊猫肠道微生物无显著影响。在门水平上,以厚壁菌门和变形菌门为主。目前,利用转录组学技术对动物胃肠道菌群的研究较少,Neves等[21]通过转录组学研究发现公牛瘤胃中的优势菌门为拟杆菌门、厚壁菌门和变形菌门,约占细菌总丰度的80%。对人体肠道转录组研究发现厚壁菌门(49.18%)和拟杆菌门(31.42%)的丰度较高[22]。而对大熊猫肠道微生物的转录组学研究还未见报道,但部分学者通过不同研究方法也得出了相似结果,如Fang等[23]通过RFLP分析了大熊猫肠道内优势菌门,Tun等[8]通过16S rRNA高通量测序检测成年和老年大熊猫肠道内菌群,Yang等[3]通过宏基因组测序方法对圈养、野化培训和野生大熊猫肠道菌群进行了研究,均发现厚壁菌门和变形菌门为大熊猫肠道内的优势菌门。在属水平上,以梭菌属、链球菌属和Intestinibacter属的细菌为主,其中梭菌包含纤维素和半纤维素消化酶的编码基因,有助于大熊猫消化利用竹子中的消化纤维素与半纤维素[24]。目前,对Intestinibacter属的细菌相关研究还较少,对人体肠道内Intestinibacter相关功能分析显示,它能抵抗氧化应激、降解岩藻糖,还原硫酸盐,还能间接参与黏液降解[25]。已有学者通过高通量测序检测到亚成年大熊猫肠道内以梭菌(39.68%)、埃希杆菌(Escherichia)(20.94%)、明串珠菌(Leuconostoc)(8.75%)、假单胞菌(Pseudomonas)(5.08%)和刺骨鱼菌(Epulopiscium)(4.69%)属为主[2],而Yang等[3]通过宏基因组测序发现大熊猫肠道内优势菌属包括埃希杆菌(41.1%)、链球菌(15.6%)、假单胞菌(10.7%)、耶尔森菌(Yersinia) (8.9%)、乳球菌(Lactococcus) (4.8%)。这与本试验结果有一定差异,可能与检测方法有关。通过宏基因组学测序可获得样品采集时活性和死亡微生物信息,而宏转录测序以样品中全部活性微生物为研究对象,更能真实、全面地反映出特定时期样品中高表达活性的微生物。
担子菌门、子囊菌门和毛霉亚门的真菌在大熊猫肠道内的相对丰度较高,这些菌门也在大熊猫肠道真菌相关的研究中被发现为优势菌门[2-3, 8]。与细菌相比,真菌在肠道内所占比例较小,但肠道真菌可能影响肠道细菌菌群结构,改变肠道内关键代谢物的产生和消耗,甚至影响宿主免疫发育和造成疾病[1, 26-27]。Sokol等[28]研究发现,炎症性肠病患者和健康对照中的优势菌门均为担子菌门和子囊菌门,但炎症性肠病患者肠道内担子菌门和子囊菌门比例明显升高,表明真菌群落结构的改变可能由炎症引起或参与了炎症发生过程。此外,在大熊猫肠道内发现的病毒以噬菌体为主,包括肌尾噬菌体科、短尾噬菌体科、长尾噬菌体科和光滑噬菌体科的噬菌体,这与Yang等[3]对大熊猫肠道内噬菌体研究结果相似。这些噬菌体以细菌为主要宿主,并对肠道菌群结构起着调节作用[29],研究表明肠道噬菌体组与人体某些疾病有着密切关系,同时噬菌体可用于人和动物的细菌感染防治[30]。因此,对大熊猫肠道噬菌体的研究可为大熊猫疾病的诊断和治疗提供新思路。
圈养大熊猫由于使用抗生素治疗疾病,与人类接触以及耐药菌株传播等因素,促使其肠道菌群获得大量抗生素耐药基因[31]。本研究在不同大熊猫肠道内菌群中检测出大量的共有耐药基因,提示应重视耐药性的水平传播。在耐药基因大类中,外排泵基因的种类最多、相对丰度最高。同时,通过全基因组测序发现大熊猫源大肠杆菌中耐药基因以外排泵基因为主[32],表明外排泵系统可能为大熊猫产生耐药性的主要因素。多肽类和elfamycins类耐药基因也具有较高的相对丰度。其中,多肽类耐药基因pmrF参与合成并连接氨基阿拉伯糖到革兰阴性菌外膜的脂类A上, 增加外膜的正电荷电量以提高对阳离子抗菌肽的抗性,可导致细菌对多黏菌素耐药性的产生[33]。目前,关于elfamycins类耐药在大熊猫肠道菌群中的研究还较少,elfamycins类为窄谱抗生素,包括phenelfamycins、kirromycin和pulvomycin等,这类抗生素通过与蛋白质合成的延伸因子EF-Tu结合从而抑制细菌蛋白质合成、阻碍细菌生长,而耐药基因Streptomyces_cinnamoneus_EF-Tu在各个样本中的相对丰度之和最高,可以通过影响延长因子EF-Tu从而使细菌产生抗生素抗性[34-35]。此外,四环素类、β-内酰胺类和喹诺酮类耐药基因具有较多种类和较高相对丰度,其在大熊猫源细菌耐药性研究中均有报道,为大熊猫体内细菌常见的耐药基因类型[32, 36]。通过对耐药基因与物种丰度进行相关性分析,发现耐药基因大类及优势耐药基因与大部分优势菌属呈正相关,提示这些菌属为耐药基因的宿主菌,介导相关抗生素耐药性。此外,部分抗生素大类与链球菌、埃希杆菌和肠球菌属呈显著正相关性,已有相关报道指出链球菌和埃希杆菌为大熊猫潜在致病菌[36-37],因此,在治疗大熊猫相关细菌性疾病时,应选择对病原菌耐药性较低的抗生素进行治疗,同时应合理用量,控制耐药性的增加。
由于活动空间相对较小、同一生境伴生动物交叉传播、人类介入以及食物等影响因素,使圈养大熊猫肠道寄生虫的感染较为普遍,而胃肠道寄生虫病是危害大熊猫的一类常见、多发性疾病。本研究发现大熊猫肠道中优势虫种为线虫,这些寄生虫可引起钩虫病、丝虫病和类圆线虫病等多种寄生虫病[38]。其中,钩虫是一种最常见的由土壤传播的寄生虫,可引起哺乳动物严重的缺铁性贫血和蛋白营养不良症[39]。目前,已有学者在野生大熊猫的肠道内多次发现钩虫属线虫,推测其为寄生于大熊猫的固定虫种[40]。类圆线虫病可威胁人类和动物的健康,研究发现90%的类圆线虫病由粪类圆线虫(S. stercoralis)引起,可导致机体胃肠道和肺部疾病的发生[41]。感染乳突类圆线虫(S. papillosus)后的动物常出现腹泻、贫血、厌食等症状,严重时可引起麻痹性肠梗阻[42]。蛔虫是大熊猫肠道内一种常见的大型寄生线虫,冯文和张安居[43]对野外大熊猫死亡原因进行分析,得出野生大熊猫蛔虫感染率为100%,大熊猫感染蛔虫的直接、间接死亡率为66.67%,严重危及大熊猫的生存。目前对大熊猫肠道内绦虫和吸虫以及其引起的相关疾病报道不多,但由棘球属绦虫引起的棘球蚴病被列为我国三大人兽共患寄生虫病之一[44]。因此,在圈养大熊猫的饲养和管理过程中,应保证食物、圈舍及周围环境的清洁卫生,及时妥善处理粪便。此外,通过对寄生虫的监测,并根据各种寄生虫的生长发育规律做好驱虫工作,可有效防治大熊猫寄生虫病。尽管通过序列比对可获得大量寄生虫信息,但部分寄生虫在幼虫期存在于中间宿主(如蚂蚁、甲壳虫和其他软体动物)中[3, 45],这些中间宿主在进入大熊猫肠道后可能立即排出,并未定植于大熊猫肠道,因此,在下一步研究中可结合离心沉淀法、饱和盐水漂浮法以及免疫荧光检测技术等对大熊猫肠道寄生虫种类进行确认。
4 结论利用宏转录组学技术对圈养成年大熊猫粪便中的微生物组成及功能进行了研究,结果表明,大熊猫粪便中微生物参与相关代谢活动,性别对成年大熊猫粪便微生物组成无显著影响。通过与数据库比对,阐明了对大熊猫粪便中耐药基因和寄生虫种类与丰度情况。目前,通过宏转录组学对大熊猫粪便中微生物的研究还较少,本研究结果可为大熊猫源微生物研究提供基础数据,为大熊猫疾病的合理用药及寄生虫病的防治提供依据。
| [1] | SAM Q H, CHANG M W, CHAI L Y A. The fungal mycobiome and its interaction with gut bacteria in the host[J]. Int J Mol Sci, 2017, 18(2): 330. DOI: 10.3390/ijms18020330 |
| [2] |
晋蕾, 周应敏, 李才武, 等. 野化培训与放归、野生大熊猫肠道菌群的组成和变化[J]. 应用与环境生物学报, 2019, 25(2): 344–350.
JIN L, ZHOU Y M, LI C W, et al. Composition and variation of gut microbiome of trained, preparatory reintroduced, reintroduced and wild giant pandas[J]. Chinese Journal of Applied and Environmental Biology, 2019, 25(2): 344–350. (in Chinese) |
| [3] | YANG S Z, GAO X, MENG J H, et al. Metagenomic analysis of bacteria, fungi, bacteriophages, and helminths in the gut of giant pandas[J]. Front Microbiol, 2018, 9: 1717. DOI: 10.3389/fmicb.2018.01717 |
| [4] | KELLY D, CONWAY S, AMINOV R. Commensal gut bacteria: mechanisms of immune modulation[J]. Trends Immunol, 2005, 26(6): 326–333. DOI: 10.1016/j.it.2005.04.008 |
| [5] |
高琰宇, 毕文静, 吴新颜, 等. 细菌耐药影响肠道菌群及其宿主免疫调控[J]. 生物工程学报, 2018, 34(8): 1259–1269.
GAO Y Y, BI W J, WU X Y, et al. Bacterial resistance influences intestinal flora and host immune regulation[J]. Chinese Journal of Biotechnology, 2018, 34(8): 1259–1269. (in Chinese) |
| [6] | ZHANG L, YANG X Y, WU H, et al. The parasites of giant pandas: individual-based measurement in wild animals[J]. J Wildlife Dis, 2011, 47(1): 164–171. DOI: 10.7589/0090-3558-47.1.164 |
| [7] |
徐梦, 沈玉娟. 蠕虫及肠道原虫感染与肠道菌群关系研究进展[J]. 中国血吸虫病防治杂志, 2019, 31(1): 77–85, 93.
XU M, SHEN Y J. Progress of research on the interplay between helminth and intestinal proto-zoa and gut microbiota[J]. Chinese Journal of Schistosomiasis Control, 2019, 31(1): 77–85, 93. (in Chinese) |
| [8] | TUN H M, MAUROO N F, YUEN C S, et al. Microbial diversity and evidence of novel homoacetogens in the gut of both geriatric and adult giant pandas (Ailuropoda melanoleuca)[J]. PLoS One, 2014, 9(1): e79902. DOI: 10.1371/journal.pone.0079902 |
| [9] |
余泽晖, 梁志伟, 李浩聪, 等. 厌氧消化污泥微生物组研究方法及应用[J]. 微生物学通报, 2019, 46(8): 2053–2068.
YU Z H, LIANG Z W, LI H C, et al. Anaerobic sludge digestion microbiome - analytical methods and applications[J]. Microbiology China, 2019, 46(8): 2053–2068. (in Chinese) |
| [10] |
牛化欣, 常杰, 胡宗福, 等. 基于组学技术研究反刍动物瘤胃微生物及其代谢功能的进展[J]. 畜牧兽医学报, 2019, 50(6): 1113–1122.
NIU H X, CHANG J, HU Z F, et al. Research progress on rumen microbiomes and their metabolic functions in ruminants based on omics technology[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(6): 1113–1122. (in Chinese) |
| [11] |
马海霞, 张丽丽, 孙晓萌, 等. 基于宏组学方法认识微生物群落及其功能[J]. 微生物学通报, 2015, 42(5): 902–912.
MA H X, ZHANG L L, SUN X M, et al. Understanding microbial communities and their functions by meta-omics approaches[J]. Microbiology China, 2015, 42(5): 902–912. (in Chinese) |
| [12] |
张国华, 王伟, 涂建, 等. 基于宏转录组学技术解析传统酸面团中微生物代谢机理[J]. 中国粮油学报, 2019, 34(11): 10–16.
ZHANG G H, WANG W, TU J, et al. Analysis of microbial metabolism mechanism in traditional Sourdough fermentation based on macro- transcriptomics technology[J]. Journal of the Chinese Cereals and Oils Association, 2019, 34(11): 10–16. (in Chinese) |
| [13] | GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nat Biotechnol, 2011, 29: 644–652. DOI: 10.1038/nbt.1883 |
| [14] | THOMAS M, FENSKE G J, ANTONY L, et al. Whole genome sequencing-based detection of antimicrobial resistance and virulence in non-typhoidal Salmonella enterica isolated from wildlife[J]. Gut Pathog, 2017, 9: 66. DOI: 10.1186/s13099-017-0213-x |
| [15] |
李星, 曹振辉, 林秋叶, 等. 肠道微生物及其代谢产物对动物免疫机能的影响[J]. 动物营养学报, 2019, 31(2): 553–559.
LI X, CAO Z H, LIN Q Y, et al. Effects of gut microbiota and its metabolites on animal immune function[J]. Chinese Journal of Animal Nutrition, 2019, 31(2): 553–559. (in Chinese) |
| [16] | WIKOFF W R, ANFORA A T, LIU J, et al. Metabolomics analysis reveals large effects of gut microflora on mammalian blood metabolites[J]. Proc Natl Acad Sci U S A, 2009, 106(10): 3698–3703. DOI: 10.1073/pnas.0812874106 |
| [17] | POROYKO V, WHITE J R, WANG M, et al. Gut microbial gene expression in mother-fed and formula-fed piglets[J]. PLoS One, 2010, 5(8): e12459. DOI: 10.1371/journal.pone.0012459 |
| [18] | BAARS A, OOSTING A, LOHUIS M, et al. Sex differences in lipid metabolism are affected by presence of the gut microbiota[J]. Sci Rep, 2018, 8: 13426. DOI: 10.1038/s41598-018-31695-w |
| [19] |
刘选珍, 李明喜, 余建秋, 等. 圈养大熊猫排泄的粘液成分分析[J]. 应用与环境生物学报, 2005, 11(5): 584–587.
LIU X Z, LI M X, YU J Q, et al. Composition analysis of the muces excreted by captive giant panda[J]. Chinese Journal of Applied and Environmental Biology, 2005, 11(5): 584–587. (in Chinese) |
| [20] |
何永果, 晋蕾, 李果, 等. 基于高通量测序技术研究成年大熊猫肠道菌群[J]. 应用与环境生物学报, 2017, 23(5): 771–777.
HE Y G, JIN L, LI G, et al. Gut microbiome of adult giant pandas based on high-throughput sequencing technology[J]. Chinese Journal of Applied and Environmental Biology, 2017, 23(5): 771–777. (in Chinese) |
| [21] | NEVES A L A, LI F Y, GHOSHAL B, et al. Enhancing the resolution of rumen microbial classification from metatranscriptomic data using kraken and mothur[J]. Front Microbiol, 2017, 8: 2445. DOI: 10.3389/fmicb.2017.02445 |
| [22] | GOSALBES M J, DURBÁN A, PIGNATELLI M, et al. Metatranscriptomic approach to analyze the functional human gut microbiota[J]. PLoS One, 2011, 6(3): e17447. DOI: 10.1371/journal.pone.0017447 |
| [23] | FANG W, FANG Z M, ZHOU P, et al. Evidence for lignin oxidation by the giant panda fecal microbiome[J]. PLoS One, 2012, 7(11): e50312. DOI: 10.1371/journal.pone.0050312 |
| [24] | ZHU L F, WU Q, DAI J Y, et al. Evidence of cellulose metabolism by the giant panda gut microbiome[J]. Proc Natl Acad Sci U S A, 2011, 108(43): 17714–17719. DOI: 10.1073/pnas.1017956108 |
| [25] | FORSLUND K, HILDEBRAND F, NIELSEN T, et al. Disentangling type 2 diabetes and metformin treatment signatures in the human gut microbiota[J]. Nature, 2015, 528(7581): 262–266. DOI: 10.1038/nature15766 |
| [26] | PATERSON M J, OH S, UNDERHILL D M. Host-microbe interactions: commensal fungi in the gut[J]. Curr Opin Microbiol, 2017, 40: 131–137. DOI: 10.1016/j.mib.2017.11.012 |
| [27] | FORBES J D, BERNSTEIN C N, TREMLETT H, et al. A fungal world: Could the gut mycobiome be involved in neurological disease[J]. Front Microbiol, 2018, 9: 3249. |
| [28] | SOKOL H, LEDUCQ V, ASCHARD H, et al. Fungal microbiota dysbiosis in IBD[J]. Gut, 2017, 66(6): 1039–1048. DOI: 10.1136/gutjnl-2015-310746 |
| [29] | BROWN-JAQUE M, MUNIESA M, NAVARRO F. Bacteriophages in clinical samples can interfere with microbiological diagnostic tools[J]. Sci Rep, 2016, 6: 33000. DOI: 10.1038/srep33000 |
| [30] |
王悦, 陈倩. 肠道噬菌体组与人体健康[J]. 微生物与感染, 2019, 14(5): 317–322.
WANG Y, CHEN Q. Intestinal phageome and human health[J]. Journal of Microbes and Infections, 2019, 14(5): 317–322. (in Chinese) |
| [31] | ZOU W C, LI C W, YANG X, et al. Frequency of antimicrobial resistance and integron gene cassettes in Escherichia coli isolated from giant pandas (Ailuropoda melanoleuca) in China[J]. Microb Pathog, 2018, 116: 173–179. DOI: 10.1016/j.micpath.2018.01.034 |
| [32] |
邓雯文, 李才武, 赵思越, 等. 大熊猫源致病大肠杆菌CCHTP全基因组测序及耐药和毒力基因分析[J]. 遗传, 2019, 41(12): 1138–1147.
DENG W W, LI C W, ZHAO S Y, et al. Whole genome sequencing reveals the distribution of resistance and virulence genes of pathogenic Escherichia coli CCHTP from giant panda[J]. Hereditas, 2019, 41(12): 1138–1147. (in Chinese) |
| [33] |
陈福, 罗玉萍, 龚熹, 等. 抗菌肽耐药性研究进展[J]. 微生物学通报, 2008, 35(11): 1786–1790.
CHEN F, LUO Y P, GONG X, et al. Mechanism of antimicrobial peptide resistance[J]. Microbiology, 2008, 35(11): 1786–1790. (in Chinese) |
| [34] | BRÖTZ E, KULIK A, VIKINESWARY S, et al. Phenelfamycins G and H, new elfamycin-type antibiotics produced by Streptomyces albospinus Acta 3619[J]. J Antibiot, 2011, 64(3): 257–266. DOI: 10.1038/ja.2010.170 |
| [35] | PREZIOSO S M, BROWN N E, GOLDBERG J B. Elfamycins: Inhibitors of elongation factor-Tu[J]. Mol Microbiol, 2017, 106(1): 22–34. |
| [36] | GUO L J, LONG M, HUANG Y, et al. Antimicrobial and disinfectant resistance of Escherichia coli isolated from giant pandas[J]. J Appl Microbiol, 2015, 119(1): 55–64. |
| [37] | GUO M, CHEN J W, LI Q F, et al. Dynamics of gut microbiome in giant panda cubs reveal transitional microbes and pathways in early life[J]. Front Microbiol, 2018, 9: 3138. DOI: 10.3389/fmicb.2018.03138 |
| [38] | STOLTZFUS J D, MINOT S, BERRIMAN M, et al. RNAseq analysis of the parasitic nematode Strongyloides stercoralis reveals divergent regulation of canonical dauer pathways[J]. PLoS Negl Trop Dis, 2012, 6(10): e1854. DOI: 10.1371/journal.pntd.0001854 |
| [39] | XIE Y, HOBERG E P, YANG Z J, et al. Ancylostoma ailuropodae n. sp. (Nematoda: Ancylostomatidae), a new hookworm parasite isolated from wild giant pandas in Southwest China[J]. Parasit Vectors, 2017, 10(1): 277. |
| [40] |
张同富, 杨光友, 卢明科, 等. 四川动物体内发现的钩口线虫[J]. 四川动物, 2005, 24(2): 177–179.
ZHANG T F, YANG G Y, LU M K, et al. Hookworms found in animals of Sichuan province[J]. Sichuan Journal of Zoology, 2005, 24(2): 177–179. (in Chinese) |
| [41] | VARATHARAJALU R, RAO K V. Strongyloides stercoralis: current perspectives[J]. Rep Parasitol, 2016, 5: 23–33. |
| [42] |
林海, 杨光友. 人和动物的类圆线虫病[J]. 中国人兽共患病学报, 2016, 32(5): 477–484, 505.
LIN H, YANG G Y. Strongyloidiasis of human and animals[J]. Chinese Journal of Zoonoses, 2016, 32(5): 477–484, 505. (in Chinese) |
| [43] |
冯文和, 张安居.
大熊猫繁殖与疾病研究[M]. 成都: 四川科学技术出版社, 1991: 244-248.
FENG W H, ZHANG A J. A study on breeding and disease of the giant panda[M]. Chengdu: Sichuan Science and Technology Press, 1991: 244-248. (in Chinese) |
| [44] |
陈启军, 尹继刚, 刘明远. 重视人兽共患寄生虫病的研究[J]. 中国基础科学, 2008, 10(6): 3–11.
CHEN Q J, YIN J G, LIU M Y. The importance of zoonotic parasitoses in China[J]. China Basic Science, 2008, 10(6): 3–11. (in Chinese) |
| [45] |
唐崇惕. 蚂蚁与人类寄生虫病[J]. 中国媒介生物学及控制杂志, 2005, 16(3): 165–168.
TANG C T. Ants and human parasites diseases[J]. Chinese Journal of Vector Biology and Control, 2005, 16(3): 165–168. (in Chinese) |



