乳腺的发生从胚胎时期开始,在出生后经历青春期的快速生长,进入妊娠期后乳腺发育再次加速直至泌乳期发育完全。在妊娠早期导管分支开始增多,腺泡系统开始发育,小叶间脂肪组织减少,细胞开始增殖分化且代谢活跃[1]。妊娠中期血管和神经不断增加,腺泡及导管体积不断增加,腺泡腔开始出现[2]。妊娠晚期乳腺上皮细胞内的内质网和线粒体大量增加[3],腺泡发育完全,乳腺细胞开始具备分泌功能。妊娠期乳腺的发育是一个极为复杂的生理变化过程,受到很多信号通路调控,如Wnt信号通路参与调节乳腺导管和腺泡的发育[4],有研究表明,Wnt-2在乳腺导管发育时期高表达,而Wnt-5a和Wnt-5b在妊娠期的表达显著升高[5];TGF-β信号通路参与调节乳腺上皮细胞的增殖与凋亡[6];Notch信号通路调节乳腺上皮细胞的分化[7]等。此外,乳腺的发育还涉及到激素(如雌激素、孕激素、催乳素及生长激素和胰岛素生长因子等)和神经的调节[8],以及免疫细胞的参与[9]。
加权基因共表达网络分析(weighted gene co-expression network analysis, WGCNA)可以将基因表达量与表型数据结合分析,更适用于复杂过程的数据分析。其算法自2008年发表以来,在不同组学(如转录组学[10]、蛋白组学和代谢组学[11])和不同生物(动物、植物和微生物)中都广泛应用,最主要是用于肿瘤等疾病的分子标记物筛选[12]、发病机制探究[13]及预后影响因子筛选[14]的研究。WGCNA将表达模式相似的基因聚为一类称为模块,这些模块内的基因往往参与了相同的功能或通路[15]。因而WGCNA还可以用作基因功能的注释[16]和复杂生理过程的研究[17]等,但采用WGCNA分析妊娠期至泌乳早期奶山羊乳腺发育数据的研究鲜有报道。
因此,本试验采用WGCNA对不同生理阶段的奶山羊乳腺微阵列数据进行综合分析,并结合蛋白互作网络等方法,最终得到13个目标基因,其中UQCR主要与妊娠前期线粒体呼吸相关,FZR1和UBE2L3通过泛素化在妊娠中期参与调控细胞周期。在妊娠后期及末期主要是与免疫相关的基因(TNF、ITGB2、GPR18、JAK1)表达。CSN2作为酪蛋白基因在妊娠末期至泌乳早期阶段被筛选得到。这为进一步阐明奶山羊的乳腺发育及奶山羊的育种提供理论基础。
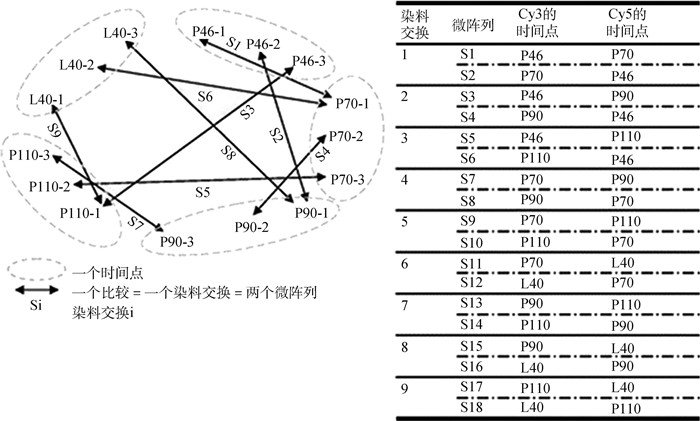
1 材料与方法 1.1 数据的获取及预处理本试验所采用的数据集为GSE14008[18],来源于NCBI的GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)。该数据集是对山羊5个生理阶段乳腺组织实质的微阵列测序数据,每个生理阶段测3只山羊,采用双通道循环试验设计(图 1)[18],共产生18个样本。所选择的5个生理阶段包括4个妊娠期和1个哺乳期,分别是妊娠46天、70天、90天、110天和产后40天,分别用P46、P70、P90、P110和L40表示。基于平台文件GPL2853对该数据集进行注释,将探针名字转换为基因名字,对于两个探针对应一个基因的情况取平均值,对于缺失值采用K近邻(k-nearest neighbor,KNN)算法进行补充。根据样本数据的详细介绍(ln2(signal median Cy5/signal median Cy3))对其取逆运算,得到双通道测序的原始数据比值,用于后续分析。
1.2 共表达模块的构建首先使用R语言WGCNA包[19]的hclust函数对样本进行聚类,剔除离群样本。随后使用pickSoftThreshold函数从备选软阈值中选择最佳软阈值,以无标度拓扑拟合指数(R2)大于0.8,且网络具有较好的平均连通性为条件进行筛选。随后采用adjacency函数构建TOM矩阵,并根据TOM矩阵的相异度,使用hclust函数对基因进行聚类。采用主成分分析法计算每个模块的特征值(module eigengene,ME),根据ME进行聚类,对相似度大于0.75的模块进行合并。
1.3 目标模块的选择与分析首先构建不同生理阶段的表型矩阵,由于生理阶段并不是数量表型,因而采用0和1来代表是否属于该生理阶段,该方法已经在癌症的分析中用于代表男女不同性别[20]。随后将合并后的模块重新计算ME,与表型矩阵进行关联分析,选择与目标阶段最相关的模块为目标模块。随后计算每个目标模块内的连接度,选出连接度排名前30的枢纽基因(hub gene)。随后计算目标模块内每个基因的基因身份(module membership,MM)与基因显著性(gene significance, GS)的关系,再次验证所选模块的正确性。采用DAVID[21]网站(http://david.abcc.ncifcrf.gov/)对目标模块进行基因本体论(GO)富集分析,并使用Cytoscape[22]软件对目标模块内权重前200的基因连接进行可视化。
1.4 核心基因的选择采用String 11.0网站[23]对目标模块进行蛋白互作网络(protein-protein interaction network,PPI network)的构建,选取Bos taurus为参考物种,并使用Cytoscape软件的cytoHubba插件[24],根据PPI网络中节点的Degree选出前20个基因为核心基因(core gene),并与模块内的枢纽基因(hub gene)取交集得到目标基因(target gene)。
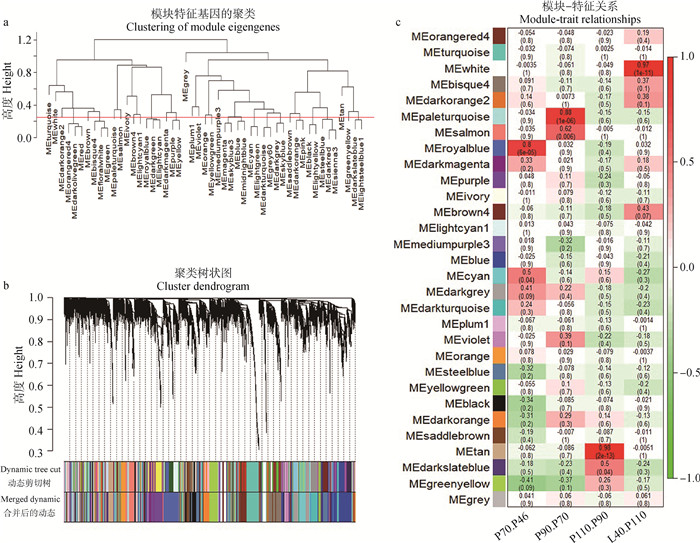
2 结果 2.1 共表达模块的构建数据预处理后得到8 443个基因全部用于后续分析。对于18个样本进行聚类,并未发现离群样本。以R2>0.8且具有一定连通性为标准选择软阈值,根据结果选择10,得到49个模块。根据ME进行模块聚类(图 2a),合并相似度大于0.75的模块,最后得到30个模块(图 2b)。灰色模块内的基因是不能归入任何其他模块内的共表达趋势不明显的基因。
|
a.模块聚类图,红色线的高度是0.25,在线以下的模块是相似度较大需要合并的模块;b.基因动态剪切聚类树,每个颜色代表一个模块,第一行的颜色是首次聚类的结果,第二行的颜色是模块经过合并后的结果;c.模块与生理阶段关联分析,红色代表正相关,绿色代表负相关,颜色越深相关性越强 a.Module cluster diagram, the height of the red line is 0.25, the module below the line is a large degree of similarity needs to merge the module; b. Gene dynamic cut clustering tree, each color represents a module, the color of the first row is the result of the first clustering, and the color of the second row is the result of the merging of the modules; c. Module and physiological stage correlation analysis, red represents a positive correlation, green represents a negative correlation, the deeper the color, the stronger the correlation 图 2 加权基因共表达网络分析 Fig. 2 The weighted gene co-expression network analysis |
双通道测序芯片的基因表达量为两个生理时期基因表达量的比值(cy5/cy3),所选择的目标生理过程cy3和cy5是连续时期,且cy3时期在cy5时期后面,即P70.P46(妊娠70天相对于妊娠46天基因表达量),P90.P70(妊娠90天相对于妊娠70天基因表达量),P110.P90(妊娠110天相对于妊娠90天基因表达量),L40.P110(产后40天相对于妊娠110天基因表达量)。模块的ME与表型矩阵进行关联分析,得到每个模块与不同生理过程的相关性及P值(图 2c),选择与目标生理过程相关性最强且P < 0.05的模块为目标模块。最终选择了4个模板模块,分别是“royalblue”(r=0.8,P=6×10-5)、“paleturquoise”(r=0.88,P=1×10-6)、“tan”(r=0.98,P=2×10-13)、“white”(r=0.97,P=1×10-11),这4个模块包含的基因数分别为538、127、229、140个。分别对每个模块内基因与表型相关性进行分析得到MM和GS的散点图,均呈现明显线性相关,进一步说明了所选模块的可靠性。
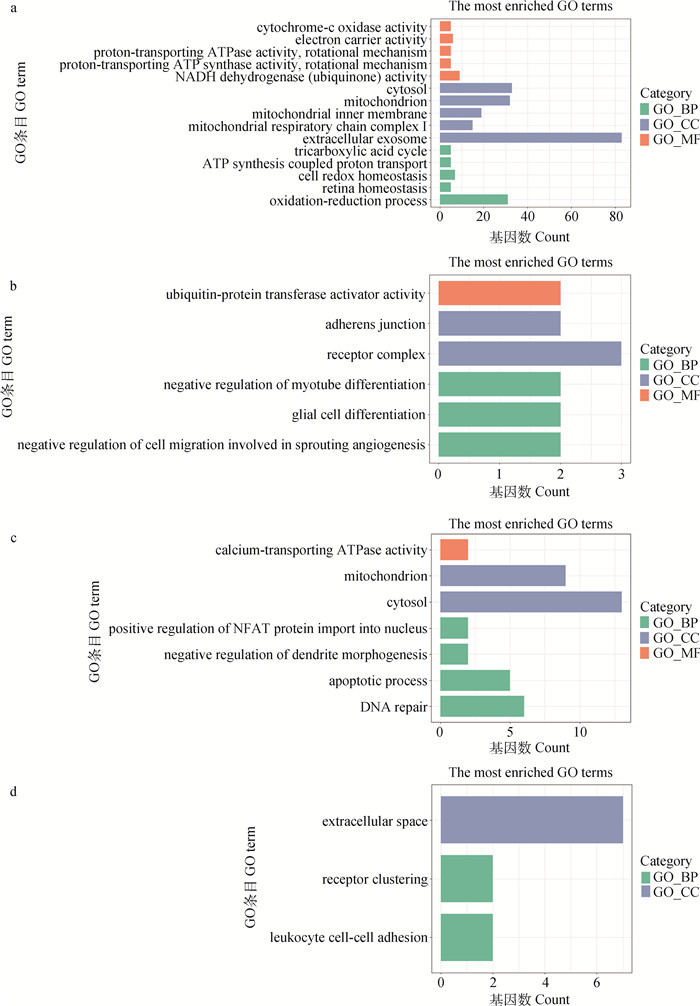
GO富集结果包括3部分:分子功能(molecular function,MF)、生物过程(biological process,BP)、和细胞组成(cellular component,CC),每部分富集结果仅在图中展示前5个,不足5个的全部展示。妊娠46~70天GO富集的结果主要集中在氧化还原、线粒体呼吸链复合体I、NADH脱氢酶(泛醌)活性等(图 3a),妊娠70~90天主要集中在细胞迁移参与新生血管生成的负调控、神经胶质细胞分化、肌管分化负调控等(图 3b),妊娠90~110天主要集中在DNA修复、凋亡过程、活化T细胞转录因子(NFAT)蛋白进入细胞核的正向调节、染色质结合的负调控等(图 3c),妊娠110天到产后40天主要集中在白细胞粘附、受体集群和细胞外间隙等过程(图 3d)。
|
a.妊娠40~70天;b.妊娠70~90天;c.妊娠90~110天;d.妊娠110天到产后40天 a.P46-P70;b.P70-P90;c.P90-P110;d.P110-L40 图 3 目标模块的GO富集结果 Fig. 3 GO enrichment results of the target modules |
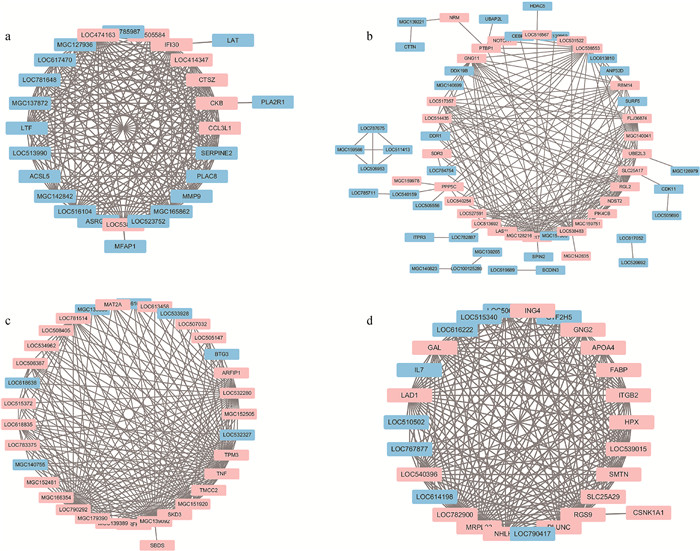
选择每个模块内权重前200的基因连接进行可视化,并将模块内连接度前30的基因定义为枢纽基因并用粉红色标出(图 4a-d)。
|
a.royalblue模块;b. paleturquoise模块;c. tan模块;d. white模块。每条线代表一个连接,其中粉色的是枢纽基因 a.royalblue module; b. paleturquoise module; c. tan module; d. white module. Each line represents a connection, and the pink one is hub gene 图 4 连接度前200的基因可视化结果 Fig. 4 Visualization results of the top 200 genes for degrees of connectivity |
在构建PPI网络时,因royalblue模块基因数目较多,所以选择的最低互作标准(minimum required interaction score)为0.7而非默认的0.4,其他3个模块基因数目较少所选择的标准为0.15。PPI网络的核心基因互作见图 5。
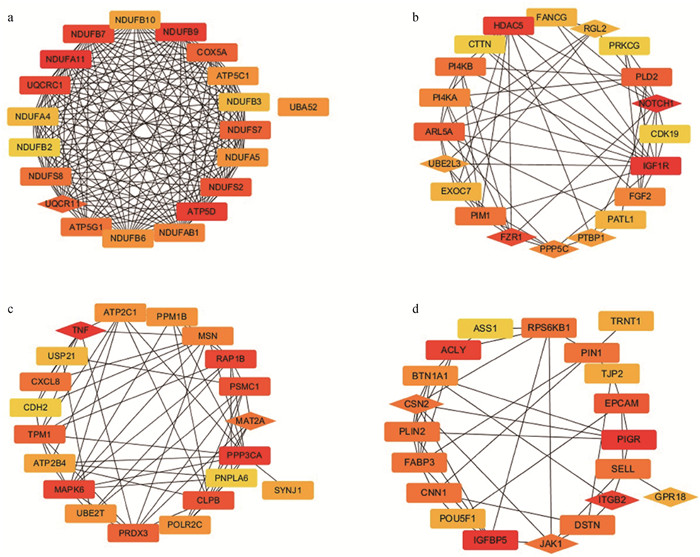
|
a.royalblue模块;b. paleturquoise模块;c. tan模块;d. white模块。红色越深说明基因连接度越高,菱形的基因代表核心基因与枢纽基因取交集得到的目标基因 a.royalblue module; b. paleturquoise module; c. tan module; d. white module. The deeper the red, the higher the degree of gene connectivity, the diamond-shaped genes represent the target genes obtained by the intersection of the core genes and the hub genes 图 5 PPI网络核心基因互作图 Fig. 5 PPI network core genes interaction diagram |
PPI网络的核心基因代表其在蛋白水平发挥关键作用,而模块的枢纽基因代表其在转录组水平发挥关键作用。将两者取交集得到目标基因后,就进一步确定了目标基因在相应乳腺发育时期的关键作用。妊娠46~70天的目标基因为UQCR;妊娠70~90天的目标基因为RGL2、NOTCH1、PTBP1、PPP5C、FZR1、UBE2L3;妊娠90~110天的目标基因为TNF、MAT2A;妊娠110天到产后40天的目标基因为ITGB2、GPR18、JAK1、CSN2。
3 讨论本研究通过对妊娠期和泌乳期奶山羊乳腺组织的微阵列数据分析,得到了与不同妊娠期乳腺发育相关的4个模块,及13个乳腺发育相关的目标基因,这将有助于对奶山羊妊娠期乳腺发育过程的进一步研究。
与妊娠前期(46~70 d)密切相关的royalblue模块的富集结果表明,在妊娠早期乳腺并没有进入一个快速增殖分化的阶段,而是代谢和呼吸作用加强。这与Hurley[8]的研究结果中所提到的母猪妊娠早期乳腺生长缓慢,随后生长加速是一致的。该模块PPI网络的核心基因(core gene)与共表达网络的枢纽基因(hub gene)取交集,仅得到1个目标基因UQCR11,可能原因是制作PPI网络时所选择的参考物种是牛,有相当一部分基因没有匹配到蛋白,且选择的最低互作标准较高。根据PPI网络文件,UQCR11是UQCR基因匹配到的,该基因编码辅酶细胞色素C还原酶复合物,是线粒体呼吸链的一部分。张庆林[25]的研究中也预测到该基因是乳腺癌上皮和间质细胞中线粒体的差异表达基因。作为一个能量代谢基因,UQCR在妊娠早期乳腺发育的过程中起着重要作用。Butt等[26]的研究表明,HSPC111基因是雌激素和c-myc的靶基因,而雌激素是调节乳腺发育尤其是增殖分化过程的重要激素。Wnt信号通路活化可导致下游c-myc基因的转录[27],而HSPC111作为c-myc的下游转录调节基因可以通过影响核糖体的生物合成而影响细胞增殖[28]。HSPC111是WGCNA模块的枢纽基因,表明影响细胞增殖的基因在妊娠前期已经开始表达并处于核心位置,但该基因并非PPI网络的核心基因,也没有在GO富集中得到与细胞增殖相关的条目,造成这些结果的原因还有待进一步分析。
与妊娠中期(70~90 d)密切相关的paleturquoise模块的富集结果表明,奶山羊在妊娠70天时已经开始大量进行细胞的增殖、分化与迁移,这与李真[29]的研究结果中所阐述的奶山羊妊娠中期乳房内血管和神经纤维开始增生,腺泡和导管开始出现且体积不断增大相吻合。目标基因NOTCH1是Notch通路中的一个重要基因,参与正常乳腺的导管增生[30]。除此之外,PTBP1通过调控可变剪切影响细胞的增殖、凋亡等过程[31],PPP5C通过参与蛋白磷酸化调控细胞增殖和分化[32],这些目标基因均在妊娠中期的乳腺发育中占据核心位置。
与妊娠后期(90~110 d)密切相关的tan模块的富集结果表明,乳腺发育过程中的免疫反应和DNA的增长发生在这一时期,这与Sciascia等[33]的研究结果一致。肿瘤坏死因子TNF主要参与细胞免疫反应,并促进c-myc、c-fos等基因的表达从而参与到细胞的增殖。MAT2A基因可以编码甲硫氨酸腺苷转移酶MATII的一个亚基,在生物体的甲基化修饰中起到重要作用[34]。有趣的是TNF-α可以通过Nf-κB和AP-1调控MAT2A的转录激活[35],这两者均为所筛选到的目标基因。
与妊娠末期到泌乳早期(P110 d~L40 d)密切相关的white模块的富集结果较少,但仍包括与炎症反应中的白细胞黏附及细胞组成中的胞外间隙相关的基因。由于该数据最后是将妊娠末期与泌乳早期结合在一起,因而不能确定该模块的免疫反应是存在于妊娠末期还是泌乳早期或者一直存在。目标基因ITGB2参与细胞的黏附及细胞表面的信号介导,该基因的缺失会导致白细胞黏附的缺陷[36],有研究表明, 其与荷斯坦奶牛的乳房炎抗性及产奶性状相关[37]。Terzi等[38]研究表明,枢纽基因RGS9-2可以调节和感觉情绪相关的神经性疼痛症状,该基因或许也与吮吸乳头排出乳汁过程中的神经传导有关。
通过本研究的分析发现,妊娠期乳腺发育是一个涉及多基因调控的复杂过程,不同妊娠阶段乳腺发育的侧重点不同,起到核心调控作用的基因也不同。此外,免疫反应的消失时间还不确定,目标基因的具体作用机制还有待进一步研究。
4 结论本研究发现,奶山羊妊娠前期乳腺发育主要是呼吸代谢作用增强,UQCR基因在这一时期起着重要作用。中期时细胞开始大量的增殖、分化、迁移,并有新的血管生成,这一时期Notch信号通路与PTBP1、PPP5C等基因发挥重要作用。免疫反应在妊娠后期开始出现,免疫过程中TNF和ITGB2起核心调控作用。这些基因都为进一步阐明乳腺发育机制提供了理论基础。
| [1] | CAIXETA L S, GIESY S L, KRUMM C S, et al. Fibroblast growth factor-21 (FGF21) administration to early-lactating dairy cows.Ⅱ.Pharmacokinetics, whole-animal performance, and lipid metabolism[J]. J Dairy Sci, 2019, 102(12): 11597–11608. DOI: 10.3168/jds.2019-16696 |
| [2] | ROOK W, JOHNSON C D, CONEY A M, et al. Prenatal hypoxia leads to increased muscle sympathetic nerve activity, sympathetic hyperinnervation, premature blunting of neuropeptide Y signaling, and hypertension in adult life[J]. Hypertension, 2014, 64(6): 1321–1327. DOI: 10.1161/HYPERTENSIONAHA.114.04374 |
| [3] |
曲波, 闫宏博, 佟慧丽, 等. 奶山羊不同发育时期乳腺上皮细胞中内质网和线粒体的变化[J]. 细胞生物学杂志, 2008, 30(6): 798–802.
QU B, YAN H B, TONG H L, et al. Changes of endoplasmic reticulum and mitochondria in mammary epithelium cells during mammogenesis of dairy goat[J]. Chinese Journal of Cell Biology, 2008, 30(6): 798–802. (in Chinese) |
| [4] | KESSENBROCK K, SMITH P, STEENBEEK S C, et al. Diverse regulation of mammary epithelial growth and branching morphogenesis through noncanonical Wnt signaling[J]. Proc Natl Acad Sci U S A, 2017, 114(12): 3121–3126. DOI: 10.1073/pnas.1701464114 |
| [5] | BVHLER T A, DALE T C, KIEBACK C, et al. Localization and quantification of Wnt-2 gene expression in mouse mammary development[J]. Dev Biol, 1993, 155(1): 87–96. DOI: 10.1006/dbio.1993.1009 |
| [6] | KHANEHSAR M A, HOSEINBEYKI M, TAHA M F, et al. Repression of TGF-β signaling in breast cancer cells by miR-302/367 cluster[J]. Cell J, 2020, 21(4): 444–450. |
| [7] | DING L N, SHUNKWILER L B, HARPER N W, et al. Deletion of Cdkn1b in ACI rats leads to increased proliferation and pregnancy-associated changes in the mammary gland due to perturbed systemic endocrine environment[J]. PLoS Genet, 2019, 15(3): e1008002. DOI: 10.1371/journal.pgen.1008002 |
| [8] | HURLEY W L. Review:mammary gland development in swine:embryo to early lactation[J]. Animal, 2019, 13(S1): S11–S19. DOI: 10.1017/S1751731119000521 |
| [9] |
赵冰, 李庆章. 奶山羊乳腺发育过程中肥大细胞的分布[J]. 东北农业大学学报, 2012, 43(9): 47–50.
ZHAO B, LI Q Z. Distribution of mast cells in development of dairy goat mammary gland[J]. Journal of Northeast Agricultural University, 2012, 43(9): 47–50. (in Chinese) |
| [10] | ZHU M D, XIE H J, WEI X J, et al. WGCNA analysis of salt-responsive core transcriptome identifies novel hub genes in rice[J]. Genes (Basel), 2019, 10(9): 719. DOI: 10.3390/genes10090719 |
| [11] | PEI G, CHEN L, ZHANG W. WGCNA application to proteomic and metabolomic data analysis[J]. Methods Enzymol, 2017, 585: 135–158. DOI: 10.1016/bs.mie.2016.09.016 |
| [12] | MO X G, LIU W, YANG Y, et al. NCF2, MYO1F, S1PR4, and FCN1 as potential noninvasive diagnostic biomarkers in patients with obstructive coronary artery:a weighted gene co-expression network analysis[J]. J Cell Biochem, 2019, 120(10): 18219–18235. DOI: 10.1002/jcb.29128 |
| [13] | SONG Z Y, CHAO F, ZHUO Z Y, et al. Identification of hub genes in prostate cancer using robust rank aggregation and weighted gene co-expression network analysis[J]. Aging (Albany NY), 2019, 11(13): 4736–4756. |
| [14] | WAN Q, TANG J, HAN Y, et al. Co-expression modules construction by WGCNA and identify potential prognostic markers of uveal melanoma[J]. Exp Eye Res, 2018, 166: 13–20. DOI: 10.1016/j.exer.2017.10.007 |
| [15] | GALÁN-VÁSQUEZ E, PEREZ-RUEDA E. Identi-fication of modules with similar gene regulation and metabolic functions based on co-expression data[J]. Front Mol Biosci, 2019, 6: 139. DOI: 10.3389/fmolb.2019.00139 |
| [16] |
宗剑.基于RNA-seq技术的AD相关lncRNA的识别与功能分析[D].哈尔滨: 哈尔滨工业大学, 2018.
ZONG J.Identification and functional analysis of long non-coding RNAs associated with Alzheimer's disease based on RNA-seq technology[D].Harbin: Harbin Institute of Technology, 2018.(in Chinese) |
| [17] |
石田培, 侯浩宾, 王欣悦, 等. 中国美利奴羊胚胎骨骼肌发育的加权基因共表达网络分析[J]. 畜牧兽医学报, 2020, 51(3): 452–464.
SHI T P, HOU H B, WANG X Y, et al. Weighted gene Co-expression network analysis for embryo development of skeletal muscle in Chinese Merino Sheep[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(3): 452–464. (in Chinese) |
| [18] | FAUCON F, REBOURS E, BEVILACQUA C, et al. Terminal differentiation of goat mammary tissue during pregnancy requires the expression of genes involved in immune functions[J]. Physiol Genomics, 2009, 40(1): 61–82. DOI: 10.1152/physiolgenomics.00032.2009 |
| [19] | LANGFELDER P, HORVATH S. WGCNA:an R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9(1): 559. |
| [20] | DI Y, CHEN D S, YU W, et al. Bladder cancer stage-associated hub genes revealed by WGCNA co-expression network analysis[J]. Hereditas, 2019, 156(1): 7. DOI: 10.1186/s41065-019-0083-y |
| [21] | WEI H D, SHERMAN B T, LEMPICKI R A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44–57. DOI: 10.1038/nprot.2008.211 |
| [22] | SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape:a software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003, 13(11): 2498–2504. DOI: 10.1101/gr.1239303 |
| [23] | SZKLARCZYK D, GABLE A L, LYON D, et al. STRING v11:protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Res, 2019, 47(D1): D607–D613. DOI: 10.1093/nar/gky1131 |
| [24] | CHIN C H, CHEN S H, WU H H, et al. cytoHubba:identifying hub objects and sub-networks from complex interactome[J]. BMC Syst Biol, 2014, 8(S4): S11. |
| [25] |
张庆林.乳腺癌上皮和间质细胞中差异表达的线粒体相关基因及其功能模块研究[D].合肥: 中国科学技术大学, 2013.
ZHANG Q L.Differentially expressed nuclear encoded mitochondrial genes in breast cancer epithelial and stromal cells and functional module analysis[D].Hefei: University of Science and Tech-nology of China, 2013.(in Chinese) |
| [26] | BUTT A J, SERGIO C M, INMAN C K, et al. The estrogen and c-Myc target gene HSPC111 is over-expressed in breast cancer and associated with poor patient outcome[J]. Breast Cancer Res, 2008, 10(2): R28. DOI: 10.1186/bcr1985 |
| [27] | REN Z F, DU M F, FU H, et al. MiR-200c promotes proliferation of papillary thyroid cancer cells via Wnt/β-catenin signaling pathway[J]. Eur Rev Med Pharmacol Sci, 2020, 24(10): 5512–5518. |
| [28] | ZHANG C W, YIN C Y, WANG L, et al. HSPC111 governs breast cancer growth by regulating ribosomal biogenesis[J]. Mol Cancer Res, 2014, 12(4): 583–594. DOI: 10.1158/1541-7786.MCR-13-0168 |
| [29] |
李真.奶山羊乳腺发育过程中激素及其受体表达规律的研究[D].哈尔滨: 东北农业大学, 2009.
LI Z.Law and role of hormones and their receptors in development of dairy goat mammary gland[D].Harbin: Northeast Agricultural University, 2009.(in Chinese) |
| [30] |
李想.受孕激素调控的miR-31在乳腺发育中的功能与分子机制[D].北京: 中国农业大学, 2017.
LI X.The in vivo function of progestogen responsive miR-31 in mammary gland development[D].Beijing: China Agricultural University, 2017.(in Chinese) |
| [31] | HUAN L, GUO T A, WU Y J, et al. Hypoxia induced LUCAT1/PTBP1 axis modulates cancer cell viability and chemotherapy response[J]. Mol Cancer, 2020, 19(1): 11. DOI: 10.1186/s12943-019-1122-z |
| [32] | FENG L, SUN P, LI Z Y, et al. Knockdown of PPP5C inhibits growth of hepatocellular carcinoma cells in vitro[J]. Appl Biochem Biotechnol, 2015, 175(1): 526–534. |
| [33] | SCIASCIA Q L, VAN DER LINDEN D S, SALES F A, et al. Parenteral administration of l -arginine to twin-bearing Romney ewes during late pregnancy is associated with reduced milk somatic cell count during early lactation[J]. J Dairy Sci, 2019, 102(4): 3071–3081. DOI: 10.3168/jds.2018-15433 |
| [34] | HU X, ZHOU Y J. Curcumin reduces methionine adenosyltransferase 2B expression by interrupting phosphorylation of p38 MAPK in hepatic stellate cells[J]. Eur J Pharmacol, 2020, 886: 173424. DOI: 10.1016/j.ejphar.2020.173424 |
| [35] | YANG H, SADDA M R, YU V, et al. Induction of human methionine adenosyltransferase 2A expression by tumor necrosis factor α role of NF-κB and AP-1[J]. J Biol Chem, 2003, 278(51): 50887–50896. DOI: 10.1074/jbc.M307600200 |
| [36] | TIPU H N, RAZA R, JAFFAR S, et al. β2 Integrin Gene (ITGB2) mutation spectra in Pakistani families with leukocyte adhesion deficiency type 1 (LAD1)[J]. Immunobiology, 2020, 225(3): 151938. DOI: 10.1016/j.imbio.2020.151938 |
| [37] |
杨宇泽, 冯涛, 路永强, 等. 中国荷斯坦奶牛ITGB2基因多态性及其与乳房炎抗性和产奶性状的相关分析[J]. 中国畜牧杂志, 2018, 54(10): 58–61, 111.
YANG Y Z, FENG T, LU Y Q, et al. Polymorphisms of ITGB2 gene and its association with somatic cell score and milk production traits in Chinese Holstein cows[J]. Chinese Journal of Animal Science, 2018, 54(10): 58–61, 111. (in Chinese) |
| [38] | TERZI D, GASPARI S, MANOURAS L, et al. RGS9-2 modulates sensory and mood related symptoms of neuropathic pain[J]. Neurobiol Learn Mem, 2014, 115: 43–48. DOI: 10.1016/j.nlm.2014.08.005 |