产志贺毒素大肠埃希菌(Shiga toxin-producing Escherichia coli,STEC)是一种能够引起全球性人畜共患疾病暴发的食源性病原体[1],因携带前噬菌体编码的一种或两种志贺毒素基因,具有很高的致病性。人感染STEC以后常出现胃肠道疾病,例如水样或血样腹泻、出血性结肠炎、溶血性尿毒综合征(hemolytic uremic syndrome,HUS)[2]。自从1980年,已报道的全世界STEC分离株中鉴定出400多种血清型[3],其中,O157 STEC是导致STEC暴发的主要致病血清型[4-6]。近些年,不定时暴发的非O157 STEC不仅危害人类,也危害动物,并会引起严重的疾病。STEC每年引起的疾病中非O157 STEC(O26、O104、O45、O103、O111、O121和O145)占64%以上[7],2011年,德国暴发STEC O104:H4的疫情,仅2个月的时间就蔓延到了16个国家[8],有22%以上的患者发生HUS。STEC主要通过粪-口途径传播[9],大量研究表明,STEC可污染肉类、蔬菜及水等。牛、羊、猪等是STEC主要宿主,提示未来STEC通过污染动物来源食品而造成人传染,将会成为公共卫生领域面临的一个重要问题。
能够引起STEC发病机制毒力因素有很多,其中志贺毒素(Stx,也称为Vero毒素)被认为是最重要的因素。STEC菌株主要产生两种Stx类型,即Stx1和Stx2,它们进一步分为3个子类型Stx1(Stx1a、Stx1c、Stx1 d)和7个子类型Stx2(Stx2a、Stx2b、Stx2c、Stx2 d、Stx2e、Stx2f、Stx2 g)[10]。除志贺毒素之外,还有能引起人致病性的STEC通常携带eae基因,该基因位于编码黏附素功能致病岛(LEE)。STEC也存在编码α-溶血素的hlyA基因[11]。
耐药性STEC菌株出现和传播已成为全球范围关注的公共卫生问题,因形成耐药性STEC菌株,可通过从事动物源性相关工作人员直接接触或通过动物粪便沿着生产链途径从牛羊体内间接传给人[12]。因此,对STEC抗药性监测可以提供有关该菌株抗菌药滥用、抗菌药传播和抗菌药发展的动态信息。
尽管许多家养和野生动物都可以作为STEC菌株的贮藏库,据相关报道,牛携带STEC数量是0.2%~74.0%,羊携带STEC数量达到32.1%~75.3%。牛、羊携带如此高的STEC数值离不开其养殖环节-屠宰环节-销售环节交叉传播[13]。牛羊养殖业是新疆现代畜牧业发展的主导产业,因STEC菌株在贮存宿主牛羊体内稳定存在,潜在影响了动物性食品质量安全和环境卫生安全。笔者对新疆部分地区牛、羊源不同生产环节STEC污染情况进行了调查以及耐药性检测,并采用ERIC-PCR基因分型方法对分离菌株的遗传进化关系进行分析。
1 材料与方法 1.1 材料1.1.1 样品来源 试验收集的431份样品分别来自新疆伊犁、昌吉和库尔勒牛、羊养殖场场内新鲜粪便或者直肠肛拭子、饲槽内饲料、水槽内饮水和市场、屠宰场胴体拭子或者新鲜肉样。牛、羊直肠肛拭子:用灭菌棉拭子采集到新鲜粪便样品置于2 mL灭菌肉汤内;粪便:用一次性PE手套抓取不同圈舍粪便;饲料:用灭菌自封袋采集每个圈舍不同饲槽内饲料;饮水:用灭菌2 mL EP管采集不同圈舍饮水;胴体拭子:用灭菌棉拭子采集到新鲜肉样置入2 mL灭菌肉汤内;肉样:用灭菌剪刀剪取2 g肉样置于灭菌自封袋内,并根据不同采样批次对样品分组编号,置于盛有冰块的保温盒内运回实验室检测(表 1)。
|
|
表 1 样品来源 Table 1 Sample source |
1.1.2 主要试剂与仪器 Taq MasterMix、DNA 2000 marker、DNA1500 marker、50×TAE购自新疆宝信生物技术有限公司;琼脂糖购自新疆伟博鑫生物有限公司;伊红美蓝培养基、麦康凯培养基、营养琼脂均购自北京奥博星生物技术有限公司;Gen Green核酸染料购自北京鼎国昌盛生物技术有限责任公司;营养肉汤、革兰阴性杆菌药敏试剂盒均购自杭州天和微生物试剂有限公司;氯化钠、氢氧化钠均购自天津市致远化学试剂有限公司;革兰染色液试剂盒购自北京陆桥技术有限责任公司。
移液器、PCR仪、高速离心机均购自Eppendorf公司;电凝胶成像仪购自美国伯乐公司;DYY-6C型电泳仪购自北京六一仪器厂。
1.1.3 引物设计合成 毒力基因引物、基因分型检测引物分别参考Bai等[14]和曹芸[15]的报道设计,均由上海生工生物工程股份有限公司合成(表 2)。
|
|
表 2 毒力基因及引物序列 Table 2 Virulence genes and primer sequences |
1.2.1 菌株的分离 样品按照食品卫生微生物检验标准GB/T 4789.3—2008[16]检验方法中规定进行处理,在伊红美蓝培养基上呈现出紫黑色有金属光泽圆形的菌落,或在麦康凯培养基上呈现光滑圆形桃红色的菌落进行分离培养。
1.2.2 DNA模板制备 将分离纯化的大肠杆菌用营养肉汤37 ℃过夜培养,吸取1 mL培养液至1.5 mL离心管中,13 000 r·min-1离心10 min,弃去上清液,用生理盐水洗菌1次,同样方法离心,弃去上清液。沉淀重悬于100 μL双蒸水,100 ℃水浴煮沸10 min,置于冰浴中使DNA从细胞中充分释放出来[17]。10 000 r·min-1离心10 min,收集上清液,-20 ℃保存DNA模板。
1.2.3 毒力基因检测 分别对分离的大肠杆菌菌株4种毒力基因stx1、stx2、hlyA和eae进行检测。检测STEC菌株,需要通过扩增stx1、stx2基因来判定。25 μL PCR反应体系:Taq MasterMix 10 μL,上、下游引物各0.5或1 μL,DNA模板1 μL,ddH2O 12或13 μL。PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s,Tm值退火30 s,72 ℃延伸1 min,共35个循环;72 ℃延伸10 min;PCR产物进行1.0%琼脂糖凝胶电泳检测。
1.2.4 ERIC-PCR基因分型 通过PCR方法成功扩增出3条带和3条带以上的菌株使用Bio Numerics软件绘制UPGMA聚类树,并进行遗传进化关系分析。反应体系25 μL:Taq Master Mix 10 μL,20 μmol·L-1上游引物ERIC-1R与下游引物ERIC-2各为1.5 μL,菌株DNA模板1 μL,ddH2O 11 μL。PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s,50 ℃退火1 min,72 ℃延伸2 min,共35个循环;72 ℃延伸10 min。扩增后的PCR产物进行1.0%琼脂糖凝胶电泳检测。根据相似度≥60%为同一簇的条件来判定产生的多态性条带。
1.2.5 药敏纸片检测 以大肠杆菌ATCC 25922为质控菌株,对64份菌株进行鉴定和17种常用抗生素的药物敏感试验,结果依照CLSI判定标准进行判定。用酶标仪测量STEC菌液浓度,将菌液浓度稀释在0.5麦氏比浊浓度左右。分别吸取STEC菌液600 μL加入到Mueller-Hinton培养基中,用灭菌的涂布棒将培养基中的菌液均匀的涂开,用灭菌的镊子在每个平皿贴6个药敏纸片,倒置放于37 ℃温箱中培养24 h后,观察结果。
1.2.6 数据分析 采用BioNumerics软件的非加权配对平均法(unweighted pair group method with arithmetic mean,UPGMA)对ERIC-PCR指纹图谱进行分析,条件位置差异容许度选择1.5%,优化值1.5%,相似系数使用Dice系数(F值)表示。公式:F=2nxy/(nx+ny)。其中,nx表示菌株X的条带数,ny表示菌株Y的条带数,nxy表示菌株X和Y相同的电泳条带数,F值在0~1,值越大则表示相似程度越高。相似度≥80%者为同一亚型,相似度<80%者为不同的基因亚型[18]。
2 结果 2.1 产志贺毒素大肠杆菌的分离鉴定根据stx(64株)检测情况统计,STEC共有64株,养殖场阳性分离率最高,分离率23.0%、其次是屠宰场,分离率为5.0%,市场分离率最低,分离率为2.9%(表 3),养殖场明显高于屠宰场和市场。
|
|
表 3 STEC不同环节分离鉴定结果 Table 3 Separation and identification results of different links of STEC |
stx1和stx2 2种毒素均产生的有31株(48.4%),只产生stx1的有29株(45.3%),只产生stx2的有4株(6.3%),毒力基因hlyA分离率最高为51.5%, 其次eae分离率为4.7%,4种毒力基因stx1、stx2、eae、hlyA同时存在的有1株,毒力基因的检测结果见表 4。对来自养殖场、屠宰场、市场的64株STEC毒力基因检测结果显示(图 1、2展示部分结果),养殖场基因阳性数达到58株,阳性率90.6%,屠宰场阳性数有4株,阳性率为6.3%,市场基因阳性数2株,阳性率为3.1%。其中阳性分布情况见表 5。养殖场阳性率明显高于屠宰场和市场。
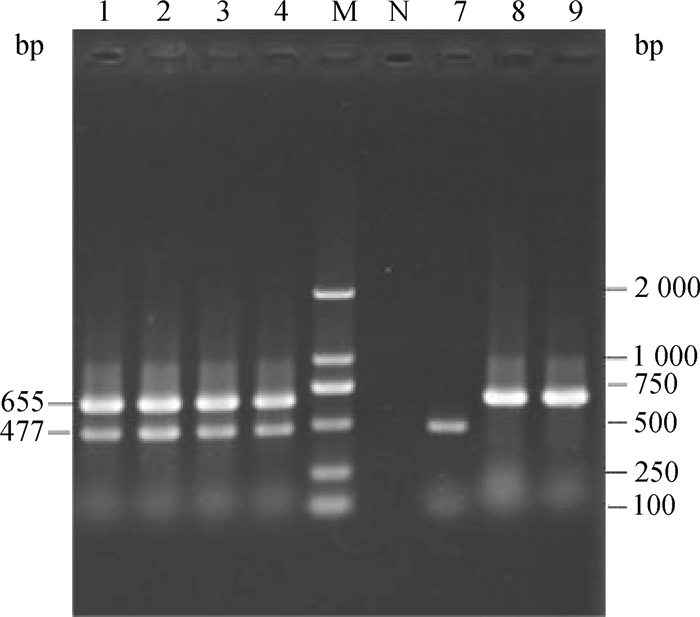
|
1~3、7~9.部分临床菌株;4.阳性对照;M. DNA相对分子质量标准;N.阴性对照 1-3, 7-9. Some clinical strains; 4. Positive control; M. DNA marker; N. Negative control 图 1 stx1和stx2基因PCR产物电泳 Fig. 1 Electrophoresis of PCR products of stx1 and stx2 genes |
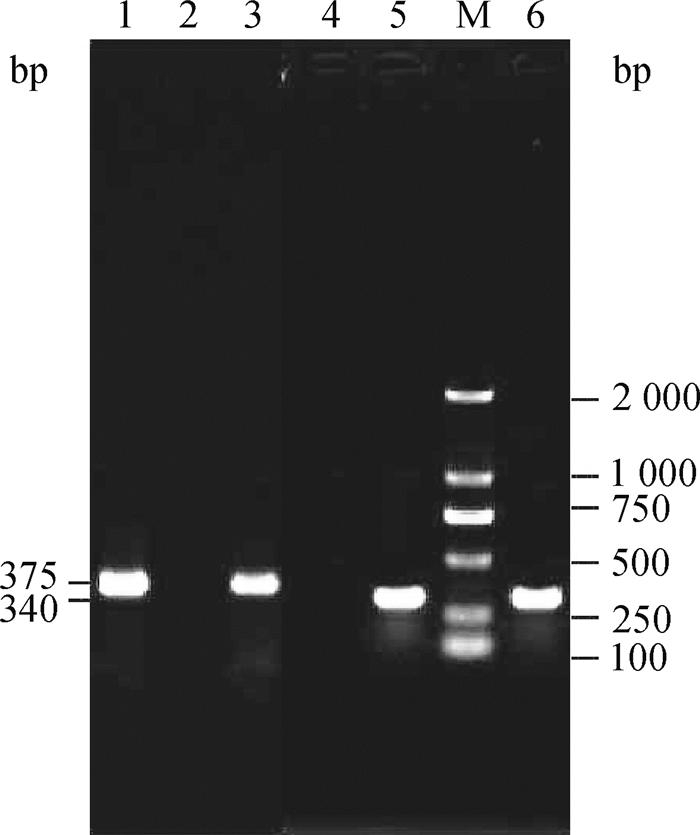
|
1、5.携带hlyA和eae临床菌株;2、4.阴性对照;3、6.阳性对照;M. DNA相对分子质量标准 1, 5. Clinical strains carrying hlyA, eae; 2, 4. Negative control; 3, 6. Positive control; M. DNA marker 图 2 eae基因和hlyA基因PCR产物电泳图 Fig. 2 Electrophoresis of PCR products of eae and hlyA genes |
|
|
表 4 64株STEC毒力基因检测 Table 4 Results of STEC virulence genes |
|
|
表 5 不同来源的64株STEC毒力基因检测 Table 5 Test results of virulence genes of 64 strains of STEC from different sources |
将64株STEC菌株分为A和B两个簇(图 3),A簇共有36株菌,相似度61.5%~100.0%,有6组的菌株ERIC-PCR指纹图谱相同,分别为菌株B34-3和H8-1,M15-1和M15-2,J12和M33-3,d12-4和d36-3、G27,M1-2和M22-3、M22-4、M22-5,6-111和M22-1。

|
图 3 ERIC-PCR扩增结果 Fig. 3 ERIC-PCR amplification results |
B簇共有28株菌,相似度65.3%~100.0%,有4组的菌株ERIC-PCR指纹图谱相同,分别为M23-2和M6-2,G11和G15,M15-4和M38-3、M38-5、M4-1、M4-4、M6-1、Q6-2,B21和B34、d26-2、d36-2、d36-4、G22、L14、M15-3、M2-1。
ERIC-PCR图谱相似度达到80%以上的菌株可以判定菌株的亲缘关系较近。在不同地区牛源和羊源养殖场分离菌株中存在相同图谱,如J12(羊)和M33-3(牛),d12-4(牛)、d36-3(牛)和G27(羊)。在不同地区的屠宰场和养殖场中存在相同的指纹图谱,6-111(屠宰场)和M22-1(养殖场)。
2.4 药敏试验2.4.1 耐药性结果 利用17种抗生素分别对64株产志贺毒素大肠杆菌进行药敏试验,耐药性结果见表 6。由表 6可知,分离的64株产志贺毒素大肠杆菌对麦迪霉素(61%)、头孢噻吩(4.7%)、头孢西丁(4.7%)、氨苄西林(3.1%)、哌拉西林(1.6%)、妥布霉素(1.6%)、头孢唑啉(1.6%)7种抗生素耐药,其中STEC对麦迪霉素耐药率最高,对氨曲南、氧氟沙星、链霉素、卡那霉素、头孢呋辛、头孢曲松、头孢吡肟、头孢他啶、头孢哌酮、头孢噻肟10种抗生素耐药率为0。
|
|
表 6 64株STEC对17种常用抗生素的敏感性结果 Table 6 Sensitivity results of 64 strains of STEC to 17 commonly used antibiotics |
2.4.2 多重耐药性结果 进一步鉴定64株STEC中耐药菌株,结果显示:共有8株存在多重耐药,占耐药菌株数的14%,其中2株存在三重耐药,耐药模式为MID+CEP+FOX、MID+CZO+FOX,6株存在二重耐药,耐药模式为MID+FOX、TOB+CEP、MID+AMP(2)、MID+PIP、MID+CEP。
3 讨论STEC是一种能引起人畜共患的食源性致病菌,人感染后能导致胃肠炎甚至出现HUS。有资料显示[19],人感染STEC是因食入的动物源性食品在养殖、生产、加工、包装、储藏、运输、销售等各个环节受该菌株污染[20]。因此,在本研究中,收集了牛羊养殖场、屠宰场、市场431份样品,从中分离出64株STEC并阐述新疆部分地区STEC污染牛羊肉品的程度。
本研究结果显示,在养殖场中,STEC分离率最高,其次是屠宰场,市场分离率最低,符合食品生产环节中污染逐渐降低的发展模式。通过考察发现,在养殖场和屠宰场中存在环境恶劣、管理落后、密度大等情况,这些情况极大地增加了细菌的传播风险,应该加强建立现代食品安全的控制体系,对食品的生产、加工、包装、储运、销售、进出口等各个环节都进行严格的监管。
在本研究中,所有主要毒力基因(stx1、stx2、hlyA和eae)被检测到。据报道,通过应用多重PCR技术检测菌株stx1、stx2、hlyA、eae等基因,发现hlyA和eae的存在形式较为复杂,有hlyA+eae、hlyA、eae和二者均阴性等4种携带类型[21-22]。本研究结果显示,hlyA阳性率为51.5%,eae阳性率为4.7%,明显低于诸明欣等[23]采用PCR方法检测的大肠杆菌hlyA基因阳性率(96.0%)。与张力国[24]报道的牛源大肠杆菌eae阳性率(14.8%)相比稍低,这可能与不同地区有关。与Bai等[25]检测的我国青藏高原STEC分离株毒力基因明显不同,stx1+stx2基因型占的比例最多(48.4%),stx1、stx2基因型分别占45.3%、6.3%。
ERIC-PCR基因分型方法建立后一直用于大肠杆菌[26]、沙门菌[27]、李斯特菌[28]、副溶血性弧菌[29]等单个菌株基因组多态性的分析。基于ERIC-PCR以其能反映出细菌整个基因组结构的差异和能清晰地区别包含有ERIC(一段长126 bp的反向重复序列)这些重复序列细菌的不同种和不同菌株,具有很强的鉴别种乃至菌株的能力的特点而广泛应用[30]。何力等[31]采用ERIC-PCR方法构建大肠杆菌的DNA指纹图谱库,用Info Quest FPTM(Bio-rad)软件对指纹图进行聚类分析来确定其可能来源。林朝洪[32]利用ERIC-PCR基因分型方法聚类分析把26株菌分为明确的3个类群(A、B、C),这3类群相似率范围分别为52%~76%、40%~92%和37%~73%。本研究中ERIC-PCR结果中,STEC分为A、B两个簇。在昌吉地区的羊源和伊犁州的牛源养殖场分离菌株中发现相同图谱,如J12(羊)和M33-3(牛),d12-4(牛)、d36-3(牛)和G27(羊)。这一发现可能预示着STEC传播不仅存在同一地区同一农场内部,也有可能通过牛、羊群在不同地区不同农场之间的买卖交易或者来源同一育种中心而引起STEC菌株交叉传播。
通过调查牛、羊源STEC的药物敏感性及多重耐药性现状,为进一步指导临床用药提供参考。对头孢类、单环β内酰胺类、氨基糖苷类、青霉素类、喹诺酮类进行药敏检测,结果显示,耐药率很低,因此抗菌效果较理想的原因,可能来源于样本地理位置和类型的差异。陈月月等[33]对新疆伊犁地区牛源大肠杆菌对氨苄西林耐药率为8.5%,对头孢噻吩耐药率为0,而本试验中对氨苄西林耐药率为3.1%,与其相比较低,但对头孢噻吩耐药率较高,这可能与药物使用频率和时间不同而引起的。进一步研究50株耐药菌株,发现共有8株存在多重耐药,占耐药菌株数的14%,其中2株存在三重耐药,6株存在二重耐药。邹立扣等[34]对2010年四川省猪肉源大肠杆菌进行17种抗生素耐药性分析,结果显示耐3种及3种以上抗生素的菌株占44.1%,明显高于本试验结果,这可能是与地域和动物来源不同有关。在对STEC的流行性研究中,细菌的分离鉴定、抗生素敏感性检测等为基础性研究内容,通过这些研究从源头可以控制细菌性疾病的发生和发展。
4 结论STEC在养殖场、屠宰场和市场各个环节都存在不同程度的污染,养殖场污染最严重。在各个生产环节中,牛源STEC检出均略高于羊源STEC,说明牛源污染较为严重。64株STEC总体耐药率较低,对麦迪霉素耐药率最高,对青霉素类(氨苄西林、哌拉西林)、氨基糖胺类(妥布霉素)、头孢类(头孢噻吩、头孢唑啉、头孢西丁)6种抗生素呈现不同浓度的轻微耐药,并存在多重耐药情况。从图谱中发现,牛源和羊源之间亲缘关系较近。养殖场环节中STEC和屠宰场环节中的STEC存在相同指纹图谱,亲缘关系较近。
| [1] | YANG X, BAI X N, ZHANG J, et al. Escherichia coli strains producing a novel Shiga toxin 2 subtype circulate in China[J]. Int J Med Microbiol, 2020, 310(1): 151377. DOI: 10.1016/j.ijmm.2019.151377 |
| [2] | GRIFFIN P M, TAUXE R V. The epidemiology of infections caused by Escherichia coli O157:H7, other enterohemorrhagic E. coli, and the associated hemolytic uremic syndrome[J]. Epidemiol Rev, 1991, 13(1): 60–98. DOI: 10.1093/oxfordjournals.epirev.a036079 |
| [3] | FAN R Y, SHAO K, YANG X, et al. High prevalence of non-O157 Shiga toxin-producing Escherichia coli in beef cattle detected by combining four selective agars[J]. BMC Microbiol, 2019, 19(1): 213. |
| [4] | KING L A, MAILLES A, MARIANI-KURKDJIAN P, et al. Community-wide outbreak of Escherichia coli O157:H7 associated with consumption of frozen beef burgers[J]. Epidemiol Infect, 2009, 137(6): 889–896. DOI: 10.1017/S0950268808001490 |
| [5] |
巴鹏斌, 孟琼, 白向宁, 等. 非O157产志贺毒素大肠埃希菌研究进展[J]. 中国人兽共患病学报, 2017, 33(2): 156–162.
BA P B, MENG Q, BAI X N, et al. Research progress on non-O157 Shiga toxin-producing Escherichia coli[J]. Chinese Journal of Zoonoses, 2017, 33(2): 156–162. (in Chinese) |
| [6] |
白向宁, 赵爱兰, 夏胜利, 等. 非O157产志贺毒素大肠杆菌分离株的多位点序列分型研究[J]. 中国人兽共患病学报, 2012, 28(6): 544–548, 560.
BAI X N, ZHAO A L, XIA S L, et al. Multilocus sequence typing of non-O157 Shiga toxin-producing Escherichia coli isolates[J]. Chinese Journal of Zoonoses, 2012, 28(6): 544–548, 560. (in Chinese) |
| [7] | PENG Z, LIANG W, HU Z Z, et al. O-serogroups, virulence genes, antimicrobial susceptibility, and MLST genotypes of Shiga toxin-producing Escherichia coli from swine and cattle in Central China[J]. BMC Vet Res, 2019, 15(1): 427. DOI: 10.1186/s12917-019-2177-1 |
| [8] | FRANK C, WERBER D, CRAMER J P, et al. Epidemic profile of Shiga-toxin-producing Escherichia coli O104:H4 outbreak in Germany[J]. N Engl J Med, 2011, 365(19): 1771–1780. DOI: 10.1056/NEJMoa1106483 |
| [9] |
白向宁, 王红, 赵爱兰, 等. 食源性产志贺毒素大肠杆菌的分离及菌株特征分析[J]. 中国食品卫生杂志, 2014, 26(4): 312–317.
BAI X N, WANG H, ZHAO A L, et al. Isolation and characterization of Shiga toxin-producing Escherichia coli isolates in foods[J]. Chinese Journal of Food Hygiene, 2014, 26(4): 312–317. (in Chinese) |
| [10] | BOONE J T, CAMPBELL D E, DANDRO A S, et al. A rapid immunoassay for detection of Shiga toxin-producing Escherichia coli directly from human fecal samples and its performance in detection of toxin subtypes[J]. J Clin Microbiol, 2016, 54(12): 3056–3063. DOI: 10.1128/JCM.01785-16 |
| [11] | MENG Q, BAI X N, ZHAO A L, et al. Characterization of Shiga toxin-producing scherichia coli isolated from healthy pigs in China[J]. BMC Microbiol, 2014, 14(1): 5. |
| [12] | KARAMA M, MAINGA A O, CENCI-GOGA B T, et al. Molecular profiling and antimicrobial resistance of Shiga toxin-producing Escherichia coli O26, O45, O103, O121, O145 and O157 isolates from cattle on cow-calf operations in South Africa[J]. Sci Rep, 2019, 9(1): 11930. DOI: 10.1038/s41598-019-47948-1 |
| [13] | GALARCE N, ESCOBAR B, SÁNCHEZ F, et al. Virulence genes, Shiga toxin subtypes, serogroups, and clonal relationship of Shiga toxin-producing Escherichia coli strains isolated from livestock and companion animals[J]. Animals, 2019, 9(10): 733. DOI: 10.3390/ani9100733 |
| [14] | BAI J F, SHI X R, NAGARAJA T G. A multiplex PCR procedure for the detection of six major virulence genes in Escherichia coli O157:H7[J]. J Microbiol Methods, 2010, 82(1): 85–89. DOI: 10.1016/j.mimet.2010.05.003 |
| [15] |
曹芸.检测副猪嗜血杆菌的乳胶凝集试验及ERIC-PCR方法的建立与应用[D].武汉: 华中农业大学, 2009.
CAO Y. Development and application of a simple latex agglutination test and ERIC-PCR method for detection of Haemophilus parasuis[D]. Wuhan: Huazhong Agricultural University, 2009. (in Chinese) |
| [16] |
中华人民共和国卫生部, 中国国家标准化管理委员会. GB/T 4789. 3-2008食品卫生微生物学检验大肠菌群计数[S].北京: 中国标准出版社, 2009: 1-8.
Ministry of Health of the PRC, Standardization Administration of China. GB/T 4789. 3-2008 Microbiological examination of food hygiene-enumeration of coliforms[S]. Beijing: China Standard Press, 2009: 1-8. (in Chinese) |
| [17] |
赫然, 张颖, 王强, 等. 利用微波炉和煮沸法快速制备大肠杆菌基因组DNA PCR模板[J]. 中山大学学报:自然科学版, 2004, 43(S1): 80–81.
HE R, ZHANG Y, WANG Q, et al. Rapid preparation of PCR templates of genomic DNA from Escherichia coli by using microwave and boiling[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni, 2004, 43(S1): 80–81. (in Chinese) |
| [18] | THONG K L, LAI K S, GANESWRIE R, et al. Pulsed-field gel electrophoresis of multidrug-resistant and -sensitive strains of Pseudomonas aeruginosa from a Malaysian hospital[J]. Jan J Infect Dis, 2004, 57(5): 206–209. |
| [19] |
孙晖, 白向宁, 赵爱兰, 等. 牦牛携带的产志贺毒素大肠杆菌分离株的多位点序列分型研究[J]. 中国人兽共患病学报, 2013, 29(12): 1137–1142.
SUN H, BAI X N, ZHAO A L, et al. Multilocus sequence typing of Shiga toxin-producing Escherichia coli isolated from yaks[J]. Chinese Journal of Zoonoses, 2013, 29(12): 1137–1142. (in Chinese) |
| [20] |
杨育斌. 畜产品安全分析及应对措施[J]. 甘肃农业, 2004(1): 34–35.
YANG Y B. Animal product safety analysis and countermeasures[J]. Gansu Agriculture, 2004(1): 34–35. (in Chinese) |
| [21] |
张锦, 马宏, 夏胜利, 等. 产志贺毒素大肠杆菌毒力基因检测[J]. 中国公共卫生, 2003, 19(12): 56–57.
ZHANG J, MA H, XIA S L, et al. Detection of toxicity genes of Shiga toxigenic Escherichia coli[J]. Chinese Journal of Public Health, 2003, 19(12): 56–57. (in Chinese) |
| [22] |
薛涛, 李丽, 高清清, 等. 牛源产志贺毒素大肠杆菌分离株的毒力基因分布和遗传进化分析[J]. 中国畜牧兽医, 2017, 44(10): 2878–2885.
XUE T, LI L, GAO Q Q, et al. Study on the virulence gene distribution and genetic evolution of cattle Shiga toxin-producing Escherichia coli isolates[J]. China Animal Husbandry & Veterinary Medicine, 2017, 44(10): 2878–2885. (in Chinese) |
| [23] |
诸明欣, 张焕容, 汤承, 等. 四川阿坝州牦牛源溶血性大肠杆菌血清型鉴定及毒力相关基因检测[J]. 中国预防兽医学报, 2018, 40(1): 9–13.
ZHU M X, ZHANG H R, TANG C, et al. Serotype identification and virulence-related genes detection of hemolytic E. coli isolated from yak in Sichuan Aba state[J]. Chinese Journal of Preventive Veterinary Medicine, 2018, 40(1): 9–13. (in Chinese) |
| [24] |
张力国.牛源大肠杆菌毒素基因、黏附素基因、O血清型及耐药性调查[D].哈尔滨: 东北农业大学, 2015.
ZHANG L G. Investigation of toxin gene, adhesin, O serotype and drug resistant of cattle Escherichia coli[D]. Harbin: Northeast Agricultural University, 2015. (in Chinese) |
| [25] | BAI X N, ZHANG W, TANG X Y, et al. Shiga toxin-producing Escherichia coli in Plateau Pika (Ochotona curzoniae) on the Qinghai-Tibetan Plateau, China[J]. Front Microbiol, 2016, 7: 375. |
| [26] |
陈迎春, 曹又方, 赵立平. 大肠杆菌MG1655菌株ERIC-PCR图谱主带序列组成分析[J]. 微生物学通报, 2002, 29(6): 28–32.
CHEN Y C, CAO Y F, ZHAO L P, et al. Non-random nature of genomic DNA amplification of E.coli K-12 MG1655 via ERIC-PCR[J]. Microbiology, 2002, 29(6): 28–32. (in Chinese) |
| [27] | KUMAR R, SURENDRAN P K, THAMPURAN N. Molecular fingerprinting of Salmonella enterica subsp. enterica typhimurium and Salmonella enterica subsp. enterica Derby isolated from tropical seafood in South India[J]. Mol Biotechnol, 2008, 40(1): 95–100. |
| [28] | LACIAR A, VACA L, LOPRESTI R, et al. DNA fingerprinting by ERIC-PCR for comparing Listeria spp. strains isolated from different sources in San Luis, Argentina[J]. Rev Argent Microbiol, 2006, 38(2): 55–60. |
| [29] | BHOWMICK P P, KHUSHIRAMANI R, RAGHUNATH P, et al. Molecular typing of Vibrio parahaemolyticus isolated from seafood harvested along the south-west coast of India[J]. Lett Appl Microbiol, 2008, 46(2): 198–204. |
| [30] |
段会勇, 柴同杰, 蔡玉梅, 等. ERIC-PCR对鸡舍大肠杆菌气溶胶向舍外环境传播的鉴定[J]. 中国科学C辑:生命科学, 2008, 51(2): 164–173.
DUAN H Y, CHAI T J, CAI Y M, et al. Transmission identification of Escherichia coli aerosol in chicken houses to their environments using ERIC-PCR[J]. Science in China Series C: Life Sciences, 2008, 51(2): 164–173. (in Chinese) |
| [31] |
何力, 傅玲琳, 冯立芳, 等. 大肠埃希氏菌ERIC-PCR指纹图谱构建及贝类污染微生物源示踪[J]. 中国食品学报, 2012, 12(8): 163–169.
HE L, FU L L, FENG L F, et al. ERIC-PCR fingerprinting of fecal Escherichia coli and microbial source tracking in non-point pollution of the shellfish culture area of East China sea[J]. Journal of Chinese Institute of Food Science and Technology, 2012, 12(8): 163–169. (in Chinese) |
| [32] | LIN Z H. Identification, screening and ERIC-PCR differentiation of lactobacilli isolated from weaning piglet[J]. Ya'an: Sichuan Agricultural University, 2007. |
| [33] |
陈月月, 王凯, 王舒丰, 等. 新疆伊犁地区不同动物源大肠杆菌耐药性调查[J]. 新疆农业科学, 2018, 55(8): 1560–1568.
CHEN Y Y, WANG K, WANG S F, et al. Investigation into drug resistance of Escherichia coli from different animal sources in Yili, Xinjiang[J]. Xinjiang Agricultural Sciences, 2018, 55(8): 1560–1568. (in Chinese) |
| [34] |
邹立扣, 蒲妍君, 杨莉, 等. 四川省猪肉源大肠杆菌和沙门氏菌的分离与耐药性分析[J]. 食品科学, 2012, 33(13): 202–206.
ZOU L K, PU Y J, YANG L, et al. Isolation and drug resistance analysis of Escherichia coli and Salmonella spp. in pork from Sichuan province[J]. Food Science, 2012, 33(13): 202–206. (in Chinese) |



