2. 兰州大学生命科学学院, 兰州 730000
2. School of Life Sciences, Lanzhou University, Lanzhou 730000, China
低压低氧、低温和强紫外辐射是高海拔地区最显著的生态限制因子,而低压低氧是高原动物生存所面临的最大挑战之一[1-2]。氧气是动物生长发育不可或缺的条件,较低的氧分压导致机体组织中的氧气供应不足,从而影响动物正常的生理功能。然而,高原低氧适应是指长期居住在高海拔环境中的生物为了能够在高海拔低氧环境中生存下来而产生的一种非可逆但可稳定遗传的表观形态及生理行为等方面的改变。与平原地区动物相比,生活在高原极端环境下的动物对低氧、低温具有独特的适应机制,例如机体利用生理、分子等水平上的变化修饰,使其呼吸速率、红细胞数量以及血容量增加,从而维持氧气运输[3];机体还可通过抑制体内某些物质代谢活性[2],增强氧气摄入和转运能力[4],维持组织间氧气水平,促进其生长、发育和繁殖,同时可降低缺氧对机体的损害[5];此外,高原动物还可通过调控参与线粒体氧化代谢相关的信号通路,使其发生一系列的生理生化反应,确保动物能在低温、缺氧的极端环境中正常生活[6]。青藏高原是世界上最大、最年轻的高原,低温、缺氧、强紫外辐射等独特的环境特征,使世居于此的高原土著动物如牦牛、藏猪、藏羊等,历经数万年的自然选择,在生理和遗传等方面适应了青藏高原极端自然环境[5, 7-10],为研究动物高原适应性提供了科研材料,更为高原牧民提供了衣食住行等生产和生活资料。因此,全面、系统地解析高原动物适应性进化的分子机制和遗传原理,有利于耐低温、低氧和强紫外辐射的动物新品种培育;更有利于生物学、生物医学和人类医学的发展。为此,本文以青藏高原特有的低氧、低温和强紫外辐射为线索,并结合相关微生物的研究成果,综述了近几十年关于高原动物适应性进化的研究进展,以期为培育耐寒、耐低氧和抗紫外辐射的动物新品种提供理论和技术依据,也为临床预防和治疗高原性疾病提供新思路。
1 高原土著动物低氧适应性进化的研究进展低氧是高海拔地区生态环境的主要特征之一。研究发现,随着海拔的逐渐升高,海平面氧分压百分比逐渐下降,而且呼吸氧分压也随即下降[3](图 1)。经过漫长的生物进化,生活于高原之上的土著动物已能很好的适应高原低氧极端环境,并形成了独特的高原适应性遗传机制,比如拥有了较高的静息代谢率(resting metabolic rate, RMR)、非颤抖性产热(non-shivering thermogenesis, NST)及氧利用率[11-12]。因此,以氧为线索,分别从生理和遗传机制等方面综述高原动物低氧适应性的研究进展。
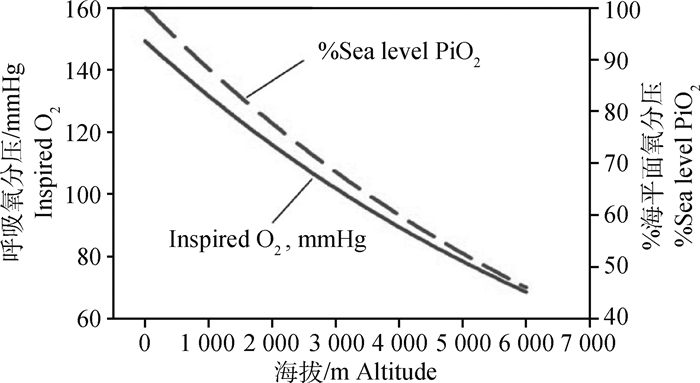
|
实线表示呼吸氧分压;虚线表示%海平面氧分压 The solid line indicates the respiratory oxygen partial pressure; the dashed line indicates the sea level oxygen partial pressure 图 1 不同海拔氧分压变化情况[3] Fig. 1 Changes in oxygen partial pressure at different altitudes[3] |
作为氧气循环的第一阶段——心肺组织,它是机体血液循环及氧气运输最重要的调控中心。在长期的自然选择和进化过程中,生活于高原低氧极端环境中的动物,为了适应极端生存环境,其肺部结构发生了一系列适应性变化,增加了氧气的摄入、转运和扩散能力,以便更好地适应高原低氧的环境[13]。研究发现,高原鼢鼠的肺部微血管密度及单位肺泡表面积增加,且肺和体重比明显增大[14];高原鼠兔肺血管内皮细胞及平滑肌无低氧性损伤并维持一氧化氮正常释放,这对维持肺血管的低张力起到了重要作用[15]。藏猪肺支气管周围无炎性细胞浸润,肺泡腔内尘细胞极少,肺泡壁和肺间质血管较丰富,此类组织结构有利于血氧结合,增加氧气的摄入[16]。藏羊肺泡较小,单位面积中肺泡数量多,肺泡隔内毛细血管呈开放状态,管径较粗,毛细血管内红细胞数量较多,单位面积中血管数量较多,这些结构有利于其肺部气体交换,增加氧气的摄入和转运[17]。藏羚羊小支气管的黏膜上皮为假复层纤毛柱状上皮,杯状细胞明显,纤毛清晰可见,且部分小支气管的外膜中,存在一层较厚的、完整的纵行平滑肌层,并分布有明显的透明软骨,这些特征有利于肺部气体的交换和无效气体的排出[18]。作为“高原之舟”的牦牛,其肺部也产生了显著的适应性改变,如各级支气管管壁和血管壁内都有丰富的弹性纤维分布,杯状细胞大量分布于各级支气管和细支气管黏膜上皮中,其分泌的黏液有助于牦牛在干旱环境下保护呼吸道的通气量,同时气——血屏障的厚度明显比体格较小的其它家畜还要薄,这有利于气体交换时增加氧的弥散量[19]。
随着生物技术的快速发展,科研人员尝试在基因组水平上结合组织学研究解析高原动物如何适应高原极端环境。Brutsaert[20]研究了不同高原地区人的静息通气量,发现在低氧极端环境条件下,不同高原地区人的静息通气量是不同的,该研究揭示了在低氧环境下肺通气受基因、环境及其相互间作用的影响。近期的研究也指出,在低氧诱导途径中,阿片样受体激动剂的编码基因OPRM1具有刺激呼吸的重要作用,能够增加心肺呼吸功能和高原低氧适应性[21]。马俊兴等[22]研究发现,LRP6和VEGFR2不仅在不同年龄段的牦牛肺组织中表达量具有差异性,而且它们对高原环境下牦牛肺适应低氧的结构形成和维持可能的调控具有重要意义。
1.2 氧之转运——心血管系统心血管系统作为氧气循环的第二阶段,也是血氧结合和转运的关键部位。该系统由心、动脉、静脉、毛细血管及其流淌于其中的血液组成,而心作为心血管系统的动力器官,是维持机体血氧稳定的关键因素。通过该系统将血液泵入机体的各大血管系统,氧气及各种营养物质被运送至机体全身细胞利用,并带走细胞代谢废物。氧作为动物机体新陈代谢的重要物质基础,也是限制高原农牧业发展的主要因素之一。高原动物在长期自然选择和自身适应性进化过程中,其心血管系统也发生了显著的改变,以便维持机体的血氧平衡及能量代谢需求进而适应低氧环境。对生活在高海拔地区的牦牛、藏猪等高原动物的研究发现,高原动物为适应极端环境拥有发达的心和丰富的毛细血管系统[23-24]。
作为机体氧气运输者——血液,其中的氧气携带者红细胞(RBC)和血红蛋白(hemoglobin, Hb)主要负责机体内氧气的结合与释放。RBC和Hb含量的增多是许多高原动物为适应高原低氧环境,增强机体运氧能力最明显的血液学特征[25]。若只是增加RBC数量,会导致血液黏滞性增加,血流阻力增强,心负担加重,不利于机体健康[26]。研究发现,与平原动物相比,高原动物血液中的RBC数量与Hb含量发生了显著增加,但血液中的平均红细胞容积(MCV)和血细胞比容(HCT)较低,而平均红细胞血红蛋白浓度(MCHC)则较高,使得高原动物血液中的RBC拥有了更大的表面积,大大地提高了氧气结合、运输与交换能力,缓解了因RBC和Hb的增加所造成的血液黏稠,对于减小血液循环阻力、减轻心负担、提高氧气输送效率等具有重要意义[27-28]。李美荃等[29]对云南高原乌金猪与约大乌猪低氧适应研究发现,乌金猪低氧适应生理表征参数明显高于约大乌猪,在多数组织约大乌猪低氧适应基因的表达高于乌金猪。对于牦牛、蜂鸟、斑头雁等高原动物血液学的研究也证明了这一点,同时还发现,RBC氧亲和能力的提升也是一种动物适应高海拔地区低氧环境的重要途径[30-32]。
此外,高原动物也会通过调控一些与低氧适应性相关基因的表达来调整机体代谢水平以便适应低氧环境[8]。最新研究发现,低氧诱导因子(hypoxia-inducible factor, HIF)通路总共含有200多个相关基因,其中EPAS1和EGLN1基因最为关键[33]。EPAS1与低氧诱导的正性调节有关,是慢性缺氧相关基因的关键调节因素,主要参与促红细胞生成素(EPO)、血管内皮生长因子(VEGF)、内皮型一氧化氮合酶(eNOS)相关基因的上游调节[33]。而EGLN1的作用刚好相反,它主要与正常氧分压下HIF-1α和EPAS1蛋白的降解有关;在缺氧时,EGLN1基因的改变导致降解能力下降,间接提高了EPAS1蛋白的表达水平,增加了与缺氧调控有关物质的产量[33-34]。在人和小鼠上,EPAS1和EGLN1己被验证主要参与低氧诱导因子转录通路,对氧感受、协调机体对低氧的应答起主要作用[35]。
通过对藏猪和家猪基因组水平的基因选择性清除分析,鉴定出了家猪肌肉生长、脂肪沉积、免疫等受到人工强烈选择的基因516个,筛选出的268个与高海拔适应性相关的基因发生了快速进化,其中ALB、GLDC、SPTLC2、ECE1、PIK3C2G、GNG2等基因在藏猪中呈现极强的正向选择信号,而这些基因可以促进Hb的生成,并增强Hb与氧气的结合,对藏猪的高原适应性具有重要作用[36-37]。Qiu等[38]对青藏高原野生牦牛与普通黄牛进行全基因组测序发现,ADAM17、ARG2、MMP3等多个基因在牦牛上发生了适应性变异,这些变异有助于牦牛适应低氧环境,调整机体能量代谢,以便更好地生存于高原极端环境;而Camk2b、Gcnt3、Hsd17b12、Whsc1、Glu1的变异有助于牦牛在冷季食物匮乏的青藏高原极端环境中获得生命活动必须的能量。Wei等[39]通过对藏羊的全基因组分析发现,EPAS1、CRYAA、LONP1、NF1、DPP4、SOD1、PPARG、SOCS2等在藏羊适应高原低氧极端环境中具有重要作用,其中EPAS1在低氧适应中的作用尤为重要。如图 2所示,对其他高原土著动物的研究中,也发现了大量与高原低氧适应性相关的基因,通过对这些基因信息的深入挖掘,有助于解析动物高原适应性进化的遗传机制[40-43]。大量研究发现,参与调控低氧适应性的大部分基因都集中在HIF通路,主要参与调控机体的RBC生成、血管舒缩和生长、应激反应和糖代谢等生理过程,提高了机体氧的摄入和利用,进而增强了机体的高原低氧适应能力[44],通过对高原土著动物的研究证明了这一点[36-43]。同时,Bigham等[44-45]对不同高海拔地区人的高原适应性研究成果也证明了这一点。目前鉴定出的HIFs的靶基因有EPO、VEGF、NOSs等[46]。Verma等[47]利用转录组分析发现,在Ladakhi奶牛中与高海拔缺氧和应激反应相关的INHBC、ITPRI、HECA、ABI3、GPR171和HIF-1α基因的表达显著上调;同时qPCR分析发现,在高海拔牛类型中,DEGs如HIF-1、EPAS-1、VEGFA、NOS2和GLUT-1/SLC2A1的表达增加,表明这些基因对它们在高海拔极端环境中生存具有关键作用。另外,当平原动物进入高原地区后会出现明显的有氧呼吸抑制和无氧呼吸增强的现象,具体表现为线粒体数量和大小降低、氧化磷酸化功能的抑制和解偶联、ATP含量的降低、细胞色素c酶活性变化,通过线粒体这一系列的代偿性功能调控来实现对高原低氧环境的短期适应[48-49]。
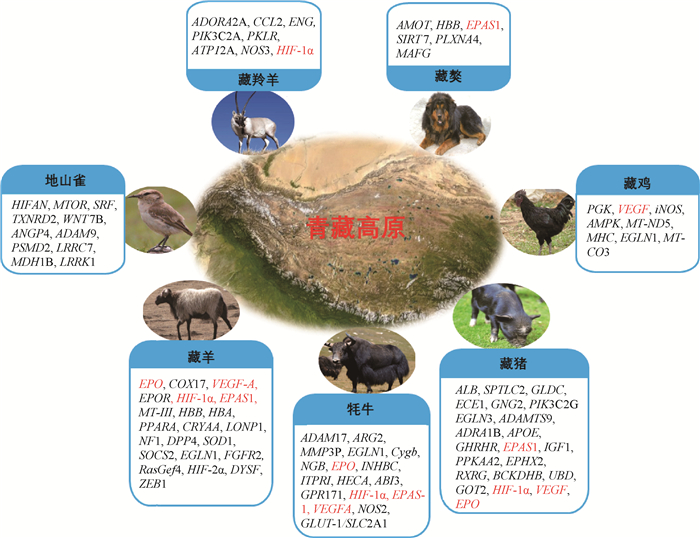
|
图中所示基因均为不同动物目前已研究发现的,其中红色表示不同动物所共有的基因;黑色表示不同动物所特有的基因 The genes showed in the figure have been discovered in different animals, in which the genes represented by red are the genes shared by different animals; the genes represented by black are the genes unique to different animals 图 2 青藏高原土著动物低氧适应性基因研究 Fig. 2 Study on hypoxia adaptation genes of indigenous animals in Qinghai-Tibet Plateau |
作为氧在机体的最终利用者——线粒体,它是机体有氧呼吸氧化磷酸化反应的最终场所,是机体各种生命活动的能量“供给站”,也是机体维持动物体温恒定的“供热站”。动物线粒体基因(mitochondrial DNA,mtDNA)是闭合环状的双链结构,大约含有16 500个碱基对;线粒体基因组由37个基因组成,这些基因都是OXPHOS、电子传递、线粒体蛋白合成所必需的[50],同时还可以编码氧化磷酸化体系中的13个核心蛋白质[51-53]。近年来,随着人们对青藏高原关注的不断提升,以线粒体为研究对象进行高原适应性研究已成为当下热点。1994年,Torroni等[54]关于高海拔地区人群的mtDNA分析指出,高海拔地区人群与普通低海拔地区人群相比具有50%以上的特异性mtDNA,证实mtDNA的变异可能是影响高原适应性的重要因素之一。2007年,Xu等[55]对藏马mtDNA进行分析,发现在所有的基因中,ND6基因的非同义变异最高,这一基因可能在高海拔适应中起作用。2008年,Luo等[56]对高原鼠兔的线粒体基因组分析得到15个特异的氨基酸变异位点,其中3个位于细胞色素c氧化酶基因中,这些氨基酸位点的变异可能对鼠兔适应低氧和寒冷环境产生影响。2010年,李莉等[57-58]研究发现,牦牛心肌、骨骼肌线粒体抗氧化能力随海拔的升高而增强,表明牦牛对高原低氧环境具有显著的适应性;同时研究还发现,随着海拔的升高,牦牛心肌、骨骼肌线粒体标志酶SDH、ATP表达量增加,肌组织有氧代谢能力增强;另外研究发现,生活在海拔4 200 m的牛和平原牛相比,高海拔牛心肌线粒体数目增加40%,而线粒体大小相似。2011年,Luo等[59]研究发现,藏族人群和汉族人群在nt3010G和nt3970C位点有显著区别,随后在mtDNA上发现了另外两个突变位点3397A/G和3552T/A,通过试验发现,HAPE患者的3397G和3552A频率显著高于对照组[60]。2013年,Luo等[61]提出,mtDNA应该作为高原适应性一个新的突破点。2011年,王召锋[62]对牦牛及其近缘物种北美野牛的线粒体蛋白编码序列进行了适应性进化分析,发现在牦牛线粒体蛋白跨膜螺旋区域中苏氨酸残基明显增加,而这一残基的增加有利于提高呼吸过程中的氧气结合效率以及氧化呼吸效率,从而对青藏高原的高海拔环境有很好的适应。张勤文等[63]研究发现,大通牦牛骨骼肌组织线粒体在出生时已获得良好的组织遗传学特性,表现为骨骼肌线粒体平均体积小、面数密度、体密度高的特点;而在个体生长发育过程中,表现为骨骼肌线粒体平均体积先降低再升高、面数密度先升高再降低,体密度逐渐增加。
李娜等[64]对不同海拔的几种沙蜥高海拔适应性研究发现,高海拔沙蜥线粒体基因组中蛋白编码基因的进化速率最快,其中ATP8基因的进化速率最快;同时发现,ATP8基因存在明显的正选择信号(P < 0.05, ω1),而且存在2个正选择位点。这些结果说明,ATP8基因可能在高海拔沙蜥的低氧适应中起到了重要的作用。李莉等[65]探讨了高原低氧环境下大通牦牛发育早期线粒体标志酶-琥珀酸脱氢酶(SDH)在不同组织中的表达模式,发现SDH基因表现为4月龄胎儿大脑组织和出生1日龄犊牦牛心肌组织中SDHA、SDHB、SDHC和SDHD 4个亚基转录水平均上调。
2017年,Shi等[66]研究发现,编码NADH脱氢酶的2个亚基的2个线粒体基因MT-ND1和MT-ND2可作为与高海拔适应性相关的候选基因。通过对藏羚羊、牦牛、高原鼠兔线粒体基因组进行进化分析后,发现细胞色素氧化酶1(COX1)基因都有高的非同义突变速率,表明COX1基因在动物适应高原高寒缺氧环境方面具有重要作用[67],同时又将COX1作为进行动物分类的一个标识符。另外,研究还发现,高原鼠兔出现了细胞色素c氧化基因[54]、青藏高原野生牦牛出现了Adam17、Arg2、Mmp3等多个线粒体基因的适应性变异[68],这些变异的线粒体基因在低氧反应中都发挥着重要作用。
线粒体是动物机体糖类、脂肪和氨基酸最终氧化释放能量的场所,同时也是研究群体遗传进化的重要手段。近年来对高原动物进行线粒体基因组测序分析,发现它们的线粒体基因组在ATP、COX、ND基因均发生变异,暗示mtDNA变异可能与高原适应相关。最新研究发现,43.6%的藏族胃癌患者中,8个mtDNA中发现了8个突变,预示着这些突变可能与胃癌的发现有关[69]。目前大多数mtDNA序列均是由小样本中测序得到的,对其序列进行生物信息学分析,发现它们与高原适应相关,但是缺乏直接的证据和功能学试验证明线粒体基因组变异与高原适应的关系。因此,应该通过DNA-RNA-蛋白质水平深入分析高原动物mtDNA的变化,并结合细胞融合试验研究这些变化与高原适应的关系,以期深入揭示线粒体基因组变异在高原环境适应的作用。
2 高原土著动物低温适应性进化的研究进展青藏高原素有“地球第三极”之称,其年均气温常在0 ℃以下,最暖月平均温度也不足10 ℃,其特殊的低温极端环境成为高原土著动物面临的主要生存挑战之一,而世代生活于此的高原土著动物经长期进化,已具备了一系列适应高原低温极端环境的生理、形态及行为特征,如鸟类会利用羽毛和迁徙抵抗寒冷;有些动物会利用增加毛绒和脂肪抵抗寒冷;有些动物可以利用冬眠抵抗寒冷;有些动物甚至可以根据环境温度的变化调节体温适应环境等。
恒温动物对低温环境的适应除了改变基础代谢率、减少热传导和增加毛皮的厚度以外,还可依靠两种不同的产热方式维持体温恒定:颤抖性产热(shivering thermogenesis, ST)和非颤抖性产热(non-shivering thermogenesis, NST),其中ST是通过骨骼肌无自律性收缩增加产热,而NST是通过体内物质代谢快速产热,是寒冷环境中机体维持体温恒定的主要方式之一[70]。研究发现,褐色脂肪组织(brown adipose tissue, BAT)是哺乳动物热发生的重要部位,是体内进行NST的主要器官,其活性对小型哺乳动物在寒冷环境中生存和冬眠觉醒至关重要[71-73]。热量主要来源于细胞内储存的脂质在燃烧过程中线粒体解偶联[74],而BAT中含有大量线粒体,位于线粒体内膜的解偶联蛋白(uncoupling protein, UCP)的活性和含量是决定产热的关键因素[75]。这种蛋白通过促进氧化呼吸链驱出的质子重新进入线粒体,在进行氧化磷酸化解偶联过程中使能量绕过ATP合成而以热能的形式散发出来,同时它受饮食、环境温度、去甲肾上腺素、甲状腺激素、胰岛素等因素的调控,但游离脂肪酸(free fatty acids, FFA)是影响UCP表达的主要因素[76]。
目前已知的UCP包括UCP1、UCP2、UCP3、UCP4、BMCP1以及植物中的UCP等,所有的UCP均属于线粒体阴离子载体蛋白家族(MACP),MACP中包括二磷酸腺苷、三磷酸腺苷载体蛋白、磷酸载体蛋白和其他载体蛋白[77]。ucp基因是由一个多基因家族构成,其中ucp1主要在BAT中表达,而UCP1是BAT解偶联呼吸产热的核心因素[78],其在食物资源匮乏和冷适应等条件下的调节产热作用已得到印证,例如剔除ucp1基因的小鼠将在寒冷中无法维持体温恒定[79]。UCP1所诱导的寒冷产热分为长期和短期两种,其中长期反应一般为几小时或几天,而短期反应一般在几分钟之内完成。在长期反应中PKA激活一系列级联反应,最终导致ucp1表达增加、新线粒体合成和褐色脂肪组织增生,为长时间产热做准备;而短期反应的机制是寒冷可诱导交感神经系统释放去甲肾上腺素(norepinephrine, NE),然后通过BAT细胞上的β-肾上腺素能受体(β-adrenergic receptor, β-AR)刺激环磷酸腺苷(cyclic Adenosine monophosphate, cAMP)的产生,cAMP激活蛋白激酶A(protein kinase A, PKA),PKA促进脂解的发生,释放出FFA,FFA促进UCP1对H+的运输,使其快速产热[80](图 3)。利用不同的抗生素配方处理小鼠以清除小鼠的肠道菌群,发现缺失肠道菌群的小鼠体温调控机制受到破坏,进一步研究发现,肠道菌群的缺失减弱了BAT中UCP-1蛋白表达量的增加,降低了WAT的棕色化水平,而且该结果在无菌小鼠中也得到了证实,说明肠道菌群在低温条件下对动物的体温调节过程起到了重要作用[81]。研究发现,2个UCP1(UCP1 rs6536991和UCP1 rs2071415)多态性与欧洲青少年的肥胖相关[82]。
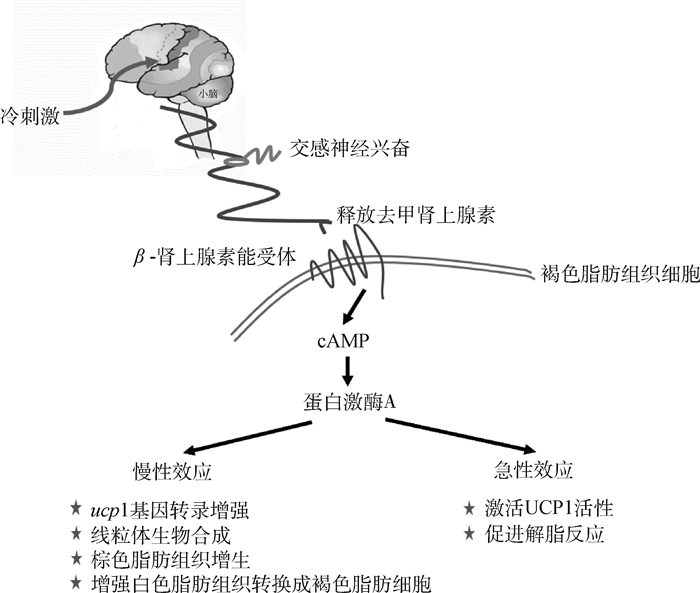
|
图 3 UCP1冷诱导适应性产热机制 Fig. 3 Mechanism of cold-induced adaptive thermogenesis induced by UCP1 |
ucp2基因在哺乳动物的大多数组织中均有表达,广泛分布于全身各组织器官如心、白色脂肪和免疫系统组织等[83];通过转基因和敲除小鼠的ucp2基因,发现在高脂饮食下ucp2表达与小鼠血糖和胰岛素分泌相关[84]。ucp3主要在小鼠的骨骼肌、BAT中表达,研究表明, ucp3和ucp2的表达与脂肪酸代谢密切相关,可通过改变活性氧(reactive oxygen species, ROS),控制脂毒性和氧化损害作用,阻止脂肪酸氧化及脂质积累[85];但剔除小鼠ucp2或ucp3在寒冷条件下对体温的维持没有影响[86-87],表明在寒冷诱导的适应性产热过程中ucp3和ucp2并未起重要作用;但研究发现,藏猪脂肪细胞中UCP3能显著提高脂肪细胞氧化呼吸速率,说明UCP3是藏猪抗寒产热的一个重要基因,并且推翻了UCP1是唯一产热解耦联蛋白的说法[88]。最新研究发现,ucp1和ucp3之间的互作对肥胖和肥胖症具有重要影响[89]。ucp4和bmcp1主要表达于脑中,其中UCP4是大脑特有的线粒体UCP同族体[83],它是一种神经内分泌激素,对神经结构、代谢率和脑的适应性产热有调控作用,并与控制食欲有关。
除UCP外,研究人员又发现了一种主要来源于脂肪组织,且在控制食物摄入、能量消耗和性行为等方面发挥重要作用的激素——瘦素[90-92]。研究发现,血清瘦素水平与体重和体脂质量呈正相关,却与能量摄入和UCP1含量呈负相关,且分泌水平呈现季节性波动,秋冬季明显低于春夏季[11, 93-94],造成这种差异的原因可能是秋冬季节气温降低、营养匮乏,在保障维持需要的前提下,并未有多余的营养物质用于转化生成脂肪,反而增加了脂肪的降解,用于维持体温恒定,故血清瘦素水平降低,而UCP含量却增加,两者出现了一种拮抗作用。
综上所述,在寒冷环境中,通过UCP的解偶联作用产热,对机体维持体温恒定﹑能量平衡、代谢以及体重调节有重要作用;同时,瘦素、LDH等对高原低温适应性研究提供了新思路,有望通过研究低温适应性,了解脂肪转化产热代谢机理,找到治疗人肥胖症的方法和思路。
3 高原土著动物抗辐射适应性进化的研究进展近30年来,随着工农业的快速发展,大气中氯氟烃、一氧化二氮等污染物质不断增多,导致平流层中臭氧层逐渐变薄,到达地表的太阳紫外辐射(ultraviolet radiation, UVR)增强,受太阳紫外辐射引起的生物效应已成为全球广泛关注的主要环境问题之一。因此,探索紫外辐射引起的生物学效应,不仅对医疗事业有重要的实用价值,同时对生物多样性和保护生命起源以及空间生物学等方面的研究也具有重要的参考价值[95-96]。少量的紫外辐射作为自然界中的重要诱变源,可促使生物遗传物质的突变,形成自然界丰富的生物多样性,也可诱导动物的孤雌生殖[97],同时也有助于人体维生素D的合成。
过量的紫外辐射会对生物体产生一定程度的损伤,Setlow等[98]和Caldwell[99]较早地研究了UVR对人体的伤害以及对动物的生物作用。地表UVR增强对地表生物、生态系统和人类活动将产生深远的影响,它会引起人类多种疾病,包括白内障及皮肤癌发病率增高[100]。同时,过量紫外线辐射还将导致鱼类孵化率降低,幼鱼死亡率升高[101],伤害水中浮游生物,大大降低水生产力,破坏鱼类的食物链基础,导致水中蛋白质大量减少,进一步导致人类食物供应短缺[102]。同时,还有大量证据证实,全球两栖动物种群数量正在下降,可能与过量紫外线辐射造成胚胎基因发生突变,引起机体癌变、细胞周期停滞、凋亡,两栖动物胚胎致死和亚致死有重要关系[103-104]。青藏高原上强烈的太阳紫外辐射,为研究强紫外辐射对动物生理生化等方面的影响提供了天然试验材料,也为寻求抗紫外辐射的相关生理机制提供了可能。通过对藏猪和家猪比较基因组研究发现,藏猪进化出了在强紫外线下保护皮肤的特有基因,为研究强紫外线加速皮肤老化和抗“光衰老”等人类相关疾病提供了重要线索[36]。通过对高原动物抗紫外辐射的研究,有望找到某些与抗紫外辐射的相关基因或技术用于预防和治疗人类皮肤癌等疾病。
4 其他相关研究目前,大多数高原适应性研究都集中于低氧、低温适应性研究,主要从与氧气的摄入、运输、利用以及能量代谢等相关组织和基因上进行研究,却很少关注与其共同进化的微生物群落。随着近年来测序技术的飞速发展,人们对机体内微生物基因组信息的关注越来越多。因此,有些研究人员从微生物角度解释高原动物如何适应高原极端环境的,并取得了一定成果。Zhang等[105]利用超深宏基因组测序揭示了高海拔反刍动物瘤胃微生物基因向VFA产生途径的显著富集。同时,对RNA转录组的分析发现了与高海拔反刍动物瘤胃上皮细胞中VFA转运和吸收相关的36个基因的显著上调。同时,体外模拟瘤胃发酵试验发现,高原反刍动物产生更多宿主可利用的挥发性脂肪酸和更少的甲烷,说明高原反刍动物的能量利用更高效,从侧面揭示了高原反刍动物具有更好的高原适应性[106]。最新研究发现,肠道菌群在低温条件下对动物的体温调节过程起着重要作用[81]。由于微生物无处不在,对处于特殊环境各种微生物的研究,可以发现某些具有重大意义的成果,比如对低温微生物的研究有助于冷冻食品的质量安全控制[107]。因此,随着研究工作的不断深入,基因工程、酶固定化等生物技术手段的运用,低温微生物具有诱人的应用前景。最近,Liu等[108]研究组装了青藏高原特有物种——黑斑原鮡的基因组,为进一步研究鱼类高海拔适应性分子机制,解析高原鱼类物种形成机制及后期种质资源保护提供了理论支持。Sun等[109]通过比较分析了生活于青藏高原上的青蛙和沙蜥,发现与DNA修复和能量代谢相关的基因在高原适应性进化过程中具有重要作用;同时,Gao等[110]利用不同高原沙蜥种类揭示了爬行动物卵生和胎生繁殖模式进化的遗传机制,为阐述卵生如何进化为胎生提供了重要的理论基础。
5 展望目前,尽管国内外关于动物对高原极端环境低氧、低温及太阳辐射适应性进化的相关研究已取得丰硕成果,但仍未全面解析高原动物适应高原极端环境的分子机制,且大多数研究都集中在其中某一方面,并未全面考虑高原低氧、低温和强紫外辐射对高原动物适应性的影响。随着各项科学技术的快速发展,有关高原动物适应性进化的相关研究需整合目前已发现的所有与高原适应性相关基因的分子机制和生理基础,综合考虑各项研究成果,全面、系统的从DNA—RNA—蛋白质水平深入解析高原动物适应性进化的分子机制和遗传原理。同时,还需从高原动物消化道微生物出发,寻找不同微生物对动物高原适应性进化做出的贡献。通过对高原土著动物适应性进化的系统研究,不仅为培育耐低温、低氧及强紫外辐射的动物新品种提供理论基础,也为临床预防和治疗高原性疾病提供新思路,更为提高藏区家畜生产性能提供技术支撑。
| [1] | BENCOWITZ H Z, WAGNER P D, WEST J B. Effect of change in P50 on exercise tolerance at high altitude:a theoretical study[J]. J Appl Physiol Respir Environ Exerc Physiol, 1982, 53(6): 1487–1495. |
| [2] | RAMIREZ J M, FOLKOW L P, BLIX A S. Hypoxia tolerance in mammals and birds:from the wilderness to the clinic[J]. Annu Rev Physiol, 2007, 69: 113–143. DOI: 10.1146/annurev.physiol.69.031905.163111 |
| [3] | BEALL C M. Two routes to functional adaptation:Tibetan and Andean high-altitude natives[J]. Proc Natl Acad Sci U S A, 2007, 104(Suppl 1): 8655–8660. |
| [4] | LABIE D, ELION J. Genetics of adaptation to high altitude[M]. Chichester: John Wiley & Sons, Ltd, 2012: 1413-1426. |
| [5] | BEALL C M. Andean, Tibetan, and Ethiopian patterns of adaptation to high-altitude hypoxia[J]. Integr Comp Biol, 2006, 46(1): 18–24. DOI: 10.1093/icb/icj004 |
| [6] | KAELIN W G Jr, RATCLIFFE P J. Oxygen sensing by metazoans:the central role of the HIF hydroxylase pathway[J]. Mol Cell, 2008, 30(4): 393–402. DOI: 10.1016/j.molcel.2008.04.009 |
| [7] | SIMONSON T S, MCCLAIN D A, JORDE L B, et al. Genetic determinants of Tibetan high-altitude adaptation[J]. Hum Genet, 2012, 131(4): 527–533. DOI: 10.1007/s00439-011-1109-3 |
| [8] | BIGHAM A W, WILSON M J, JULIAN C G, et al. Andean and Tibetan patterns of adaptation to high altitude[J]. Am J Hum Biol, 2013, 25(2): 190–197. DOI: 10.1002/ajhb.22358 |
| [9] | DING X Z, LIANG C N, GUO X, et al. Physiological insight into the high-altitude adaptations in domesticated yaks (Bos grunniens) along the Qinghai-Tibetan Plateau altitudinal gradient[J]. Livest Sci, 2014, 162: 233–239. DOI: 10.1016/j.livsci.2014.01.012 |
| [10] | WANG Y D, ZHANG J, LI C H, et al. Molecular cloning, sequence characteristics, and tissue expression analysis of ECE1 gene in Tibetan pig[J]. Gene, 2015, 571(2): 237–244. DOI: 10.1016/j.gene.2015.06.054 |
| [11] | YANG J, WANG Z L, ZHAO X Q, et al. Natural selection and adaptive evolution of leptin in the Ochotona family driven by the cold environmental stress[J]. PLoS One, 2008, 3(1): e1472. DOI: 10.1371/journal.pone.0001472 |
| [12] | LI Q F, SUN R Y, HUANG C X, et al. Cold adaptive thermogenesis in small mammals from different geographical zones of China[J]. Comp Biochem Physiol A Mol Integr Physiol, 2001, 129(4): 949–961. DOI: 10.1016/S1095-6433(01)00357-9 |
| [13] | SHAO B, LONG R, DING Y, et al. Morphological adaptations of yak (Bos grunniens) tongue to the foraging environment of the Qinghai-Tibetan Plateau[J]. J Anim Sci, 2010, 88(8): 2594–2603. DOI: 10.2527/jas.2009-2398 |
| [14] |
王晓君, 魏登邦, 魏莲, 等. 高原鼢鼠和高原鼠兔肺细叶的结构特征[J]. 动物学报, 2008, 54(3): 531–539.
WANG X J, WEI D B, WEI L, et al. Characteristics of pulmonary acinus structure in the plateau zokor Myospalax baileyi and plateau pika Ochotona curzniae[J]. Acta Zoologica Sinica, 2008, 54(3): 531–539. (in Chinese) |
| [15] |
祁玥.高原鼠兔骨骼肌VEGF基因的特异性研究[D].西宁: 青海大学, 2012.
QI Y.Study on specificity of VEGF gene in plateau pika (Ochotona curzoniae) skeletal muscle tissue[D].Xi'ning: Qinghai University, 2012. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10743-1012034111.htm |
| [16] |
许永华, 张东辉, 许琴, 等. 西藏小型猪心脏、呼吸系统组织学观察[J]. 中国比较医学杂志, 2009, 19(8): 61–62.
XU Y H, ZHANG D H, XU Q, et al. Histological observation of heart and respiratory system in Tibet minipigs[J]. Chinese Journal of Comparative Medicine, 2009, 19(8): 61–62. DOI: 10.3969/j.issn.1671-7856.2009.08.016 (in Chinese) |
| [17] |
贾荣莉. 移居高原小尾寒羊与藏羊肺组织形态学比较研究[J]. 四川畜牧兽医, 2001, 28(5): 25, 27.
JIA R L. The study on morphology of lung tissue of migrated to plateau Xiao Wei Han-sheep and Tibetan-sheep[J]. Sichuan Animal & Veterinary Sciences, 2001, 28(5): 25, 27. (in Chinese) |
| [18] |
张先钧, 张晓岩, 蒲小燕. 探讨藏羚羊在高寒低氧环境肺脏的光镜结构[J]. 现代预防医学, 2011, 38(12): 2377–2378.
ZHANG X J, ZHANG X Y, PU X Y. Investating the ultrastructure of lung of hide-gazella who stag for a long time in highautitude district in hypso-frigidity and hypoxemia environment[J]. Modern Preventive Medicine, 2011, 38(12): 2377–2378. (in Chinese) |
| [19] |
陈秋生, 冯霞, 姜生成. 牦牛肺脏高原适应性的结构研究[J]. 中国农业科学, 2006, 39(10): 2107–2113.
CHEN Q S, FENG X, JIANG S C. Structural study on plateau adaptability of yak lung[J]. Scientia Agricultura Sinica, 2006, 39(10): 2107–2113. DOI: 10.3321/j.issn:0578-1752.2006.10.024 (in Chinese) |
| [20] | BRUTSAERT T D. Population genetic aspects and phenotypic plasticity of ventilatory responses in high altitude natives[J]. Respir Physiol Neurobiol, 2007, 158(2-3): 151–160. DOI: 10.1016/j.resp.2007.03.004 |
| [21] | JI L D, QIU Y Q, XU J, et al. Genetic adaptation of the hypoxia-inducible factor pathway to oxygen pressure among Eurasian human populations[J]. Mol Biol Evol, 2012, 29(11): 3359–3370. DOI: 10.1093/molbev/mss144 |
| [22] |
马俊兴, 何俊峰, 崔燕, 等. LRP6和VEGFR2在牦牛肺的分布[J]. 畜牧兽医学报, 2018, 49(7): 1550–1557.
MA J X, HE J F, CUI Y, et al. The distribution of LRP6 and VEGFR2 in yak lungs[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(7): 1550–1557. (in Chinese) |
| [23] |
李朝旭, 段德勇, 崔燕. 牦牛心室普肯耶纤维的分布和结构特点[J]. 解剖学报, 2013, 44(1): 79–84.
LI C X, DUAN D Y, CUI Y. Distribution and structural characteristics of the ventricular Purkinje fibers of yaks[J]. Acta Anatomica Sinica, 2013, 44(1): 79–84. DOI: 10.3969/j.issn.0529-1356.2013.01.015 (in Chinese) |
| [24] |
黄文耀, 张鏸予, 李平, 等. 甘孜州藏猪内脏器官发育特征研究[J]. 西南大学学报:自然科学版, 2017, 39(11): 37–44.
HUANG W Y, ZHANG H Y, LI P, et al. The characteristics of the development of the visceral organs of the Tibetan pig in Ganzi of Sichuan[J]. Journal of Southwest University:Natural Science Edition, 2017, 39(11): 37–44. (in Chinese) |
| [25] | CRAIT J R, PRANGE H D, MARSHALL N A, et al. High-altitude diving in river otters:coping with combined hypoxic stresses[J]. J Exp Biol, 2012, 215(2): 256–263. DOI: 10.1242/jeb.059774 |
| [26] |
齐新章, 袁琦, 曹兴玲, 等. 岩羊血液对低氧环境的适应特性研究[J]. 安徽农业科学, 2013, 41(8): 3392, 3406.
QI X Z, YUAN Q, CAO X L, et al. Adaptation of the blood of blue sheep (Pseudois nayaur) to hypoxic conditions[J]. Journal of Anhui Agricultural Sciences, 2013, 41(8): 3392, 3406. (in Chinese) |
| [27] | SUN K Q, ZHANG Y J, D'ALESSANDRO A, et al. Sphingosine-1-phosphate promotes erythrocyte glycolysis and oxygen release for adaptation to high-altitude hypoxia[J]. Nat Commun, 2016, 7: 12086. DOI: 10.1038/ncomms12086 |
| [28] |
黄文超. 高原红细胞增多与高血粘度发生的相关分析[J]. 西南军医, 2010, 12(6): 1047–1048.
HUANG W C. Correlation analysis of polycythemia and the occurrence of high blood viscosity at high altitude[J]. Journal of Military Surgeon in Southwest China, 2010, 12(6): 1047–1048. (in Chinese) |
| [29] |
李美荃, 张春勇, 安清聪, 等. 云南乌金猪与约大乌猪低氧适应性的研究[J]. 畜牧兽医学报, 2015, 46(6): 965–973.
LI M Q, ZHANG C Y, AN Q C, et al. The study of hypoxia adaptive differences of Yunnan Wujin and Yuedawu pigs[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(6): 965–973. (in Chinese) |
| [30] | ISHIZAKI T, KOIZUMI T, RUAN Z H, et al. Nitric oxide inhibitor altitude-dependently elevates pulmonary arterial pressure in high-altitude adapted yaks[J]. Respir Physiol Neurobiol, 2005, 146(2-3): 225–230. DOI: 10.1016/j.resp.2004.12.002 |
| [31] | PROJECTO-GARCIA J, NATARAJAN C, MORIYAMA H, et al. Repeated elevational transitions in hemoglobin function during the evolution of Andean hummingbirds[J]. Proc Natl Acad Sci U S A, 2013, 110(51): 20669–20674. DOI: 10.1073/pnas.1315456110 |
| [32] | LIU X Z, LI S L, JING H, et al. Avian haemoglobins and structural basis of high affinity for oxygen:structure of bar-headed goose aquomet haemoglobin[J]. Acta Crystallogr D Biol Crystallogr, 2001, 57(6): 775–783. DOI: 10.1107/S0907444901004243 |
| [33] | HU C J, WANG L Y, CHODOSH L A, et al. Differential roles of hypoxia-inducible factor 1α (HIF-1α) and HIF-2α in hypoxic gene regulation[J]. Mol Cell Biol, 2003, 23(24): 9361–9374. DOI: 10.1128/MCB.23.24.9361-9374.2003 |
| [34] | PATEL S A, SIMON M C. Biology of hypoxia-inducible factor-2α in development and disease[J]. Cell Death Differ, 2008, 15(4): 628–634. DOI: 10.1038/cdd.2008.17 |
| [35] | PETOUSI N, ROBBINS P A. Human adaptation to the hypoxia of high altitude:the Tibetan paradigm from the pregenomic to the postgenomic era[J]. J Appl Physiol, 2014, 116(7): 875–884. DOI: 10.1152/japplphysiol.00605.2013 |
| [36] | LI M Z, TIAN S L, JIN L, et al. Genomic analyses identify distinct patterns of selection in domesticated pigs and Tibetan wild boars[J]. Nat Genet, 2013, 45(12): 1431–1438. DOI: 10.1038/ng.2811 |
| [37] |
金龙.藏猪高原适应性相关基因的鉴定及其DNA甲基化调控规律[D].雅安: 四川农业大学, 2015.
JIN L.Genome-wide detection of high-altitude related genes and DNA methylation regulation of gene expression in Tibetan pigs[D].Ya'an: Sichuan Agricultural University, 2015. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10626-1016052066.htm |
| [38] | QIU Q, ZHANG G J, MA T, et al. The yak genome and adaptation to life at high altitude[J]. Nat Genet, 2012, 44(8): 946–949. DOI: 10.1038/ng.2343 |
| [39] | WEI C H, WANG H H, LIU G, et al. Genome-wide analysis reveals adaptation to high altitudes in Tibetan sheep[J]. Sci Rep, 2016, 6: 26770. DOI: 10.1038/srep26770 |
| [40] | GOU X, WANG Z, LI N, et al. Whole-genome sequencing of six dog breeds from continuous altitudes reveals adaptation to high-altitude hypoxia[J]. Genome Res, 2014, 24(8): 1308–1315. DOI: 10.1101/gr.171876.113 |
| [41] | WANG M S, LI Y, PENG M S, et al. Genomic analyses reveal potential independent adaptation to high altitude in Tibetan chickens[J]. Mol Biol Evol, 2015, 32(7): 1880–1889. DOI: 10.1093/molbev/msv071 |
| [42] | QU Y H, ZHAO H W, HAN N J, et al. Ground tit genome reveals avian adaptation to living at high altitudes in the Tibetan plateau[J]. Nat Commun, 2013, 4: 2071. DOI: 10.1038/ncomms3071 |
| [43] | GE R L, CAI Q L, SHEN Y Y, et al. Draft genome sequence of the Tibetan antelope[J]. Nat Commun, 2013, 4: 1858. DOI: 10.1038/ncomms2860 |
| [44] | BIGHAM A W, LEE F S. Human high-altitude adaptation:forward genetics meets the HIF pathway[J]. Gene Dev, 2014, 28(20): 2189–2204. DOI: 10.1101/gad.250167.114 |
| [45] | BIGHAM A W. Genetics of human origin and evolution:high-altitude adaptations[J]. Curr Opin Genet Dev, 2016, 41: 8–13. DOI: 10.1016/j.gde.2016.06.018 |
| [46] | PUGH C W, RATCLIFFE P J. Regulation of angiogenesis by hypoxia:role of the HIF system[J]. Nat Med, 2003, 9(6): 677–684. DOI: 10.1038/nm0603-677 |
| [47] | VERMA P, SHARMA A, SODHI M, et al. Transcriptome analysis of circulating PBMCs to understand mechanism of high altitude adaptation in native cattle of ladakh region[J]. Sci Rep, 2018, 8: 7681. DOI: 10.1038/s41598-018-25736-7 |
| [48] | CHITRA L, BOOPATHY R. Altered mitochondrial biogenesis and its fusion gene expression is involved in the high-altitude adaptation of rat lung[J]. Respir Physiol Neurobiol, 2014, 192: 74–84. DOI: 10.1016/j.resp.2013.12.007 |
| [49] | ZHANG H X, LUO Q B, SUN J, et al. Mitochondrial genome sequences of Artemia tibetiana and Artemia urmiana:assessing molecular changes for high plateau adaptation[J]. Sci China Life Sci, 2013, 56(5): 440–452. DOI: 10.1007/s11427-013-4474-4 |
| [50] |
张世扬, 肖振贤, 赵建龙, 等. 基于连接酶检测反应的并行分型系统检测AGT M235T和ACE I/D基因多态性[J]. 华东理工大学学报:自然科学版, 2006, 32(9): 1050–1054.
ZHANG S Y, XIAO Z X, ZHAO J L, et al. A typing system based on ligase detection reaction for AGT M235T and ACE I/D gene polymorphism[J]. Journal of East China University of Science and Technology:Natural Science Edition, 2006, 32(9): 1050–1054. (in Chinese) |
| [51] | KEDDIE E M, HIGAZI T, UNNASCH T R. The mitochondrial genome of Onchocerca volvulus:sequence, structure and phylogenetic analysis[J]. Mol Biochem Parasitol, 1998, 95(1): 111–127. DOI: 10.1016/S0166-6851(98)00102-9 |
| [52] | SBISÀ E, TANZARIELLO F, REYES A, et al. Mammalian mitochondrial D-loop region structural analysis:identification of new conserved sequences and their functional and evolutionary implications[J]. Gene, 1997, 205(1-2): 125–140. DOI: 10.1016/S0378-1119(97)00404-6 |
| [53] | HIKOSAKA K, WATANABE Y I, TSUJI N, et al. Divergence of the mitochondrial genome structure in the apicomplexan parasites, Babesia and Theileria[J]. Mol Biol Evol, 2010, 27(5): 1107–1116. DOI: 10.1093/molbev/msp320 |
| [54] | TORRONI A, MILLER J A, MOORE L G, et al. Mitochondrial DNA analysis in Tibet:implications for the origin of the Tibetan population and its adaptation to high altitude[J]. Am J Phys Anthropol, 1994, 93(2): 189–199. DOI: 10.1002/ajpa.1330930204 |
| [55] | XU S Q, LUOSANG J B, HUA S, et al. High altitude adaptation and phylogenetic analysis of Tibetan horse based on the mitochondrial genome[J]. J Genet Genomics, 2007, 34(8): 720–729. DOI: 10.1016/S1673-8527(07)60081-2 |
| [56] | LUO Y J, GAO W X, GAO Y Q, et al. Mitochondrial genome analysis of Ochotona curzoniae and implication of cytochrome c oxidase in hypoxic adaptation[J]. Mitochondrion, 2008, 8(5-6): 352–357. DOI: 10.1016/j.mito.2008.07.005 |
| [57] |
李莉. 不同海拔地区牦牛心肌、骨骼肌线粒体氧自由基代谢的研究[J]. 畜牧与兽医, 2010, 42(9): 65–68.
LI L. Metabolism of mitochondrial oxygen free radicals in yak heart muscle and skeletal muscle at different altitudes[J]. Animal Husbandry & Veterinary Medicine, 2010, 42(9): 65–68. (in Chinese) |
| [58] |
李莉, 王勇, 荆海霞, 等. 不同海拔地区牦牛心肌、骨骼肌线粒体标志酶的测定[J]. 畜牧与兽医, 2013, 45(1): 69–71.
LI L, WANG Y, JING H X, et al. Determination of mitochondrial marker enzymes in yak myocardium and skeletal muscle at different altitudes[J]. Animal Husbandry & Veterinary Medicine, 2013, 45(1): 69–71. (in Chinese) |
| [59] | LUO Y J, GAO W X, LIU F Y, et al. Mitochondrial nt3010G-nt3970C haplotype is implicated in high-altitude adaptation of Tibetans[J]. Mitochondrial DNA, 2011, 22(5-6): 181–190. DOI: 10.3109/19401736.2011.632771 |
| [60] | LUO Y J, GAO W X, CHEN Y, et al. Rare mitochondrial DNA polymorphisms are associated with high altitude pulmonary edema (HAPE) susceptibility in Han Chinese[J]. Wilderness Environ Med, 2012, 23(2): 128–132. DOI: 10.1016/j.wem.2012.02.003 |
| [61] | LUO Y J, YANG X H, GAO Y Q. Mitochondrial DNA response to high altitude:a new perspective on high-altitude adaptation[J]. Mitochondrial DNA, 2013, 24(4): 313–319. DOI: 10.3109/19401736.2012.760558 |
| [62] |
王召锋.牦牛驯化和适应: 线粒体基因组证据[D].兰州: 兰州大学, 2011.
WANG Z F.Yak domestication and adaptation: evidence from mitochondrial genome[D].Lanzhou: Lanzhou University, 2011. (in Chinese) paperuri: (03dd30b46e090849633ec2bcf02d3aaa) |
| [63] |
张勤文, 俞红贤, 荆海霞, 等. 基于骨骼肌线粒体超微结构研究生长期牦牛低氧适应性[J]. 畜牧兽医学报, 2013, 44(3): 447–452.
ZHANG Q W, YU H X, JING H X, et al. Adaptation mechanism of developmental yak to hypoxia base on skeletal muscle mitochondria ultrastructure[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(3): 447–452. (in Chinese) |
| [64] |
李娜, 杨伟钊, 傅金钟. 基于线粒体基因组的沙蜥高海拔适应研究[J]. 四川动物, 2015, 34(6): 810–816.
LI N, YANG W Z, FU J Z. High-altitude adaptation of genus Phrynocephalus based on mitochondrial genome[J]. Sichuan Journal of Zoology, 2015, 34(6): 810–816. (in Chinese) |
| [65] |
李莉, 柳梦琪, 胡佩莹, 等. 大通牦牛胎盘与胎儿组织线粒体SDH基因分析[J]. 畜牧兽医学报, 2016, 47(2): 284–289.
LI L, LIU M Q, HU P Y, et al. The succinate dehydrogenase genetic analysis of placenta and fetus tissue mitochondria in Datong yak[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(2): 284–289. (in Chinese) |
| [66] | SHI Y, HU Y S, WANG J, et al. Genetic diversities of MT-ND1 and MT-ND2 genes are associated with high-altitude adaptation in yak[J]. Mitochondrial DNA Part A, 2018, 29(3): 485–494. DOI: 10.1080/24701394.2017.1307976 |
| [67] |
徐树青.线粒体基因组的变异与哺乳动物高原适应性[D].昆明: 中国科学院昆明动物研究所, 2007.
XU S Q.Variation of mitochondrial genome and adaptability of mammalian plateau[D].Kunming: Kunming Institute of Zoology, Chinese Academy of Sciences, 2007. (in Chinese) |
| [68] |
刘强.从线粒体细胞色素b基因和D-LOOP控制区序列差异研究野牦牛和家牦牛的系统进化关系[D].杭州: 浙江大学, 2005.
LIU Q.Phylogenetic relationship between wild yak and domestic yak from mitochondrial cytochrome b gene and D-LOOP control region sequence differences[D].Hangzhou: Zhejiang University, 2005. (in Chinese) http://cdmd.cnki.com.cn/article/cdmd-10335-2005128132.htm |
| [69] | JIANG J, ZHAO J H, WANG X L, et al. Analysis of mitochondrial DNA in Tibetan gastric cancer patients at high altitude[J]. Mol Clin Oncol, 2015, 3(4): 875–879. DOI: 10.3892/mco.2015.539 |
| [70] | JANSKY L. Non-shivering thermogenesis and its thermoregulatory significance[J]. Biol Rev Camb Philos Soc, 1973, 48(1): 85–132. |
| [71] | KHEDOE P P S J, HOEKE G, KOOIJMAN S, et al. Brown adipose tissue takes up plasma triglycerides mostly after lipolysis[J]. J Lipid Res, 2015, 56(1): 51–59. DOI: 10.1194/jlr.M052746 |
| [72] | HOEKE G, KOOIJMAN S, BOON M R, et al. Role of brown fat in lipoprotein metabolism and atherosclerosis[J]. Circ Res, 2016, 118(1): 173–182. DOI: 10.1161/CIRCRESAHA.115.306647 |
| [73] | VAN DEN BERG B, KOOIJMAN S, NOORDAM R, et al. A diurnal rhythm in brown adipose tissue causes rapid clearance and combustion of plasma lipids at wakening[J]. Cell Rep, 2018, 22(13): 3521–3533. DOI: 10.1016/j.celrep.2018.03.004 |
| [74] | BERBÉ J F P, BOON M R, KHEDOE P P S J, et al. Brown fat activation reduces hypercholesterolaemia and protects from atherosclerosis development[J]. Nat Commun, 2015, 6: 6356. DOI: 10.1038/ncomms7356 |
| [75] | KAJIMURA S, SAITO M. A new era in brown adipose tissue biology:molecular control of brown fat development and energy homeostasis[J]. Annu Rev Physiol, 2014, 76: 225–249. DOI: 10.1146/annurev-physiol-021113-170252 |
| [76] |
柴艳, 李琦华, 田允波, 等. 冷环境中解偶联蛋白对动物体温的调节作用[J]. 家畜生态学报, 2008, 29(3): 104–108.
CAI Y, LI Q H, TIAN Y B, et al. Uncoupling proteins regulation on body temperature in cold environment[J]. Acta Ecologae Animalis Domastici, 2008, 29(3): 104–108. DOI: 10.3969/j.issn.1673-1182.2008.03.025 (in Chinese) |
| [77] | RICQUIER D, BOUILLAUD F. The uncoupling protein homologues:UCP1, UCP2, UCP3, StUCP and AtUCP[J]. Biochem J, 2000, 345(2): 161–179. DOI: 10.1042/bj3450161 |
| [78] | NEDERGAARD J, GOLOZOUBOVA V, MATTHIAS A, et al. UCP1:the only protein able to mediate adaptive non-shivering thermogenesis and metabolic inefficiency[J]. Biochim Biophys Acta, 2001, 1504(1): 82–106. DOI: 10.1016/S0005-2728(00)00247-4 |
| [79] |
肖放, 孙野青. 解偶联蛋白及功能研究进展[J]. 生命的化学, 2003, 23(1): 14–17.
XIAO F, SUN Y Q. Progress in the study of uncoupling proteins and their functions[J]. Chemistry of Life, 2003, 23(1): 14–17. DOI: 10.3969/j.issn.1000-1336.2003.01.007 (in Chinese) |
| [80] | NIBBELINK M, MOULIN K, ARNAUD E, et al. Brown fat UCP1 is specifically expressed in uterine longitudinal smooth muscle cells[J]. J Biol Chem, 2001, 276(50): 47291–47295. DOI: 10.1074/jbc.M105658200 |
| [81] | LI B G, LI L, LI M, et al. Microbiota depletion impairs thermogenesis of brown adipose tissue and browning of white adipose tissue[J]. Cell Rep, 2019, 26(10): 2720–2737. DOI: 10.1016/j.celrep.2019.02.015 |
| [82] | PASCUAL-GAMARRA J M, SALAZAR-TORTOSA D, MARTINEZ-TELLEZ B, et al. Association between UCP1, UCP2, and UCP3 gene polymorphisms with markers of adiposity in European adolescents:the HELENA study[J]. Pediatr Obes, 2019, 14(6): e12504. DOI: 10.1111/ijpo.12504 |
| [83] | BOUILLAUD F, COUPLAN E, PECQUEUR C, et al. Homologues of the uncoupling protein from brown adipose tissue (UCP1):UCP2, UCP3, BMCP1 and UCP4[J]. Biochim Biophys Acta, 2001, 1504(1): 107–119. DOI: 10.1016/S0005-2728(00)00241-3 |
| [84] |
冉林武, 陈菲, 鲁晓丽, 等. UCP2+/+和UCP2-/-小鼠的鉴定及表型初步分析[J]. 江苏医药, 2018, 44(1): 5–8.
RAN L W, CHEN F, LU X L, et al. Identification and elementary phenotype analysis of UCP2+/+and UCP2-/-mice[J]. Jiangsu Medical Journal, 2018, 44(1): 5–8. (in Chinese) |
| [85] | SAMEC S, SEYDOUX J, RUSSELL A P, et al. Skeletal muscle heterogeneity in fasting-induced upregulation of genes encoding UCP2, UCP3, PPARγ and key enzymes of lipid oxidation[J]. Pflugers Arch, 2002, 445(1): 80–86. DOI: 10.1007/s00424-002-0879-9 |
| [86] | ARSENIJEVIC D, ONUMA H, PECQUEUR C, et al. Disruption of the uncoupling protein-2 gene in mice reveals a role in immunity and reactive oxygen species production[J]. Nat Genet, 2000, 26(4): 435–439. DOI: 10.1038/82565 |
| [87] | VIDAL-PUIG A J, GRUJIC D, ZHANG C Y, et al. Energy metabolism in uncoupling protein 3 gene knockout mice[J]. J Biol Chem, 2000, 275(21): 16258–16266. DOI: 10.1074/jbc.M910179199 |
| [88] | LIN J, CAO C W, TAO C, et al. Cold adaptation in pigs depends on UCP3 in beige adipocytes[J]. J Mol Cell Biol, 2017, 9(5): 364–375. DOI: 10.1093/jmcb/mjx018 |
| [89] | LEE K H, CHAI V Y, KANACHAMY S S, et al. Association of UCP1 -3826A/G and UCP3 -55C/T gene polymorphisms with obesity and its related traits among multi-ethnic Malaysians[J]. Ethn Dis, 2015, 25(1): 65–71. |
| [90] | AHIMA R S, FLIER J S. Leptin[J]. Annu Rev Physiol, 2000, 62: 413–437. DOI: 10.1146/annurev.physiol.62.1.413 |
| [91] | LI X S, WANG D H. Regulation of body weight and thermogenesis in seasonally acclimatized Brandt's voles (Microtus brandti)[J]. Horm Behav, 2005, 48(3): 321–328. DOI: 10.1016/j.yhbeh.2005.04.004 |
| [92] | VAN DIJK G. The role of leptin in the regulation of energy balance and adiposity[J]. J Neuroendocrinol, 2010, 13(10): 913–921. |
| [93] | NIEMINEN P, HYVARINEN H. Seasonality of leptin levels in the BAT of the common shrew (Sorex araneus)[J]. Z Naturforsch C, 2000, 55(5-6): 455–460. DOI: 10.1515/znc-2000-5-623 |
| [94] | KLINGENSPOR M, NIGGEMANN H, HELDMAIER G. Modulation of leptin sensitivity by short photoperiod acclimation in the Djungarian hamster, Phodopus sungorus[J]. J Comp Physiol B, 2000, 170(1): 37–43. |
| [95] | DE GRUIJL F R, VAN DER LEUN J C. Environment and health:3.Ozone depletion and ultraviolet radiation[J]. CMAJ, 2000, 163(7): 851–855. |
| [96] | SOLOMON K R. Effects of ozone depletion and UV-B radiation on humans and the environment[J]. Atmos Ocean, 2008, 46(1): 185–202. DOI: 10.3137/ao.460109 |
| [97] |
吴健, 戴桂馥, 张凤秋, 等. UV辐射对金鱼受精卵和幼胚发育的影响[J]. 辐射研究与辐射工艺学报, 2005, 23(4): 229–232.
WU J, DAI G F, ZHANG F Q, et al. Effects of UV-C irradiation on development of goldfish embryos[J]. Journal of Radiation Research and Radiation Processing, 2005, 23(4): 229–232. DOI: 10.3969/j.issn.1000-3436.2005.04.009 (in Chinese) |
| [98] | SETLOW R B, SWENSON P A, CARRIER W L. Thymine dimers and inhibition of DNA synthesis by ultraviolet irradiation of cells[J]. Science, 1963, 142(3598): 1464–1466. DOI: 10.1126/science.142.3598.1464 |
| [99] | CALDWELL M M. Plant life and ultraviolet radiation:some perspective in the history of the earth's UV climate[J]. Bioscience, 1979, 29(9): 520–525. DOI: 10.2307/1307719 |
| [100] | CÉSARINI J P.The sun and ultraviolet radiation[M]//PERRIN A, SOUQUES M.Electromagnetic Fields, Environment and Health.Paris: Springer, 2012: 109-125. |
| [101] | KOUWENBERG J H M, BROWMAN H I, CULLEN J J, et al. Biological weighting of ultraviolet (280-400 nm) induced mortality in marine zooplankton and fish.I.Atlantic cod (Gadus morhua) eggs[J]. Mar Biol, 1999, 134(2): 269–284. DOI: 10.1007/s002270050545 |
| [102] | BLAUSTEIN A R, ROMANSIC J M, KIESECKER J M, et al. Ultraviolet radiation, toxic chemicals and amphibian population declines[J]. Divers Distrib, 2003, 9(2): 123–140. DOI: 10.1046/j.1472-4642.2003.00015.x |
| [103] | KIESECKER J M, BLAUSTEIN A R, BELDEN L K. Complex causes of amphibian population declines[J]. Nature, 2001, 410(6829): 681–684. DOI: 10.1038/35070552 |
| [104] |
陈智强, 俞耀飞, 李雪, 等. 中波紫外辐射对两栖动物的生物学效应研究进展[J]. 生物学教学, 2017, 42(3): 2–3.
CHEN Z Q, YU Y F, LI X, et al. Advances in research on biological effects of medium-wave ultraviolet radiation on amphibians[J]. Biology Teaching, 2017, 42(3): 2–3. (in Chinese) |
| [105] | ZHANG Z G, XU D M, WANG L, et al. Convergent evolution of rumen microbiomes in high-altitude mammals[J]. Curr Biol, 2016, 26(14): 1873–1879. DOI: 10.1016/j.cub.2016.05.012 |
| [106] |
许东明.青藏高原哺乳动物高原适应的遗传机制[D].北京: 中国科学院大学, 2016.
XU D M.Genetic mechanism of mammalian plateau adaptation in the Qinghai-Tibet Plateau[D].Beijing: University of Chinese Academy of Sciences, 2016. (in Chinese) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y3189864 |
| [107] |
叶晶鑫, 杨胜平, 程颖, 等. 食品中低温微生物的适冷机制研究进展[J]. 微生物学杂志, 2018, 38(4): 114–119.
YE J X, YANG S P, CHENG Y, et al. Advances in cryo-adaptation mechanism of food-related microorganisms[J]. Journal of Microbiology, 2018, 38(4): 114–119. DOI: 10.3969/j.issn.1005-7021.2018.04.016 (in Chinese) |
| [108] | LIU H P, LIU Q Y, CHEN Z P, et al. Draft genome of Glyptosternon maculatum, an endemic fish from Tibet plateau[J]. GigaScience, 2018, 7(9): giy104. |
| [109] | SUN Y B, FU T T, JIN J Q, et al. Species groups distributed across elevational gradients reveal convergent and continuous genetic adaptation to high elevations[J]. Proc Natl Acad Sci U S A, 2018, 115(45): E10634–E10641. DOI: 10.1073/pnas.1813593115 |
| [110] | GAO W, SUN Y B, ZHOU W W, et al. Genomic and transcriptomic investigations of the evolutionary transition from oviparity to viviparity[J]. Proc Natl Acad Sci U S A, 2019, 116(9): 3646–3655. DOI: 10.1073/pnas.1816086116 |



