硫辛酸(lipoic acid, LA)又称α-硫辛酸,是高效抗氧化剂,广泛存在于动物、植物及微生物中,在有机体的能量代谢过程中起着重要作用。LA是生物体能量代谢过程中多种多酶复合物的辅酶,包括α-酮戊二酸脱氢酶(ketoglutaric dehydrogenase complex, KGDHC)、丙酮酸脱氢酶(pyruvate dehydrogenase complex, PDHC)以及甘氨酸裂解系统(glycine cleavage system, GCV),这些多酶复合物在生物体能量代谢、脂肪酸合成以及氨基酸降解中发挥重要作用[1]。对病原微生物的研究表明,了解硫辛酸代谢途径特点对于了解病原微生物的生长特性以及致病机制均具有重要意义。通过缺失病原微生物的硫辛酸代谢酶可以使其致病性减弱[2]。这些研究对于开发烈性病原微生物的弱毒疫苗以及药物靶标的筛选具有重要的指导意义。
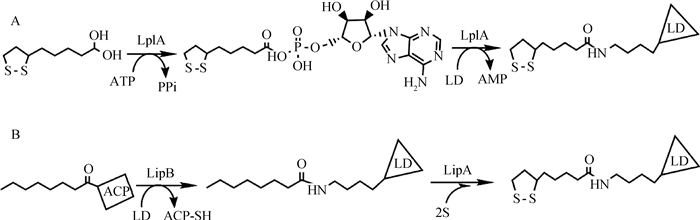
生物体中LA的代谢主要是在相关酶的催化作用下,以共价键方式连接到一类含有硫辛酸结合结构域(lipoyl domain, LD)的蛋白分子上,该过程又称为硫辛酸修饰[3]。大肠杆菌中,LA的代谢研究得比较透彻,它可以分为外源性LA和内源性LA两种代谢途径(图 1)。外源性LA代谢分两步进行,首先,硫辛酸分子在LplA的作用下发生活化生成lipoyl-AMP,然后再在LplA的作用下将lipoyl-AMP的硫辛酰基部分转移到PDHC、KGDHC的E2亚基或甘氨酸裂解系统H蛋白(GcvH)的LD上[4]。内源性的LA代谢也分两步进行,第一步是在N-辛酰转移酶(octyl transferase, LipB)的作用下将辛酰载体蛋白(Oct-ACP)的辛酰部分转移到E2的LD上,然后再在硫辛酰基合成酶(lipoyl synthase, LipA)的作用下,在C6和C8上插入两个硫原子以形成硫辛酰基,以此完成硫辛酸内源性代谢,得到硫辛酰化的LD[5]。
|
A.外源性LA代谢:硫辛酸在LplA的作用下活化成lipoyl-AMP,活化后的lipoyl-AMP在LplA的作用下将硫辛酰部分转移到LD上;B.内源性LA代谢:LipB将Oct-ACP的辛酰部分直接转移到E2的LD上,然后在LipA的作用下迅速插入两个硫原子完成硫辛酸内源性代谢反应生成硫辛酰基 A.Metabolism of LA provided in the environment:LA is activated to lipoyl-AMP by LplA, LplA then transfers the lipoyl moiety on the activated lipoyl-AMP to the LD; B. Metabolism of endogenous LA:LipB transfers the octanoyl moiety on the octanoyl-ACP to the LD, then LipA catalyzes two sulfur atoms to insert into the octanoylated domains 图 1 大肠杆菌硫辛酸代谢途径 Fig. 1 Lipoic acid metabolism pathways in E.coli |
尽管硫辛酸代谢对了解病原微生物的生长特性及致病特点至关重要,但仅少数病原微生物的硫辛酸代谢途径研究得较为透彻。由于基因操作系统的限制,一些病原微生物参与硫辛酸代谢的酶的研究无法在自身体内进行,而且不同病原微生物的LA代谢相关酶和底物的功能具有高度保守性[6],因此不同物种的硫辛酸蛋白连接酶可以将大肠杆菌的底物LD进行硫辛酰化。利用Red同源重组的方法[7-8],作者成功构建大肠杆菌lplA及lipB双基因缺失的DH5α突变株,其LA外源性及内源性代谢途径均被阻断,在M9基础培养基中无法正常生长。但是当突变株回补lplA或lipB后,相应的代谢通路均可以恢复。该突变株的成功构建为探究其他病原微生物硫辛酸修饰过程奠定了基础。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒试验中所用到的菌株大肠杆菌DH5α,购自南京诺唯赞生物科技有限公司;pKD46、pKD3、pCP20质粒均由本实验室保存;pBAD322G质粒由伊利诺伊大学教授John E. Cronan惠赠;pMD18-T载体购自大连宝生物工程有限公司。其中pKD46为温敏质粒,在阿拉伯糖诱导下能够表达三个重组酶Gam、Bet、Exo,重组酶促进同源重组的发生。pKD3质粒为同源重组片段的扩增提供氯霉素抗性基因,在氯霉素抗性基因的两侧存在FRT位点。pCP20质粒用于抗性基因的去除,由于pCP20能够表达重组酶FLP,FLP能够作用于FRT位点,从而将氯霉素抗性基因去除。pBAD322G质粒用于回补质粒的构建,由于pBAD322G含有大肠杆菌编码AraC调控的基因蛋白,当有阿拉伯糖诱导时AraC为转录激活剂,同时pBAD322G中的ROP基因对拷贝数进行负调控,可以有效避免由于基因大量表达对细胞产生毒性[9]。
1.1.2 试剂和仪器氨苄青霉素、氯霉素购自北京索莱宝科技有限公司;L-阿拉伯糖购自Sigma公司;DL2000 Marker、聚合酶及限制性内切酶均购自大连宝生物工程有限公司;核酸胶电泳回收试剂盒及质粒提取试剂盒购自OMEGA生物科技有限公司;兔源anti-lipoic acid抗体购自Abcam生物公司,IRDye 680RD山羊抗兔IgG抗体购自LI-COR Bioscinece公司;电转化仪购自Bio-Rad公司。
1.1.3 引物设计与合成根据GenBank中收录的大肠杆菌MG1655菌株lplA和lipB基因序列设计基因重组引物及鉴定引物(表 1)。所有引物均由哈尔滨博士生物科技有限公司合成。其中P1/P2用于扩增含lplA基因同源臂,中间含有FRT和氯霉素抗性的基因片段,P7/P8分别用于扩增两端为lipB基因同源臂,中间为FRT和氯霉素抗性的基因片段,P3/P4用于lplA基因第一次同源重组的检测,P5/P6用于lplA基因第二次同源重组后无抗突变的检测,P9/P10用于lipB基因突变菌株的检测,P11/P12用于构建回补质粒pBAD322G-lplA时扩增lplA,P13/P14用于构建回补质粒pBAD322G-lipB时扩增lipB,P15/P16为pBAD322G通用引物,用于回补株鉴定。
|
|
表 1 引物和序列 Table 1 Primer sequences |
以P1/P2、P7/P8为引物,pKD3质粒为模板,分别扩增出两端为lplA、lipB基因上下游同源序列、中间为含有FRT和氯霉素抗性的DNA片段,分别命名为lplA-UP-cat-Down和lipB-UP-cat-Down。PCR产物经琼脂糖凝胶电泳送测序。
1.2.2 电击感受态细胞的制备将pKD46质粒化转至DH5α感受态细胞中,得到pKD46~DH5α,挑取单菌落在30 ℃ 200 r·min-1振荡培养12 h,然后1:100转接到30 mL含氨苄青霉素的LB培养基中,在30 ℃继续振荡培养至OD600nm≈0.2时,加入终浓度为30 mmol·L-1的L-阿拉伯糖继续培养2 h,使pKD46中的重组酶Exo、Bet、Gam能够充分表达,离心收集菌体,按照常规方法[10]制备电转感受态细胞,并将其放置在-80 ℃保存备用。
1.2.3 △lplA△lipB~DH5α双基因缺失株的构建及鉴定将150 ng纯化的lplA-UP-cat-Down电转入pKD46~DH5α感受态细胞中,将转化产物涂布于氯霉素抗性的LB平板上,在30 ℃培养24 h后,通过PCR方法鉴定氯霉素阳性重组菌,然后将鉴定正确的重组菌株在氯霉素抗性的LB平板上连续划线传代,37 ℃培养数代以丢失温敏质粒pKD46,得到lplA基因缺失突变株记为△lplA::Cat~DH5α。将△lplA::Cat~DH5α制备电击感受态细胞,电转入pCP20质粒以去除突变株△lplA::Cat~DH5α中的氯霉素抗性基因,将转化产物涂布于氨苄青霉素抗性的LB平板上,在37 ℃培养12 h挑取单克隆菌落,通过PCR扩增和序列分析的方法筛选成功去除氯霉素抗性的△lplA~DH5α基因缺失株,多次传代培养以去除pCP20质粒。在突变株△lplA~DH5α制备电转感受态细胞后,将同源重组片段lipB-UP-cat-Down电转入△lplA~DH5α感受态细胞,参照上述操作继续缺失lipB基因,最终获得无抗性基因的双基因缺失株△lplA△lipB~DH5α。
1.2.4 △lplA△lipB~DH5α双基因缺失株的稳定性试验将构建的缺失株△lplA△lipB~DH5α在LB无抗性平板上连续划线传20代,分别于第1、5、10、15和20代时,挑取单菌落用鉴定引物(P5/P6,P9/P10)进行PCR鉴定,检测其遗传稳定性[11]。
1.2.5 lplA、lipB基因回补株的构建及鉴定使用CaCl2法制备缺失株△lplA△lipB~DH5α的化学转化感受态细胞[12]。以DH5α基因组为模板,分别以P11/P12和P13/P14为引物扩增lplA、lipB基因,将它们克隆至表达载体pBAD322G中,得到重组质粒pBAD322G-lplA、pBAD322G-lipB。经序列测定确定正确后,将重组质粒和pBAD322G分别转化到△lplA△lipB~DH5α中,得到回补株ClplA-△lplA△lipB~DH5α、ClipB-△lplA△lipB~DH5α以及pBAD322G-△lplA△lipB~DH5α,用引物P15/P16进行PCR鉴定,检测重组质粒是否导入缺失株。
1.2.6 △lplA△lipB~DH5α补偿试验将构建的回补株ClplA-△lplA△lipB~DH5α、ClipB-△lplA△lipB~DH5α和pBAD322G-△lplA△lipB~DH5α的单菌落接种于添加有5 mmol·L-1醋酸盐、琥珀酸的M9基础培养基中,在37 ℃培养24 h后,将菌液在含有0.2% L-阿拉伯糖的M9平板上进行划线,补加LA的L-阿拉伯糖M9培养平板作为对照,在37 ℃培养48 h后观察菌落生长情况[13-14]。
1.2.7 缺失株及回补株底物硫辛酰化的分析为分析缺失株及回补株底物硫辛酰化情况,将野生株DH5α、突变株△lplA△lipB~DH5α、回补株(ClplA-△lplA△lipB~DH5α、ClipB-△lplA△lipB~DH5α和pBAD322G-△lplA△lipB~DH5α)的单菌落接种于LB中,37 ℃培养12 h,同时在补加LA的培养基中进行培养作为对照[15]。将培养的菌株进行SDS-PAGE电泳(两个重复,一块胶用于Western blot检测,一块胶进行考马斯亮蓝染色),将蛋白转印至NC膜上,用5%脱脂乳封闭NC膜,然后分别以兔源anti-lipoic acid为一抗(1:8 000),IRDye 680RD山羊抗兔IgG抗体为二抗(1:8 000)进行孵育,利用近红外荧光扫描成像系统对菌株硫辛酰化蛋白进行Western blot分析鉴定。Western blot阳性条带即为菌株中硫辛酰化的底物,将考马斯亮蓝染色PAGE胶上对应的位置切下送北京华大蛋白质研究中心进行质谱分析。
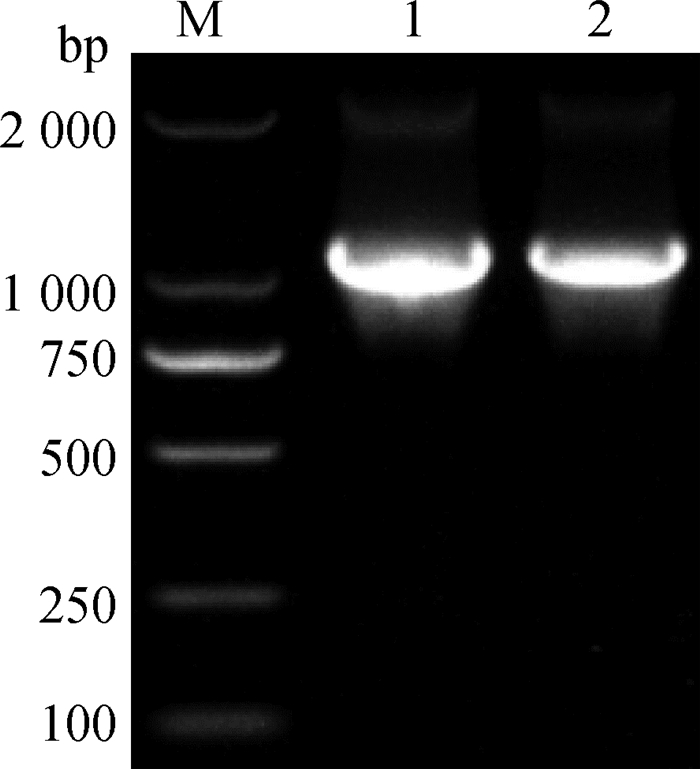
2 结果 2.1 lplA和lipB同源重组片段的扩增以P1/P2、P7/P8为引物,通过PCR的方法从质粒pKD3中扩增氯霉素基因,扩增的产物lplA-UP-cat-Down和lipB-UP-cat-Down两端分别携带50 bp的lplA和lipB基因上下游同源序列,经琼脂糖凝胶电泳检测,片段大小均为1 152 bp,同预期结果一致(图 2),通过测序显示获得的PCR产物中的氯霉素抗性基因序列完全正确,其两端分别包含了lplA和lipB两端的同源臂序列。
|
M. DL2000 DNA相对分子质量标准;1. PCR产物lplA-UP-cat-Down;2. PCR产物lipB-UP-cat-Down M. DL2000 DNA marker; 1. lplA-UP-cat-Down fragment; 2. lipB-UP-cat-Down fragment 图 2 lplA和lipB同源重组片段的鉴定 Fig. 2 Amplification of homologous recombinant fragment of lplA and lipB |
将同源重组片段lplA-UP-cat-Down电转化至pKD46~DH5α中,经氯霉素抗性筛选出阳性克隆,以P3/P4为引物进行PCR鉴定,结果显示,从△lplA::Cat~DH5α重组菌扩增的片段大小约为1 060 bp,而从DH5α扩增反应为阴性(图 3A),表明融合PCR产物成功替换了DH5α基因组中的lplA基因。为了消除缺失株中的氯霉素基因,将筛选出的重组菌株制备电转感受态细胞,然后将pCP20质粒电转入△lplA::Cat~DH5α感受态细胞中,利用pCP20表达的重组酶去除氯霉素抗性基因。从培养板中挑取单克隆用引物P5/P6进行PCR鉴定,结果显示,从突变株扩增的片段约610 bp,DH5α扩增片段约为1 627 bp(图 3B),将从突变株扩增出的片段进行测序,结果表明DH5α菌株中的lplA基因已缺失成功,并且不含氯霉素基因,因此,成功得到不含抗性基因的lplA基因突变株△lplA~DH5α。

|
A. △lplA::Cat~DH5α的PCR鉴定;B. △lplA~DH5α的PCR鉴定;C. △lplA△lipB::Cat~DH5α的PCR鉴定;D.△lplA△lipB~DH5α的PCR鉴定;M. DL2000 DNA相对分子质量标准;1.相应缺失株的PCR结果;2. DH5α菌株的PCR结果 A. Identification of △lplA::Cat~DH5α by PCR; B. Identification of △lplA~DH5α by PCR; C. Identification of △lplA△lipB::Cat~DH5α by PCR; D. Identification of △lplA△lipB~DH5α by PCR; M. DL2000 DNA marker; 1. PCR products amplified from corresponding gene-deleted strain; 2. PCR amplification of DH5α 图 3 △lplA△lipB~DH5α的鉴定 Fig. 3 Identification of △lplA△lipB~DH5α |
为了进一步缺失lipB基因,将同源重组片段lipB-UP-cat-Down转入△lplA~DH5α感受态细胞,通过氯霉素抗性筛选出lipB缺失的克隆△lplA△lipB::Cat~DH5α。用P9/P10引物进行PCR鉴定显示,从△lplA△lipB::Cat~DH5α重组菌扩增的片段大小约为1 445 bp,而从DH5α扩增的片段大小约为1 035 bp(图 3C),因此△lplA~DH5α缺失株基因组中的lipB基因替换成功。为了去除缺失株中的氯霉素,将pCP20质粒电转入△lplA△lipB::Cat~DH5α感受态细胞中,PCR鉴定结果显示,突变株和野生株的PCR产物大小分别为393和1 035 bp(图 3D),测序结果显示突变株氯霉素抗性基因完全消除。这些结果表明lplA及lipB双基因缺失的无抗缺失株△lplA△lipB~DH5α构建成功。
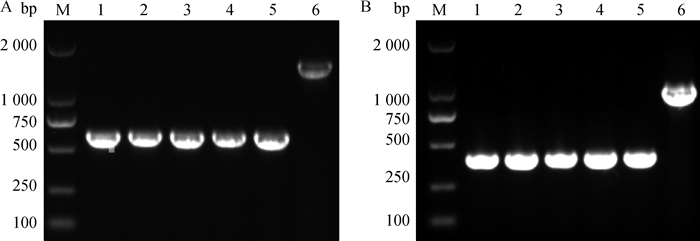
2.3 △lplA△lipB~DH5α双基因缺失株的稳定性试验为了检测lplA和lipB双基因缺失株的稳定性,将△lplA△lipB~DH5α菌株在LB无抗性平板上连续划线培养20代,分别于第1、5、10、15和20代时,挑取单菌落分别对缺失的lplA和lipB进行PCR鉴定,结果显示两基因均未发生回复突变(图 4),表明缺失株△lplA△lipB~DH5α遗传性状非常稳定。
|
A.缺失lplA遗传稳定的PCR鉴定结果(引物P5/P6);B.缺失lipB遗传稳定的PCR鉴定结果(引物P9/P10);M. DL2000 DNA相对分子质量标准;1~5.第1、5、10、15和20代△lplA△lipB~DH5α的PCR鉴定结果;6. DH5α菌株的PCR结果 A. Identification of genetic stability for the deleted lplA by PCR assay (using primers P5/P6); B. Identification of genetic stability for the deleted lipB by PCR assay(using primers P9/P10); M. DL2000 DNA marker; 1-5. PCR products from 1, 5, 10, 15 and 20 generation of △lplA△lipB~DH5α; 6. PCR amplification of DH5α 图 4 缺失株遗传稳定性试验的PCR鉴定 Fig. 4 Identification of genetic stability of deleted genes using PCR assay |
将重组质粒pBAD322G-lplA、pBAD322G-lipB和空质粒pBAD322G分别转化到△lplA△lipB~DH5α中,得到回补株ClplA-△lplA△lipB~DH5α、ClipB-△lplA△lipB~DH5α和pBAD322G-△lplA△lipB~DH5α,通过PCR方法进行鉴定,结果显示,以pBAD322G-△lplA△lipB~DH5α、ClplA-△lplA△lipB~DH5α和ClipB-△lplA△lipB~DH5α为模板,扩增的片段大小分别为350、1 458和992 bp,而对照菌△lplA△lipB~DH5α和DH5α的PCR为阴性(图 5), 测序结果显示目的质粒成功导入缺失株。

|
M. DL2000 DNA相对分子质量标准;1. pBAD322G;2. ClplA-△lplA△lipB~DH5α;3. ClipB-△lplA△lipB~DH5α;4. △lplA△lipB~DH5α;5. DH5α M. DL2000 DNA marker; 1. pBAD322G; 2. ClplA-△lplA△lipB~DH5α; 3. ClipB-△lplA△lipB~DH5α; 4. △lplA△lipB~DH5α; 5. DH5α 图 5 lplA、lipB基因回补株的鉴定 Fig. 5 Identification of lplA and lipB in complemented strains |
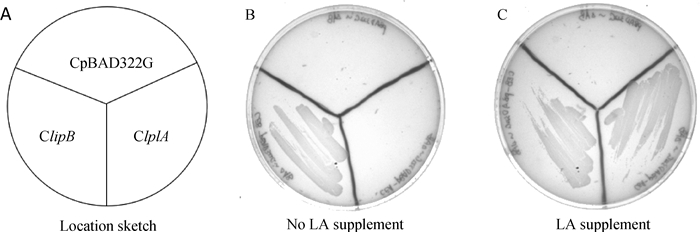
在添加L-阿拉伯糖的M9琼脂平板上,回补株pBAD322G-△lplA△lipB~DH5α和ClplA-△lplA△lipB~DH5α不生长,而回补株ClipB-△lplA△lipB~DH5α能够正常生长;在添加L-阿拉伯糖和LA的M9琼脂平板上,pBAD322G-△lplA△lipB~DH5α不生长,回补株ClplA-△lplA△lipB~DH5α及ClipB-△lplA△lipB~DH5α均能生长(图 6)。这些结果表明,缺失株△lplA△lipB~DH5α同时表现出了缺失lplA或lipB基因的相应表型,且回补质粒导入的情况下,能够恢复对应的生物学功能。
|
A.位置示意;B.回补株在未添加LA平板上的生长特性(回补株pBAD322G-△lplA△lipB~DH5α和ClplA-△lplA△lipB~DH5α不能生长,回补株ClipB-△lplA△lipB~DH5α能够生长);C.回补株在添加LA平板上的生长特性(回补株pBAD322G-△lplA△lipB~DH5α不能生长,ClplA-△lplA△lipB~DH5α和ClipB-△lplA△lipB~DH5α能够生长) A. Location sketch; B. The growth of complemented strains on the plate without LA (The complemented strains of pBAD322G-△lplA△lipB~DH5α and ClplA-△lplA△lipB~DH5α cannot grow, but ClipB-△lplA△lipB~DH5α can grow); C. The growth of complemented strains on the plate with LA (The complemented strains of pBAD322G-△lplA△lipB~DH5α cannot grow, ClplA-△lplA△lipB~DH5α and ClipB-△lplA△lipB~DH5α can grow) 图 6 缺失株补偿试验 Fig. 6 Complementation experiment of deleted strains |
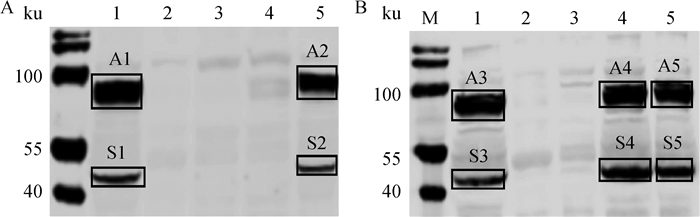
将野生株DH5α、突变株△lplA△lipB~DH5α和回补株ClplA-△lplA△lipB~DH5α、ClipB-△lplA△lipB~DH5α、pBAD322G-△lplA△lipB~DH5α的单菌落接种于LB中,将培养的菌株进行Western blot分析,试验结果显示,突变株△lplA△lipB~DH5α以及回补株ClplA-△lplA△lipB~DH5α中均无硫辛酰化底物,回补株ClipB-△lplA△lipB~DH5α中检测到与亲本株一致的硫辛酰化底物(图 7A);当培养基中补加LA后突变株未检测到硫辛酰化底物,回补株ClplA-△lplA△lipB~DH5α和ClipB-△lplA△lipB~DH5α硫辛酰化底物与亲本株保持一致(图 7B)。表明突变株△lplA△lipB~DH5α回补lplA及lipB后,分别恢复LplA和LipB相应生物学功能。将野生株和回补株硫辛酰化的底物(图 7A、7B中A1~A5、S1~S5)切下进行质谱分析,结果显示A1~A5蛋白为AceF(UniProtKB/Swiss-Prot:P06959,大肠杆菌PDHC E2亚基),S1~S5蛋白为SucB(UniProtKB/Swiss-Prot:P0AFG6,大肠杆菌KGDHC E2亚基)。AceF、SucB分别携带了PDHC、KGDHC的LD,质谱结果也进一步说明了缺失株和回补株构建成功。这些结果表明ClplA-△lplA△lipB~DH5α和ClipB-△lplA△lipB~DH5α构建成功,而且缺失株完全失去了对E2亚基进行硫辛酸化修饰的能力,但是当回补了lplA或lipB后,可恢复相应的硫辛酸修饰功能。
|
A.菌株底物硫辛酰化分析;B.菌株添加LA后底物硫辛酰化分析;M.预染蛋白质相对分子质量标准;1. DH5α;2.缺失株△lplA△lipB~DH5α;3.回补株pBAD322G-△lplA△lipB~DH5α;4.回补株ClplA-△lplA△lipB~DH5α;5.回补株ClipB-△lplA△lipB~DH5α A. Analysis of sulfur octyl acylation of strain substrates; B. Analysis of sulfur octyl acylation of strain substrates when LA was added; M. Prestained protein ladder; 1. DH5α; 2. △lplA△lipB~DH5α; 3. pBAD322G-△lplA△lipB~DH5α; 4. ClplA-△lplA△lipB~DH5α; 5. ClipB-△lplA△lipB~DH5α 图 7 缺失株及回补株底物硫辛酰化的分析 Fig. 7 Analysis of substrate octyl acylation in mutant and supplemented strains |
LA是PDHC、KGDHC等多种多酶复合物的辅酶,与多酶复合物酶的活性关系密切[16-17],是许多生命体的生长限制因子。研究表明生物体LA都是同重要蛋白质共价结合。相关研究已经证实,肺炎链球菌中硫辛酸蛋白连接酶(lipoate protein ligase, LPL)的同系物对于菌株黏附宿主细胞有抑制作用[18]。李斯特杆菌中LPL的同系物为其毒力因子,它的缺失使菌株不能从宿主摄取LA,继而影响细菌的生长和毒力[2]。这些研究结果对于致弱病原微生物具有重要的指导意义。LPL参与的硫辛酸修饰显著提高了猪胰岛素同受体的结合能力[19]。结核分枝杆菌中,硫辛酸修饰的蛋白作为还原剂能够还原硫氧还原蛋白,达到维持病原体稳态的目的[20]。因此,与LA代谢相关的LPL同系物对于LA在生物体中的存在形式及其功能可能发挥重要作用,因此展开LPL相关蛋白的研究有助于理解一些病原的致病分子机制及其生长特性。
大肠杆菌中硫辛酸的代谢通过两个途径完成,并且受LplA、LipB和LipA三种酶调控。在外源性LA存在的情况下,只需要LplA单独来完成LA的代谢;如果外部环境不存在LA时,大肠杆菌就利用LipB和LipA启动体内合成途径来完成LA的代谢。无论是外源性还是内源性代谢途径,均是将LA转移到KGDHC、PDHC的E2亚基或者GcvH的LD上[21]。如果将lplA和lipB进行缺失,则LA的两种代谢途径均被阻断,大肠杆菌的生长将会受到严重的影响。本研究通过Red同源重组技术在DH5α菌株中成功将lplA和lipB进行了缺失,该缺失株在营养缺陷型M9基础培养基中失去了生长能力,通过质谱分析进一步证明了突变株△lplA△lipB~DH5α失去了对E2亚基进行硫辛酰化的能力。但是当缺失株导入lplA或lipB基因时,缺失株生长和对底物的硫辛酰修饰功能得到了恢复。因此△lplA△lipB~DH5α可以视为硫辛酸代谢缺陷型的生物体,保留了表达硫辛酰化底物的能力,但自身不能对底物进行硫辛酸修饰,这些底物以脱辅基的形式存在,不能发挥相应的生物学功能,又由于生物体硫辛酸代谢中的各种酶和底物高度保守,可以将待研究物种的硫辛酸代谢相关酶以回补质粒为载体,转入△lplA△lipB~DH5α菌株中,通过生长特性及硫辛酰化底物分析,对回补酶的功能有初步的了解。因此,△lplA△lipB~DH5α可以为其他物种的硫辛酸代谢酶提供反应环境,用于一些病原微生物硫辛酸代谢的体内研究。例如,由于猪肺炎支原体目前缺乏质粒操作系统,研究它的LA代谢酶无法在其体内进行缺失研究,因此可以利用笔者建立的△lplA△lipB~DH5α展开体内功能研究。本体系的建立为研究动物重要病原微生物的硫辛酸代谢酶的功能提供了重要的平台。
4 结论成功在DH5α菌株中对lplA和lipB进行了双基因缺失,获得的双基因缺失株△lplA△lipB~DH5α失去了对E2亚基进行硫辛酸修饰的功能,回补株能够恢复对E2亚基的硫辛酸修饰功能。由于不同微生物硫辛酸修饰相关酶的功能具有高度保守性,因此△lplA△lipB~DH5α缺失菌株可以用于其他微生物硫辛酸代谢中各种酶功能的研究。
| [1] | REED L J. A trail of research from lipoic acid to α-keto acid dehydrogenase complexes[J]. J Biol Chem, 2001, 276(42): 38329–38336. DOI: 10.1074/jbc.R100026200 |
| [2] | O′RIORDAN M, MOORS M A, PORTNOY D A. Listeria intracellular growth and virulence require host-derived lipoic acid[J]. Science, 2003, 302(5644): 462–464. DOI: 10.1126/science.1088170 |
| [3] |
贺华良, 宾淑英, 廖泓之, 等. 黄曲条跳甲线粒体源硫辛酸蛋白连接酶的序列分析及表达[J]. 华南农业大学学报, 2012, 33(3): 351–355.
HE H L, BIN S Y, LIAO H Z, et al. Sequencing and expression profile of the gene encodinglipoate protein ligase in Phyllotreta striolata[J]. Journal of South China Agricultural University, 2012, 33(3): 351–355. DOI: 10.3969/j.issn.1001-411X.2012.03.016 (in Chinese) |
| [4] | MORRIS T W, REED K E, CRONAN J E Jr. Identification of the gene encoding lipoate-protein ligase A of Escherichia coli. Molecular cloning and characterization of the lplA gene and gene product[J]. J Biol Chem, 1994, 269(23): 16091–16100. |
| [5] | MORRIS T W, REED K E, CRONAN J E Jr. Lipoic acid metabolism in Escherichia coli: the lplA and lipB genes define redundant pathways for ligation of lipoyl groups to apoprotein[J]. J Bacteriol, 1995, 177(1): 1–10. DOI: 10.1128/jb.177.1.1-10.1995 |
| [6] | CHRISTENSEN Q H, CRONAN J E. Lipoic acid synthesis:a new family of octanoyltransferases generally annotated as lipoate protein ligases[J]. Biochemistry, 2010, 49(46): 10024–10036. DOI: 10.1021/bi101215f |
| [7] |
于艳超, 王莉莉, 潘巧, 等. 肠炎沙门菌luxS缺失株的构建及其生物学特性研究[J]. 畜牧兽医学报, 2018, 49(6): 1291–1298.
YU Y C, WANG L L, PAN Q, et al. Construction and biological characteristics of Salmonella enterica serovar enteritidis luxS mutant[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(6): 1291–1298. (in Chinese) |
| [8] |
苏洋洋, 黄骏, 吴白, 等. 鼠伤寒沙门菌rpoN基因缺失株构建及生物学特性研究[J]. 畜牧兽医学报, 2016, 47(4): 771–778.
SU Y Y, HUANG J, WU B, et al. Construction and biological characterization of rpoN gene deletion mutants of Salmonella typhimurium[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(4): 771–778. (in Chinese) |
| [9] | CHAKRAVARTTY V, CRONAN J E. A series of medium and high copy number arabinose-inducible Escherichia coli expression vectors compatible with pBR322 and pACYC184[J]. Plasmid, 2015, 81: 21–26. DOI: 10.1016/j.plasmid.2015.03.001 |
| [10] | BERG M, SEEMÜLLER E. Chromosomal organization and nucleotide sequence of the genes coding for the elongation factors G and Tu of the apple proliferation phytoplasma[J]. Gene, 1999, 226(1): 103–109. DOI: 10.1016/S0378-1119(98)00552-6 |
| [11] |
司微, 于申业, 陈利苹, 等. 肠炎沙门氏菌tu-fA基因缺失株的构建及其生物学特性研究[J]. 中国预防兽医学报, 2015, 37(5): 361–365.
SI W, YU S Y, CHEN L P, et al. Construction and biological identification of a tu-fA gene deletion mutant from Salmonella enterica[J]. Chinese Journal of Preventive Veterinary Medicine, 2015, 37(5): 361–365. DOI: 10.3969/j.issn.1008-0589.2015.05.09 (in Chinese) |
| [12] | CASTUMA C E, HUANG R P, KORNBERG A, et al. Inorganic polyphosphates in the acquisition of competence in Escherichia coli[J]. J Biol Chem, 1995, 270(22): 12980–12983. DOI: 10.1074/jbc.270.22.12980 |
| [13] | FUJIWARA K, MAITA N, HOSAKA H, et al. Global Conformational change associated with the two-step reaction catalyzed by Escherichia coli lipoate-protein ligase A[J]. J Biol Chem, 2010, 285(13): 9971–9980. DOI: 10.1074/jbc.M109.078717 |
| [14] | CAO X Y, CRONAN J E. The Streptomyces coelicolor lipoate-protein ligase is a circularly permuted version of the Escherichia coli enzyme composed of discrete interacting domains[J]. J Biol Chem, 2015, 290(11): 7280–7290. DOI: 10.1074/jbc.M114.626879 |
| [15] | CRONAN J E. A family of arabinose-inducible Escherichia coli expression vectors having pBR322 copy control[J]. Plasmid, 2006, 55(2): 152–157. DOI: 10.1016/j.plasmid.2005.07.001 |
| [16] | CRONAN J E, ZHAO X, JIANG Y F. Function, attachment and synthesis of lipoic acid in Escherichia coli[J]. Adv Micro Physiol, 2005, 50: 103–146. DOI: 10.1016/S0065-2911(05)50003-1 |
| [17] | PERHAM R N. Swinging arms and swinging domains in multifunctional enzymes: catalytic machines for multistep reactions[J]. Ann Rev Biochem, 2000, 69(69): 961–1004. |
| [18] |
杨晓亮, 尹楠林, 庞丹, 等. 假想的脂蛋白连接酶对肺炎链球菌毒力的影响[J]. 微生物学报, 2010, 50(6): 774–779.
YANG X L, YIN N L, PANG D, et al. Contributions of putative lipoate-protein ligase to the virulence of Streptococcus pneumoniae[J]. Acta Microbiologica Sinica, 2010, 50(6): 774–779. (in Chinese) |
| [19] |
黄涛, 黄开勋. 硫辛酸修饰胰岛素与胰岛素受体的结合能力[J]. 中国生化药物杂志, 2006, 27(2): 65–68.
HUANG T, HUANG K X. The binding potency oflipoic acid modified insulin with insulin receptor[J]. Chinese Journal of Biochemical Pharmaceutics, 2006, 27(2): 65–68. DOI: 10.3969/j.issn.1005-1678.2006.02.001 (in Chinese) |
| [20] | BRYK R, LIMA C D, ERDJUMENT-BROMAGE H, et al. Metabolic enzymes of mycobacteria linked to antioxidant defense by a thioredoxin-like protein[J]. Science, 2002, 295(5557): 1073–1077. DOI: 10.1126/science.1067798 |
| [21] | OKAMURA-IKEDA K, HOSAKA H, MAITA N, et al. Crystal structure of aminomethyltransferase in complex with dihydrolipoyl-H-protein of the glycine cleavage system: implications for recognition of lipoyl protein substrate, disease-related mutations, and reaction mechanism[J]. J Biol Chem, 2010, 285(24): 18684–18692. DOI: 10.1074/jbc.M110.110718 |



