2. 国家胶类中药工程技术研究中心, 东阿阿胶股份有限公司, 聊城 252201
2. National Engineering Research Center for Gelatin-based Traditional Chinese Medicine, Dong-E-E-Jiao Corporation Co. Ltd., Liaocheng 252201, China
NCAPG基因作为哺乳动物浓缩素I复合体的调节亚基,在细胞有丝分裂过程中起重要作用[1]。斑马鱼NCAPG基因突变会导致神经视网膜祖细胞的高度增殖[2]。DCAF16基因可作为CUL4-DDB1 E3泛素蛋白连接酶复合物的底物受体,参与促进蛋白质修饰和泛素化两条途径[3]。大量研究表明,NCAPG-DCAF16区域与生物体的生长发育相关。全基因组关联分析(GWAS)揭示了该区域与婴儿的初生重[4]、体长[5]及成年人的身高[6-9]显著相关。对家畜的研究表明,该区域与牛的初生重、断奶重、1岁重[10]、体尺[11]等显著相关,NCAPG I442M(rs109570900)位点的突变对牛出生前后的生长发育有一定影响[11-13],研究指出,该位点影响生长性状潜在的机制可能是由于血浆中精氨酸水平的升高[14],通路分析还找到了一些关键基因与该位点相互作用进而调控牛初情期的生长[15]。后期研究表明,NCAPG I442M位点对公牛在初情期开始时的饲料转化率和采食量有显著的影响[16]。在西门塔尔牛中,利用GWAS找到了与日增重极显著相关的两个位点,均定位在NCAPG-DCAF16区域,能够解释表型变异的4.01%[17]。在胎牛和成年牛背最长肌中,NCAPG基因的表达水平有显著差异,说明该基因可能参与肌肉发育[18]。此外,该区域还与绵羊[19-22]、兔[23]、马[24-26]的体重、体型等重要的生长发育性状相关[20]。在西班牙纯种马群体中验证了NCAPG-LCORL区域存在1个SNP位点,该位点CC基因型的个体要比TT基因型的体高和体长分别增加3.6%、4.5%[27]。
德州驴作为我国优良的大型驴种之一,具有体格高大、耐粗饲、抗病力强等特点,与其他品种相比具有较好的肉用性能[28]。该驴种现已列入《国家畜禽资源保护名录》。目前,我国驴饲养量逐年下降,驴规模化程度较低,种业基础非常薄弱,大部分驴场没有育种,少部分驴场也只是通过表型性状进行简单选种,造成驴的遗传进展非常缓慢。
本研究利用混池测序、质谱分型技术对德州驴群体中NCAPG-DCAF16基因区域的多态性SNPs位点进行检测,利用SPSS24.0一般线性模型(GLM)分析基因型及组合基因型与德州驴初生、3月龄、6月龄不同时期生长性状的关联性,以期找到与德州驴生长发育性状显著相关的SNP位点,进而为开展德州驴分子育种奠定理论基础。
1 材料与方法 1.1 试验动物和表型数据选取227头健康、统一舍饲的德州驴为试验动物,其中公驴122头,母驴105头,用5 mL EDTK一次性真空采血管进行颈静脉采血,轻微振荡后保存于-20 ℃冰箱,用于DNA的提取。试验样品来自山东东阿国家黑毛驴繁育中心。
对227头德州驴的初生、3月龄和6月龄生长性状进行测量,测量指标包括体重、体高、体长、胸宽、胸深、胸围、管围、尻高、尻长和尻宽,具体测量方法参照马体重和体尺的测定[29]。
1.2 德州驴基因组DNA提取及检测德州驴基因组DNA采用DNA提取试剂盒(Tiangen生物技术有限公司)提取。利用NANO DROP检测DNA样本的浓度,1.25%的琼脂糖凝胶电泳检测DNA质量。
1.3 DNA混池测序所用PCR扩增引物的设计根据德州驴的系谱记录,选择亲缘关系较远的30个DNA样本,均匀稀释为30 ng·μL-1,每个样本取7 μL,均匀混合,构建德州驴DNA混池。根据NCBI数据库的候选基因序列,以目的序列为模板,对候选区域的外显子和上下游调控区(400~800 bp)设计引物(Primer 5.0软件),并进行扩增,引物信息见表 1。混池DNA经过PCR扩增后进行测序。PCR总反应体系为25 μL:其中DNA模板2 μL,正反向引物各1 μL,12.5 μL 2×Taq PCR MasterMix,ddH2O 8.5 μL;PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,退火温度(表 1)退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min;4 ℃保存。采用基质辅助激光解吸电离飞行时间质谱技术(MALDI-TOF MS, Squenom MassARRAY,Bioyong Technologeies Inc. HK)对227头德州驴群体的基因型进行检测(表 1)。
|
|
表 1 NCAPG、DCAF16基因PCR扩增引物信息 Table 1 PCR primers used for amplifying the NCAPG and DCAF16 genes |
使用Excel 2007统计各个位点的等位基因频率、基因型频率、多态信息含量(PIC)、纯合度(Ho)、杂合度(He)和有效等位基因数(Ne),并进行Hardy-Weinberg平衡定律适合性检验。根据NCAPG-DCAF16基因区域SNP位点信息,使用Haploview4.2软件进行位点之间的连锁不平衡及单倍型分析。使用SPSS24.0软件对个体基因型与初生、3月龄和6月龄的表型数据进行关联分析,结果表示为“最小二乘均值±标准误”。
所采用的分析模型:y=μ+G+S+e
其中,y为表型测定值;μ为群体均值;G为基因型效应;S为性别效应;e为随机残差。
2 结果 2.1 德州驴血液基因组DNA提取经检测本研究所提取DNA浓度均超过20 ng·μL-1,OD值在1.7~1.9之间;1.25%琼脂糖凝胶电泳检测结果显示,DNA质检合格,可用于后续PCR扩增反应(图略)。
2.2 德州驴DNA混池测序通过DNA混池测序发现,NCAPG基因第9内含子上发生T→C突变(rs1),rs1图为反向测序的峰图与T→C互补,第11内含子发生了A→G突变(rs2),第19内含子上发现了1个C→G突变(rs4),第8内含子上存在C→G突变(rs5);DCAF16基因第2外显子存在A→G同义突变(rs008),基因结构图红色区域代表NCAPG基因的21个外显子,黄色区域代表DCAF16基因的2个外显子(图 1)。

|
图 1 NCAPG、DCAF16基因结构及混池测序峰图 Figure 1 NCAPG and DCAF16 gene structures and DNA pool sequencing |
质谱分型结果统计了各基因型频率和等位基因频率的大小。统计结果表明,rs1位点优势基因型为TT;rs2位点优势基因型为AA;rs4位点优势基因型为CC;rs5位点优势基因型为CC;rs008位点优势基因型为AA(表 2)。
|
|
表 2 NCAPG、DCAF16不同多态位点基因型频率和等位基因频率 Table 2 The genotype and allele frequencies of NCAPG and DCAF16 genes polymorphic loci |
在NCAPG基因中,rs1、rs2、rs4、rs5 4个SNPs位点的多态信息含量均处于低度多态(PIC < 0.25),DCAF16基因rs008位点的多态信息含量处于中度多态性(0.25<PIC < 0.50),χ2结果显示,这5个SNPs位点均处于Hardy-Weinberg平衡状态(P>0.05)(表 3)。
|
|
表 3 NCAPG、DCAF16基因群体遗传参数和Hardy-Weinberg平衡检验 Table 3 The genetic diversity and Hardy-Weinberg equilibrium test of NCAPG and DCAF16 genes |
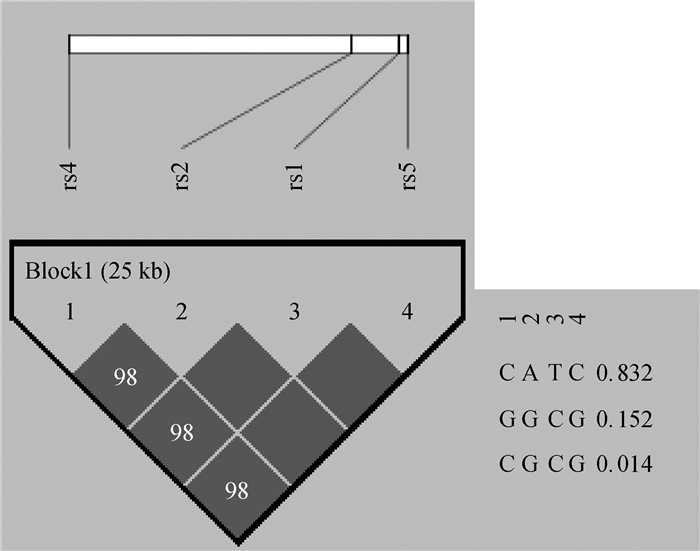
根据NCAPG基因中SNPs信息,利用Haploview4.2软件进行连锁分析,并推测其单倍型。结果发现,rs1、rs2、rs4、rs5之间强连锁,其中rs1、rs2、rs5之间完全连锁,在德州驴群体中共构建了CATC、GGCG、CGCG 3种单倍型,3种单倍型的频率分别为0.832、0.152、0.014,其中CATC为优势单倍型(图 2)。
|
图 2 NCAPG基因多态位点连锁不平衡分析 Figure 2 Linkage disequilibrium analysis of polymorphism loci of NCAPG gene |
根据连锁不平衡分析结果,rs1、rs2和rs5之间完全不平衡连锁,rs1位点的基因型CC、CT、TT分别对应rs2位点的GG、GA、AA基因型以及rs5位点的GG、GC、CC基因型,所以相对应的关联分析结果是相同的。试验结果表明,在初生时期,rs1位点基因型CC个体的平均尻长显著高于CT基因型个体(P < 0.05);在3月龄时期,3种基因型的表型平均值之间无显著差异(P>0.05);在6月龄时期,基因型为TT的个体的平均胸宽与CC基因型个体相比差异极显著(P < 0.01),同时,TT、CT基因型个体的平均管围显著高于CC基因型(P < 0.05)。rs4位点关联分析揭示了GG基因型个体初生时期的平均胸深显著高于GC基因型个体;在3月龄时期,各个基因型之间的平均表型值无显著差异(P>0.05);CC基因型个体6月龄时期的平均胸宽和管围显著高于GG基因型(P < 0.05)。rs008位点关联分析揭示了各基因型之间初生时期的平均表型值无显著差异(P>0.05);AA、GA基因型个体3月龄时期的体重、体高和尻高都显著高于GG基因型(P < 0.05);AA、GA基因型个体6月龄时期的体高和胸围都极显著地高于GG基因型(P < 0.01),GA基因型与GG基因型个体间胸深、尻高的差异达到极显著水平(P < 0.01),GA基因型个体的尻宽与GG、AA基因型个体差异显著(P < 0.05)(表 4、表 5、表 6)。
|
|
表 4 NCAPG、DCAF16基因各位点与出生时期生长性状关联分析 Table 4 Association analysis between single nucleotide polymorphism of NCAPG and DCAF16 genes and growth traits at birth |
|
|
表 5 NCAPG、DCAF16基因各位点与3月龄生长性状关联分析结果 Table 5 Association analysis between single nucleotide polymorphism of NCAPG and DCAF16 genes and growth traits at 3 months old |
|
|
表 6 NCAPG、DCAF16基因各位点与6月龄生长性状关联分析 Table 6 Association analysis between single nucleotide polymorphism of NCAPG and DCAF16 genes and growth traits at 6 months old |
连锁不平衡分析结果表明,rs1、rs2、rs4和rs5这4个位点的组合基因型在德州驴群体中有5种,分别为CCGGGGGG、CTGACCGC、CTGAGCGC、TTAACCCC和CTGAGGGC,由于组合基因型CTGAGGGC个体数为1,无统计学意义,因此在后续的关联分析中舍去。关联分析揭示了在初生和3月龄时期各组合基因型间的各生长性状无显著差异(表 7、表 8),TTAACCCC基因型个体6月龄时期的平均胸宽显著高于CCGGGGGG组合基因型个体(P < 0.05)(表 9)。
|
|
表 7 rSl、rS2、rS5和rs4组合基因型与初生时期生长性状关联分析 Table 7 Asscociation analysis between rsl, rs2, rs4, rs5 combined genotypes and growth traits at birth |
|
|
表 8 rSl、rS2、rS5和rs4组合基因型与3月龄生长性状关联分析 Table 8 Asscociation analysis between rsl, rs2, rs5, rs4 combined genotypes and growth traits at 3 months old |
|
|
表 9 rsl、rS2、rS5和rS4组合基因型与6月龄生长性状关联分析 Table 9 Asscociation analysis between rsl, rs2, rs5, rs4 combined genotypes and growth traits at 6 months old |
本研究报道了德州驴NCAPG-DCAF16基因区域上5个SNPs位点,利用NCBI上公布的序列信息发现,NCAPG基因上4个SNPs位点均位于内含子上,DCAF16基因上的1个SNP位于第2外显子。Hardy-Weinberg平衡检验结果表明,这5个SNPs位点均处于平衡状态(P>0.05)。群体遗传特征分析显示,NCAPG基因4个SNPs位点的多态信息含量均处于低度多态性(PIC < 0.25),杂合度较低,纯合基因型积累,表明这4个位点的遗传变异较小,可能与该群体的长期本品种选育以及缺少与其它种群的基因交流有关。
3.2 德州驴NCAPG、DCAF16基因多态性与生长性状的关联分析本研究在NCAPG基因上发现的4个SNPs位点均位于内含子上,目前关于NCAPG基因多态性的研究多见于人、牛和马,在德州驴群体中,4个位点处于强连锁状态,其中rs1、rs2、rs5位点完全连锁,构建了3种单倍型,其中优势单倍型CATC的频率为0.832。有研究报道,在遗传疾病及性状关联分析研究方面,单倍型可能比单个SNP标记具有更大的效应[30]。在牛上的验证群体中,该基因的SNP彼此处于强连锁不平衡状态[31],说明该基因在不同的物种中可能发挥同样的作用,对生长性状的机制也有待进一步进行功能基因组学的研究。在人、牛、马上研究的NCAPG、DCAF16基因大都是通过GWAS筛到的,人研究多采用大样本,牛和马多利用不同品种以及较大的样本量,个体间的表型值差别较大。对牛的研究发现了一个影响活体重和胴体重的致因错义突变[13],马该区域的变异能够引起体尺的变化[32]。在西门塔尔牛群体中,基于SNP的GWAS研究结果表明,与日增重极显著相关的SNP位点定位于DCAF16基因,基于单倍型的GWAS研究表明,极显著相关的单倍型位于DCAF16基因的下游,同时DCAF16和NCAPG基因在背最长肌的表达也与日增重显著相关[17]。
本试验发现,NCAPG基因位于内含子上的4个SNPs标记及其组合基因型均能够引起生长性状的变化,这与之前报道的内含子可以通过影响基因的表达来影响动物表型的结论是一致的,这种影响可能是增强基因的表达[33],也有可能是抑制基因的表达[34]。尽管内含子区域的变异并不能通过直接导致蛋白质结构的变异而影响生长发育性状的变化,但是内含子区域的变异可能与一些致因突变位点处于连锁不平衡状态有关;另一方面,90%以上与复杂性状相关的常见遗传变异位于基因间和内含子区域,调节并参与基因表达,但却不能直接改变蛋白质结构。也有研究表明,真核生物内含子可以参与基因的可变剪切机制,导致基因的编码蛋白质发生改变,进而调控生物体的生长发育[35]。Hir等[36]在2003年已经证明了内含子能够调控和影响基因的表达以及mRNA的剪接,这一结论可以解释内含子突变能够影响驴的生长发育性状,但其深层次的机制仍需要进一步研究。中国科学院最新研究发现,利用CRISPR-Cas9技术对巴马猪IGF基因第3内含子1个保守的SNP进行编辑,可以抑制转录因子ZBED6的结合并提高其生长性状[37],证明通过编辑基因组非编码区域可以显著提高家畜的生产经济性状。DCAF16基因第二外显子的rs008位点与德州驴体重和体高等重要生长性状具有显著的相关性,并且AA、GA基因型个体的表型在整体上都优于GG基因型,AA、GA基因型个体在3月龄时期的体重比GG基因型个体分别增加了5.33和4.91 kg,体高分别增长了2.46和2.10 cm,6月龄时期的体重各基因型之间的差异虽然不显著,但比GG基因型分别增加了6.01和6.22 kg,体高显著高出2.65和2.36 cm,因此在德州驴的选育过程中应加强对AA和GA两种基因型个体的选择。rs008位点突变为同义突变,不能直接导致编码氨基酸的改变,并且该位点对初生时期的表型无显著影响。此外,由于生长性状是受微效多基因调控的复杂的数量性状,除遗传因素外还受环境、性别等相关因素的影响,因此,下一步可以从转录组或者蛋白质组水平进行进一步挖掘和功能验证。
4 结论德州驴群体中NCAPG-DCAF16基因区域5个SNPs位点与生长性状均有显著的相关性,特别是rs008位点与德州驴体重、体高等重要生长性状达到极显著相关,因此,该分子标记可以用于后续德州驴的标记辅助选择,提高选择的准确性。
| [1] | DEJ K J, AHN C, ORR-WEAVER T L. Mutations in the drosophila condensin subunit dCAP-G:defining the role of condensin for chromosome condensation in mitosis and gene expression in interphase[J]. Genetics, 2004, 168(2): 895–906. DOI: 10.1534/genetics.104.030908 |
| [2] | SEIPOLD S, PRILLER F C, GOLDSMITH P, et al. Non-SMC condensin I complex proteins control chromosome segregation and survival of proliferating cells in the zebrafish neural retina[J]. BMC Dev Biol, 2009, 9(1): 40. DOI: 10.1186/1471-213X-9-40 |
| [3] | WEN X Y, DUUS K M, FRIEDRICH T D, et al. The HIV1 protein Vpr acts to promote G2 cell cycle arrest by engaging a DDB1 and Cullin4A-containing ubiquitin ligase complex using VprBP/DCAF1 as an adaptor[J]. J Biol Chem, 2007, 282(37): 27046–27057. DOI: 10.1074/jbc.M703955200 |
| [4] | HORIKOSHI M, YAGHOOTKAR H, MOOK-KANAMORI D O, et al. New loci associated with birth weight identify genetic links between intrauterine growth and adult height and metabolism[J]. Nat Genet, 2013, 45(1): 76–82. DOI: 10.1038/ng.2477 |
| [5] | VAN DER VALK R J P, KREINER-MVLLER E, KOOIJMAN M N, et al. A novel common variant in DCST2 is associated with length in early life and height in adulthood[J]. Hum Mol Genet, 2015, 24(4): 1155–1168. |
| [6] | WEEDON M N, LANGO H, LINDGREN C M, et al. Genome-wide association analysis identifies 20 loci that influence adult height[J]. Nat Genet, 2008, 40(5): 575–583. DOI: 10.1038/ng.121 |
| [7] | SORANZO N, RIVADENEIRA F, CHINAPPEN-HORSLEY U, et al. Meta-analysis of genome-wide scans for human adult stature identifies novel loci and associations with measures of skeletal frame size[J]. PLoS Genet, 2009, 5(4): e1000445. DOI: 10.1371/journal.pgen.1000445 |
| [8] | OKADA Y, KAMATANI Y, TAKAHASHI A, et al. A genome-wide association study in 19633 Japanese subjects identified LHX3-QSOX2 and IGF1 as adult height loci[J]. Hum Mol Genet, 2010, 19(11): 2303–2312. DOI: 10.1093/hmg/ddq091 |
| [9] | N'DIAYE A, CHEN G K, PALMER C D, et al. Identification, replication, and fine-mapping of loci associated with adult height in individuals of African ancestry[J]. PLoS Genet, 2011, 7(10): e1002298. DOI: 10.1371/journal.pgen.1002298 |
| [10] | SNELLING W M, ALLAN M F, KEELE J W, et al. Genome-wide association study of growth in crossbred beef cattle[J]. J Anim Sci, 2010, 88(3): 837–848. DOI: 10.2527/jas.2009-2257 |
| [11] | SETOGUCHI K, WATANABE T, WEIKARD R, et al. The SNP c.1326T>G in the non-SMC condensin I complex, subunit G (NCAPG) gene encoding a p.Ile442Met variant is associated with an increase in body frame size at puberty in cattle[J]. Anim Genet, 2011, 42(6): 650–655. DOI: 10.1111/age.2011.42.issue-6 |
| [12] | EBERLEIN A, TAKASUGA A, SETOGUCHI K, et al. Dissection of genetic factors modulating fetal growth in cattle indicates a substantial role of the non-SMC condensin I complex, subunit G (NCAPG) gene[J]. Genetics, 2009, 183(3): 951–964. DOI: 10.1534/genetics.109.106476 |
| [13] | SETOGUCHI K, FURUTA M, HIRANO T, et al. Cross-breed comparisons identified a critical 591-kb region for bovine carcass weight QTL (CW-2) on chromosome 6 and the Ile-442-Met substitution in NCAPG as a positional candidate[J]. BMC Genet, 2009, 10(1): 43. DOI: 10.1186/1471-2156-10-43 |
| [14] | WEIKARD R, ALTMAIER E, SUHRE K, et al. Metabolomic profiles indicate distinct physiological pathways affected by two loci with major divergent effect on Bos taurus growth and lipid deposition[J]. Physiol Genomics, 2010, 42A(2): 79–88. DOI: 10.1152/physiolgenomics.00120.2010 |
| [15] | WIDMANN P, REVERTER A, FORTES M R S, et al. A systems biology approach using metabolomic data reveals genes and pathways interacting to modulate divergent growth in cattle[J]. BMC Genomics, 2013, 14(1): 798. DOI: 10.1186/1471-2164-14-798 |
| [16] | WIDMANN P, REVERTER A, WEIKARD R, et al. Systems biology analysis merging phenotype, metabolomic and genomic data identifies Non-SMC Condensin I Complex, Subunit G (NCAPG) and cellular maintenance processes as major contributors to genetic variability in bovine feed efficiency[J]. PLoS One, 2015, 10(4): e0124574. DOI: 10.1371/journal.pone.0124574 |
| [17] | ZHANG W G, LI J Y, GUO Y, et al. Multi-strategy genome-wide association studies identify the DCAF16-NCAPG region as a susceptibility locus for average daily gain in cattle[J]. Sci Rep, 2016, 6: 38073. DOI: 10.1038/srep38073 |
| [18] | LIU Y, DUAN X Y, CHEN S, et al. NCAPG is differentially expressed during longissimus muscle development and is associated with growth traits in Chinese Qinchuan beef cattle[J]. Genet Mol Biol, 2015, 38(4): 450–456. DOI: 10.1590/S1415-475738420140287 |
| [19] | AL-MAMUN H A, KWAN P, CLARK S A, et al. Genome-wide association study of body weight in Australian Merino sheep reveals an orthologous region on OAR6 to human and bovine genomic regions affecting height and weight[J]. Genet Sel Evol, 2015, 47(1): 66. DOI: 10.1186/s12711-015-0142-4 |
| [20] | GUTIÉRREZ-GIL B, ESTEBAN-BLANCO C, WIENER P, et al. High-resolution analysis of selection sweeps identified between fine-wool Merino and coarse-wool Churra sheep breeds[J]. Genet Sel Evol, 2017, 49(1): 81. DOI: 10.1186/s12711-017-0354-x |
| [21] | FARIELLO M I, SERVIN B, TOSSER-KLOPP G, et al. Selection signatures in worldwide sheep populations[J]. PLoS One, 2014, 9(8): e103813. DOI: 10.1371/journal.pone.0103813 |
| [22] | ROCHUS C M, TORTEREAU F, PLISSON-PETIT F, et al. Revealing the selection history of adaptive loci using genome-wide scans for selection:an example from domestic sheep[J]. BMC Genomics, 2018, 19(1): 71. DOI: 10.1186/s12864-018-4447-x |
| [23] | CARNEIRO M, HU D, ARCHER J, et al. Dwarfism and altered craniofacial development in rabbits is caused by a 12.1 kb deletion at the HMGA2 locus[J]. Genetics, 2017, 205(2): 955–965. DOI: 10.1534/genetics.116.196667 |
| [24] | SIGNER-HASLER H, FLURY C, HAASE B, et al. A genome-wide association study reveals loci influencing height and other conformation traits in horses[J]. PLoS One, 2012, 7(5): e37282. DOI: 10.1371/journal.pone.0037282 |
| [25] | TETENS J, WIDMANN P, KVHN C, et al. A genome-wide association study indicates LCORL/NCAPG as a candidate locus for withers height in German Warmblood horses[J]. Anim Genet, 2013, 44(4): 467–471. DOI: 10.1111/age.2013.44.issue-4 |
| [26] | BOYKO A R, BROOKS S A, BEHAN-BRAMAN A, et al. Genomic analysis establishes correlation between growth and laryngeal neuropathy in Thoroughbreds[J]. BMC Genomics, 2014, 15(1): 259. DOI: 10.1186/1471-2164-15-259 |
| [27] | SEVANE N, DUNNER S, BOADO A, et al. Polymorphisms in ten candidate genes are associated with conformational and locomotive traits in Spanish Purebred horses[J]. J Appl Genet, 2017, 58(3): 355–361. DOI: 10.1007/s13353-016-0385-y |
| [28] |
周楠, 韩国才, 柴晓峰, 等. 驴的产肉、理化指标及加工特性比较研究[J]. 畜牧兽医学报, 2015, 46(12): 2314–2321.
ZHOU N, HAN G C, CHAI X F, et al. A comparative study of donkey meat production, physicochemical indicators and processing properties[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(12): 2314–2321. (in Chinese) |
| [29] |
侯文通.
现代马学[M]. 北京: 中国农业出版社, 2013.
HOU W T. Modern equine science[M]. Beijing: China Agriculture Press, 2013. (in Chinese) |
| [30] | AKEY J, JIN L, XIONG M M. Haplotypes vs single marker linkage disequilibrium tests:what do we gain?[J]. Eur J Hum Genet, 2001, 9(4): 291–300. DOI: 10.1038/sj.ejhg.5200619 |
| [31] | LINDHOLM-PERRY A K, SEXTEN A K, KUEHN L A, et al. Association, effects and validation of polymorphisms within the NCAPG-LCORL locus located on BTA6 with feed intake, gain, meat and carcass traits in beef cattle[J]. BMC Genet, 2011, 12(1): 103. DOI: 10.1186/1471-2156-12-103 |
| [32] | STAIGER E A, AL ABRI M A, PFLUG K M, et al. Skeletal variation in Tennessee Walking Horses maps to the LCORL/NCAPG gene region[J]. Phys Genomics, 2016, 48(5): 325–335. DOI: 10.1152/physiolgenomics.00100.2015 |
| [33] | CHANG K C. Critical regulatory domains in intron 2 of a porcine sarcomeric myosin heavy chain gene[J]. J Muscle Res Cell Motil, 2000, 21(5): 451–461. DOI: 10.1023/A:1005625302409 |
| [34] | BEAUCHAMP N J, DALY M E, MAKRIS M, et al. A novel mutation in intron K of the PROS1 gene causes aberrant RNA splicing and is a common cause of protein S deficiency in a UK thrombophilia cohort[J]. Thromb Haemost, 1998, 79(6): 1086–1091. DOI: 10.1055/s-0037-1615020 |
| [35] | HIRSCHEY M D, SHIMAZU T, GOETZMAN E, et al. SIRT3 regulates mitochondrial fatty-acid oxidation by reversible enzyme deacetylation[J]. Nature, 2010, 464(7285): 121–125. DOI: 10.1038/nature08778 |
| [36] | HIR H L, NOTT A, MOORE M J. How introns influence and enhance eukaryotic gene expression[J]. Trends Biochem Sci, 2003, 28(4): 215–220. DOI: 10.1016/S0968-0004(03)00052-5 |
| [37] | XIANG G H, REN J L, HAI T, et al. Editing porcine IGF2 regulatory element improved meat production in Chinese Bama pigs[J]. Cell Mol Life Sci, 2018, 75(24): 4619–4628. DOI: 10.1007/s00018-018-2917-6 |



