2. 青藏高原动物遗传资源保护与利用教育部重点实验室, 成都 610041;
3. 青藏高原动物遗传资源保护与利用四川省重点实验室, 成都 610041;
4. 西南民族大学现代生物技术国家民委重点实验室, 成都 610041
2. Key Laboratory of Ministry of Education for Qinghai-Tibetan Plateau Animal Genetic Resource Reservation and Exploitation, Chengdu 610041, China;
3. Qinghai-Tibetan Plateau Animal Genetic Resource Reservation and Utilization Key Laboratory of Sichuan Province, Chengdu 610041, China;
4. Key Laboratory of Modern Biotechnology of State Ethnic Affairs Commission, Southwest Minzu University, Chengdu 610041, China
组蛋白乙酰化是一类重要的表观遗传修饰方式[1],细胞内组蛋白乙酰化水平主要由组蛋白乙酰化转移酶(histone acetyltransferases,HATs)及去乙酰化酶(histone deacetylases,HDACs)共同调节[2]。哺乳动物的HDACs共有3类17个成员,Ⅰ类包括HDAC1、2、3和8,Ⅱ类包括HDAC4、5、6、7、9和10,Ⅲ类则主要是SIRT家族成员[3]。HDAC主要通过对已经添加乙酰基(acetyl,ac)的组蛋白赖氨酸(lysine,K)位点进行去乙酰化,进而增加组蛋白表面的负电荷,并增强组蛋白与DNA的结合能力,使染色质处于凝集状态,从而对基因的表达起负调控作用[4]。Sirtuin家族是一类具有烟酰胺嘌呤核苷酸依赖性的组蛋白去乙酰化酶,包括7个成员:SIRT1~SIRT7,其中每个成员均包含一个由275个氨基酸组成的催化核心区域,其在细胞中的亚定位也各不相同。SIRT1、SIRT6和SIRT7主要定位于细胞核中, SIRT2定位于细胞质,SIRT3、SIRT4和SIRT5则主要定位于线粒体。哺乳动物Sirtuin可与多个蛋白如NF-κB、p53、FOXO、Ku70及PGC-1等互作进而影响细胞应激反应、代谢、衰老和凋亡等过程[5-8]。
SIRT3是目前研究最多的线粒体Sirtuin蛋白,在进化过程中高度保守,广泛分布于各组织中,通过对组蛋白、线粒体靶蛋白或其他蛋白去乙酰化从而调控基因的转录或蛋白的活性[9]。SIRT3可以对线粒体中的ATP合成酶去乙酰化,缺失SIRT3会导致多个器官ATP的本底水平降低[10]。在能量代谢中的不同应激状态下,SIRT3可以通过去乙酰化超氧化物歧化酶2(superoxide dismutase 2,SOD2)和异柠檬酸脱氢酶2(isocitrate dehydrogenase 2,IDH2)减少细胞内ROS的水平,并保护细胞免受氧化应激诱导的死亡[11-13],在心肌细胞中,SIRT3可以将Ku70去乙酰化,去乙酰化的Ku70能够与促凋亡蛋白BAX互作并抑制后者转移到线粒体,进而保护线粒体的完整性[14]。SIRT3还可以通过去乙酰化在转录水平抑制低氧诱导因子-1(hypoxia inducible factor-1,HIF-1)的表达,进而抑制氧化应激相关通路及肿瘤细胞的生长[15]。
牦牛是生活在青藏高原低氧环境下的特有畜种,能为牧民提供优质畜产品,是当地人民不可或缺的生活生产资料, 素有“高原之舟”和“全能家畜”的美誉,但牦牛繁殖能力较低,对于雄性牦牛,其性腺的发育始于胚胎期,而跨背交配行为通常发生在幼年阶段的1~2岁,3~5岁进入性成熟期并且具有较强的配种能力,7~9岁逐渐进入老年期,其采精量及交配能力明显下降[16-18]。牦牛的繁殖特性严重阻碍了其生产效率。迄今为止,对于SIRT3基因在牦牛的功能研究尚未见报道。本试验利用RT-PCR技术,克隆获得牦牛SIRT3基因序列,并对所得序列进行生物信息学分析。通过qRT-PCR技术检测SIRT3在牦牛不同组织和4个不同发育阶段(胎牛期、幼年期、性成熟期及老年期)睾丸中的表达水平,本研究为深入探讨SIRT3基因对低氧环境下牦牛的生长发育、尤其是睾丸组织发育的调控提供了一定的试验依据。
1 材料与方法 1.1 组织样本的采集样本来源于四川广汉市盛大食品加工厂,根据齿龄将牦牛划分为胎牛组(5~6个月)、幼年组(1~2岁)、性成熟组(3~5岁)及老年组(7~9岁)。经颈部放血致死后剖腹,立即用无菌剪刀采集健康牦牛(3~5岁)的不同组织,包括肝、心、肺、脾、脑、肾、肌肉、小肠、睾丸和大肠,并采集胎牛期、幼年期、性成熟期及老年期牦牛睾丸组织(胎牛期牦牛睾丸组织需以外科去势手术采集), 其中来源于同一年龄阶段的3个组织样本视为生物学重复。采集的组织用灭菌生理盐水冲洗后,再用剪刀剪成约为1 cm×1 cm×0.5 cm大小的组织块,放入冻存管中,置于液氮罐中带回实验室,-80 ℃保存。
1.2 主要试剂和仪器Trizol购自Invitrogen公司;PrimeScriptTM RT Reagent Kit反转录试剂盒、Premix TapTM DNA聚合酶、SYBR®Premix Ex TaqTM Ⅱ试剂盒、pMD19-T载体由TaKaRa公司生产;DNA胶回收试剂盒由康宁生命科学有限公司生产;DEPC、感受态细胞DH5α和DNA marker均购自天根生化科技有限公司,其他无特殊说明的试剂均为国产分析纯。
1.3 牦牛各组织总RNA的提取和反转录按照Trizol方法从牦牛各个组织提取RNA,使用紫外分光光度计测定RNA浓度和OD值,将符合要求的RNA按照反转录试剂盒的说明方法合成cDNA,将所得cDNA于-20 ℃保存备用。
1.4 牦牛SIRT3基因的克隆测序参照GenBank中黄牛SIRT3基因序列(登录号:NM_001206669.1),通过Primer Premier 5.0软件设计PCR扩增引物(表 1),送南京金斯瑞生物科技有限公司合成。PCR扩增体系为25 μL:2×LA Taq Master Mix 12.5 μL,上、下游引物各1 μL,cDNA模板1 μL,ddH2O 9.5 μL。PCR反应条件:95 ℃预变性4 min;95 ℃变性30 s,62 ℃退火45 s,72 ℃延伸45 s,38个循环;72 ℃延伸7 min,4 ℃保存。使用浓度为1.5%的琼脂糖凝胶进行PCR电泳检测,并将目的条带切胶回收。在16 ℃的金属浴中将胶回收产物与pMD19-T载体连接15 h,之后与感受态细胞混合,并在LB液体培养基中培养1 h后离心。将沉淀菌液均匀涂布到含有0.1%氨苄的LB固体培养基上,置于37 ℃恒温培养箱中培养12 h,选取白色单菌落接种于液态LB培养基中,并放入摇床震荡培养8 h后,通过PCR筛选阳性菌株,交由Sangon Biotech公司测序。
|
|
表 1 PCR引物序列及产物长度 Table 1 PCR primer sequences and products length |
利用NCBI中BLAST在线对比工具检索SIRT3基因的核苷酸序列,通过ORF Finder预测其氨基酸序列。通过ExPASy在线软件中的ProtParam、ProtScale、PredictProtein和SWISS_MODEL分别预测牦牛SIRT3蛋白的基本理化性质、亲疏水性、二级结构和三级结构。通过Megalign软件进行同源性比对并构建进化树。
1.6 牦牛SIRT3基因组织表达谱分析根据已克隆出的牦牛SIRT3基因序列设计实时荧光定量PCR引物(表 1),以GAPDH作为内参基因,其引物根据GenBank中黄牛GAPDH基因序列(登录号:NM_001034034.2)设计(表 1),并通过实时荧光定量PCR仪检测该基因在各组织的表达量。反应总体系为20 μL:Green Supermix 10 μL、上下游引物各0.5 μL、cDNA模板1 μL、ddH2O 8 μL。反应条件:95 ℃预变性2 min;95 ℃变性10 s,60 ℃退火30 s,72 ℃延伸30 s,40个循环,然后以0.5 ℃/10 s的速率从62 ℃缓慢升温到98 ℃。熔解曲线:95 ℃ 1 min,55 ℃ 1 min。每个样品重复检测3次。
1.7 qRT-PCR检测SIRT3基因在牦牛睾丸不同发育阶段的表达同样以GAPDH为内参基因,利用qRT-PCR检测SIRT3 mRNA在不同年龄牦牛睾丸中的表达水平,反应体系同1.6,每个样品重复3次。
1.8 统计学分析以脾的Ct值作为不同组织间差异定量分析的参照,采用2-ΔΔCt法进行分析,通过SPSS统计软件对数据进行显著性分析,结果用“平均值±标准差”表示,P < 0.05表示差异显著,P < 0.01表示差异极显著。
2 结果 2.1 牦牛SIRT3基因的克隆以牦牛肝组织cDNA为模板,通过RT-PCR对SIRT3基因的CDS区进行扩增,经琼脂糖凝胶电泳检测, 获得含有目的基因1 019 bp的条带(图 1), 测序获得其核苷酸序列,通过NCBI在线分析,开放阅读框(open reading frame, ORF)为1 002 bp,共编码333个氨基酸。
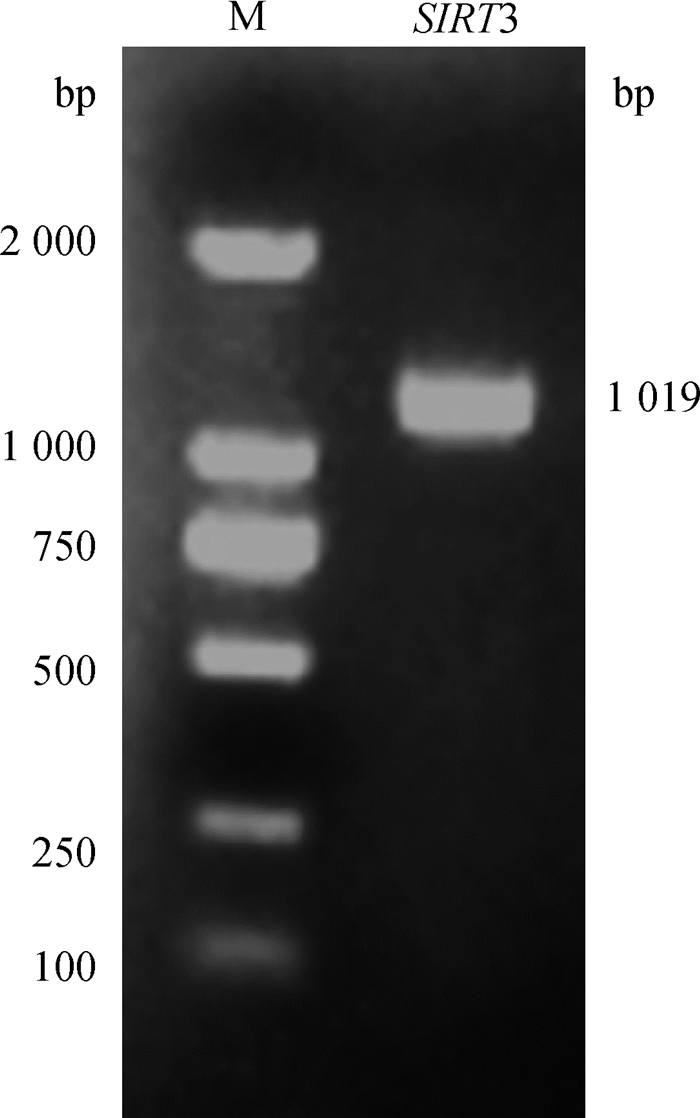
|
图 1 牦牛SIRT3基因扩增结果 Fig. 1 PCR amplification for SIRT3 gene of yak |
利用Megalign软件,将牦牛SIRT3基因与其他物种进行相似性比对,并构建系统进化树(图 2),结果表明,牦牛SIRT3基因CDS区高度保守,牦牛与黄牛SIRT3基因的亲缘关系最近, 同源性达到99.9%,其次是绵羊,达到96.7%,与其他物种的相似性也均达到85%以上(表 2)。由此可见,SIRT3基因在物种进化过程中高度保守,并且,其中碱基突变也多为同义突变。
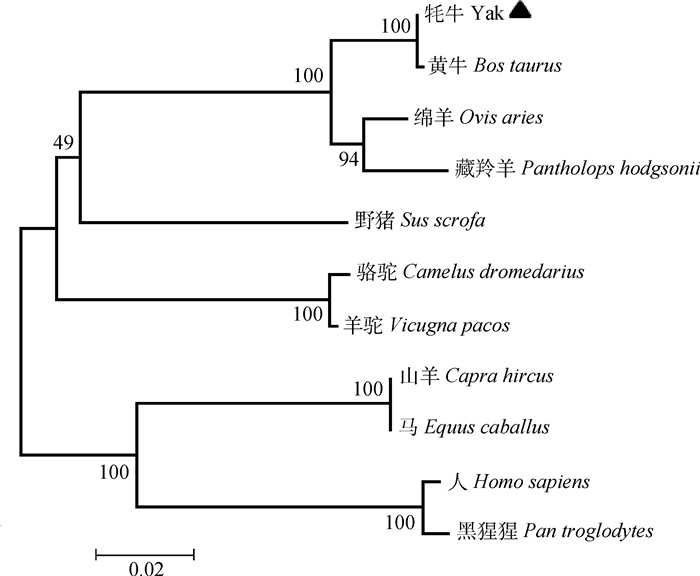
|
图 2 SIRT3基因核苷酸进化树分析 Fig. 2 Nucleotide phylogenetic tree analysis of SIRT3 gene |
|
|
表 2 牦牛与其他物种SIRT3基因的同源性比对 Table 2 Alignment of SIRT3 gene sequences between yak and other species |
SIRT3蛋白的理化性质通过ExPASy在线工具进行预测。得到该蛋白分子量为36.991 ku,分子式为C1686H2611N459O460S10,脂肪族系数为91.38,半衰期为30 h,等电点为8.86,不稳定系数为36.53,推测该蛋白为碱性不稳定蛋白。SIRT3蛋白包含20种氨基酸,其中出现频率较高的有Leu(12.0%)、Gly(9.0%)、Ala(7.8%)和Arg(7.8%)。带正电荷氨基酸(Arg+Lys)有39个,带负电荷氨基酸(Asp+Glu)有34个,由此可得,SIRT3蛋白整体上带正电。亲疏水性分析发现,牦牛SIRT3蛋白在第51个氨基酸位点存在最小亲水指数-2.222,在第77个氨基酸位点存在最大疏水指数2.689,平均亲水指数为-0.053。Signal P和TM pred预测发现,SIRT3蛋白为无跨膜结构和信号肽的非分泌蛋白。
SIRT3蛋白的二级结构预测结果显示(图 3),该蛋白中α-螺旋占35.14%,β-转角占5.71%,延伸链占17.12%,无规卷曲占42.04%,同时,SIRT3蛋白的三级结构进一步验证了二级结构的预测(图 4)。
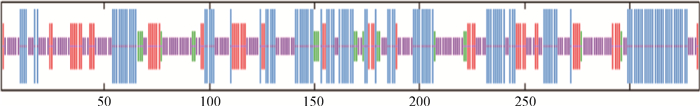
|
最短线(紫色)代表自由卷曲;最长线(蓝色)代表α-螺旋;次长线(红色)代表延伸链;次短线(绿色)代表β-转角 The shortest lines(purple) stand for random coil; the longest lines (blue) stand for alpha helix; and the longer lines(red) stand for beta sheet; the shorter lines (green) stand for beta turn 图 3 牦牛SIRT3蛋白二级结构预测 Fig. 3 Predicted secondary structure of yak SIRT3 protein |
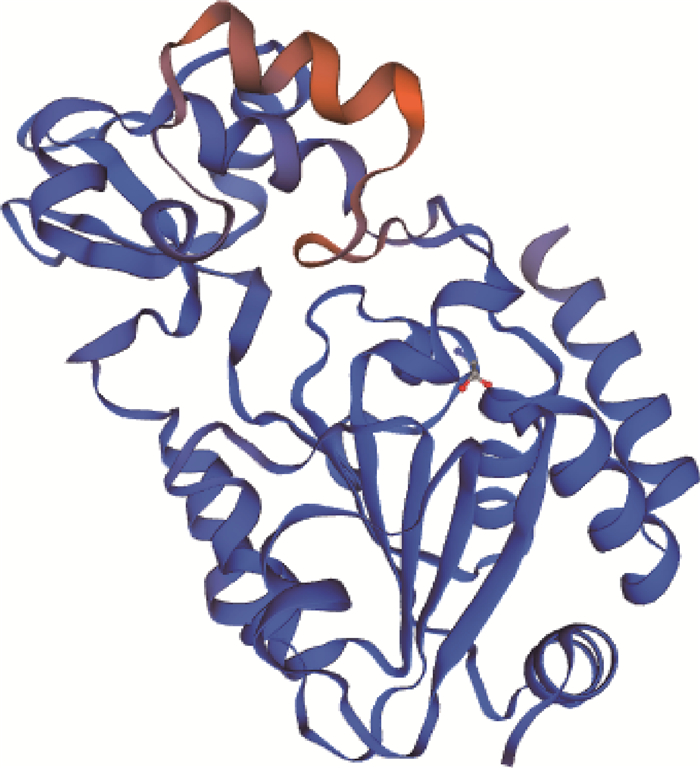
|
图 4 牦牛SIRT3蛋白三级结构预测 Fig. 4 Predicted tertiary structure of yak SIRT3 protein |
利用qRT-PCR检测SIRT3基因在牦牛各组织中的表达差异,结果显示,SIRT3基因在所测组织中均有表达(图 5),其中在脾的表达相对较高,在肌肉、睾丸、肺和肾中次之,在脑、大肠、小肠、心和肝中表达较弱。
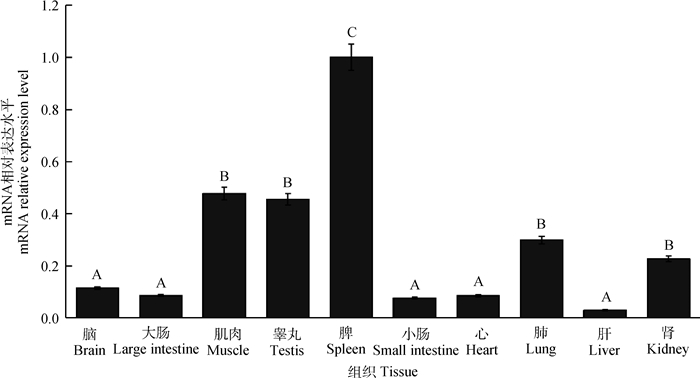
|
相同字母表示差异不显著(P>0.05),不同字母表示差异极显著(P < 0.01), 下同 The same letter indicates that the difference is not significant (P>0.05), and the different letters indicate that the difference is significant (P < 0.01).The same as below 图 5 牦牛SIRT3 mRNA的组织表达谱 Fig. 5 The mRNA expression profile of SIRT3 in tissues of yak |
以GAPDH为内参基因,通过qRT-PCR法检测SIRT3基因在胎牛期、幼年期、性成熟期及老年期牦牛睾丸中的表达水平(图 6),结果显示, 随年龄的增长,SIRT3基因在牦牛睾丸中的表达呈先升高后下降的趋势,在性成熟期的表达最高。
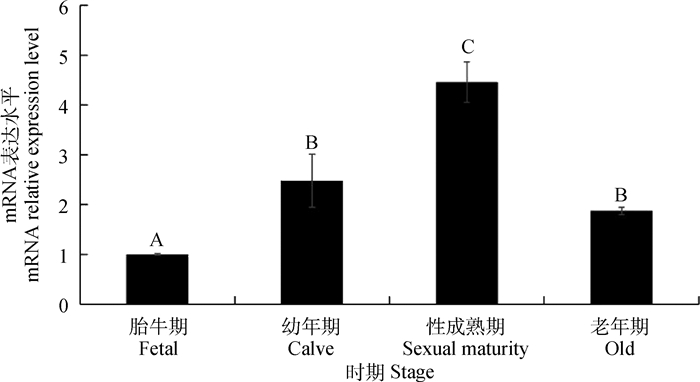
|
图 6 SIRT3 mRNA在不同发育时期牦牛睾丸中的表达 Fig. 6 The mRNA expression of SIRT3 in yak testis at different development stages |
SIRT3属于沉默信息调节因子SIRT蛋白家族成员,是主要的线粒体去乙酰化酶之一[19],已报道其在恶性肿瘤、肺动脉高压、心室重构等的发生发展中发挥了重要作用[20-22]。SIRT3可激活心肌细胞叉头框蛋白3a (forkhead box 03a,FOXO3a)、超氧化物歧化酶(superoxide dismutase,SOD)及过氧化氢酶, 从而清除氧自由基, 抑制下游磷脂酰肌醇3激酶/蛋白激酶B(phosphatidylinositol 3′-OH kinase(PI3K) /protein kinase B(PKB))及丝裂原活化蛋白激酶/细胞外调节蛋白激酶(mitogen-activated protein kinases/extracellular signal-regulated kinase,MAPK/ERK)等的活性[23],SIRT3还能够抑制NF-κB信号途径,促进其下游的B细胞淋巴瘤2(B-cell lymphoma-2,Bcl-2)、SOD2蛋白的表达, 抑制Bcl-2关联X蛋白(Bcl-2-associated X protein,BAX)的表达, 具有抑制抗氧化应激诱导的心肌细胞损伤及抗凋亡作用[24]。
本试验成功克隆了牦牛SIRT3的基因序列。通过比对分析,得出该序列为牦牛SIRT3基因的全长编码区序列。编码区序列比较分析结果显示,在哺乳动物中SIRT3基因的同源性较高,表明SIRT3基因在物种进化过程中高度保守。同时发现,牦牛与黄牛的氨基酸序列高度匹配,由此说明SIRT3在蛋白质结构水平上也较为保守。推测在物种进化过程中,SIRT3氨基酸序列及其功能的稳定性对协调生物体适应其生存环境是必要的。
本试验检测了SIRT3基因在牦牛不同组织中的表达,结果表明,该基因mRNA在牦牛的各类组织中广泛表达,其在脾中的表达量最高。研究表明,对大鼠喂食养生药膏—琼玉膏之后,其脾衰老的速度明显减缓,脾中SIRT3的表达量明显升高,而MAPK/ERK信号通路受到抑制[25],牦牛脾中高水平的SIRT3是否通过MAPK/ERK信号通路能够延缓脾的衰老有待进一步研究。此外,SIRT3在代谢活跃的组织如肌肉、肺和肾中的表达较高[26-27],推测可能与其负责的维持能量稳态的作用有关。
在不同年龄牦牛睾丸中SIRT3基因的表达呈动态变化,在性成熟期之前SIRT3的表达随年龄增加而升高,推测这可能与睾丸组织中活性氧水平以及SIRT3的抗氧化能力有关。随着年龄的增加,睾丸内各类细胞开始不断进行旺盛的细胞代谢活动并产生大量活性氧,而高水平活性氧被认为会抑制青春期睾丸大量分泌的雄性激素-睾酮的合成[28-29],研究表明,多种抗氧化剂如辅酶Q10及N-乙酰半胱氨酸(NAC)等均能通过降低睾丸组织内的ROS从而提高睾酮水平[30-31],因此推测,此阶段具有抗氧化作用的SIRT3的高表达可能是为了清除睾酮生成时产生的活性氧,从而使睾丸组织维持在一个较低的活性氧水平,并保持睾酮的正常生成。此外,本研究结果表明,老年期牦牛睾丸中SIRT3的表达量显著低于性成熟期,而在人和小鼠的一些组织或器官,如卵巢、肾和脂肪中存在类似的现象[32],对人的研究表明,老龄化卵巢中低水平的SIRT3可能和线粒体内另一种重要的酶-谷氨酸脱氢酶的活性有关[33],因此推测,老年期牦牛睾丸中SIRT3表达的下降可能与此阶段线粒体功能的衰退有关。
4 结论本研究成功克隆获得了牦牛SIRT3基因的cDNA序列,生物信息学分析表明,SIRT3蛋白为亲水、不存在跨膜区、无信号肽的蛋白质,并且其编码区在生物进化中高度保守,qRT-RCR检测SIRT3 mRNA在牦牛不同组织中的表达,其在脾的表达量最高。在不同年龄的牦牛睾丸中SIRT3的表达随其年龄的增长先升高后下降,在性成熟期的表达最高。本试验为进一步研究SIRT3基因对牦牛的生长发育,尤其是雄性生殖系统的发育机制提供了试验依据。
| [1] | SHEN Y, WEI W, ZHOU D X. Histone acetylation enzymes coordinate metabolism and gene expression[J]. Trends Plant Sci, 2015, 20(10): 614–621. DOI: 10.1016/j.tplants.2015.07.005 |
| [2] | YIN S, JIANG X H, JIANG H W, et al. Histone acetyltransferase KAT8 is essential for mouse oocyte development by regulating reactive oxygen species levels[J]. Development, 2017, 144(12): 2165–2174. DOI: 10.1242/dev.149518 |
| [3] | MILLARD C J, WATSON P J, FAIRALL L, et al. Targeting class I histone deacetylases in a "Complex" environment[J]. Trends Pharmacol Sci, 2017, 38(4): 363–377. DOI: 10.1016/j.tips.2016.12.006 |
| [4] | ALVES-FERNANDES D K, JASIULIONIS M G. The role of SIRT1 on DNA damage response and epigenetic alterations in cancer[J]. Int J Mol Sci, 2019, 20(13): 3153. DOI: 10.3390/ijms20133153 |
| [5] | ZHOU L, XU D Y, SHA W G, et al. Long non-coding RNA MALAT1 interacts with transcription factor Foxo1 to regulate SIRT1 transcription in high glucose-induced HK-2 cells injury[J]. Biochem Biophys Res Commun, 2018, 503(2): 849–855. DOI: 10.1016/j.bbrc.2018.06.086 |
| [6] | MAHMUD Z, GOMES A R, LEE H J, et al. EP300 and SIRT1/6 co-regulate lapatinib sensitivity via modulating FOXO3-acetylation and activity in breast cancer[J]. Cancers, 2019, 11(8): 1067. DOI: 10.3390/cancers11081067 |
| [7] | BOTTA G, DE SANTIS L P, SALADINO R. Current advances in the synthesis and antitumoral activity of SIRT1-2 inhibitors by modulation of p53 and pro-apoptotic proteins[J]. Curr Med Chem, 2012, 19(34): 5871–5884. DOI: 10.2174/092986712804143303 |
| [8] | KOSCIUK T, WANG M, HONG J Y, et al. Updates on the epigenetic roles of sirtuins[J]. Curr Opin Chem Biol, 2019, 51: 18–29. DOI: 10.1016/j.cbpa.2019.01.023 |
| [9] | KATWAL G, BARAL D, FAN X L, et al. SIRT3 a major player in attenuation of hepatic ischemia-reperfusion injury by reducing ROS via its downstream mediators:SOD2, CYP-D, and HIF-1α[J]. Oxid Med Cell Longev, 2018, 2018: 2976957. |
| [10] | STORDER J, RENARD P, ARNOULD T. Update on the role of Sirtuin 3 in cell differentiation:a major metabolic target that can be pharmacologically controlled[J]. Biochem Pharmacol, 2019, 169: 113621. DOI: 10.1016/j.bcp.2019.08.023 |
| [11] | CUI X X, LI X, DONG S Y, et al. SIRT3 deacetylated and increased citrate synthase activity in PD model[J]. Biochem Biophys Res Commun, 2017, 484(4): 767–773. DOI: 10.1016/j.bbrc.2017.01.163 |
| [12] | LIU X H, ZHANG L, WANG P, et al. Sirt3-dependent deacetylation of SOD2 plays a protective role against oxidative stress in oocytes from diabetic mice[J]. Cell Cycle, 2017, 16(13): 1302–1308. DOI: 10.1080/15384101.2017.1320004 |
| [13] | YU W, DITTENHAFER-REED K E, DENU J M. SIRT3 protein deacetylates isocitrate dehydrogenase 2 (IDH2) and regulates mitochondrial redox status[J]. J Biol Chem, 2012, 287(17): 14078–14086. DOI: 10.1074/jbc.M112.355206 |
| [14] | PARODI-RULLÁN R M, CHAPA-DUBOCQ X R, JAVADOV S. Acetylation of mitochondrial proteins in the heart:the role of SIRT3[J]. Front Physiol, 2018, 9: 1094. |
| [15] | WEI L, ZHOU Y, QIAO C, et al. Oroxylin a inhibits glycolysis-dependent proliferation of human breast cancer via promoting SIRT3-mediated SOD2 transcription and HIF1α destabilization[J]. Cell Death Dis, 2015, 6(4): e1714. DOI: 10.1038/cddis.2015.86 |
| [16] |
权凯.牦牛超数排卵技术研究[D].兰州: 甘肃农业大学, 2004.
QUAN K.Study on superovulation of yak[D]. Lanzhou: Gansu Agricultural University, 2004.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10733-2005013262.htm |
| [17] |
蔡雯祎, 熊显荣, 陈通, 等. KDM2B基因克隆及其在牦牛组织和卵母细胞减数分裂过程中的表达研究[J]. 畜牧兽医学报, 2018, 49(3): 534–541.
CAI W Y, XIONG X R, CHEN T, et al. Molecular cloning of KDM2B gene and its expression pattern in tissues and the process of oocyte meiosis in yak[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(3): 534–541. (in Chinese) |
| [18] |
阮崇美.不同发育期白牦牛睾丸蛋白质组学分析及生殖相关候选基因HSP60生物学研究[D].兰州: 甘肃农业大学, 2017.
RUAN C M.Proteomic analysis of white yak testis in different developmental stages and reproductive candidate gene HSP60 biology research[D]. Lanzhou: Gansu Agricultural University, 2017.(in Chinese) |
| [19] | SALVATORI I, VALLE C, FERRI A, et al. SIRT3 and mitochondrial metabolism in neurodegenerative diseases[J]. Neurochem Int, 2017, 109: 184–192. DOI: 10.1016/j.neuint.2017.04.012 |
| [20] | QUAN Y Z, WANG N T, CHEN Q Q, et al. SIRT3 inhibits prostate cancer by destabilizing oncoprotein c-MYC through regulation of the PI3K/Akt pathway[J]. Oncotarget, 2015, 6(28): 26494–26507. |
| [21] | WEI T, HUANG G J, GAO J, et al. Sirtuin 3 deficiency accelerates hypertensive cardiac remodeling by impairing angiogenesis[J]. J Am Heart Assoc, 2017, 6(8): e006114. |
| [22] | PAULIN R, DROMPARIS P, SUTENDRA G, et al. Sirtuin 3 deficiency is associated with inhibited mitochondrial function and pulmonary arterial hypertension in rodents and humans[J]. Cell Metab, 2014, 20(5): 827–839. DOI: 10.1016/j.cmet.2014.08.011 |
| [23] | PENG Y H, YANG C, SHI X L, et al. Sirt3 suppresses calcium oxalate-induced renal tubular epithelial cell injury via modification of FoxO3a-mediated autophagy[J]. Cell Death Dis, 2019, 10(2): 34. DOI: 10.1038/s41419-018-1169-6 |
| [24] | CHEN C J, FU Y C, YU W, et al. SIRT3 protects cardiomyocytes from oxidative stress-mediated cell death by activating NF-κB[J]. Biochem Biophys Res Commun, 2013, 430(2): 798–803. DOI: 10.1016/j.bbrc.2012.11.066 |
| [25] |
方妙玉.琼玉膏经MAPK/ERK1/2信号通路延缓大鼠脾脏衰老机制研究[D].广州: 广州中医药大学, 2016.
FANG M Y.Study on Qiongyugao aging mechanisms via MAPK/ERK1/2 signal pathway on the spleen of aging rats[D]. Guangzhou: Guangzhou University of Chinese Medicine, 2016.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10572-1016208773.htm |
| [26] | CORONA J C, DUCHEN M R. PPARγ and PGC-1α as therapeutic targets in Parkinson's[J]. Neurochem Res, 2015, 40(2): 308–316. DOI: 10.1007/s11064-014-1377-0 |
| [27] | EDGETT B A, HUGHES M C, MATUSIAK J B L, et al. SIRT3 gene expression but not SIRT3 subcellular localization is altered in response to fasting and exercise in human skeletal muscle[J]. Exp Physiol, 2016, 101(8): 1101–1113. DOI: 10.1113/EP085744 |
| [28] | GUERRIERO G, TROCCHIA S, ABDEL-GAWAD F K, et al. Roles of reactive oxygen species in the spermatogenesis regulation[J]. Front Endocrinol (Lausanne), 2014, 5: 56. |
| [29] | DI-LUOFFO M, BROUSSEAU C, BERGERON F, et al. The transcription factor MEF2 is a novel regulator of gsta gene class in mouse MA-10 leydig cells[J]. Endocrinology, 2015, 156(12): 4695–4706. DOI: 10.1210/en.2015-1500 |
| [30] | BANIHANI S A. Effect of coenzyme Q10 supplementation on testosterone[J]. Biomolecules, 2018, 8(4): 172. DOI: 10.3390/biom8040172 |
| [31] | MALMIR M, SOLEIMANI MEHRANJANI M, NADERI NOREINI S, et al. Protective antioxidant effects of N-acetylcysteine against impairment of spermatogenesis caused by paranonylphenol[J]. Andrologia, 2018, 50(10): e13114. DOI: 10.1111/and.13114 |
| [32] | KWON Y, KIM J, LEE C Y, et al. Expression of SIRT1 and SIRT3 varies according to age in mice[J]. Anat Cell Biol, 2015, 48(1): 54–61. DOI: 10.5115/acb.2015.48.1.54 |
| [33] | TATONE C, DI EMIDIO G, VITTI M, et al. Sirtuin functions in female fertility:possible role in oxidative stress and aging[J]. Oxid Med Cell Longev, 2015, 2015: 659687. |



