2. 中国农业科学院兰州兽医研究所, 兰州 730046
2. Lanzhou Veterinary Research Institute, Chinese Academy of Agricultural Sciences, Lanzhou 730046, China
布鲁菌病是由布鲁菌引起的一种重要的人畜共患细菌性传染病,该病在世界范围内流行,给家畜的健康养殖和人类公共卫生安全带来严重危害。由于布鲁菌能够经呼吸道、胃肠道、结膜的黏膜入侵人和动物体并被黏膜巨噬细胞和树突状细胞(DC)吞噬,因此,巨噬细胞和树突状细胞被认为是布鲁菌先天性免疫的关键细胞。在布鲁菌入侵宿主后最初几小时内巨噬细胞和树突状细胞能够杀死90%左右的布鲁菌,得以存活的布鲁菌通过各种毒力因子调节和逃避宿主先天性免疫应答及适应性免疫应答使其能够在单核吞噬细胞内存活和复制[1]。布鲁菌已经进化出多种逃避宿主免疫识别的机制进入细胞并抑制宿主免疫应答,进而建立慢性感染使其在细胞内长期生存,然而布鲁菌具体的免疫逃逸机制仍不明确。
蛋白质泛素化修饰是一种动态多功能的翻译后修饰,几乎涉及真核生物的所有生物过程。通过连续的酶原激活,由E1泛素活化酶通过其活性位点中的半胱氨酸残基与泛素C末端结合,该过程需要ATP的参与;然后将硫酯连接的泛素转移至泛素缀合酶E2的半胱氨酸残基,泛素缀合酶E2负责组装不同拓扑结构的多聚泛素链;最终由泛素连接酶E3通过募集靶蛋白来控制底物蛋白质的泛素化特异性。一旦76个氨基酸的泛素连接到底物上将会对底物蛋白质进行进一步修饰并产生不同生物级联信号。泛素可以通过七个赖氨酸残基或N-末端的甲基进行泛素连接,形成具有拓扑异构的多聚泛素链。免疫应答复杂性、多功能性和特异性不仅仅由多种类型免疫细胞及其受体实现,还需要通过参与免疫信号传导途径的蛋白质翻译后修饰来实现,越来越多证据表明蛋白质的泛素化及其去泛素化在免疫反应的启动、维持和终止中起到重要作用。相关研究表明多种细菌可以调控宿主泛素化或去泛素化,进而影响宿主细胞蛋白质的降解与信号传导,如假结核耶尔森菌(Y. pseudotuberculosis)通过效应子YopJ作为一种类似于去泛素化酶CYLD对宿主NF-κB信号通路上Traf6/2的K63多聚泛素进行切割,进而抑制NF-κB的激活[2];弗氏志贺菌(Shigella flexneri)效应子OspG通过干扰IκBα的蛋白酶体依赖性降解来负调节NF-κB炎症反应[3]。然而,关于布鲁菌及其毒力因子对宿主泛素化修饰调控进而影响免疫应答的分子机制尚不清楚。
为了探究布鲁菌感染宿主细胞后对宿主免疫应答相关蛋白质泛素化修饰的影响,笔者基于Label-free和泛素化富集技术以及高分辨率LC-MS/MS联用的定量蛋白质组学技术,对布鲁菌16M感染后巨噬细胞和未感染的巨噬细胞开展了泛素化修饰蛋白质组学定量研究。预筛选出布鲁菌16M感染后宿主细胞免疫应答受抑制的关键泛素化修饰蛋白质,为进一步揭示布鲁菌感染后造成宿主免疫抑制并逃避宿主免疫识别的分子机制提供理论依据,为布鲁菌新型疫苗的设计及药物靶点的筛选提供技术支撑。
1 材料与方法 1.1 菌株、细胞和试剂细菌:在TSA固体培养基上划线羊布鲁菌16M(军事兽医研究所王兴龙研究员馈赠)。37 ℃恒温培养箱中培养72 h后挑取单菌落于5 mL TSB中,在恒温摇床中37 ℃、170 r·min-1培养72 h,并进行菌落计数。
细胞:复苏液氮中保存的小鼠单核巨噬细胞白血病细胞RAW264(武汉普诺赛生命科学有限公司)。用含有10% FBS的DMEM于37 ℃、5% CO2恒温细胞培养箱中培养,待细胞密度达到80%时按照MOI为1:100接种布鲁菌16M,布鲁菌在感染巨噬细胞初期的6~12 h是其LPS依赖期和T4SS依赖期,因此选择感染总时长为11 h(先胞外侵染5 h后PBS洗3遍,再使用100 μg·mL-1的含庆大霉素的DMEM完全培养基继续培养6 h),对照组在同等时间节点使用不含FBS的DMEM代替布鲁菌使用相同方法处理,16M感染组细胞和未感染组细胞样本独立进行两个生物学重复,并对两个生物学重复样本进行三个技术重复。
乙腈、三氟乙酸、碘代乙酰胺、二硫苏糖醇、尿素、乙二胺四乙酸、烟酰胺和TSA购自Sigma,甲酸和胰酶购自Fluka,蛋白酶抑制剂和BCA试剂盒购自碧云天。
1.2 蛋白质提取、胰酶酶解对感染11 h的巨噬细胞和未感染的对照组细胞分别加入4体积裂解缓冲液(8 mol·L-1尿素,1%蛋白酶抑制剂,3 μmol·L-1 TSA,50 mmol·L-1 NAM和2 mmol·L-1 EDTA),超声裂解。4 ℃,12 000 g离心10 min,去除细胞碎片,上清液转移至新的离心管,利用BCA试剂盒进行蛋白浓度测定。蛋白溶液中加入二硫苏糖醇使其终浓度为5 mmol·L-1,56 ℃还原30 min。之后加入碘代乙酰胺使其终浓度为11 mmol·L-1,室温避光孵育15 min。最后将样品的尿素浓度稀释至低于2 mol·L-1。以1:50的质量比例(胰酶:蛋白)加入胰酶,酶解过夜。再以1:100的质量比例(胰酶:蛋白)加入胰酶,继续酶解4 h。
1.3 亲和富集将肽段溶解在IP缓冲溶液中(100 mmol·L-1 NaCl, 1 mmol·L-1 EDTA, 50 mmol·L-1 Tris-HCl, 0.5% NP-40, pH 8.0),转移上清液至提前洗涤好的泛素化树脂中(树脂货号PTM-1 104,来源于杭州景杰生物科技有限公司,PTM Bio),放置于4 ℃环境的旋转摇床上,温和摇晃并过夜孵育。孵育结束后依次使用IP缓冲溶液洗涤树脂4次,去离子水洗涤两次。最后使用0.1%三氟乙酸洗脱液,将树脂结合的肽段洗脱下来,共洗脱三次,收集洗脱液并真空冷冻抽干。抽干后按照C18 ZipTips说明书除盐,真空冷冻抽干后供液质联用分析。
1.4 液相色谱-质谱联用分析肽段用液相色谱流动相A相溶解后使用EASY-nLC 1000超高效液相系统进行分离。流动相A为含0.1%甲酸和2%乙腈的水溶液;缓冲液B为含0.1%甲酸和90%乙腈的水溶液。液相梯度设置:0~60 min,6%~24% B;60~82 min,24%~36% B;82~86 min,36%~80% B;86~90 min,80% B,流速维持在350 nL·min-1。
肽段经由超高效液相系统分离后被注入NSI离子源中进行电离然后进OrbitrapFusionTM质谱进行分析。离子源电压设置为2.0 kV,肽段母离子使用高分辨的Orbitrap进行检测和分析。一级质谱扫描范围设置为350~1 550 m/z,扫描分辨率设置为60 000;二级质谱扫描范围则固定起点为100 m/z,并且二级离子进入离子阱检测。数据采集模式使用数据依赖型扫描(DDA)程序,即在一级扫描后选择信号强度最高的20个母离子使用35%的碎裂能量进行HCD碎裂,同样依次进行二级质谱分析。为了提高质谱的有效利用率,设置自动增益控制(AGC)设置为1E4,信号阈值设置为5 000 ions·s-1,最大注入时间设置为100 ms,串联质谱扫描的动态排除时间设置为15 s避免母离子的重复扫描。
1.5 数据库搜索二级质谱数据使用Maxquant (v1.5.2.8)进行检索。检索参数设置:数据库为SwissProtmouse(16 839条序列),添加了反库以计算随机匹配造成的假阳性率(FDR),并且在数据库中加入了常见的污染库,用于消除鉴定结果中污染蛋白的影响;酶切方式设置为Trypsin/P;漏切位点数设为4;肽段最小长度设置为7个氨基酸残基;肽段最大修饰数设为5;First search和Main search的一级母离子质量误差容忍度分别设为2×10-5和5×10-6,二级碎片离子的质量误差容忍度为0.5 u。将半胱氨酸烷基化设置为固定修饰,可变修饰为甲硫氨酸的氧化,蛋白N端的乙酰化和赖氨酸的泛素化。蛋白鉴定、PSM鉴定的FDR都设置为1%。
1.6 生物信息学分析将质谱鉴定得到差异表达的蛋白数据分别通过GO分析,蛋白结构域注释,KEGG通路注释,亚细胞定位的方法进行蛋白注释。根据蛋白注释分类分别进行GO富集,通路富集和蛋白结构域富集,其中GO富集将蛋白质分成三大类,即生物进程、细胞组成、分子功能。Kyoto Encyclopedia of Genes and Genomes(KEGG)数据库被用于通路的富集分析。InterPro数据库用于分析差异表达蛋白的功能结构域的富集情况。这些富集检验均以P-value值小于0.05为差异显著。
2 结果 2.1 样品重复性检验本次试验共设立了两个生物学重复和三个技术重复,并对两个生物学重复和三个技术重复样品的定量结果是否符合统计学上的一致性进行了检验。通过计算所有样本两两之间的皮尔森相关系数(Pearson’s correlation coefficient)绘制成了热图。此系数是度量两组数据线性相关程度的值:当皮尔森系数越接近-1为负相关,使用绿色表示;当皮尔森系数越接近1为正相关,使用红色表示。结果表明两个生物学重复和三个技术重复样品的定量结果重复性高且符合统计学上的一致性(图 1)。
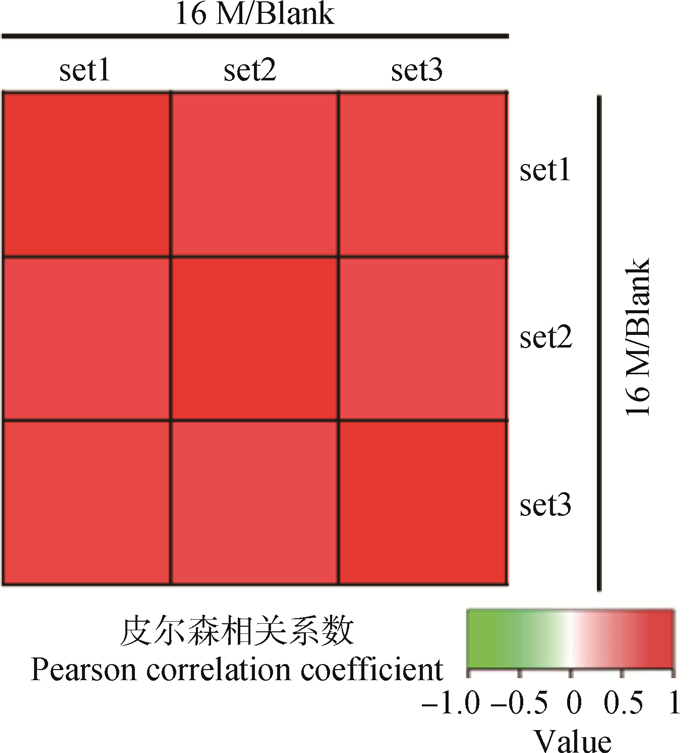
|
图 1 样品重复性检验 Fig. 1 Sample repeatability test |
对质谱数据进行了质控检测,结果显示检测的所有鉴定到的肽段的质量偏移(mass error),质量误差以0为中轴并且集中在低于1×10-5的范围内,说明质量误差符合要求(图 2A)。其次,绝大部分的肽段长度分布在8~20个氨基酸残基之间,符合胰酶消化肽段的规律(图 2B),说明样品制备达到标准。
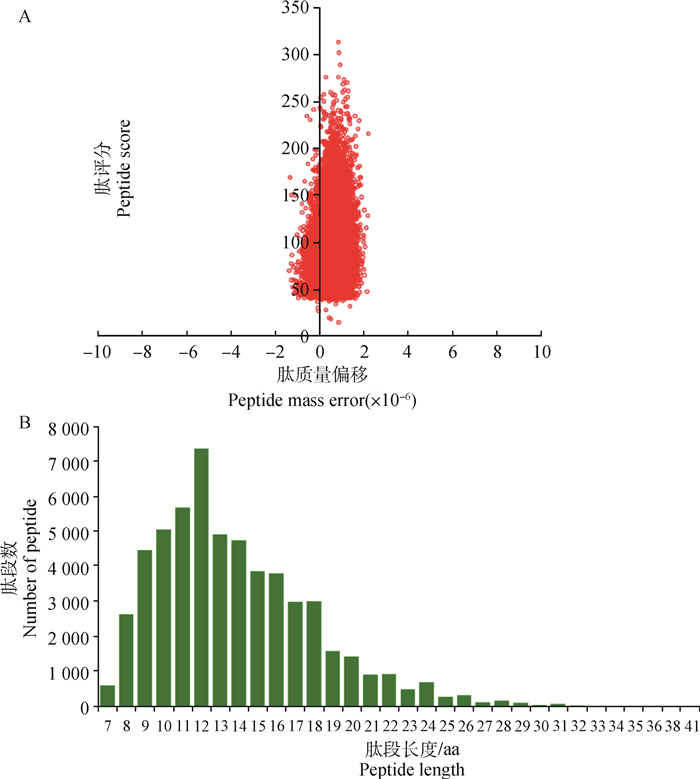
|
A.鉴定肽段的质量偏移分布;B.鉴定肽段的长度分布 A. Mass offset distribution of identification peptide segment; B. Length distribution of peptide segments 图 2 质谱数据的指控检测 Fig. 2 Testing results of mass spectrometry data |
通过对鉴定到的两组样品中所有泛素化位点前后氨基酸序列(从位置-10到+10)的规律进行统计,并计算出发生泛素化位点区域内氨基酸序列的规律趋势。分析修饰的位点序列特征,从而推测或者确定修饰相关的酶。通过对鉴定到的所有泛素化修饰位点上下游氨基酸的基序富集热图分析差异表达的泛素化位点区域内氨基酸序列的规律。这些被检测到的基序被解释为KubxxxxxxxR、RxxxxxxxKub、KubxxxxxxR、KubxxxxxR和KubG。Kub是赖氨酸的泛素化,x代表随机氨基酸残基。对这些基序的分布分析中发现在这些基序中仅发现了两种特定氨基酸残基。这两种保守的氨基酸残基分别是精氨酸(R)和甘氨酸(G)(图 3A)。如热图所示,这些基序主要可分为两大类,第一种其位置+1是甘氨酸残基,另一种其位置+10、+9、+8、+7、+6、+5、-8、-9是精氨酸残基。这些结果表明,泛素化体系倾向于修饰赖氨酸残基,并且容易被+10到-10同样带正电荷的精氨酸残基包围(图 3B)。
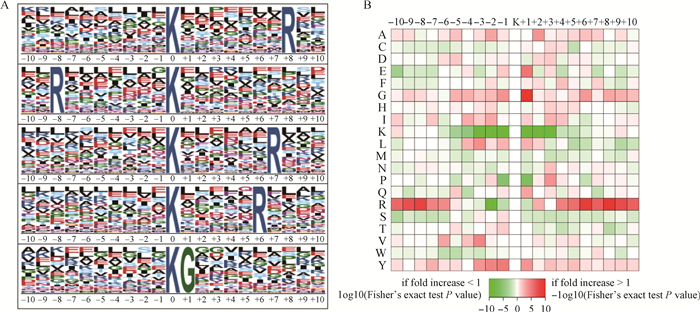
|
A.中心(位置0)K表示泛素化赖氨酸。所有周围的氨基酸残基用不同高度的字母表示,这与它们在各自位置的频率一致;B.泛素化修饰位点上下游氨基酸序列富集热图 A.The central K (at position 0) indicates the Ubiquitination lysine. All the surrounding amino acid residues are indicated with the letters in different heights which is consistent with their frequencies in respective positions; B.Sequence enrichment heat map of upstream and downstream amino acids at ubiquitinized modified sites 图 3 用Motif-x软件构建泛素化基序 Fig. 3 Ubiquitination motifs were constructed with Motif-X software |
通过数据库搜索共鉴定到349个蛋白质上的580个泛素化位点(FDR≤1%),其中16M感染组细胞相对于对照组细胞上调泛素化位点259个,对应的蛋白质167个;下调泛素化位点321个,对应的蛋白质212个。通过GO二级注释分类的生物信息学分析方法将GO注释分为三个一级大类:生物进程、细胞组成和分子功能,我们着重对生物进程进行了比对分析,分析结果发现泛素化修饰差异表达蛋白质主要集中在cellular process、metabolic process、biological regulation、signaling、immune system process、developmental process、response to stimulus和other 8个二级分类中(图 4),其中16M感染后宿主细胞免疫应答相关蛋白质泛素化修饰下调表达27个,泛素化修饰上调蛋白质11个。
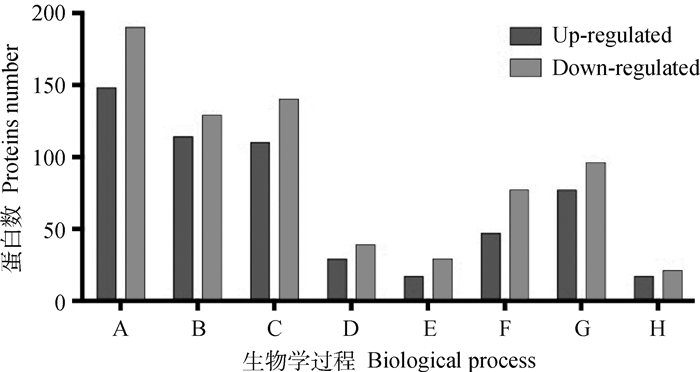
|
A.细胞过程;B.代谢过程;C.生物调控;D.信号;E.免疫系统过程;F.发育过程;G.对刺激的反应;H.其它 A. Cellular process; B. Metabolic process; C. Biological regulation; D. Signaling; E. Immune system process; F.Developmental process; G. Response to stimulus; H. Other 图 4 16M/Blank上调和下调泛素化修饰蛋白质生物学过程富集 Fig. 4 Biological process enrichment of up-regulated and down-regulated ubiquitinated modified proteins by 16M/Blank |
布鲁菌16M感染后巨噬细胞与未感染巨噬细胞的比对,下调表达的泛素化位点对应的蛋白质共212个,其中与免疫应答相关的蛋白质有27个,有调控细胞凋亡的Bcap31[4]、参与MHCⅠ类抗原递呈的Tubb5[5]、对细菌反应的正调节的Grn[6]、参与MAPK信号级联的Ywhae[7]、调控细胞对IL-4的反应的Hsp90ab1[8]和介导细胞程序性死亡的Pkm[9]等蛋白质(表 1[4-29]);同时布鲁菌感染后宿主免疫应答相关蛋白质的泛素化修饰上调11个,有促进T细胞活化的Cd44[30]、负调控Toll样受体3信号通路活化的Ubqln1、调控自噬体成熟的Ubqln4[31]以及促进胞内细菌清除的Irgm1[32]等蛋白质(表 2)。
|
|
表 1 免疫应答相关泛素化修饰下调表达的位点对应的蛋白质 Table 1 Proteins corresponding to down-regulated expression sites of immune response-related ubiquitination modification |
|
|
表 2 免疫应答相关泛素化修饰上调表达的位点对应的蛋白质 Table 2 Proteins corresponding to up-regulated expression sites of immune response-related ubiquitination modification |
根据STRING数据库对鉴定出的与免疫应答相关差异表达的35个泛素化修饰蛋白质,包括泛素化位点上调表达对应的11个蛋白质,泛素化位点下调表达对应的27个蛋白质,3个既存在上调表达的泛素化修饰位点同时又存在下调表达的泛素化修饰位点的蛋白质,通过Cytoscape组成蛋白质与蛋白质之间的网络互作图(confidence score>0.7)(图 5),通过蛋白质间的网络互作关系,笔者发现布鲁菌16M感染宿主后能够影响多个参与免疫应答相关的蛋白质泛素化修饰的下调表达进而起到抑制免疫应答的作用,其中作为T细胞和B细胞抗原受体信号传导的重要调节器的ptprc[11];参与T细胞的抗原加工和递呈的Icam1[13];参与TLR9信号通路中NF-κB激活的btk[15]等27个正调控免疫应答的蛋白质泛素化修饰均受到了抑制;同时布鲁菌感染宿主细胞后还能促进Ubqln1的泛素化修饰的上调表达,而Ubqln1能够通过自噬途径抑制降低TICAM1的表达,负调控Toll受体3信号通路的激活;Ubqln4作为抑制自噬体成熟的重要分子,其泛素化修饰也显著增强。
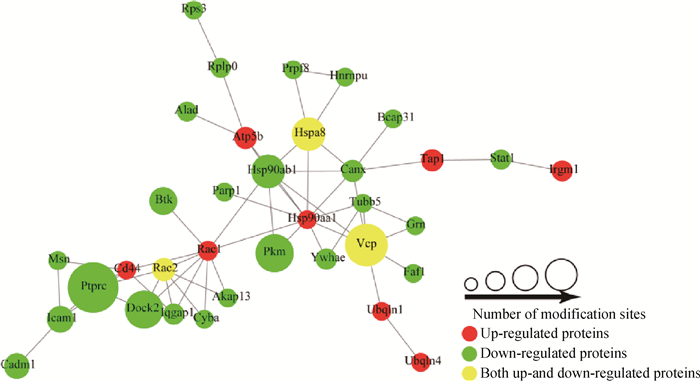
|
图 5 免疫应答相关差异表达泛素化修饰蛋白质互作网络 Fig. 5 Protein-protein interaction network diagram of differentially expressed ubiquitinated modified proteins |
泛素化是所有真核生物的基本蛋白质翻译后修饰,泛素化包括将一个或几个泛素结合到靶蛋白上。单泛素化可以触发定位或蛋白质活性的改变,多泛素化又分为非降解型泛素化和触发靶向26S蛋白酶体降解型泛素化。蛋白质也可以通过与泛素样蛋白质的共价连接来修饰,例如与泛素相关的小修饰物SUMO和NEDD8的相互作用[36]。泛素具有7个赖氨酸残基,可以形成7种不同连接类型的可能性,即K6、K11、K27、K29、K33、K48和K63。此外,供体泛素也可以通过泛素氨基酸链末端的甲硫氨酸(Met1)与受体蛋白质连接,形成Met1连接的线性泛素链,通过不同氨基酸位点的连接总共可以产生8种不同的泛素连接类型的泛素链,并且在所有真核生物内泛素化和去泛素化是动态共存的。本研究对布鲁菌感染宿主细胞后泛素化修饰蛋白质组学研究中对鉴定到与免疫应答的35个泛素化修饰蛋白质进行了泛素化类型的富集分析。分析结果发现布鲁菌感染巨噬细胞后,其泛素化修饰差异表达蛋白泛素化修饰类型主要分为10种类型(图 6),并且多以K63多聚泛素化、泛素样蛋白的结合和蛋白酶体-泛素依赖性蛋白质分解三大类为主。K63多聚泛素化在细胞内作为非降解型泛素化修饰参与多种免疫信号的传递和DNA损伤的修复[37];泛素样蛋白是类似于泛素的一类蛋白质,泛素样蛋白与靶蛋白之间的连接能够促进靶蛋白与其他蛋白质之间也发生相互作用,这种泛素化修饰主要通过形成蛋白质复合物发挥功能作用[38];而蛋白酶体-泛素依赖性蛋白质分解系统(ubiquitin-proteasome system,UPS)作为细胞内蛋白质和肽的主要降解机制,参与细胞内80%以上的蛋白质降解。

|
图 6 免疫应答相关蛋白质泛素化类型富集分析 Fig. 6 Ubiquitous type enrichment map of immune response related proteins |
布鲁菌能够通过多种毒力因子逃避宿主天然免疫识别进入细胞内并抑制宿主免疫应答,进一步建立慢性感染使其在细胞内长期生存,然而具体机制还不明确。同时,越来越多研究表明蛋白质的泛素化及其去泛素化在免疫应答过程中起到重要作用。为此,筛选鉴定布鲁菌在LPS依赖性和T4SS依赖性的感染初期,宿主免疫应答相关泛素化修饰差异表达蛋白质,对研究布鲁菌及其毒力因子调节宿主免疫反应的分子机制和药物靶点的筛选具有重要意义。本研究通过非标定量技术,泛素化肽段的富集技术以及基于质谱的定量蛋白质组学技术等一系列前沿技术的有机结合,对布鲁菌16M感染小鼠巨噬细胞RAW264.7细胞样本进行泛素化定量组学进行研究,比较感染前后小鼠巨噬细胞中泛素化修饰显著差异表达蛋白质,共获得349个蛋白质上580个泛素化位点,其中,布鲁菌16M感染组167个蛋白质上259个位点泛素化修饰水平发生上调,212个蛋白质上321个位点泛素化修饰水平发生下调(差异倍数>1.5,P < 0.05);其中35个蛋白质与免疫应答相关,并且其泛素化修饰发生了差异表达,35个免疫相关蛋白质中下调表达的泛素化修饰蛋白质有27个,上调表达泛素化修饰蛋白质仅11个。该结果说明布鲁菌感染后可能通过抑制宿主细胞的免疫相关蛋白质的泛素化修饰进而调控宿主的免疫应答。
为了分析布鲁菌16M感染后对宿主整个生物过程的影响,笔者对鉴定到的所有差异表达泛素化位点对应的蛋白质进行了生物过程分析,发现差异表达的泛素化修饰主要影响宿主细胞的细胞生物进程、水解过程、生物调节、信号传导、免疫应答过程、细胞生长过程和对应激的应答过程(图 4)。为了进一步分析布鲁菌16M对生物过程中的免疫应答的影响,我们对参与免疫应答过程的35个差异表达的泛素化修饰蛋白质进行了功能注释和分类,其中下调泛素化修饰蛋白质的功能又可以分以下6大类:抗原加工和MHC抗原递呈(Tubb5和Canx)、对细菌或细菌脂多糖反应(Grn、Alad、Stat1、Prpf8和Cyba)、参与MAPK或NF-κB信号的传递(Ywhae、Btk、Akap13和Rps3)、对细胞因子的反应(Hsp90ab1、Rplp0、Iqgap1和Hurnpu)、细胞程序性死亡(Bcap31、pkm、Parp1、Faf1和Vcp)、T细胞的活化与增殖(Dock2、Ptprc、Rac2、Icam1、Cadm1、Msn和Hspa8)。我们推测布鲁菌可能通过调控上述27个下调表达的泛素化修饰蛋白质进而抑制宿主在免疫应答过程中的抗原加工和MHC抗原递呈、自噬与凋亡、MAPK或NF-κB信号传导进而影响细胞因子的分泌。在相关研究中发现Btk作为非受体酪氨酸激酶对B细胞的发育分化以及信号传导都是必不可少的,其作为BCR-NF-κB信号通路中必需调节因子,在Btk缺失细胞系(DT40.BTK)中,NF-κB的激活受到了显著抑制,同时在TLR途径中Btk是调节脾B细胞TLR9信号通路激活的关键分子[15]。Faf1的N-端UBA结构域可与泛素结合,其C-端UBX结构域又可与Vcp相关联,三者结合共同参与泛素-蛋白酶体途径调节蛋白质的降解,而泛素-蛋白酶体途径作为细胞凋亡和自噬过程中重要环节直接促进细胞的凋亡和自噬,同时Faf1自身的过表达也能够直接导致细胞的死亡[26]。此外,布鲁菌感染后还能通过上调toll样受体3信号通路过程中负调控蛋白Ubqln1的泛素化修饰,而Ubqln1的过表达能够导致TLR3信号通路的抑制,同时Ubqln1和Ubqln4相互作用能够促进自噬体的成熟[31]。而在本研究中通过对布鲁菌感染前后免疫应答相关蛋白质泛素化修饰差异表达的富集分析与功能分类,发现布鲁菌感染后能够调控宿主细胞的免疫信号通路、抗原的识别和加工、T细胞的活化与增殖和细胞的自噬与凋亡等免疫应答过程中关键蛋白质的泛素化修饰的差异表达,这些分子可能致使布鲁菌在感染初期达到“隐身”状态,并使得其在宿主细胞成功的生存复制。
通过对35个免疫应答相关蛋白质的泛素化修饰类型的富集分析,笔者发现布鲁菌能够抑制多个蛋白质的非降解型的K63多聚泛素化传递免疫信号,如Ywhae能够通过形成K63多聚泛素链促进MAPK信号通路的激活[13];Btk、Akap13和Rps3的K63多聚泛素化能够促进NF-κB上游信号的级联[39]。自噬的过程离不开泛素-蛋白酶体系统,在对免疫应答相关的蛋白质富集泛素化修饰类型分析中,笔者还发现布鲁菌能够通过促进Vcp和Ubqln4的泛素化,而在其他文献中报道Vcp和Ubqln4的泛素化可能影响蛋白酶体-泛素系统进而影响细胞自噬。细胞凋亡是细胞程序性死亡最重要的调节方式之一,对胞内细菌的清除具有优异的作用,然而组学结果发现布鲁菌能够通过下调表达宿主细胞的Parp1和Faf1的泛素化修饰抑制细胞的凋亡,从而维持布鲁菌在细胞内的生态位复制。
4 结论通过泛素化修饰蛋白质组学研究,发现布鲁菌16M能够抑制宿主细胞MAPK和NF-κB信号通路中正调控蛋白Ywhae、Btk、Akap13和Rps3的泛素化修饰,促进负调控蛋白Ubqln1的泛素化修饰;布鲁菌16M还能够抑制宿主细胞自噬和凋亡相关蛋白质Vcp、Parp1和Faf1的泛素化修饰;并且布鲁菌16M对T细胞的增殖与活化等多种免疫应答过程中Dock2、Ptprc、Icam1和Cadm1等多个调控蛋白质的泛素化修饰均具有抑制作用;这些免疫应答过程中相关蛋白质的泛素化修饰差异表达可能有助于布鲁菌在感染宿主后逃避宿主免疫应答并为其在细胞内成功复制生存提供稳态环境。
| [1] |
周玉成, 张海威, 程世鹏, 等. 布鲁菌感染早期抗免疫机制研究进展[J]. 动物医学进展, 2019, 40(1): 88–93.
ZHOU Y C, ZHANG H W, CHENG S P, et al. Advances in anti-immune mechanism of early infection with Brucella[J]. Progress in Veterinary Medicine, 2019, 40(1): 88–93. DOI: 10.3969/j.issn.1007-5038.2019.01.018 (in Chinese) |
| [2] | ZHOU H L, MONACK D M, KAYAGAKI N, et al. Yersinia virulence factor YopJ acts as a deubiquitinase to inhibit NF-κB activation[J]. J Exp Med, 2005, 202(10): 1327–1332. DOI: 10.1084/jem.20051194 |
| [3] | KIM D W, LENZEN G, PAGE A L, et al. The Shigella flexneri effector OspG interferes with innate immune responses by targeting ubiquitin-conjugating enzymes[J]. Proc Natl Acad Sci U S A, 2005, 102(39): 14046–14051. DOI: 10.1073/pnas.0504466102 |
| [4] | NGUYEN M, BRECKENRIDGE D G, DUCRET A, et al. Caspase-resistant BAP31 inhibits fas-mediated apoptotic membrane fragmentation and release of cytochrome c from mitochondria[J]. Mol Cell Biol, 2000, 20(18): 6731–6740. DOI: 10.1128/MCB.20.18.6731-6740.2000 |
| [5] | TARAZONA R, LÓPEZ-LLUCH G, GALIANI M D, et al. HLA-B2702 (77-83/83-77) peptide binds to β-tubulin on human NK cells and blocks their cytotoxic capacity[J]. J Immunol, 2000, 165(12): 6776–6782. DOI: 10.4049/jimmunol.165.12.6776 |
| [6] | YIN F F, BANERJEE R, THOMAS B, et al. Exaggerated inflammation, impaired host defense, and neuropathology in progranulin-deficient mice[J]. J Exp Med, 2010, 207(1): 117–128. DOI: 10.1084/jem.20091568 |
| [7] | WANG X Z, GRAMMATIKAKIS N, SIGANOU A, et al. Regulation of molecular chaperone gene transcription involves the serine phosphorylation, 14-3-3ε binding, and cytoplasmic sequestration of heat shock factor 1[J]. Mol Cell Biol, 2003, 23(17): 6013–6026. DOI: 10.1128/MCB.23.17.6013-6026.2003 |
| [8] | CHU C C, PAUL W E. Expressed genes in interleukin-4 treated B cells identified by cDNA representational difference analysis[J]. Mol Immunol, 1998, 35(8): 487–502. DOI: 10.1016/S0161-5890(98)00031-5 |
| [9] | STETÁK A, VERESS R, OVÁDI J, et al. Nuclear translocation of the tumor marker pyruvate kinase M2 induces programmed cell death[J]. Cancer Res, 2007, 67(4): 1602–1608. DOI: 10.1158/0008-5472.CAN-06-2870 |
| [10] | SANUI T, INAYOSHI A, NODA M, et al. DOCK2 is essential for antigen-induced translocation of TCR and lipid rafts, but not PKC-θ and LFA-1, in T cells[J]. Immunity, 2003, 19(1): 119–129. DOI: 10.1016/S1074-7613(03)00169-9 |
| [11] | HARP J A, DAVIS B S, EWALD S J. Inhibition of T cell responses to alloantigens and polyclonal mitogens by Ly-5 antisera[J]. J Immunol, 1984, 133(1): 10–15. |
| [12] | HETZ C, MARTINON F, RODRIGUEZ D, et al. The unfolded protein response:integrating stress signals through the stress sensor IRE1α[J]. Physiol Rev, 2011, 91(4): 1219–1243. DOI: 10.1152/physrev.00001.2011 |
| [13] | SIU G, HEDRICK S M, BRIAN A A. Isolation of the murine intercellular adhesion molecule 1 (ICAM-1) gene. ICAM-1 enhances antigen-specific T cell activation[J]. J Immunol, 1989, 143(11): 3813–3820. |
| [14] | BOLES K S, BARCHET W, DIACOVO T, et al. The tumor suppressor TSLC1/NECL-2 triggers NK-cell and CD8+ T-cell responses through the cell-surface receptor CRTAM[J]. Blood, 2005, 106(3): 779–786. DOI: 10.1182/blood-2005-02-0817 |
| [15] | ANTONY P, PETRO J B, CARLESSO G, et al. B Cell receptor directs the activation of NFAT and NF-κB via distinct molecular mechanisms[J]. Exp Cell Res, 2003, 291(1): 11–24. DOI: 10.1016/S0014-4827(03)00338-0 |
| [16] | WILHELM E A, JESSE C R, ROMAN S S, et al. Hepatoprotective effect of 3-alkynyl selenophene on acute liver injury induced by D-galactosamine and lipopolysaccharide[J]. Exp Mol Pathol, 2009, 87(1): 20–26. |
| [17] | SHARMA S, FINDLAY G M, BANDUKWALA H S, et al. Dephosphorylation of the nuclear factor of activated T cells (NFAT) transcription factor is regulated by an RNA-protein scaffold complex[J]. Proc Natl Acad Sci U S A, 2011, 108(28): 11381–11386. DOI: 10.1073/pnas.1019711108 |
| [18] | DEL VESCOVO C D, COTECCHIA S, DIVIANI D. A-kinase-anchoring protein-Lbc anchors IκB kinase β to support interleukin-6-mediated cardiomyocyte hypertrophy[J]. Mol Cell Biol, 2013, 33(1): 14–27. |
| [19] | GAO X F, WAN F Y, MATEO K, et al. Bacterial effector binding to ribosomal protein S3 subverts NF-κB function[J]. PLoS Pathog, 2009, 5(12): e1000708. DOI: 10.1371/journal.ppat.1000708 |
| [20] | BHATTACHARYYA S, ZHAO Y X, KAY T W H, et al. Glucocorticoids target suppressor of cytokine signaling 1 (SOCS1) and type 1 interferons to regulate Toll-like receptor-induced STAT1 activation[J]. Proc Natl Acad Sci U S A, 2011, 108(23): 9554–9559. DOI: 10.1073/pnas.1017296108 |
| [21] | HAAS D W, SHEPHERD V L, HAGEDORN C H. Lipopolysaccharide stimulates phosphorylation of eukaryotic initiation factor-4F in macrophages and tumor necrosis factor participates in this event[J]. Second Messengers Phosphoproteins, 1992, 14(3): 163–171. |
| [22] | YANG C S, LEE J S, RODGERS M, et al. Autophagy protein Rubicon mediates phagocytic NADPH oxidase activation in response to microbial infection or TLR stimulation[J]. Cell Host Microbe, 2012, 11(3): 264–276. DOI: 10.1016/j.chom.2012.01.018 |
| [23] | LAGRESLE-PEYROU C, LUCE S, OUCHANI F, et al. X-linked primary immunodeficiency associated with hemizygous mutations in the moesin (MSN) gene[J]. J Allergy Clin Immun, 2016, 138(6): 1681–1689. DOI: 10.1016/j.jaci.2016.04.032 |
| [24] | STANELLE J, TU-RAPP H, PVTZER B M. A novel mitochondrial protein DIP mediates E2F1-induced apoptosis independently of p53[J]. Cell Death Differ, 2005, 12(4): 347–357. DOI: 10.1038/sj.cdd.4401532 |
| [25] | VIZLIN-HODZIC D, JOHANSSON H, RYME J, et al. SAF-A has a role in transcriptional regulation of Oct4 in ES cells through promoter binding[J]. Cell Reprogram, 2011, 13(1): 13–27. DOI: 10.1089/cell.2010.0075 |
| [26] | RYU S W, CHAE S K, LEE K J, et al. Identification and characterization of human Fas associated factor 1, hFAF1[J]. Biochem Biophys Res Commun, 1999, 262(2): 388–394. DOI: 10.1006/bbrc.1999.1217 |
| [27] | BENVENUTI F, HUGUES S, WALMSLEY M, et al. Requirement of Rac1 and Rac2 expression by mature dendritic cells for T cell priming[J]. Science, 2004, 305(5687): 1150–1153. DOI: 10.1126/science.1099159 |
| [28] | GAUDET P, LIVSTONE M S, LEWIS S E, et al. Phylogenetic-based propagation of functional annotations within the gene ontology consortium[J]. Brief Bioinform, 2011, 12(5): 449–462. DOI: 10.1093/bib/bbr042 |
| [29] | TAKASHIMA S, SATO N, KISHI A, et al. Involvement of peptide antigens in the cytotoxicity between 70-kDa heat shock cognate protein-like molecule and CD3+, CD4-, CD8-, TCR-alpha beta- killer T cells[J]. J Immunol, 1996, 157(8): 3391–3395. |
| [30] | KELLEHER D, MURPHY A, FEIGHERY C, et al. Leukocyte function-associated antigen 1 (LFA-1) and CD44 are signalling molecules for cytoskeleton-dependent morphological changes in activated T cells[J]. J Leukoc Biol, 1995, 58(5): 539–546. DOI: 10.1002/jlb.58.5.539 |
| [31] | SONG E J, YIM S H, KIM E, et al. Human Fas-associated factor 1, interacting with ubiquitinated proteins and valosin-containing protein, is involved in the ubiquitin-proteasome pathway[J]. Mol Cell Biol, 2005, 25(6): 2511–2524. DOI: 10.1128/MCB.25.6.2511-2524.2005 |
| [32] | SINGH S B, DAVIS A S, TAYLOR G A, et al. Human IRGM induces autophagy to eliminate intracellular mycobacteria[J]. Science, 2006, 313(5792): 1438–1441. DOI: 10.1126/science.1129577 |
| [33] | VANTOUROUT P, MARTINEZ L O, FABRE A, et al. Ecto-F1-ATPase and MHC-class Ⅰ close association on cell membranes[J]. Mol Immunol, 2008, 45(2): 485–492. |
| [34] | YOSHIDA M, XIA Y. Heat shock protein 90 as an endogenous protein enhancer of inducible nitric-oxide synthase[J]. J Biol Chem, 2003, 278(38): 36953–36958. DOI: 10.1074/jbc.M305214200 |
| [35] | RITZ U, MOMBURG F, PIRCHER H P, et al. Identification of sequences in the human peptide transporter subunit TAP1 required for transporter associated with antigen processing (TAP) function[J]. Int Immunol, 2001, 13(1): 31–41. DOI: 10.1093/intimm/13.1.31 |
| [36] | DYE B T, SCHULMAN B A. Structural mechanisms underlying posttranslational modification by ubiquitin-like proteins[J]. Annu Rev Biophys Biomol Struct, 2007, 36: 131–150. DOI: 10.1146/annurev.biophys.36.040306.132820 |
| [37] | LIU P D, GAN W J, SU S Y, et al. K63-linked polyubiquitin chains bind to DNA to facilitate DNA damage repair[J]. Sci Signal, 2018, 11(533): eaar8133. DOI: 10.1126/scisignal.aar8133 |
| [38] | KERSCHER O, FELBERBAUM R, HOCHSTRASSER M. Modification of proteins by ubiquitin and ubiquitin-like proteins[J]. Annu Rev Cell Dev Biol, 2006, 22: 159–180. DOI: 10.1146/annurev.cellbio.22.010605.093503 |
| [39] | CHEN Z J. Ubiquitin signalling in the NF-κB pathway[J]. Nat Cell Biol, 2005, 7(8): 758–765. DOI: 10.1038/ncb0805-758 |



