组蛋白的乙酰化、甲基化、磷酸化、泛素化等共价修饰在基因转录调节和基因组完整性等方面发挥关键作用[1]。随着LSD1组蛋白去甲基化功能的发现,组蛋白赖氨酸甲基化是动态变化的[2]。迄今为止,已发现超过20个组蛋白赖氨酸去甲基酶,其主要的靶位点包括H3K4、H3K9、H3K27、H3K36和H4K20[3]。基因敲除小鼠模型研究显示,适当的组蛋白甲基化对生殖细胞和胚胎发育至关重要[4-5]。敲除H3K9去甲基酶JMJD1C后,导致年龄依赖性不育,并伴随生殖细胞数量减少[6]。对KDM1A敲除小鼠研究发现,阻滞在第1次减数分裂前期的卵母细胞提前恢复减数分裂,并伴随纺锤体和染色体缺陷,导致卵母细胞凋亡[7]。
KDM2B(也称为FBXL10)是组蛋白赖氨酸去甲基化酶家族成员,含有JmjC结构域,特异催化H3K4me3、H3K36me1/2和H3K79me2/3去甲基化[8-10]。KDM2B作为PRC1的辅因子,对调节胚胎发育、形态发生、胚胎干细胞的分化等发挥关键作用。KDM2B通过CXXC结构域特异性结合未甲基化的CpG岛,进而参与调节核仁、干细胞的自我更新以及组蛋白H2A泛素化[11-13]。KDM2B与BCOR复合物共同抑制AP-2a基因表达从而调节间充质干细胞的分化[14]。Wang等[15]对KDM2B在软骨分化过程的研究结果表明,对KDM2B过表达后,将会抑制软骨分化标记物COL1、COL2和SOX9的表达,从而抑制间充质干细胞(MSC)介导的软骨再生。KDM2B与其他关键重编程因子协同作用激活早期基因组,并促进体细胞诱导产生多能干细胞[16]。Ozawa等[17]研究发现,KDM2B能调节小鼠精原细胞增殖,并确保其稳定的精子发生。最近研究发现,KDM2B通过调节EZH2的表达,抑制卵巢癌细胞的增殖和转移[18]。此外,Fukuda等[19]通过基因工程小鼠模型研究表明,KDM2B敲除的9.5 dpc胚胎出现细胞凋亡,出生后,幼鼠的存活率降低。综上表明,KDM家族与哺乳动物卵母细胞及胚胎发育息息相关,且KDM2B参与哺乳动物的繁殖生物学过程。
关于KDM2B的研究主要集中在调节细胞分化、癌症的发生等[20],而在卵母细胞及早期胚胎上尚未见相关报道。为进一步探索KDM2B在卵母细胞及胚胎发育过程中表达规律,本试验以小鼠着床前胚胎为研究对象,采用RT-qPCR检测KDM2B在小鼠早期胚胎发育过程中的表达情况,并通过免疫荧光染色分析KDM2B表达定位,旨在揭示KDM2B的时序表达规律,为进一步研究KDM2B在哺乳动物胚胎发育过程中所发挥的作用提供理论依据。
1 材料与方法 1.1 试验材料本研究的试验材料为6~8周龄性成熟昆明品系白鼠(n=20),购自成都达硕实验动物有限公司;雄性小鼠单笼饲养,雌性小鼠集中饲养,7:00-20:00光照,20:00-7:00黑暗,自由采食,温度控制在18~26 ℃。
1.2 主要试剂孕马血清(PMSG)和人绒毛膜促性腺激素(HCG)购自宁波第二激素厂;组蛋白特异性去甲基化酶KDM2B抗体购自北京博奥森生物技术有限公司;FITC-羊抗兔IgG购自博士德生物;Fluoroshield Mounting Medium With DAPI购自Abcam;微量RNA提取试剂盒(Single Cell-to-Ct TM Kit)购自Invitrogen;SYBR® Premix Ex TaqTM Ⅱ购自TaKaRa公司;polyvinyl pyrrolidone (PVP)购自Sigma;αMEM购自Gibco。
1.3 GV和MⅡ期小鼠卵母细胞的收集20~25 g雌性昆明小鼠,腹腔注射5 IU PMSG 48 h后,颈椎脱臼处死小鼠,75%酒精消毒,打开腹腔分离卵巢,置于M2操作液中,用1 mL注射器针头刺破卵巢表面的卵泡,体式显微镜下收集卵丘细胞3层及以上、胞质均匀的卵丘-卵母细胞复合体(COCs),脱颗粒细胞后,收集GV期卵母细胞;PMSG-HCG联合超数排卵16~18 h,颈椎脱臼处死小鼠,无菌条件下打开腹腔分离输卵管,置于M2操作液中,在体视显微镜下,左手用灭菌精细镊子固定,右手用1 mL注射器针头划开输卵管壶腹部,使COCs释放出来,脱颗粒细胞后,收集已排出第一极体的MⅡ期卵母细胞。
1.4 不同时期小鼠胚胎的收集选取6~8周龄雌鼠,下午16:00腹腔注射5 IU PMSG,48 h后注射5 IU HCG,然后与雄鼠1:1同笼,第2天上午7:00检查阴道栓情况,受孕雌鼠单独饲养并标记,在24、42、60 h后分离受孕小鼠输卵管,置于M2操作液中,体视显微镜下划开输卵管使胚胎流出,分别收集得到2-细胞、4-细胞、8-细胞胚胎;在3.5、4.5 dpc时分离子宫角与子宫体,1 mL注射器冲洗子宫及子宫角,收集囊胚。
1.5 小鼠卵母细胞和各时期胚胎总RNA的提取按照1.3、1.4中的方法收集GV期、MⅡ期卵母细胞各50个,2-细胞、4-细胞、8-细胞、囊胚各20个,严格按照微量RNA提取试剂盒(Single Cell-to-CtTM Kit)说明书分别提取卵母细胞及胚胎的RNA,并合成cDNA,-20 ℃保存备用。
1.6 KDM2B在小鼠卵母细胞及早期胚胎发育过程中的表达差异根据GenBank的小鼠KDM2B基因序列(NM_001003953.2)设计定量引物,以GAPDH为内参基因(表 1)。以1.5中的小鼠卵母细胞及胚胎的cDNA为模板,RT-qPCR反应体系:ddH2O 5.5 μL,SYBR®Premix Ex TaqTM Ⅱ 7.5 μL,上、下游引物各0.5 μL,模板cDNA 1 μL。反应程序:95 ℃ 4 min; (95 ℃ 10 s,60 ℃ 30 s,72 ℃ 45 s)40个循环;72 ℃延伸5 min。每个检测样本设3个重复。
|
|
表 1 RT-qPCR基因的引物信息 Table 1 The information of primers for RT-qPCR |
将一定数量的GV、MⅡ期卵母细胞和胚胎用4%多聚甲醛室温固定30 min,含0.1%PVP的αMEM清洗3次;转移至含1%Trtion-X100透膜液,37 ℃透膜15 min,在含0.1%PVP的αMEM中清洗3次后;含3%BSA的PBS中37 ℃封闭30 min;转移至3%BSA配制的一抗稀释液中(KDM2B:1:500),4 ℃孵育过夜;在0.1%PVP的αMEM中清洗3次,转移至FITC-羊抗兔IgG稀释液中(1:100),37 ℃孵育60 min,在含0.1%PVP的αMEM中清洗3次后,转移至干净的载玻片,滴加Fluoroshield Mounting Medium With DAPI封片,相同条件下进行激光共聚焦系统扫描。荧光照片利用Image-Pro Plus 6.0进行荧光平均相对密度分析。
1.8 数据分析每组试验至少重复3次,通过2-ΔΔCt法[21]计算RT-qPCR结果,数据用“平均值±标准误(mean ± SEM)”表示。所得数据通过SPSS 18.0的ANOVA进行显著性检验,P < 0.01表示差异极显著,P>0.05表示差异不显著。
2 结果 2.1 KDM2B在小鼠卵母细胞及着床前胚胎中的表达规律以GAPDH作为内参基因,采用RT-qPCR检测KDM2B在小鼠卵母细胞及早期胚胎发育过程中mRNA表达情况,结果显示,KDM2B mRNA在小鼠卵母细胞及各个时期胚胎中均能检测到表达(图 1),以GV期卵母细胞KDM2B mRNA表达量作为参照,在卵母细胞成熟过程中,KDM2B在GV期卵母细胞表达量最高,极显著高于MⅡ期卵母细胞(P < 0.01)。在早期胚胎发育过程中,KDM2B在2-~8-细胞胚胎中维持低丰度表达,且差异不显著(P>0.05),到囊胚期的表达水平升高,极显著高于其他各个时期胚胎(P < 0.01)。

|
不同大写字母表示组间差异极显著(P < 0.01),相同字母表示组间差异不显著(P>0.05),下同 The different capital letters mean extremely significant difference among groups(P < 0.01), the same letter means no significant difference among groups(P>0.05), the same as below 图 1 KDM2B在小鼠卵母细胞及早期胚胎的表达 Figure 1 The expression of KDM2B in mouse during oocyte and embryo development |
通过免疫荧光染色分析GV期、MⅡ期卵母细胞中KDM2B蛋白表达情况与定位(图 2),结果表明,在GV期卵母细胞中,KDM2B主要定位于卵母细胞细胞核,并均匀分布在整个染色质区域;在MⅡ期卵母细胞中,KDM2B主要在卵母细胞细胞核中表达。
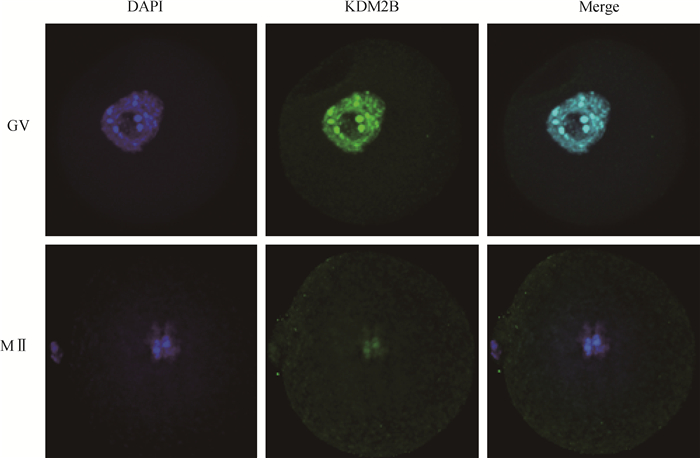
|
图 2 KDM2B蛋白在小鼠卵母细胞中的定位(100×) Figure 2 Localization of KDM2B protein in mouse oocytes(100×) |
根据免疫荧光染色分析,KDM2B在早期胚胎发育过程中的蛋白表达情况与定位如图 3、4所示,在小鼠2-细胞胚胎中,几乎检测不到KDM2B荧光信号; 在4-细胞、8-细胞胚胎KDM2B荧光信号强度低; 发育到囊胚后,KDM2B蛋白表达水平升高,并主要集中于内细胞团和滋养层细胞的细胞核;在相同曝光条件下,以GV期为对照,其相对平均光密度约是MⅡ期的3倍(P < 0.01);囊胚中KDM2B荧光信号极显著高于其他时期胚胎(P < 0.01)(图 4)。

|
图 3 KDM2B在小鼠着床前胚胎中的表达(100×) Figure 3 KDM2B expression in mouse preimplantation embryos (100×) |

|
图 4 KDM2B在小鼠卵母细胞和早期胚胎中的平均光密度值分析 Figure 4 Average optical density value analysis of KDM2B in mouse oocytes and embryos |
在卵母细胞减数分裂及胚胎发育过程中伴随表观遗传修饰的动态变化,其中组蛋白甲基化与去甲基化是重要的转录调控系统之一,参与调控卵母细胞减数分裂以及胚胎发育相关基因的表达[4]。研究表明,许多组蛋白去甲基化酶在胚胎发育和胚胎干细胞的分化方面发挥关键作用,KDM2B主要作用是调控CpG岛的甲基化状态,其缺失后,X染色体异常失活并发生特定性别相关基因表达异常,导致颅面骨畸形,神经管缺失,雌性胚胎致死率升高[22-23]。因此,KDM2B对调控卵母细胞及早期胚胎发育过程具有一定作用。
本研究利用qRT-PCR检测小鼠卵母细胞及早期胚胎中KDM2B的表达规律,结果显示,KDM2B mRNA在GV期、MⅡ期卵母细胞均表达,且在GV期卵母细胞中的表达量极显著高于MⅡ期卵母细胞(P < 0.01)。这与牛KDM2B的表达规律一致[24]。细胞周期蛋白依赖性激酶1(CDK1)是调节卵母细胞减数分裂进程的关键调节因子,研究发现,抑制CDK1的表达后,KDM2B表达上调,使多种细胞具有抗衰老性[17, 25]。GV期卵母细胞处于阻滞状态,CDK1沉默,KDM2B基因的表达量较高,CDK1激活后,GV期卵母细胞恢复减数分裂,导致KDM2B mRNA的表达量下降。因此,MⅡ期卵母细胞中KDM2B表达量降低。KDM2B在小鼠2-细胞、4-细胞、8-细胞胚胎中保持低丰度表达,在囊胚中KDM2B的表达量最高,极显著高于其他时期胚胎(P < 0.01)。在卵母细胞成熟过程中,积累了早期胚胎发育所需的母源RNA和蛋白质,因此早期胚胎发育过程中,转录处于沉默状态,这可能导致小鼠2-细胞、4-细胞、8-细胞胚胎中KDM2B表达量较低。小鼠的胚胎基因组激活发生在2-细胞阶段,虽然其调节机制尚不清楚,但研究表明,H3K4me3与激活基因表达密切相关[26],KDM2B作为H3K4me3去甲基化酶,因此也可能是2-细胞胚胎中KDM2B低表达的原因。Hosseini等[27]对牛囊胚的转录组测序显示,KDM2B在牛体外培养的囊胚中表达。同时在小鼠囊胚内细胞团的胚胎干细胞中KDM2B高丰度表达[28]。这与本研究结果基本一致。
本研究结果表明,KDM2B主要定位在GV期、MⅡ期卵母细胞细胞核,Endoh等[29]研究发现,KDM2B与靶基因结合募集PCR1/2,从而抑制生殖细胞和减数分裂相关基因的表达。卵母细胞减数分裂进程中,卵母细胞先后阻滞在GV期和MⅡ期,因此,KDM2B可能是卵母细胞阻滞的调节因子之一。Andricovich等[30]对10.5 dpc小鼠胚胎免疫荧光染色分析发现,主动脉内皮细胞、胚胎干细胞中KDM2B呈阳性。KDM2B在2-细胞、4-细胞、8-细胞胚胎中均未检测到表达,在囊胚细胞核中检测到KDM2B的表达。研究表明,H3K4甲基化异常将导致2-细胞胚胎发育停滞[31]。KDM2B是H3K4去甲基化酶之一,2-细胞胚胎中KDM2B表达抑制以维持正常的胚胎发育。在2-细胞和4-细胞胚胎透明带检测到绿色荧光,研究发现,在猪早期胚胎中H3K36me免疫荧光染色中,其透明带也存在绿色荧光[32]。这可能由于在孵育二抗后清洗不彻底,其次本研究中2-细胞和4-细胞胚胎是由核面积最大的照片叠加,因此导致了透明带荧光增强。在非洲爪蟾胚胎发生过程中,KDM2B转录起始于卵裂期胚胎,到囊胚期后,其转录水平达到最高[33]。在脊椎动物中,母源的Wnt/β-Catenin促进前背结构的规范,而合子的Wnt/β-catenin信号通路则决定胚层分化命运[34-35],当wnt信号通路激活后,KDM2B通过去甲基化作用调节核内β-catenin的稳定性从而调控胚胎发育。因此在囊胚细胞核中KDM2B荧光强度较高。综上表明,KDM2B对小鼠卵母细胞减数分裂及早期胚胎发育过程的调控发挥重要作用。
4 结论本研究分析了小鼠卵母细胞及着床前胚胎中KDM2B mRNA动态表达,并检测KDM2B蛋白表达情况与分布,结果表明,KDM2B mRNA在小鼠卵母细胞减数分裂及早期胚胎发育过程中均检测到表达,且KDM2B主要定位在卵母细胞、囊胚细胞核。提示KDM2B可能参与了卵母细胞及早期胚胎发育过程,其具体的作用机制还有待进一步研究证实。
| [1] | KOUZARIDES T. Chromatin modifications and their function[J]. Cell, 2007, 128(4): 693–705. DOI: 10.1016/j.cell.2007.02.005 |
| [2] | SHI Y J, LAN F, MATSON C, et al. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1[J]. Cell, 2004, 119(7): 941–953. DOI: 10.1016/j.cell.2004.12.012 |
| [3] | BYVOET P, SHEPHERD G R, HARDIN J M, et al. The distribution and turnover of labeled methyl groups in histone fractions of cultured mammalian cells[J]. Arch Biochem Biophys, 1972, 148(2): 558–567. |
| [4] | KOTA S K, FEIL R. Epigenetic transitions in germ cell development and meiosis[J]. Dev Cell, 2010, 19(5): 675–686. DOI: 10.1016/j.devcel.2010.10.009 |
| [5] | SAITOU M, KAGIWADA S, KURIMOTOK. Epigenetic reprogramming in mouse pre-implantation development and primordial germ cells[J]. Development, 2012, 139(1): 15–31. |
| [6] | KUROKI S, AKIYOSHI M, TOKURA M, et al. JMJD1C, a JmjC domain-containing protein, is required for long-term maintenance of male germ cells in mice[J]. Biol Reprod, 2013, 89(4): 93. |
| [7] | KIM J, SINGH A K, TAKATA Y, et al. LSD1 is essential for oocyte meiotic progression by regulating CDC25B expression in mice[J]. Nat Commun, 2015, 6: 10116. DOI: 10.1038/ncomms10116 |
| [8] | FRESCAS D, GUARDAVACCARO D, BASSERMANN F, et al. JHDM1B/FBXL10 is a nucleolar protein that represses transcription of ribosomal RNA genes[J]. Nature, 2007, 450(7167): 309–313. DOI: 10.1038/nature06255 |
| [9] | TSUKADA Y, FANG J, ERDJUMENT-BROMAGE H, et al. Histone demethylation by a family of JmjC domain-containing proteins[J]. Nature, 2006, 439(7078): 811–816. DOI: 10.1038/nature04433 |
| [10] | SEO S B, KANG J Y, KIM J Y, et al. KDM2B is a histone H3K79 demethylase and induces transcriptional repression via SIRT1-mediated chromatin silencing[J]. BioRxiv, 2017. DOI: 10.1101/228379 |
| [11] | WU X D, JOHANSEN J V, HELIN K. Fbxl10/Kdm2b recruits polycomb repressive complex 1 to CpG Islands and regulates H2A ubiquitylation[J]. Mol Cell, 2014, 49(6): 1134–1146. |
| [12] | KOTTAKIS F, FOLTOPOULOU P, SANIDAS I, et al. NDY1/KDM2B functions as a master regulator of polycomb complexes and controls self-renewal of breast cancer stem cells[J]. Cancer Res, 2014, 74(14): 3935–3946. DOI: 10.1158/0008-5472.CAN-13-2733 |
| [13] | BLACKLEDGE N P, FARCAS A M, KONDO T, et al. Variant PRC1 complex-dependent H2A ubiquitylation drives PRC2 recruitment and polycomb domain formation[J]. Cell, 2014, 157(6): 1445–1459. DOI: 10.1016/j.cell.2014.05.004 |
| [14] | DU J, MA Y S, MA P, et al. Demethylation of epiregulin gene by histone demethylase FBXL11 and BCL6 corepressor inhibits osteo/dentinogenic differentiation[J]. Stem Cells, 2013, 31(1): 126–136. DOI: 10.1002/stem.v31.1 |
| [15] | WANG J J, DONG R, WANG L P, et al. Histone demethylase KDM2B inhibits the chondrogenic differentiation potentials of stem cells from apical papilla[J]. Int J Clin Exp Med, 2015, 8(2): 2165–2173. |
| [16] | LIANG G Y, HE J, ZHANG Y. KDM2B promotes induced pluripotent stem cell generation by facilitating gene activation early in reprogramming[J]. Nat Cell Biol, 2012, 14(5): 457–466. DOI: 10.1038/ncb2483 |
| [17] | OZAWA M, FUKUDA T, SAKAMOTO R, et al. The histone demethylase FBXL10 regulates the proliferation of spermatogonia and ensures long-term sustainable spermatogenesis in mice[J]. Biol Reprod, 2016, 94(4): 92. |
| [18] | KUANG Y, LU F, GUO J, et al. Histone demethylase KDM2B upregulates histone methyltransferase EZH2 expression and contributes to the progression of ovarian cancer in vitro and in vivo[J]. Onco Targets Ther, 2017, 10: 3131–3144. DOI: 10.2147/OTT |
| [19] | FUKUDA T, TOKUNAGA A, SAKAMOTO R, et al. Fbxl10/Kdm2b deficiency accelerates neural progenitor cell death and leads to exencephaly[J]. Mol Cell Neurosci, 2011, 46(3): 614–624. |
| [20] | SUZUKI T, MINEHATA K I, AKAGI K, et al. Tumor suppressor gene identification using retroviral insertional mutagenesis in Blm-deficient mice[J]. EMBO J, 2006, 25(14): 3422–3431. DOI: 10.1038/sj.emboj.7601215 |
| [21] | LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method[J]. Methods, 2001, 25(4): 402–408. DOI: 10.1006/meth.2001.1262 |
| [22] | BOULARD M, EDWARDS J R, BESTOR T H. FBXL10 protects polycomb-bound genes from hypermethylation[J]. Nat Genet, 2015, 47(5): 479–485. DOI: 10.1038/ng.3272 |
| [23] | BOULARD M, EDWARDS J R, BESTOR T H. Abnormal X chromosome inactivation and sex-specific gene dysregulation after ablation of FBXL10[J]. Epigenetics Chromatin, 2016, 9: 22. DOI: 10.1186/s13072-016-0069-1 |
| [24] |
王帅.牛HDM家族成员在卵母细胞中的表达规律及JMJD1C在早期胚胎发育中的功能[D].长春: 吉林大学, 2014: 1-52.
WANG S.Expression pattern of HDM family members in bovine oocytes and function of JMJD1C in early embryo development[D].Changchun: Jilin University, 2014: 1-52.(in chinese) http://cdmd.cnki.com.cn/Article/CDMD-10183-1014281819.htm |
| [25] | SOLC P, SASKOVA A, BARAN V, et al. CDC25A phosphatase controls meiosis Ⅰ progression in mouse oocytes[J]. Dev Biol, 2008, 317(1): 260–269. DOI: 10.1016/j.ydbio.2008.02.028 |
| [26] | SCHNEIDER R, BANNISTER A J, MYERS F A, et al. Histone H3 lysine 4 methylation patterns in higher eukaryotic genes[J]. Nat Cell Biol, 2004, 6(1): 73–77. DOI: 10.1038/ncb1076 |
| [27] | HOSSEINI S M, DUFORT I, NIEMINEN J, et al. Epigenetic modification with trichostatin A does not correct specific errors of somatic cell nuclear transfer at the transcriptomic level; highlighting the non-random nature of oocyte-mediated reprogramming errors[J]. BMC Genomics, 2016, 17: 16. DOI: 10.1186/s12864-015-2264-z |
| [28] | HE J, SHEN L, WAN M, et al. Kdm2b maintains murine embryonic stem cell status by recruiting PRC1 complex to CpG islands of developmental genes[J]. Nat Cell Biol, 2013, 15(4): 373–384. DOI: 10.1038/ncb2702 |
| [29] | ENDOH M, ENDO T A, SHINGA J, et al. PCGF6-PRC1 suppresses premature differentiation of mouse embryonic stem cells by regulating germ cell-related genes[J]. Elife, 2017, 6: e21064. DOI: 10.7554/eLife.21064 |
| [30] | ANDRICOVICH J, KAI Y, PENG W Q, et al. Histone demethylase KDM2B regulates lineage commitment in normal and malignant hematopoiesis[J]. J Clin Invest, 2016, 126(3): 905–920. DOI: 10.1172/JCI84014 |
| [31] | SHAO G B, DING H M, GONG A H. Role of histone methylation in zygotic genome activation in the preimplantation mouse embryo[J]. In vitro Cell Dev Biol Anim, 2008, 44(3-4): 115–120. DOI: 10.1007/s11626-008-9082-4 |
| [32] | DIAO Y F, OQANI R K, LI X X, et al. Changes in histone H3 lysine 36 methylation in porcine oocytes and preimplantation embryos[J]. PLoS One, 2014, 9(6): e100205. DOI: 10.1371/journal.pone.0100205 |
| [33] | LU L, GAO Y, ZHANG Z, et al. Kdm2a/b lysine demethylases regulate canonical wnt signaling by modulating the stability of nuclear β-catenin[J]. Dev Cell, 2015, 33(6): 660–674. DOI: 10.1016/j.devcel.2015.04.006 |
| [34] | NOJIMA H, SHIMIZU T, KIM C H, et al. Genetic evidence for involvement of maternally derived Wnt canonical signaling in dorsal determination in zebrafish[J]. Mech Dev, 2004, 121(4): 371–386. DOI: 10.1016/j.mod.2004.02.003 |
| [35] | HIKASA H, SOKOL S Y. Wnt signaling in vertebrate axis specification[J]. Cold Spring Harb Perspect Biol, 2013, 5(1): a007955. DOI: 10.1101/cshperspect.a007955 |



