2. 山东农业大学动物科技学院, 泰安 271018;
3. 山东凤祥股份有限公司, 阳谷 252300
2. College of Animal Science, Shandong Agricultural University, Tai'an 271018, China;
3. Shandong Feng Xiang Co. LTD, Yanggu 252300, China
禽流感病毒(avian influenza virus,AIV)可导致家禽和野鸟的禽流感[1-3]。据病毒两种表面糖蛋白——血凝素(hemaglutinin,HA)和神经氨酸酶(neuraminidase, NA)抗原性的不同,将禽源AIVs划分为16种HA亚型和9种NA亚型。已知的HA和NA亚型均可在野鸟中分离到,因此普遍认为野鸟是AIVs的天然宿主[4-5]。AIVs基因组由8个独立的基因片段(PB2、PB1、PA、HA、NP、NA、M和NS)组成,病毒主要通过点突变和基因重组的方式发生变异,导致抗原漂移和抗原转换[6-8]。
我国于1994年首次在广东发病鸡群中分离到H9N2亚型AIV[9]。该亚型AIV致病性低,早期感染易被忽视,在鸡群中持续流行和传播,成为国内家禽的主要AIV流行亚型。该亚型病毒感染可引起鸡呼吸道炎症、免疫力下降和生产性能降低。感染鸡群易并发或继发其他疾病(如鸡传染性支气管炎和慢性呼吸道病),导致发病率和死亡率大幅增加。国内预防家禽H9N2亚型禽流感的主要手段是疫苗免疫。血凝素蛋白是AIV重要的免疫保护性抗原,在疫苗免疫后鸡体能够产生相应抗体,因此生产中普遍通过血凝抑制试验(hemagglutination inhibitation test,HI)检测鸡血清抗体水平,评价疫苗免疫效果。
系统进化分析发现,我国H9N2亚型AIV各基因隶属于多个亚系,包括A/chicken/Shanghai/F/98-like亚系、A/Quail/HongKong/G1/97-like亚系、A/Duck/HongKong/Y280/97-like亚系和A/duck/HongKong/Y439/97-like亚系(表 1,B~E)[10-11]。病毒8个基因片段重排频繁,导致病毒基因型众多[10, 12]。但是,自2010年以来G57基因型成为主要流行基因型。该基因型毒株最早于2007年分离自江苏和江西的鸡群,2009年起迅速传播开来[13]。本研究对2017年新分离自山东的6株鸡源H9N2亚型AIVs进行了系统进化、分子特征和抗原性差异分析,旨在明确病毒的系统进化状态和抗原变异情况。
|
|
表 1 本研究中的H9N2禽流感病毒分离株和经典参考毒株 Table 1 H9N2 AIV isolates and classic reference viruses in this study |
6株H9N2亚型AIV毒株(表 1)由本课题组分离、鉴定和保存。病毒于2017年2-4月分离自山东省聊城市一个发病肉鸡场,分离方法如下。病料采集后经研磨、离心、滤膜过滤和双抗处理后接种9~11日龄的SPF鸡胚。鸡胚培养96 h后收获尿囊液,HA和HI试验验证病毒为H9亚型AIVs,扩增HA和NA基因并测序,鉴定为H9N2亚型。将病毒保存于-80 ℃冰柜备用。血凝抑制试验用H9抗原来自疫苗生产用灭活抗原,由某疫苗公司提供;9~11日龄SPF鸡胚,购自山东省农业科学院家禽研究所SPF鸡研究中心。
1.2 主要试剂Trizol为Invitrogen公司产品;PrimeScript One Step RT-PCR Kit为TaKaRa公司产品;PCR产物纯化试剂盒、胶回收试剂盒和质粒提取试剂盒购自Qiagen公司;pUC57载体、DH5α感受态细胞购自上海生物工程有限公司(Sangon);IPTG (异丙基硫代-β-半乳糖苷)、X-gal (5-嗅-4-氯-3-吲哚-β-D-半乳糖苷)和氨苄青霉素购自宝生物工程(大连)有限公司;胰蛋白胨、酵母提取物和琼脂粉购自OXOID公司;其他试剂均为国产分析纯。
1.3 RNA提取和RT-PCR应用Trizol法提取含毒尿囊液的总RNA,溶解于20 μL的TE缓冲液中。以RNA为模板,分别使用病毒8个基因片段的特异引物[14]进行RT-PCR。采用25 μL反应体系:RNase Free dH2O 5.5 μL,10×Buffer 12.5 μL,Prime Script RTase 0.5 μL,Forward Primer 0.75 μL,Reverse Primer 0.75 μL RNA样品5 μL。循环参数:50 ℃反转录30 min;94 ℃预变性5 min;94 ℃ 45 s,53 ℃ 45 s,72 ℃ 1.5 min扩增35个循环;最后72 ℃延伸10 min结束反应。
1.4 扩增产物的纯化、克隆和测序取3 μL扩增产物进行1%琼脂糖凝胶电泳,若电泳影像为单一目的大小的条带,则采用PCR产物纯化试剂盒回收和纯化扩增产物;若电泳影像为包含目的片段的非单一条带,则凝胶回收目的条带,使用凝胶回收试剂盒纯化目的基因。将纯化的基因与pUC57载体连接,转化大肠杆菌DH5α感受态细胞,进行细菌培养并提取质粒。通过菌液PCR方法初步鉴定阳性质粒,每个基因取3个阳性质粒做双向测序(上海华大基因有限公司)。
1.5 序列测定及分析基因测序结果使用DNAStar的Lasergene v7.1软件包进行序列的翻译(Editseq软件)、编辑(Seqman软件)、比对以及关键氨基酸位点变异分析(MegAlign软件)。通过NCBI Blastn搜索和下载各病毒基因的最相似序列(10~20条/基因),与国内H9N2亚型AIV各进化分支的经典参考序列一起,使用MEGA7.0软件分别以Neighbor-joining(NJ)法和Maximum-likelihood法绘制各基因的系统进化树。系统进化分析所使用的基因ORF区域分别如下:PB2 1 418—2 272 bp、PB1 1 315—2 208 bp、PA 112—1 209 bp、HA 52—880 bp、NP 89—1 453 bp、NA 205—1 347 bp、M 68—784 bp以及NS 120—827 bp。
1.6 交叉血凝抑制试验检测HA抗原性差异分别使用本研究中的病毒分离株SD210、SD217和SD227抗原以及该养殖场使用的H9N2亚型疫苗株抗原制备4单位病毒,进行交叉血凝抑制试验(HI),检测和比较不同抗原与疫苗免疫鸡血清(1 000份)的反应性。
2 结果 2.1 H9N2亚型AIV分离株的基因同源性分析本研究获得了6株H9N2亚型AIV的基因序列信息(GenBank登录号见表 1)。表 1列出了6株病毒(A1~6)及其基因的GenBank登陆号,以及系统进化分析使用的经典参考毒株B~E。G57代表一株隶属于G57基因型的H9N2参考毒株。
Pairwise-distance分析表明,6株分离毒株各基因的相似性在93.4%~100%(表 2)。
|
|
表 2 H9N2亚型禽流感病毒分离株基因型和基因相似性分析结果 Table 2 The result of genotype and genic homogenous analyses of H9N2-subtype AIV isolates |
表 2显示了6株病毒分离株的基因型分类,分离株间的基因同源性(A)以及分离株和经典参考毒株(B~E和G57)的基因相似性分析结果。其中,A为6株分离株,B为CK SH F 98,C为QL HK G1 97,D为DK HK Y439 97,E为DK HK Y280 97,G57表示一株G57基因型参考株,“-”表示该基因类型来源不明。
表 3列出了与H9N2分离株各基因相似性最高的部分参考毒株的基因信息。其中,病毒分离株1~6分别为CK SD 210WZ 17、CK SD 217YY 17、CK SD 227AC 17、CK SD 306SZ 17、CK SD 321ZL 17和CK SD 413ZDM 17。NCBI Blastn分析表明,与各分离株基因同源性最高的参考序列主要来自2014年以来的病毒,基因相似性均在98%以上(表 3),其中,分离株HA和PA基因与A/duck/Japan/AQ-HE5/2015 (H9N2)相应基因的相似性最高,分离株PB2、PB1、NA、M和NS基因分别与2016年来自江西赣州的几株病毒相应基因的同源性最高(表 3)。这些参考株分离自包括山东、河北、上海、江苏、浙江、江西、湖南、广东和日本等在内的广泛地理区域。
|
|
表 3 与本研究中分离株的基因同源性最高的参考毒株信息 Table 3 Reference viruses with highest genic identities to the H9N2 genes in this study |
系统进化分析表明(图 1),NJ法和Maximum-likelihood法构建的进化树其拓扑结构相同,分离株8个基因片段均与G57基因型参考株处于同一进化亚系。结合系统进化分析(图 1)和基因同源性分析(表 2)的结果,以同一亚系内基因相似性≥90%为划分依据,发现病毒分离株的PB1、PA、NP和NS基因隶属于CK SH F 98亚系,M基因属于QL HK G1 97亚系,HA和NA基因可勉强划分到DK HK Y280 97亚系(相似性处于90%临界点附近),而分离株PB2基因隶属于一个与上述4个亚系平行进化的亚系(表 2)。
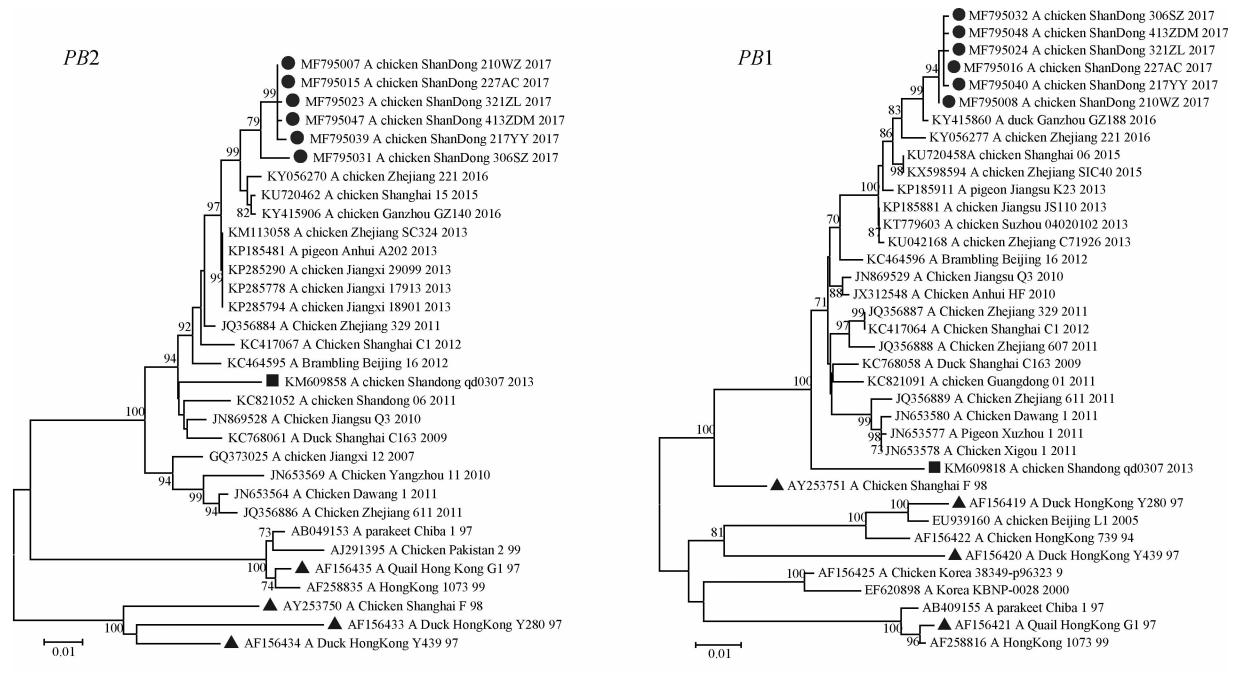
|
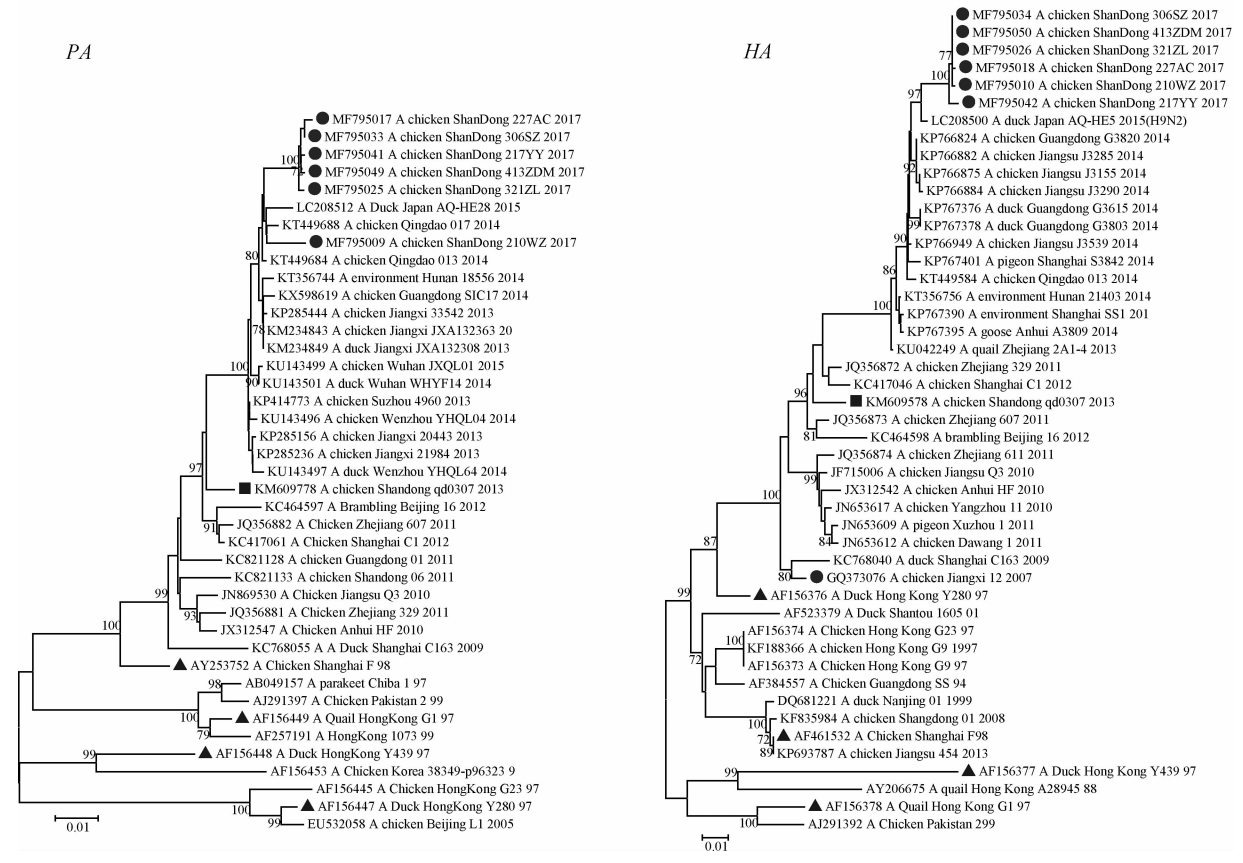
|
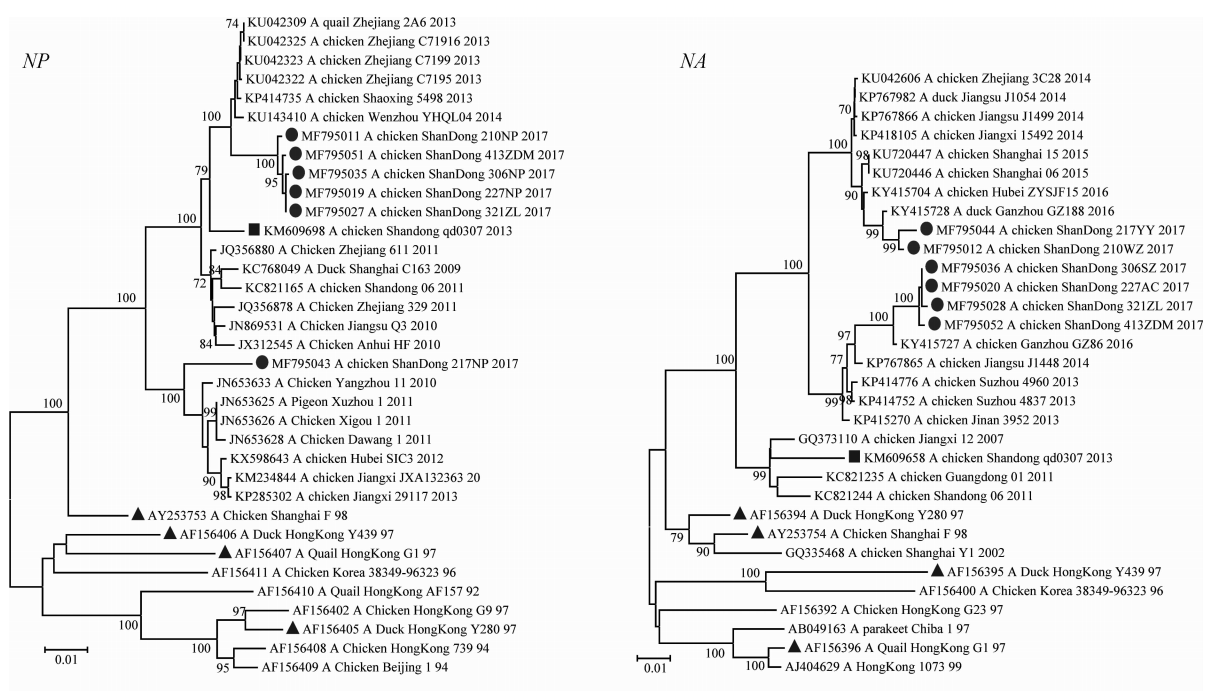
|
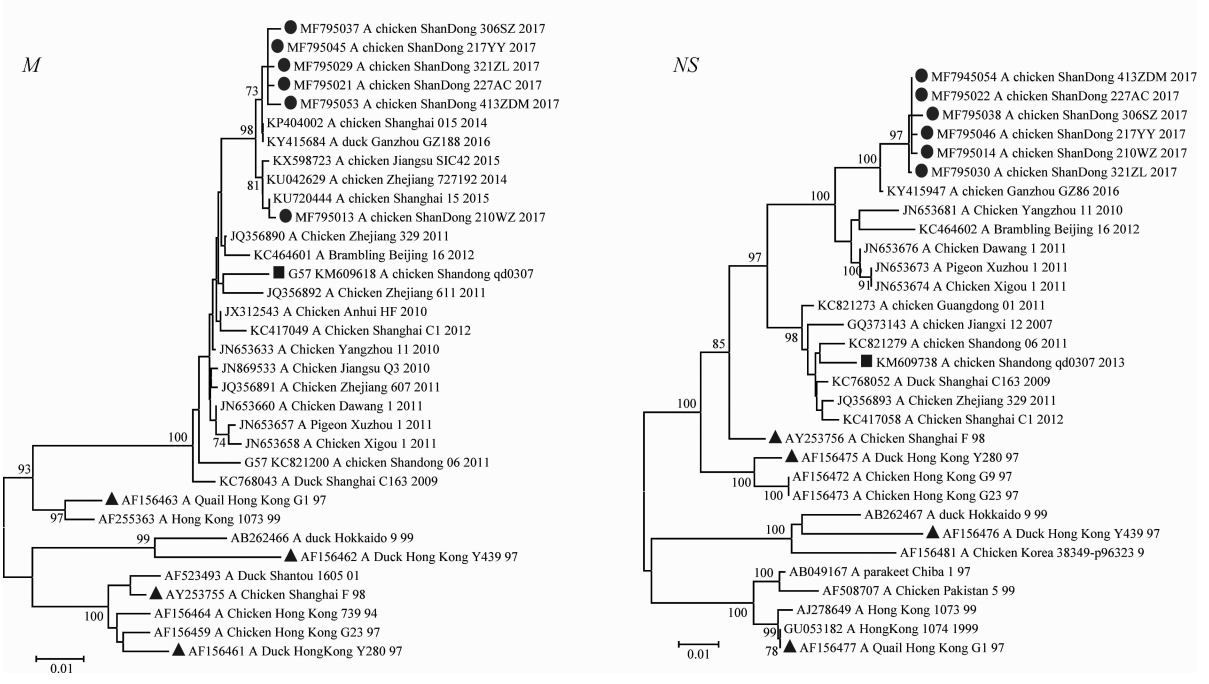
|
显示了H9N2亚型AIVs的PB2、PB1、PA、HA、NP、NA、M和NS基因系统进化树(NJ法)。其中,本研究中的6株病毒分离株以圆点表示,4个经典参考毒株(CK SH F 98、QL HK G1 97、DK HK Y439 97和DK HK Y280 97)以三角形表示,G57亚型参考株以矩形表示 Fig. 1 showed the phylogenetic trees of the 8 AIV genes (PB2, PB1, PA, HA, NP, NA, M and NS). The 6 viruses labeled in circle were the isolates in this study. The 4 viruses labeled in triangle were classical reference viruses (CK SH F 98, QL HK G1 97, DK HK Y439 97 and DK HK Y280 97). The virus labeled in rectangular was a G57-genotype reference virus 图 1 H9N2亚型AIV各基因系统进化树 Figure 1 The phylogenetic trees of genes of H9N2 avian influenza viruses in this study |
对分离株各基因的编码氨基酸进行预测分析表明,各基因编码蛋白的长度与以往报道的H9N2病毒分离株一致。PB2、PB1、PA、NP、HA和NA蛋白的长度分别为759、757、716、560、498和466个氨基酸,基质蛋白M1和离子通道蛋白M2的长度分别为252和97个氨基酸,而非结构蛋白NS1和NS2蛋白分别为217和121个氨基酸。HA蛋白关键氨基酸位点分析表明,6株病毒HA基因裂解位点的氨基酸残基均为RSSR↓GLF,符合低致病性AIV的分子特征(表 4)。构成HA上唾液酸受体结合位点的氨基酸以及HA潜在糖基化位点的氨基酸高度保守,其中CK SD 227AC 17和CK SD 306SZ 17的HA有6个潜在的糖基化位点(NX-T/S,X为除P以外的氨基酸), 而其余4个分离株有5个糖基化位点(表 4)。
|
|
表 4 H9N2毒株HA蛋白关键氨基酸位点分析 Table 4 Analysis of key sites of amino acid residues of HA protein (H9N2 isolates) |
与AIV抗药性相关的蛋白位点分析表明,本研究中的6株H9N2分离株M2蛋白的第26—34位氨基酸残基序列为LVVAANIIG,均存在S31N变异,表明病毒均对离子通道蛋白抑制剂耐药[15];分离株NA蛋白不存在R292K变异,表明病毒对神经氨酸酶抑制剂敏感[16]。此外,分离病毒其他蛋白未见有对哺乳动物模型致病性增强的单个氨基酸变异,如PB2蛋白的E627K变异和D701N变异、PB1-F2蛋白的N66S变异或NS1蛋白的D92E变异[17-20]。
2.4 AIV分离毒株的HA抗原性差异分析对1 000份免疫鸡血清进行交叉血凝抑制试验(HI)检测的结果表明,以分离株SD210为抗原比疫苗株抗原测得的免疫鸡血清HI效价平均低2 log2;而以分离株SD217和SD227为抗原比疫苗株抗原测得的血清HI效价平均低1~2 log2。表 5显示了其中20份血清的HI检测结果。
|
|
表 5 分别以H9N2亚型病毒分离株和疫苗株为抗原的血凝抑制试验结果 Table 5 Results of HI tests with H9N2 isolates or the vaccine virus as antigens |
本研究对2017年上半年自山东省肉鸡场分离到的6株H9N2亚型AIVs进行了基因组序列测定、进化分析和HA抗原性差异分析,为H9N2亚型AIVs的分子演化和抗原变异提供最新的流行病学资料。从病毒分离时间上来看,与分离毒株基因相似性最高(≥98%)的参考基因大多来自2014—2016年。相比之下,分离毒株与20世纪末分离的系统进化经典参考毒株相似性较低,其中PB2基因与4株参考株基因相似性均已低于90%,HA和NA基因与经典株基因相似性最高者也仅90%左右(表 2)。表明国内H9N2病毒流行株不断累积基因变异,与经典株的基因分化不断加剧。另外,从病毒基因型上来看,国内H9N2亚型禽流感病毒基因型自2009年起多样性骤减,G57成为绝对优势流行基因型[13]。本研究中的H9N2分离株在系统进化分类上同属于G57基因型,表明该基因型病毒继续在山东省肉鸡群中流行。
AIV的HA蛋白在合成之初以多肽HA0的形式存在,然后在适当的蛋白水解酶的作用下裂解为HA1和HA2。含单碱性氨基酸裂解位点基序的HA蛋白仅能被特异性蛋白酶(如胰酶)所裂解,因这类酶在鸟类的分布局限在呼吸道和肠道,病毒感染后仅产生温和或无症状感染;而另一方面,裂解位点为多碱性氨基酸的HA蛋白可被鸟体内广泛分布的泛素所裂解,从而导致病毒的广泛复制和多器官感染[21-22]。本研究中分离株HA蛋白裂解位点基序的单碱基氨基酸表明,各病毒仍属于低致病性范畴。
目前商品化的流感病毒药物包括M2抑制剂(金刚烷胺和金刚乙胺)和NA抑制剂(奥司他韦和扎那米韦)[23]。M2或NA蛋白单个氨基酸位点的变异即可导致流感病毒的耐药性。M2蛋白的L26I、V27I、A30P、S31N或G34E变异可导致AIV对金刚烷胺耐药性[24];NA蛋白R292K(以N2序列计数)变异可导致N1、N2和N9亚型AIV对NA抑制剂的耐药性[16, 25-26]。与耐药性相关的AIV氨基酸位点分析表明,本研究中的6株H9N2分离株对离子通道蛋白抑制剂耐受,而对神经氨酸酶(NA)抑制剂敏感。
反向遗传学研究表明,PB2蛋白的E627K突变可增强H5N1亚型AIV对小鼠的系统性感染,导致小鼠T细胞活化受损[27-28];PB2蛋白的D701N突变是促进H5N1亚型AIVs在豚鼠间传播的必要条件[29];NS1蛋白的D92E突变可增强H5N1亚型AIVs对猪的致病性[19]。此外,PB1-F2蛋白的N66S突变导致了H5N1/1997和H1N1/1918流感病毒毒株对人致病性的增强[30]。本研究中的6株分离株内部蛋白均不存在上述氨基酸变异。
交叉血凝抑制试验的结果表明,本研究中的H9N2分离株HA蛋白抗原与现有HI用标准抗原略有差异。在养禽生产中,一般将交叉HI效价相差3 log2作为流行株抗原与疫苗株抗原有显著差异的标准,据此分析,本研究中的分离株与疫苗株抗原性差异尚不显著。后续研究中我们将继续对山东省H9N2亚型AIV分子演化和抗原变异进行持续监测,以积累病毒感染、传播和进化的数据,促进AIV的有效防控。
4 结论2017年在山东省聊城市该肉鸡场流行的6株H9N2亚型禽流感病毒仍然为G57基因型,病毒对离子通道蛋白抑制剂耐药,病毒分离株的HA抗原反应性与疫苗株差异不显著。
| [1] | BOYCE W M, SANDROCK C, KREUDER-JOHNSON C, et al. Avian influenza viruses in wild birds:a moving target[J]. Comp Immunol Microbiol Infect Dis, 2009, 32(4): 275–286. DOI: 10.1016/j.cimid.2008.01.002 |
| [2] | ALEXANDER D J. A review of avian influenza in different bird species[J]. Vet Microbiol, 2000, 74(1-2): 3–13. DOI: 10.1016/S0378-1135(00)00160-7 |
| [3] | ALEXANDER D J, ALLAN W H, PARSONS D, et al. The pathogenicity of four avian influenza viruses for fowls, turkeys and ducks[J]. Res Vet Sci, 1978, 24(2): 242–247. |
| [4] | CLARK L, HALL J. Avian influenza in wild birds:status as reservoirs, and risks to humans and agriculture[J]. Ornithol Monogr, 2006, 60: 3–29. DOI: 10.1642/0078-6594(2006)60[3:AIIWBS]2.0.CO;2 |
| [5] | CHEN R B, HOLMES E C. Frequent inter-species transmission and geographic subdivision in avian influenza viruses from wild birds[J]. Virology, 2009, 383(1): 156–161. DOI: 10.1016/j.virol.2008.10.015 |
| [6] | CHEN R B, HOLMES E C. Avian influenza virus exhibits rapid evolutionary dynamics[J]. Mol Biol Evol, 2006, 23(12): 2336–2341. DOI: 10.1093/molbev/msl102 |
| [7] | BANKS J, SPEIDEL E S, MOORE E, et al. Changes in the haemagglutinin and the neuraminidase genes prior to the emergence of highly pathogenic H7N1 avian influenza viruses in Italy[J]. Arch Virol, 2001, 146(5): 963–973. DOI: 10.1007/s007050170128 |
| [8] | ZHANG L S, ZHANG Z G, WENG Z P. Rapid reassortment of internal genes in avian influenza A(H7N9) virus[J]. Clin Infect Dis, 2013, 57(7): 1059–1061. DOI: 10.1093/cid/cit414 |
| [9] | GUO X, LIAO M, XIN C. Sequence of HA gene of avian influenza A/Chicken/Guangdong/SS/1994(H9N2) virus[J]. Avian Dis, 2003, 47(S3): 1118–1121. DOI: 10.1637/0005-2086-47.s3.1118 |
| [10] | HUANG Y Y, HU B X, WEN X T, et al. Diversified reassortant H9N2 avian influenza viruses in chicken flocks in northern and eastern China[J]. Virus Res, 2010, 151(1): 26–32. DOI: 10.1016/j.virusres.2010.03.010 |
| [11] | LIU H Q, LIU X F, CHENG J, et al. Phylogenetic analysis of the hemagglutinin genes of twenty-six avian influenza viruses of subtype H9N2 isolated from chickens in China during 1996-2001[J]. Avian Dis, 2003, 47(1): 116–127. DOI: 10.1637/0005-2086(2003)047[0116:PAOTHG]2.0.CO;2 |
| [12] | SUN Y P, PU J, JIANG Z L, et al. Genotypic evolution and antigenic drift of H9N2 influenza viruses in China from 1994 to 2008[J]. Vet Microbiol, 2010, 146(3-4): 215–225. DOI: 10.1016/j.vetmic.2010.05.010 |
| [13] | PU J, WANG S G, YIN Y B, et al. Evolution of the H9N2 influenza genotype that facilitated the genesis of the novel H7N9 virus[J]. Proc Natl Acad Sci U S A, 2015, 112(2): 548–553. DOI: 10.1073/pnas.1422456112 |
| [14] |
葛菲菲, 刘健, 鞠厚斌, 等. 2007-2008年上海市H9N2亚型禽流感病毒的遗传演化[J]. 中国人兽共患病学报, 2011, 27(8): 696–699.
GE F F, LIU J, JU H B, et al. Phylogenetic analysis of H9N2 subtype avian influenza virus isolates in Shanghai area from 2007 to 2008[J]. Chinese Journal of Zoonoses, 2011, 27(8): 696–699. DOI: 10.3969/j.issn.1002-2694.2011.08.005 (in Chinese) |
| [15] | WANG J, WU Y B, MA C L, et al. Structure and inhibition of the drug-resistant S31N mutant of the M2 ion channel of influenza A virus[J]. Proc Natl Acad Sci U S A, 2013, 110(4): 1315–1320. DOI: 10.1073/pnas.1216526110 |
| [16] | YEN H L, MCKIMM-BRESCHKIN J L, CHOY K T, et al. Resistance to neuraminidase inhibitors conferred by an R292K mutation in a human influenza virus H7N9 isolate can be masked by a mixed R/K viral population[J]. mBio, 2013, 4(4): e00396–13. |
| [17] | SUBBARAO E K, LONDON W, MURPHY B R. A single amino acid in the PB2 gene of influenza A virus is a determinant of host range[J]. J Virol, 1993, 67(4): 1761–1764. |
| [18] | CONENELLO G M, TISONCIK J R, ROSENZWEIG E, et al. A single N66S mutation in the PB1-F2 protein of influenza A virus increases virulence by inhibiting the early interferon response in vivo[J]. J Virol, 2011, 85(2): 652–662. DOI: 10.1128/JVI.01987-10 |
| [19] | SEO S H, HOFFMANN E, WEBSTER R G. Lethal H5N1 influenza viruses escape host anti-viral cytokine responses[J]. Nat Med, 2002, 8(9): 950–954. DOI: 10.1038/nm757 |
| [20] | JAGGER B W, MEMOLI M J, SHENG Z M, et al. The PB2-E627K mutation attenuates viruses containing the 2009 H1N1 influenza pandemic polymerase[J]. mBio, 2010, 1(1): e00067–10. |
| [21] | SUAREZ D L, SCHULTZ-CHERRY S. Immunology of avian influenza virus:a review[J]. Dev Comp Immunol, 2000, 24(2-3): 269–283. DOI: 10.1016/S0145-305X(99)00078-6 |
| [22] | LEBARBENCHON C, STALLKNECHT D E. Host shifts and molecular evolution of H7 avian influenza virus hemagglutinin[J]. Virol J, 2011, 8: 328. DOI: 10.1186/1743-422X-8-328 |
| [23] | BEIGEL J, BRAY M. Current and future antiviral therapy of severe seasonal and avian influenza[J]. Antiviral Res, 2008, 78(1): 91–102. DOI: 10.1016/j.antiviral.2008.01.003 |
| [24] | DEYDE V M, NGUYEN T, BRIGHT R A, et al. Detection of molecular markers of antiviral resistance in influenza A (H5N1) viruses using a pyrosequencing method[J]. Antimicrob Agents Chemother, 2009, 53(3): 1039–1047. DOI: 10.1128/AAC.01446-08 |
| [25] | MCKIMM-BRESCHKIN J L, SAHASRABUDHE A, BLICK T J, et al. Mutations in a conserved residue in the influenza virus neuraminidase active site decreases sensitivity to Neu5Ac2en-derived inhibitors[J]. J Virol, 1998, 72(3): 2456–2462. |
| [26] | KISO M, OZAWA M, LE M T Q, et al. Effect of an asparagine-to-serine mutation at position 294 in neuraminidase on the pathogenicity of highly pathogenic H5N1 influenza A virus[J]. J Virol, 2011, 85(10): 4667–4672. DOI: 10.1128/JVI.00047-11 |
| [27] | HATTA M, GAO P, HALFMANN P, et al. Molecular basis for high virulence of Hong Kong H5N1 influenza A viruses[J]. Science, 2001, 293(5536): 1840–1842. DOI: 10.1126/science.1062882 |
| [28] | FORNEK J L, GILLIM-ROSS L, SANTOS C, et al. A single-amino-acid substitution in a polymerase protein of an H5N1 influenza virus is associated with systemic infection and impaired T-cell activation in mice[J]. J Virol, 2009, 83(21): 11102–11115. DOI: 10.1128/JVI.00994-09 |
| [29] | LI Z J, CHEN H L, JIAO P R, et al. Molecular basis of replication of duck H5N1 influenza viruses in a mammalian mouse model[J]. J Virol, 2005, 79(18): 12058–12064. DOI: 10.1128/JVI.79.18.12058-12064.2005 |
| [30] | CONENELLO G M, ZAMARIN D, PERRONE L A, et al. A single mutation in the PB1-F2 of H5N1(HK/97) and 1918 influenza A viruses contributes to increased virulence[J]. PLoS Pathog, 2007, 3(10): 1414–1421. |



