近年来随着传统化石燃料的临近枯竭, 加之环境问题日益突出, 致使生物能源越来越得到广泛的关注[1], 其中, 以微藻产油为代表的微藻生物燃料的生产, 尤其受到各国科学家的重视[1-2]。微藻生物燃料含油量高、生物量高、材料易培养和采收、不占用耕作用地等优点, 因此利用现代生物技术强化微藻脂质代谢途径、增加微藻的含油量、增加富油微藻生物量的累积, 来提高微藻产油的竞争力成为时下的热点[3-4]。而且以微藻为原料生产生物燃料有利于减少78%(体积分数)二氧化碳的排放, 98%(体积分数)硫排放以及50%(体积分数)燃烧后的颗粒物的排放[5]。其中莱茵衣藻(Chlamydomonas reinhardtii)是遗传背景清晰的真核单细胞藻类, 可以在平板上单克隆培养, 也易液体培养, 生长快、世代时间短、适应能力强[6-7], 已经成为微藻研究中的模式藻, 被应用在包含生物燃料研究的各个领域, 特别是研究油脂代谢基因的理想受体藻株。
莱茵衣藻的油脂合成途径[8-9]中, 甘油三酯(TAG)是油脂的主分, 代谢途径中TAG含量的多少取决于其前体物质甘油-3-磷酸(G-3-P)的积累, 关键酶甘油-3-磷酸脱氢酶(GPDH)调控G-3-P的正向合成, 直接影响TAG含量多少[8]。目前在酵母、拟南芥、油菜、莱茵衣藻中均有有关TAG代谢的研究, 证明其可以提高受体植株的油脂含量[10-12]。已有研究通过基因工程手段, 在植物中的过量表达甘油3-磷酸酰基转移酶基因(gpd1), 增强了植物Kennedy代谢途径, 提高了TAG的含量[13-14]。在Brassica napus cv.中重组表达酵母菌gpd1基因, 使种子含油量增加40%, 证实了gpd1基因对TAG合成有关键作用[14]。在莱茵衣藻的油脂代谢途径中, TAG作为油脂的重要成分和中间产物, 在合成中直接影响莱茵衣藻细胞中的油脂含量[11]。Du等人[10]关注TAG莱茵衣藻在非胁迫条件下的积累, 以基因工程技术改造的基因工程藻能在适宜生长的条件下大量积累TAG, 产油量提高。Merchant等人[11]梳理出TAG在莱茵衣藻中的代谢通路和关键酶如磷脂甘油二酯酰基转移酶和磷酸酶等的关系, 认为可以使用转录组和蛋白质组特异性分析藻类TAG代谢通路。Russa等人[12]研究了参与调控莱茵衣藻TAG代谢关键酶-脂酰辅酶a:甘油酰基转移酶(DGAT)的候选基因, 认为DGAT基因在增强mRNA水平表达中影响TAG合成。但是,不能促进脂质含量增加, 建议研究其参与的其他脂质代谢调控。因此, 借助外源gpd1基因提高莱茵衣藻TAG含量的实验有助于完善莱茵衣藻TAG调控的相关通路和其脂质积累条件的摸索。微藻中有通过在细胞壁缺陷型莱茵衣藻株系中过表达外源gpd1基因来提高藻细胞的油脂含量[15]的研究。但是,由于所选的细胞壁缺陷型藻株材料是经过人为改造所得, 使得gpd1基因的过表达研究具有一定的局限性。
莱茵衣藻作为真核转基因实验材料, 其3套基因组已经测序成功, 且都能建立外源转化体系[16], 其遗传表达方式兼备原核及真核特点来表达外源基因。核转化时具有较好效果的方法有玻璃珠法[17], 基因枪法[18-20]和农杆菌介导法[21-23]等, 其中农杆菌介导转化时, 无需细胞壁缺陷型藻株、材料操作简单、无特殊设备需求, 更具有遗传转化的优势[24]。此外, 农杆菌介导转化法可以避免珠磨法或基因枪法需要的人工改造的细胞壁缺陷型藻株的缺点, 成为一个新的研究热点[21]。然而, 目前尚未有用农杆菌介导转化外源gpd1基因到莱茵衣藻中的研究报道。
本研究选用外源gpd1基因, 借助农杆菌介导法进行莱茵衣藻的核转化来提高藻细胞的油脂表达。实验PCR克隆酵母gpd1基因, 构建农杆菌双元表达载体pRI-Ble-GPD1, 通过农杆菌转化法转化莱茵衣藻FACHB-479受体细胞, 抗性培养转化藻, 分子生物学鉴定, 筛选出过表达gpd1基因的转基因工程藻FACHB-GPD, 来提高莱茵衣藻的油脂含量, 为微藻基因工程和微藻油脂的研究提供参考。
1 材料与方法 1.1 材料藻株、菌株及培养条件:莱茵衣藻FACHB-479, 购于中科院藻种库(TAP培养基, 25±2℃, 光强1500-2000lx, 光暗1:1周期培养到细胞对数生长期); 酿酒酵母, 大肠杆菌DH5α, 农杆菌LBA4404及载体PMD-18T均为本实验室保存(常规培养使用); 载体pDBle为中国农科院生物技术所程奇教授惠赠。
主要试剂:DNA提取、纯化及连接酶试剂盒、DNA Marker及NdeⅠ等酶购自Takara公司; 质粒提取及DNA回收试剂盒购自TIANGEN公司; 引物及测序由SangonBiotech完成; 氨苄青霉素(Amp)博莱霉素(Ble)购自Sigma和Invitrogen公司。
1.2 衣藻表达载体构建载体构建方法如图 1所示。
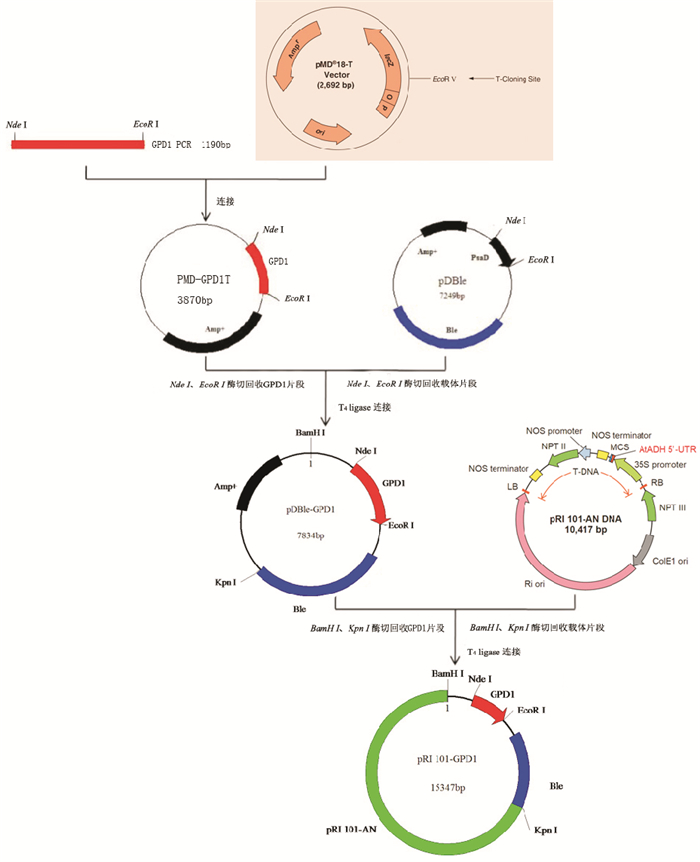
|
图 1 载体构建图 Fig. 1 Construction of expression vector |
根据gpd1基因序列(NCBI GenBank登录号为NC_001136.10), 在上下游增加酶切位点, 用Oligo7软件设计引物(表 1), 由公司合成。试剂盒法提取酿酒酵母DNA, 采用嵌套PCR方法扩增gpd1。PCR体系为20μL, 含有上下游引物终浓度各0.5μmol/L, dNTPs终浓度为125μmol/L, 10×Pyrobest Buffer 2μL, Pyrobest DNA Polymerase 1U。扩增程序为94℃ 10min, 94℃ 1min, 55℃ 1min, 72℃ 2min, 进行30个循环, 72℃ 10min。1%(质量分数)琼脂糖凝胶电泳分离第二次PCR产物, 回收gpd1基因。gpd1的3′端加碱基A连接到载体pMD18-T, 热激转入DH5α感受态细胞, 涂于附加有100mg/L Amp的LB平板, 37℃培养出单菌落, 再液体培养过夜。双酶切鉴定质粒, 测序验证阳性质粒pMD-GPD1。
|
|
表 1 实验所用引物 Tab. 1 List of the primers |
用Nde Ⅰ和EcoR Ⅰ酶37℃ 4h分别酶切质粒pMD-GPD1和pDBle, 回收产物连接, 热激转化DH5α感受态细胞, 在Amp(100mg/L)抗性的LB平板培养。阳性单菌落酶切鉴定为重组质粒pDBle-GPD1。
进行pDBle-GPD1与pRI101-AN质粒的BamH Ⅰ和Kpn Ⅰ 37℃ 2h酶切, 回收连接, 热激转化DH5α的感受态细胞, Amp(100mg/L)抗性的LB平板筛选培养, 阳性单菌落酶切鉴定命名为pRI-Ble-GPD1;冻融法[21]转化到LBA4404的感受态细胞, 在Amp(100mg/L)抗性的YEB平板筛选, 菌落PCR及公司测序验证。同时以空载体pRI 101-AN相同条件转化LBA4404的感受态细胞, 培养得到空载体转化农杆菌。
1.3 FACHB-479的博莱霉素(Ble)筛选浓度确定选用附加Ble浓度分别为0,5, 10, 20, 40, 60ng/μL的TAP固体培养基, 25±2℃, 光照强度1500-2000lx, 光暗周期1:1培养野生藻FACHB-479细胞。每皿接种量100μL(藻细胞数3×105个/mL), 每组平行3皿, 5d后观察FACHB-479细胞的Ble敏感性。
1.4 农杆菌介导转化FACHB-479细胞将FACHB-479藻液于附加100μmol/l乙酰丁香酮的TAP平板上[22]预培养, 5d的藻落接受200μL含载体pRI-Ble-GPD1的LBA4404悬浮液的均匀涂布浸染, 在25±2℃, 1500-2000lx, 光暗1:1条件共培养48h后, 收获藻细胞。同步进行FACHB-479的含空载体的农杆菌转化培养。
1.5 阳性转化藻株的筛选用灭菌玻璃片刮取藻落于TAP液体培养基中悬浮, 在TAP固体平板上Ble抗性筛选培养过夜, 再以25±2℃, 1500-2000lx, 光暗1:1培养7d, 统计阳性转化藻落。再依次将单藻落Ble抗性TAP液体活化培养至对数生长期收集。
1.6 gpd1基因转化藻株的分子生物学验证取培养至对数生长中期(细胞浓度3×105个/mL)的转化藻株, 采用改良的CTAB法[25]提取藻细胞基因组DNA, PCR检测gpd1的整合。Trizol法提取藻细胞RNA, 反转录cDNA, RT-PCR检测gpd1的表达。其中PCR与RT-PCR引物均使用gpd1-F, gpd1-R; 20μL的反应体系中, 包含模板各1μL, 上下游引物各1μL(10μmol/L), Premix Taq10μL; 反应程序为94℃ 10min, 94℃30s, 59℃ 30s, 72℃ 1min, 30个循环程序扩增, 72℃ 10min。
1.7 转gpd1基因藻株的油脂鉴定取对数生长中期(细胞浓度3×105个/mL)的gpd1转化藻株1mL, 加入800μL尼罗红贮存液(0.1g/L Nile red/Methanol)混匀(终浓度44mg/L), 用铝箔包好离心管, 23℃避光染色15min, 3000rpm, 30s收集藻细胞, 用TAP液体培养基洗去多余的染色液。激发波长485nm、发射波长590nm条件下, 激光共聚焦显微镜观察记录。同时取上述染色的莱茵衣藻细胞200μL分别加入96孔板中, 多功能酶标仪在激发波长485nm, 检测波长590nm条件下测定荧光值(a.u.), 分析不同藻株油脂情况。
2 结果与分析 2.1 衣藻表达载体的构建依据载体构建策略(图 2)构建衣藻载体过程中, 首先可以经过嵌套PCR方法扩增出gpd1基因(图 2), 电泳结果显示扩增出1175bp的DNA片段。
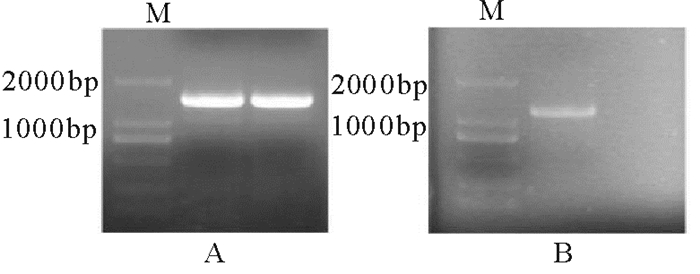
|
A 第一次PCR; B 第二次PCR; M 2kb Marker 图 2 gpd1基因套嵌PCR分析 Fig. 2 PCR analysis of gpd1 |
在验证重组质粒pMD-GPD1时, gpd1片段的PCR扩增结果(图 3A)以及Nde Ⅰ和EcoR Ⅰ双酶切结果(图 4B)均可以得到1 175bp的预期电泳条带; 公司测序验证(图 4)重组质粒pMD-GPD1中的gpd1序列与酿酒酵母gpd1基因(NCBI GenBank登录号NC_001136.10)100%吻合。
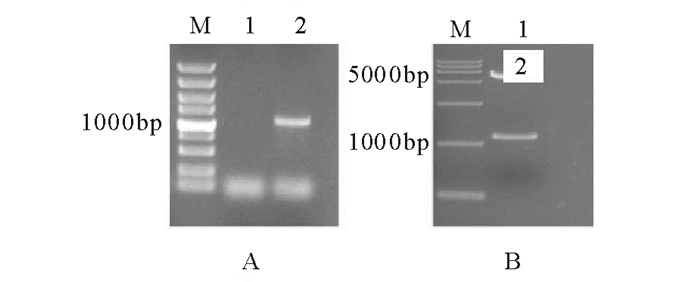
|
A PCR; B Nde I和EcoR Ⅰ双酶切 图 3 质粒pMD-GPD1鉴定 Fig. 3 Enzyme digestion analysis of pMD-GPD1 |
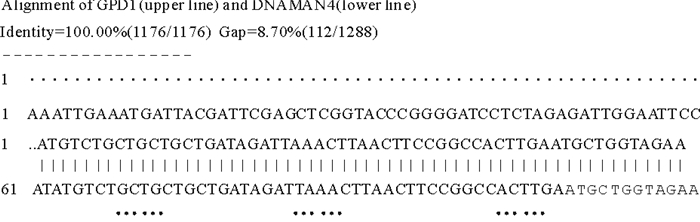
|
图 4 pMD-GPD1的gpd1测序比对 Fig. 4 gpd1 of pMD-GPD1 |
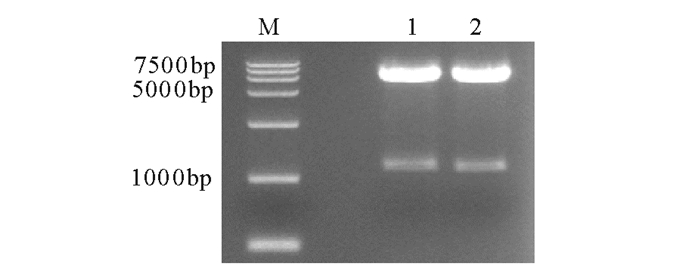
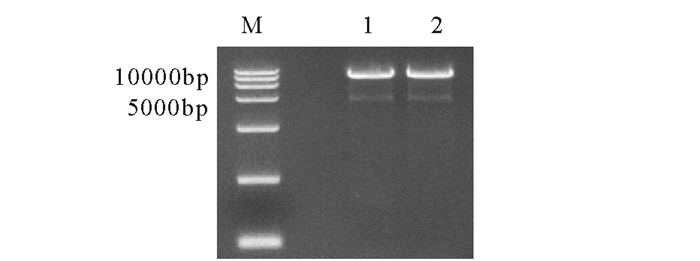
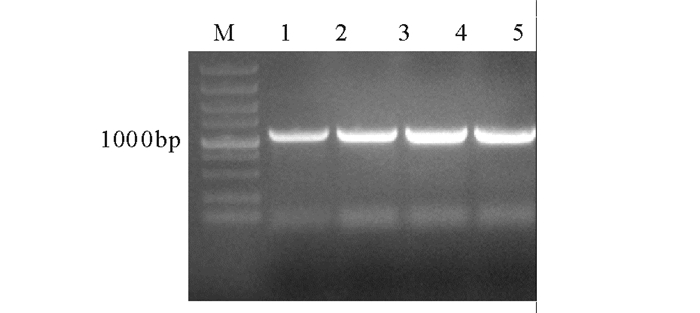
经验证, 质粒pDBle-GPD1通过Nde Ⅰ和EcoR Ⅰ双酶切可以得到1175bp的预期条带(图 5)。构建成功的质粒pRI-Ble-GPD1, 用BamH Ⅰ和Kpn Ⅰ进行双酶切得到4930bp条带(图 6)。测序验证载体gpd1序列正确(图 7), 转化LBA4404得到的阳性克隆菌PCR(图 8)可以扩增出预期的1175bp的目的片段。
|
图 5 质粒pDBle-GPD1双酶切验证 Fig. 5 Enzyme digestion analysis of pDBle-GPD1 |
|
图 6 质粒pRI-Ble-GPD1双酶切验证 Fig. 6 Enzyme digestion analysis of pRI-Ble-GPD1 |

|
图 7 质粒pRI-Ble-GPD1的gpd1测序比对 Fig. 7 gpd1 of pRI-Ble-GPD1 |
|
图 8 质粒pRI-Ble-GPD1转化LBA4404验证 Fig. 8 PCR analysis of pRI-Ble-GPD1 in LBA4404 |
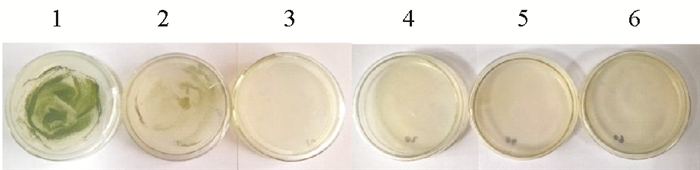
使用含Ble的TAP固体培养基培养FACHB-479(图 9), 可以看到藻细胞对Ble很敏感, 只要有Ble存在, 其生长就会受到影响。随着Ble浓度的升高, 藻细胞的生活能力迅速下降, 当Ble浓度从ng/μL增加到10ng/μL时, 藻细胞生长状态变化明显, 平板上的活细胞藻落从100%下降到一定量的Ble可以致死藻细胞, 当其浓度为5ng/μL时, 藻落大部分处于黄色状态, 生命迹象弱, 仅有少量藻落呈现绿色。Ble浓度上升到10ng/μL时完全可以抑制FACHB-479细胞的生长, 故确定衣藻遗传转化中淘汰非转化藻的Ble筛选浓度为10ng/μL。
|
1-6 Ble浓度依次为0,5,10,20,40,60 ng/μL 图 9 野生藻FACHB-479在Ble抗性TAP培养 Fig. 9 Ble resistance of wild type FACHB-479 |
衣藻载体pRI-Ble-GPD1携带gpd1基因, 经农杆菌介导转化FACHB479细胞, 历经共培养48h, Ble(10ng/μL)抗性的TAP固体筛选培养5 d, 可以收获到阳性转化藻(图 10), 单藻落编号后在Ble(10ng/μL)抗性的TAP液体继代培养, 可以收集到大量的不同转化株系的藻细胞。而野生型FACHB-479和空载体PRI101-AN(空白载体)转化藻均不能在含有10ng/μLBle的TAP培养基中上生长。

|
图 10 转化藻株在Ble抗性TAP上的培养 Fig. 10 Screening of transformants by Ble resistance |
收集单菌落液体培养(图 8)到对数生长期的藻细胞, 进行分子生物学鉴定目的gpd1基因的整合和表达。
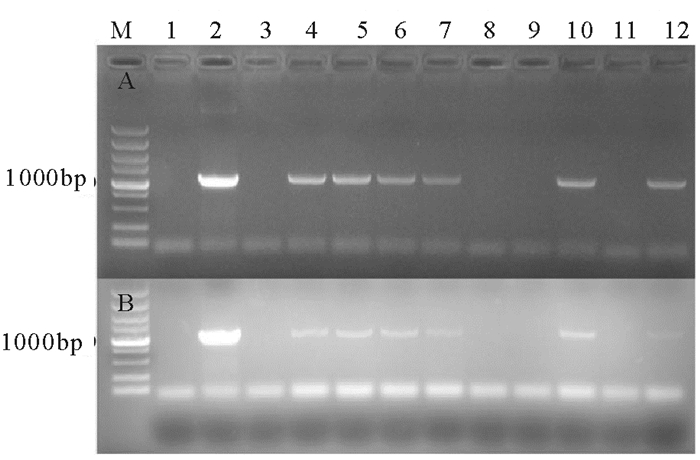
提取基因组DNA, PCR检测(图 11)筛选到的抗性藻株, 显示部分抗性藻株可扩增出约1175 bp的gpd1基因目标条带, 而野生型FACHB-479及空载体pRI 101-AN转化藻株未扩增出目标条带。提取藻株的总RNA, RT-PCR检测(图 11)到部分转化藻株的gpd1基因目标1175 bp条带清晰可见, T4,T5,T6,T7,T10,T12确定为转基因藻株。
|
图 11 不同藻株中gpd1基因的分子生物学分析 Fig. 11 PCR and RT-PCR analysis of the transformants contained gpd1 |
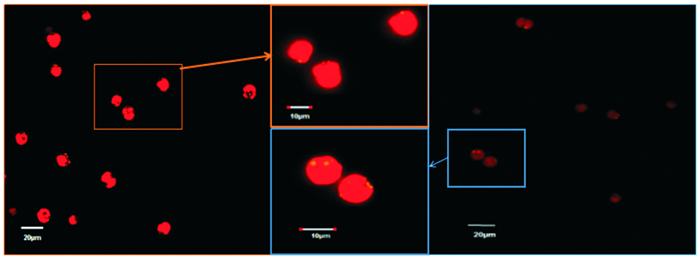
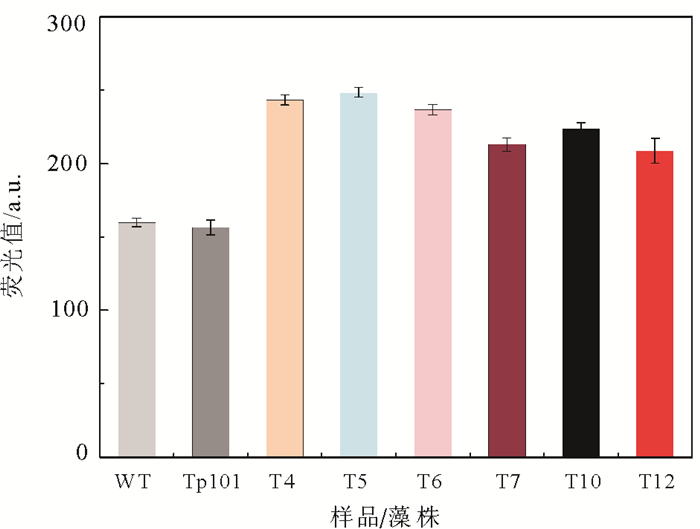
采用油脂尼罗红染色方法分析转基因藻FACHB-GPD的油脂含量。在激光共聚焦显微镜视野中(图 12), FACHB-GPD细胞整体呈红色, 亮黄色显示了细胞内存在的油脂, 大小和数目显著高于野生型藻(WT)。使用多功能酶标仪测定油脂含量, 优化的尼罗红染色荧光检测条件为终浓度44mg/L的尼罗红甲醇溶液, 23℃避光染色15min, 得到的转基因藻液的荧光值均高于野生型藻株(图 13), 检测藻株油脂含量为对照藻株的1.3~1.52倍。
|
图 12 藻细胞激光共聚焦图黄色示油脂 Fig. 12 Yellow lipid droplets observed under confocal laser scanning microscope |
|
图 13 不同藻株油脂含量测定 Fig. 13 Lipid content analysis of the different algae stains |
目前莱茵衣藻的基因组已经完成测序, 常被用作基因工程方法研究生物能源时的首选实验材料。本实验基于此,选用了莱茵衣藻藻株FACHB-479作为基因工程受体。实验中藻FACHB-479受体可以表达酵母菌gpd1基因, 得到的转基因藻株中, 油脂尼罗红染色方法分析得知油脂含量可以提高(为对照藻株的1.3~1.52倍), 推测是由于gpd1基因的过表达, 直接促进了TAG的合成, 进而增加莱茵衣藻中油脂含量, 这与王潮岗等[15]的cc-849藻的工程改造藻株的外源gpd1基因的过表达结果相似, 而他们实验仅验证了外源gpd1基因表达量相比对照株提高了75.95%, 并未提到转基因株系油脂含量是否有变化。并且, 王潮岗等[15]采用珠磨法转化莱茵衣藻, 选用的是细胞壁缺陷型藻株, 实验前期藻株的准备相对于常规有壁的莱茵衣藻, 其基因工程操作带有一定的局限性。藻株FACHB-479具有细胞壁, 为了避免准备细胞壁缺陷型的藻株过程的耗时耗力, 实验选用了适合植物转化的农杆菌介导法, 利用农杆菌转化法在不挑选受体植物细胞(有壁)的同时, 操作简单, 共培养过程时外源基因易整合的优点[26], 成功将外源gpd1基因转入细胞壁完好的FACHB-479细胞中, 说明了该方法方便快捷并具有实用性, 有望成为后期用野生富油微藻构建高产油转基因微藻的有效方法之一。
生物油脂检测通常需要很大量活体材料而且费时费力, 微藻个体小, 更是如此。传统油脂检测方法[27]需要大量微藻材料, 油脂提纯中需要防止脂质分解或氧化, 检测难度大, 费时费力。相比而言, 尼罗红染色法可以高效、定量、高通量的检测植物细胞乃至莱茵衣藻细胞的油脂含量[28], 用于转基因莱茵衣藻株系的藻细胞脂质监测, 方便快捷。与Chen等[28]的相似方法, 本实验中使用终浓度44mg/L的尼罗红-甲醇溶液染色, 23℃, 避光15min, 荧光效果较好, 对比FACHB-479细胞, 激光共聚焦显微镜视野中转基因藻FACHB-GPD细胞油脂亮黄色清晰, 大小和数量高。多功能酶标仪检测发现不同的转基因藻株中油脂含量均增多, 但是不同藻株间存在差异, 分析可能是因为外源gpd1基因在不同的转基因藻株中的表达效率不一致造成的, 有待于后续研究。
4 结论本研究构建了pRI-Ble-GPD1表达载体, 可以携带酵母gpd1基因, 通过农杆菌介导法转入受体莱茵衣藻FACHB-479细胞, 基因工程方法获得的gpd1基因过表达的转gpd1基因藻FACHB-GPD, 其细胞中油脂含量可以增高1.3~1.52倍。该研究为莱茵衣藻的基因工程改造提供理论参考的同时,也证实了gpd1基因在莱茵衣藻油脂合成方面具有促进作用, 对微藻产油有一定借鉴意义。
| [1] |
CHISTI Y. Biodiesel from microalgae beats bioethanol[J]. Trends in Biotechnology, 2008, 26(3): 126-131. DOI:10.1016/j.tibtech.2007.12.002 |
| [2] |
BHARATHIRAJA B, JAYAMUTHUNAYAI J, CHAKRAVARTHY M, et al. Algae: Promising Future Feedstock for Biofuels[M]//Development Applied Phycology. New Delhi: Springer, 2015.
|
| [3] |
MANIKANDAN G, KUMAR P S, PRAKALATHAN R. Comparison of biodiesel production from macro and micro algae[J]. International Journal of Chemtech Research, 2014, 6(9): 4143-4147. |
| [4] |
PFLASTER E L. A high-throughput fatty acid profiling screen reveals novel variations in fatty acid biosynthesis in Chlamydomonas reinhardtii and related algae[J]. Eukaryotic Cell, 2014, 13(11): 1431-1438. DOI:10.1128/EC.00128-14 |
| [5] |
BROWN L M, ZEILER K G. Aquatic biomass and carbon dioxide trapping[J]. Energy Conversion & Management, 1993, 34(9-11): 1005-1013. |
| [6] |
ROCHAIX J D. Chlamydomonas reinhardtii as the photosynthetic yeast[J]. Annual Review of Genetics, 1995, 29(4): 209-230. |
| [7] |
HARRIS E H, CHLAMYDOMONAS AS A, MODEL ORGANISM. Annual Review of Plant Physiology & Plant Molecular[J]. Biology, 2001, 52(4): 363-406. |
| [8] |
WAN L. De novo transcriptomic analysis of an oleaginous microalga: pathway description and gene discovery for production of next-generation biofuels[J]. Plos One, 2012, 7(4): 35-42. |
| [9] |
QIANG H. Microalgal triacylglycerols as feedstocks for biofuel production: perspectives and advances[J]. Plant Journal, 2008, 54(4): 621-639. DOI:10.1111/j.1365-313X.2008.03492.x |
| [10] |
DU Z Y, BENNING C. Triacylglycerol Accumulation in Photosynthetic Cells in Plants and Algae[J]. Sub-cellular Biochemistry, 2015, 86. |
| [11] |
MERCHANT S S. TAG, You′re it! Chlamydomonas as a reference organism for understanding algal triacylglycerol accumulation[J]. Current Opinion in Biotechnology, 2012, 23(3): 352-363. DOI:10.1016/j.copbio.2011.12.001 |
| [12] |
RUSSA M L. Functional analysis of three type-2 DGAT homologue genes for triacylglycerol production in the green microalga Chlamydomonas reinhardtii[J]. Journal of Biotechnology, 2012, 162(1): 13-20. DOI:10.1016/j.jbiotec.2012.04.006 |
| [13] |
SCRANTON M A. Chlamydomonas as a model for biofuels and bio-products production[J]. Plant Journal, 2015, 82(3): 523-531. DOI:10.1111/tpj.12780 |
| [14] |
VIGEOLAS H. Increasing seed oil content in oil-seed rape (Brassica napus L.) by over-expression of a yeast glycerol-3-phosphate dehydrogenase under the control of a seed-specific promoter[J]. Plant Biotechnology Journal, 2007, 5(3): 431-441. DOI:10.1111/pbi.2007.5.issue-3 |
| [15] |
王潮岗, 李逸, 胡章立. 转lpaat和gpd1基因工程藻的快速筛选l[J]. 深圳大学学报(理工版), 2016, 33(1): 33-40. |
| [16] |
BERTALAN I. A rapid modular and marker-free chloroplast expression system for the green alga Chlamydomonas reinhardtii[J]. Journal of Biotechnology, 2014, 195: 60-66. |
| [17] |
KINDLE K L. High-frequency nuclear transformation of Chlamydomonas reinhardtii[J]. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(3): 1228-1232. DOI:10.1073/pnas.87.3.1228 |
| [18] |
NEUPERT J. Genetic Transformation of the Model Green Alga Chlamydomonas reinhardtii[J]. Methods in Molecular Biology, 2012, 847: 35-47. DOI:10.1007/978-1-61779-558-9 |
| [19] |
DAWSON H N, BURLINGAME R, CANNONS A C. Stable Transformation of Chlorella: Rescue of Nitrate Reductase-Deficient Mutants with the Nitrate Reductase Gene[J]. Current Microbiology, 1998, 35(6): 356-362. |
| [20] |
SCHIEDLMEIER B. Nuclear transformation of Volvox carteri[J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(11): 5080-5084. DOI:10.1073/pnas.91.11.5080 |
| [21] |
PRATHEESH PT, VINEETHA M, KURUP G M. An efficient protocol for the Agrobacterium-mediated genetic transformation of microalga Chlamydomonas reinhardtii[J]. Molecular Biotechnology, 2014, 56(6): 507-515. DOI:10.1007/s12033-013-9720-2 |
| [22] |
KUMAR S V. Genetic transformation of the green alga & mdash; Chlamydomonas reinhardtii by Agrobacterium tumefaciens[J]. Plant Science, 2004, 166(3): 731-738. DOI:10.1016/j.plantsci.2003.11.012 |
| [23] |
CHA T S, YEE W, AZIZ A. Assessment of factors affecting Agrobacterium-mediated genetic transformation of the unicellular green alga[J]. Chlorella Vulgaris. World Journal of Microbiology & Biotechnology, 2012, 28(4): 1771-1779. |
| [24] |
LACROIX B, CITOVSKY V. The roles of bacterial and host plant factors in Agrobacterium-mediated genetic transformation[J]. International Journal of Developmental Biology, 2013, 57(6-7-8): 467-481. DOI:10.1387/ijdb.130199bl |
| [25] |
王双辉, 刘芮均, 兰利琼. 衣藻的核基因组转化及外源基因转录分析[J]. 四川大学学报(自然科学版), 2016, 53(3): 695-699. |
| [26] |
SPARKS C A, DOHERTY A, JONES H D. Genetic transformation of wheat via Agrobacterium-mediated DNA delivery[J]. Methods in Molecular Biology, 2014, 1099: 235-250. DOI:10.1007/978-1-62703-715-0 |
| [27] |
BLIGH E G, DYER W J. A rapid method of total lipid extraction and purification[J]. Canadian Journal of Biochemistry & Physiology, 1959, 37(8): 911-917. |
| [28] |
CHEN W. A high throughput Nile red method for quantitative measurement of neutral lipids in microalgae[J]. Journal of Microbiological Methods, 2009, 77(1): 41-47. DOI:10.1016/j.mimet.2009.01.001 |
 2017, Vol. 47
2017, Vol. 47

