2. 武汉大学人民医院消化内科 湖北 武汉 430060;
3. 武汉大学第一临床学院 湖北 武汉 430060
2. Dept. of Gastroentrology Renmin Hospital of Wuhan University, Wuhan 430060, China;
3. The First Clinical College, Wuhan University, Wuhan 430060, China
乳腺癌是女性肿瘤患者中,发病率最高的肿瘤之一[1]。乳腺癌,尤其是雌激素受体(estrogen receptor,ER)、孕激素受体(progesterone receptor,PR)和人表皮生长因子受体-2(human epidermalgrowth factor receptor-2,HER2)均为阴性的三阴性乳腺癌,患者往往早期就出现转移,并对化疗药物出现耐药,预后凶险;即使对于非三阴性乳腺癌,如果术后出现复发或者转移,患者的预后也往往不良[1]。结构特异性识别蛋白1(structure specific recognition protein 1,SSRP1)是一种与转录调控、DNA损伤修复以及细胞周期调控相关的蛋白[2]。SSRP1在人体正常成熟组织中表达量低,而在多种分化程度较低和癌变组织中,SSRP1则为高表达,提示SSRP1可能对维持肿瘤细胞样表达谱具有关键作用[3, 4]。本研究利用GEO公共数据集,分析SSRP1在乳腺癌中的表达情况,分析其与患者预后的关系,探寻SSRP1调控乳腺癌发展的机制,为进一步研究该基因的功能、机制和临床意义提供线索和思路。
1 材料与方法 1.1 收集数据资料我们从NCBI的GEO数据库(http://www.ncbi.nlm.nih.gov/geo)中下载基因表达谱公共数据集,包括乳腺癌样本数据集GSE3494和GSE47561。上述所有数据集的数据均导入dChip软件还原原始芯片扫描图像,之后用总荧光强度为中位数的芯片N2为基准进行标准化处理,再进行后续计算和分析。
1.2 数据集筛选和临床相关性研究GSE3494,样本量大且附带临床信息,可进行进一步的回顾性和生存期分析研究。根据表达谱数据,对样本的SSRP1的表达进行由高到低排序后,前20%的样本作为高表达组,后80%的样本作为低表达组。SSRP1的相对表达量以探针信号强度(intensity)正态分布化后的log2intensity值衡量。
1.3 基因集富集分析(GSEA)采用GSEA2.0.14版本(JAVA版本)进行分析。数据集GSE47561被纳入GSEA。肿瘤组织样本根据SSRP1的表达水平被分为高低表达两组(前50%的样本作为高表达组,后50%的样本作为低表达组),之后通过GSEA分析SSRP1的表达水平对各种基因集(gene ontology)的影响。我们从GSEA网站MsigDB数据库中获得的基因集作为参照基因集,按default weighted enrichment statistic的方法,每次分析重复1 000次。
1.4 作图及统计学处理作图及统计学处理软件使用GraphPad Prism 5。回顾性研究中,组间比较采用联列表法,采用卡方检验;生存期分析研究中,Log-Rank (Mantel-Cox)法用于生存分析。以P<0.05作为有统计学意义;在GSEA中,P<0.05及错误发现率(false discovery rates, FDR)<0.05的基因集作为显著富集基因集。
2 结果 2.1 SSRP1与乳腺癌的临床病理相关性为了探讨SSRP1的表达与乳腺癌患者临床病理资料的相关性,我们利用GSE3494中的临床资料进行了回顾性分析。在乳腺癌样本数据集GSE3494中,在不同年龄(P=0.001 8)、p53水平(P<0.000 1)、p53突变(P<0.000 1)、ER水平(P=0.000 2)、PR水平(P=0.011 6)、Elston组织学分级(P<0.000 1)分组的样本中,SSRP1的表达均有显著性差异;而SSRP1与患者的肿瘤大小及淋巴结浸润情况无显著相关性(P>0.05),见表 1。这说明SSRP1可能影响了p53的表达,又因为其是调节DNA损伤修复的关键蛋白[2],它的异常表达还可能造成p53的突变异常;SSRP1表达与ER、PR表达的显著相关性,也提示SSRP1可能是三阴性乳腺癌的重要治疗靶点。
| 表 1 SSRP1与乳腺癌患者(GSE3494)的临床病理指标的相关性 |
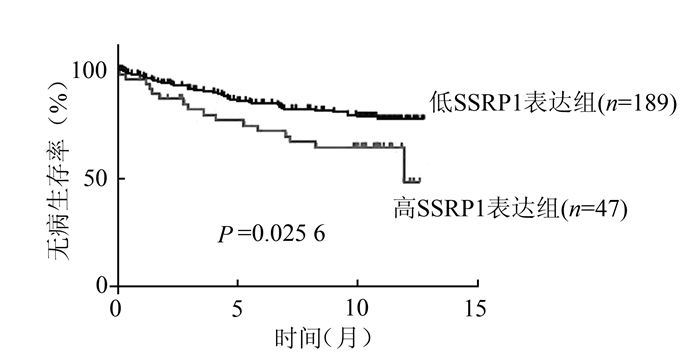
为了研究SSRP1是否能作为肿瘤预后的评价分子,我们又进行了前瞻性分析。在肝癌样本数据集GSE3494中,SSRP1高表达组患者和低表达组患者的5年生存率分别为76.57%和85.50.%,Log-Rank=4.980,P=0.025 6(图 2A)。以上结果说明SSRP1的高表达与乳腺癌患者术后早期死亡有关。
|
图 1 Kaplan-Meier法分析SSRP1表达水平与乳腺癌预后的关系(GSE3494) |
|
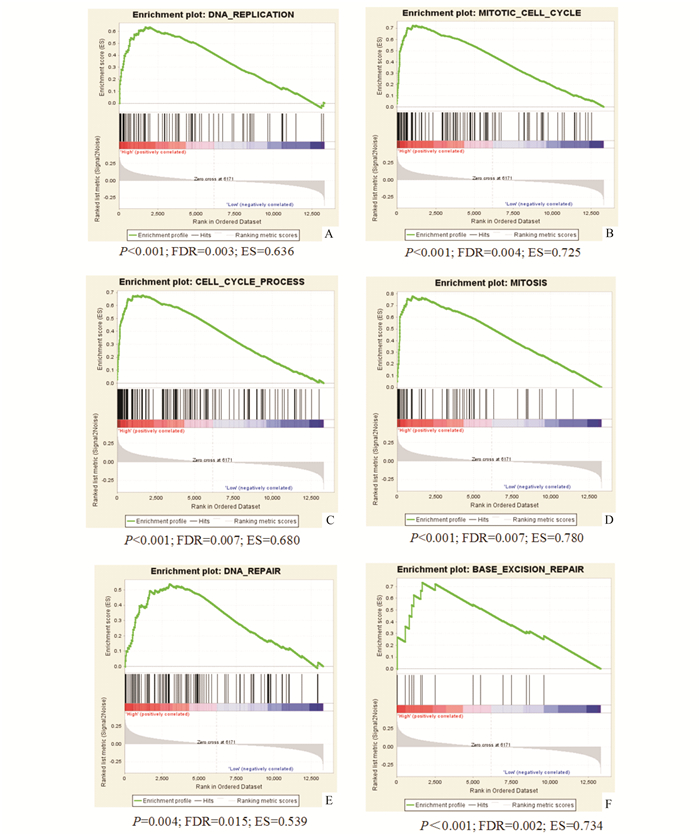
图 2 GSEA分析显示SSRP1高表达的乳腺癌样本富集了与DNA复制(A)、细胞周期(B、C)、有丝分裂(D)、DNA损伤修复(E、F)有关的基因集 P<0.05及FDR<0.05的基因集作为显著富集基因集 |
分析SSRP1与乳腺癌患者的预后关系之后,我们继而对SSRP1推动乳腺癌进展的可能方式进行了探讨。我们利用乳腺癌样本数据集GSE47561,使用GSEA方法,分析SSRP1的表达水平对各种细胞功能基因集的影响。我们使用“go ontology”作为参照基因集,可见SSRP1高表达的肿瘤样本富集了与DNA复制、有丝分裂、细胞周期和DNA损伤修复相关的基因集(图 2)。以上结果与已发表的文献中证明的SSRP1的功能相一致,说明SSRP1可能通过调节肿瘤的细胞分裂以及DNA损伤修复,影响肿瘤细胞的增殖和耐药等表型。
3 讨论SSRP1和SUPT16(suppressor of Ty 16 homolog)共同构成了FACT (Facilitates chromatin transcription)。FACT是转录和复制因子,以组蛋白分子伴侣的形式发挥生物学功能,可以与H2A/H2B组蛋白二聚体相互作用,协助DNA分子从组蛋白八聚体上解开,降低核小体的致密性,促进转录过程中延伸步骤的进行[5, 6]。另外,FACT可以通过影响RNA聚合酶Ⅰ、Ⅱ、Ⅲ影响基因转录的进程[7, 8]。鉴于以上原因,FACT对于维持细胞的转录活性至关重要。肿瘤细胞是高转录活性细胞的代表,所以FACT可能是维持肿瘤细胞生物表型的关键蛋白。有研究发现,SSRP1对基因转录的调控作用既可以通过SUPT16依赖的方式进行,也可以独立进行[9];另外,SSRP可以作为转录因子p63的共激活因子[10]。综上所述,我们推测,SSRP1是调控乳腺癌肿瘤细胞转录活性的关键分子。
有丝分裂是细胞分裂的最终步骤,有丝分裂的调节在细胞增殖、胚胎形成和肿瘤发生过程中都有重要意义。有丝分裂的完成必然伴随着DNA的复制。研究证实,FACT对于维持细胞DNA的正常复制具有重要作用--SSRP1敲除的DT40细胞,细胞周期S期进展被阻滞,增殖停止[11]。进一步探讨其机制发现,SSRP1敲除虽然不影响DNA复制的起始步骤,却导致DNA复制的延伸过程中DNA合成减少[11]。另一方面,推动有丝分裂进行的“机械装置”是由α和β微管蛋白形成的微管,因而微管的多聚化对于细胞的增殖十分关键。新近的研究证实,SSRP1是微管的绑定蛋白,可以通过促进微管的多聚化促进细胞的有丝分裂[12]。
实际上,SSRP1已经被证实为是肿瘤治疗的靶点,国外已有相应的靶向药物进入临床前实验:特异性抑制SSRP1的化合物Curaxins可以调控p53通路和NF-κB通路,从而通过诱导肿瘤细胞凋亡阻遏多种肿瘤细胞的增殖[13];有研究者利用可以自发发生乳腺癌的Her2/neu转基因小鼠为模型,检测Curaxins对乳腺癌发生的预防作用,结果显示,该靶向化合物可以减少肿瘤发生率、延缓肿瘤进展、延长小鼠的总体生存期[14]。一项新近的研究则发现,在非小细胞肺癌细胞中抑制SSRP1可以显著提高细胞对靶向药物厄洛替尼的敏感性,促进非小细胞肺癌细胞系的凋亡[15]。以上结果进一步提示了SSRP1在肿瘤发生发展中起到的作用,并展现了其抑制剂乐观的临床应用前景。
在本研究中,我们结合临床资料分析和生物信息学,证实SSRP1在乳腺癌组织中高表达,与患者的术后远期生存相关,并可以调控与DNA复制、有丝分裂、细胞周期和DNA损伤修复相关的基因集,提示其可以作为乳腺癌治疗的靶标。
| [1] | Bouchalova K, Kharaishvili G, Bouchal J, et al. Triple negative breast cancer-BCL2 in prognosis and prediction. Review[J]. Curr Drug Targets, 2014, 15(12): 1 166-1 175. DOI: 10.2174/1389450115666141106151143. |
| [2] | Dinant C, Ampatziadis-Michailidis G, Lans H, et al. Enhanced chromatin dynamics by FACT promotes transcriptional restart after UV-induced DNA damage[J]. Mol Cell, 2013, 51(4): 469-479. DOI: 10.1016/j.molcel.2013.08.007. |
| [3] | Garcia H, Fleyshman D, Kolesnikova K, et al. Expression of FACT in mammalian tissues suggests its role in maintaining of undifferentiated state of cells[J]. Oncotarget, 2011, 2(10): 783-796. DOI: 10.18632/oncotarget. |
| [4] | Garcia H, Miecznikowski JC, Safina A, et al. Facilitates chromatin transcription complex is an "accelerator" of tumor transformation and potential marker and target of aggressive cancers[J]. Cell Rep, 2013, 4(1): 159-173. DOI: 10.1016/j.celrep.2013.06.013. |
| [5] | Winkler DD, Muthurajan UM, Hieb AR, et al. Histone chaperone FACT coordinates nucleosome interaction through multiple synergistic binding events[J]. J Biol Chem, 2011, 286(48): 41 883-41 892. DOI: 10.1074/jbc.M111.301465. |
| [6] | Winkler DD, Luger K. The histone chaperone FACT: structural insights and mechanisms for nucleosome reorganization[J]. J Biol Chem, 2011, 286(21): 18 369-18 374. DOI: 10.1074/jbc.R110.180778. |
| [7] | Hsieh FK, Kulaeva OI, Patel SS, et al. Histone chaperone FACT action during transcription through chromatin by RNA polymerase Ⅱ[J]. Proc Natl Acad Sci USA, 2013, 110(19): 7 654-7 659. DOI: 10.1073/pnas.1222198110. |
| [8] | Birch JL, Tan BC, Panov KI, et al. FACT facilitates chromatin transcription by RNA polymerases Ⅰ and Ⅲ[J]. EMBO J, 2009, 28(7): 854-865. DOI: 10.1038/emboj.2009.33. |
| [9] | Li Y, Zeng SX, Landais I, et al. Human SSRP1 has Spt16-dependent and -independent roles in gene transcription[J]. J Biol Chem, 2007, 282(10): 6 936-6 945. |
| [10] | Zeng SX, Dai MS, Keller DM, et al. SSRP1 functions as a co-activator of the transcriptional activator p63[J]. EMBO J, 2002, 21(20): 5 487-5 497. DOI: 10.1093/emboj/cdf540. |
| [11] | Abe T, Sugimura K, Hosono Y, et al. The histone chaperone facilitates chromatin transcription (FACT) protein maintains normal replication fork rates[J]. J Biol Chem, 2011, 286(35): 30 504-30 512. DOI: 10.1074/jbc.M111.264721. |
| [12] | Zeng SX, Li Y, Jin Y, et al. Structure-specific recognition protein 1 facilitates microtubule growth and bundling required for mitosis[J]. Mol Cell Biol, 2010, 30(4): 935-947. DOI: 10.1128/MCB.01379-09. |
| [13] | Gasparian AV, Burkhart CA, Purmal AA, et al. Curaxins: anticancer compounds that simultaneously suppress NF-κB and activate p53 by targeting FACT[J]. Sci Transl Med, 2011, 3(95): 95ra74. |
| [14] | Koman IE, Commane M, Paszkiewicz G, et al. Targeting FACT complex suppresses mammary tumorigenesis in Her2/neu transgenic mice[J]. Cancer Prev Res (Phila), 2012, 5(8): 1 025-1 035. DOI: 10.1158/1940-6207.CAPR-11-0529. |
| [15] | Dermawan JK, Gurova K, Pink J, et al. Quinacrine overcomes resistance to erlotinib by inhibiting FACT, NF-κB, and cell-cycle progression in non-small cell lung cancer[J]. Mol Cancer Ther, 2014, 13(9): 2 203-2 214. DOI: 10.1158/1535-7163.MCT-14-0013. |
 2016, Vol. 37
2016, Vol. 37

