2. 武汉大学基础医学院免疫系, 湖北 武汉 430060;
3. 医学微生物学教研室 湖北 武汉 430071
2. Dept. of Immunology, Wuhan 430060, China;
3. Dept. of Medical Microbiology, School of Basic Medical Sciences, Wuhan University, Wuhan 430071, China
肝癌是我国常见的恶性肿瘤之一,据国家卫生和计划生育委员会统计,2012年我国城市居民的肝癌死亡率居恶性肿瘤致死率的第2位。尽管我国肝癌的发病率有所下降,但其发展趋势仍然十分严峻,2012年全球共有近80万肝癌新发病例及约75万肝癌致死病例,其中约50%发生在中国[1]。约90%的原发性肝癌属于肝细胞癌(hepatocellular carcinoma,HCC),其中约80%的HCC患者为乙肝病毒(hepatitis virus B,HBV)或丙肝病毒(hepatitis virus C,HCV)的携带者。机体免疫系统为清除病毒感染而引起的持续性的慢性肝损伤是肝炎病毒导致肝癌的主要原因。如慢性感染期间的持续性抗病毒炎症反应、病毒感染细胞的免疫清除和肝细胞再生等,都增加了病毒感染性肝病向肝癌发展的风险。然而HBV或HCV引起肝癌的机制仍未完全明确。本研究通过生物信息学方法探讨HBV、HCV相关性肝癌的差异,利用GEO公共数据库,筛选HBV、HCV相关性肝癌差异表达基因,并对其进行功能富集分析和生物功能通路富集分析,为进一步了解病毒相关性肝癌的发展机制提供线索和依据,以促进肝癌分子诊断、治疗技术发展。
1 资料与方法 1.1 资料来源在美国国立生物中心NCBI的GEO数据库http:www.ncbi.nlm.nih.gov/geo中以“HBV,HCV,HCC”作为关键词,设置研究类型为expression profiling by array,设置种属类型为homo sapiens,样本来源为肝癌组织。获得HBV、HCV相关性肝癌数据集GSE44074,含HBV、HCV相关性肝癌样本各17例[2]。该数据集另含71例HBV、HCV相关肝纤维化样本,不列入本研究中进行分析计算。
1.2 差异表达基因的筛选利用GEO2R在线工具(http://www.ncbi.nlm.nih.gov/geo/info/geo2r.html),采用Benjamini & Hochberg法进行校正,分析GSE44074数据中乙肝相关性肝癌和丙肝相关性肝癌的差异表达基因。按以下标准筛选差异表达基因:① 校正后的P值 < 0.05;② 表达数据缺失值超过50%的基因不纳入分析;③ 未知基因不纳入分析;④ 乙肝、丙肝病毒编码蛋白不纳入分析。
1.3 DAVID分析通过DAVID(https://david.ncifcrf.gov/)在线软件[3],对GSE44074数据中HBV相关性肝癌和HCV相关性肝癌的差异表达基因进行分析。提交所有差异表达基因名称,基因名对应的基因标识符为OFFICIAL_GENE_SYMBOL,勾选“人类”(HOMO SAPIENS)作为分析背景,选择“Functioal Annotation Tool”作为分析工具,获得所有差异基因的GO富集分析结果。
1.4 基因富集分析(gene sets enrichment analysis,GSEA)采用GSEA(http://www.broadinstitute.org/gsea)[4]对GSE44074进行分析,按照HBV相关性肝癌和HCV相关性肝癌分为两组,并在Molecular Signatures Database (MSigDB)上下载基因集c5.bp.v5.1、c5.cc.v5.1、c5.mf.v5.1作为参照基因集,共涉及1 454种与生物学过程(biological process,bp)、细胞组成成分(cellular component,cc)和分子功能(molecular function,mf)相关的基因集,按default weighted enrichment statistic方法进行富集分子,随机组合重复次数设置为1 000次,FDR(false discovery rate)< 25%且标准化的P < 0.05的基因集定义为阳性基因集。
2 结果 2.1 差异基因的筛选利用GSE44074数据集,通过GEO在线分析软件GEO2R对乙肝病毒相关性肝癌(乙肝组)和丙肝病毒相关性肝癌(丙肝组)间的差异表达基因进行了筛选。经Benjamini & Hochberg校正后,除HBV-Core之外,共筛选出34个差异表达基因(矫正后P < 0.05)。34个差异表达的基因中,14个为乙肝组较丙肝组表达上调的基因,20个基因为个丙肝组较乙肝组表达上调的基因。34个差异表达的基因中14个基因在两组中的表达水平的倍数差异大于2(logFC≥0.301)。差异表达基因列表及其表达水平示意热图,分别见表 1、表 2和图 1。
| 表 1 乙肝病毒相关性肝癌较丙肝病毒相关性肝癌高表达的基因 |
| 表 2 丙肝病毒相关性肝癌较乙肝病毒相关性肝癌高表达的基因 |
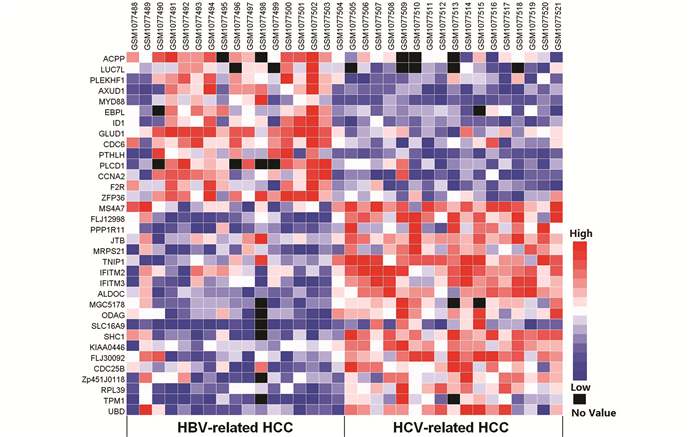
|
图 1 乙肝相关性肝癌和丙肝相关性肝癌间差异表达基因热图 |
为分析这些差异表达的基因间的功能关联及其可能共同参与的生物学作用,我们对所有差异表达基因进行了DAVID分析。分别将在HBV相关性HCC中高表达的14个基因和HCV相关性HCC高表达的20个基因上传到DAVID分析平台,除FLJ30092(HECTD4) 和MGC5178 (SLX1B)外,所有基因均得到注释。结果显示高表达于乙肝组中的基因显著富集于“生物学过程”子集中(Biological Process,BP),在Bonferroni、Benjamini矫正后,富集水平仍然显著。而在丙肝组上调基因的分析中并未发显著的基因功能交叉(表 3)。
| 表 3 乙肝相关性肝癌组上调基因的DAVID分析结果 |
为进一步探讨HBV相关性HCC和HCV相关性HCC可能的分子机制差异,我们利用GSEA分析法对GSE44074数据集中两种不同病毒相关的肝癌样本进行了分析,以全面探索二者间可能存在的功能基因集差异,而不仅局限于差异表达基因有关的生物学功能。结果显示,相较于乙肝组,丙肝组富集了结构蛋白活性(STRUCTURAL_MOLECULE_ACTIVITY)、线粒体呼吸链(MITOCHONDRIAL_RESPIRATORY_CHAIN)、细胞器内膜(ORGANELLE_INNER_MEMBRANE)及线粒体膜(MITOCHONDRIAL_MEMBRANE_PART)相关的功能基因集(表 4和图 2)。这一结果提示,在致病机制上,HCV相关的肝癌显著富集于上述功能基因集,提示HBV、HCV相关性肝癌发展过程在以上生物学过程中存在差异。
| 表 4 HBV相关性肝癌和HCV相关性肝癌GSEA分析结果 |
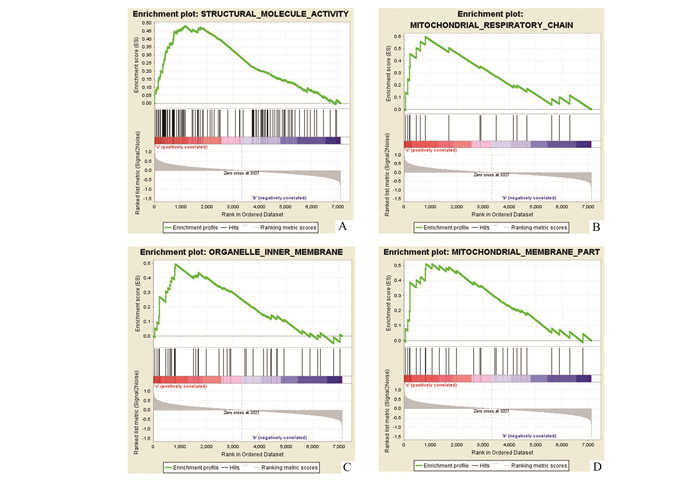
|
图 2 GSEA分析结果显示HCV相关性HCC显著富集的基因集 |
原发性肝癌的发病率较高,全球每年约有50多万例男性新发病例和20多万例女性新发病例,为男性发病率第5、女性发病率第9的癌症[1]。原发性肝癌已成为世界上第二大高致死率的癌症,其进展迅速、致死率高,据估计全球每年约有70多万患者死于肝癌[1]。乙肝和丙肝病毒所致的慢性感染是肝细胞癌(HCC)的最重要的风险因素之一。随着科技的发展,乙肝疫苗的普及和生活水平的极大改善,发展中国家HBV相关性HCC的发病率已经得到了很好的控制。而近年来,许多发达国家中由肥胖和糖尿病等与HCV感染有关的HCC的发病率却有所增加[1]。
HBV和HCV感染均与肝癌发病密切有关,二者主要的致病机理与病毒持续感染所引起的炎症反应、感染细胞的免疫清除及肝细胞再生等生物过程有关,慢性感染期的各种炎症反应等可能引起细胞在遗传或表观遗传学水平发生变化进而引起肝癌。然而,HBV和HCV的生物学分类、结构、表达产物、感染肝细胞的方式及复制方式等均截然不同,其导致HCC的机制也不尽相同。乙肝病毒属嗜肝病毒科,是一种DNA病毒,而丙肝病毒属黄病毒科肝炎病毒,为有包膜的、单正链RNA病毒。在免疫应答方面,HCV主要诱发Ⅰ型或Ⅲ型干扰素有关的炎症反应,参与抗病毒天然免疫的淋巴细胞亚型主要有NK细胞、CD8+和CD4+ T淋巴细胞等。而近来有研究认为,HBV早期感染所引起的免疫反应主要与IL-6有关,而非干扰素,而NK细胞和NKT细胞是HBV感染早期的主要免疫应答细胞[5]。
除免疫应答方式的差异外,HBV和HCV的编码产物及其复制方式在肝癌发展过程中的作用也存在差异。持续表达的野生型或截断HBx蛋白是HBV引起HCC的重要编码产物,HBx可上调多种转录因子的表达水平,进而参与细胞周期调控[6]、DNA损伤修复[7]、氧化应激反应[7]和细胞凋亡[8]等多种信号转导通路,直接参与了HCC的发展过程。此外,HBV的基因组与宿主基因组的整合也是HBV引起HCC的重要机制[9]。
不同于HBV,HCV的复制不需要进入细胞核,其基因组不会与宿主DNA发生整合。此外,虽然HCV的某些编码蛋白如HCV核心蛋白[10],及其非结构蛋白如NS5B [11]等可影响胞内信号转导、影响多种蛋白的表达水平等,进而引起细胞代谢或细胞周期失调,能够促进HCV感染的病理发展,参与HCC的发生,但目前的研究认为HCV的编码蛋白并没有直接的致癌作用。
由此可见,HBV和HCV在HCC发展过程中的作用各有不同。了解不同病毒在肿瘤等疾病发展过程中作用机制的差异,不仅对了解该病毒的致病机理十分重要,对治疗不同病毒相关性肝癌也具有重要意义。
基因芯片技术已成为多种生物、医学研究的重要工具。随着GEO、SMD(Stanford MicroArray Database)、Array Express等多个基因芯片数据库的建立,利用公用数据库进行基因表达谱的信息挖掘已成为研究多种疾病发病机制的重要手段。本研究采用生物信息学分析方法,利用公用数据库,运用在线分析软件GEO2R筛选出HBV相关性肝癌和HCV相关性肝癌的差异表达基因,并利用DAVID数据库分析了这些基因可能参与的生物过程,发现相较于HCV相关性HCC,在HBV相关型HCC高表达的基因与多细胞组织加工(regulation of multicellular organismal process)和生物学过程负向调控(negative regulation of biological process)有关。
基因组富集分析(GSEA)能在基因组水平上挖掘不同分组数据中相关的基因通路,即分析来自某一基因表达谱中处于两种不同特性的数据,了解各组在特定功能基因集中的表达状况,并确定这种表达差异是否具有统计学意义[4]。本研究利用GSEA分析方法,对HBV和HCV相关性HCC进行了基因功能集富集分析,以进一步分析二者间可能存在的功能基因集差异,而不局限于与差异表达基因有关的生物学功能。分析结果提示HBV相关性肝癌和HCV相关的肝癌在结构蛋白活性,线粒体呼吸链、细胞器膜蛋白及线粒体膜功能的基因功能集上存在显著差异,且HCV相关性HCC为上调组。
本研究利用GEO公用数据库及多种生物信息学分析手段,对HBV和HCV相关性肝癌在发病机制上可能存在的差异进行了分析。本研究中所获得的线索仍需更大量的临床样本、结合多种临床因素进行综合验证、分析,以更加深入、准确地了解HBV相关性HCC和HCV相关性HCC的致病机制和二者所存在的差别。
| [1] | Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J]. CA: a cancer journal for clinicians, 2015, 65(2): 87-108. DOI: 10.3322/caac.21262. |
| [2] | Ueda T, Honda M, Horimoto K, et al. Gene expression profiling of hepatitis b-and hepatitis c-related hepatocellular carcinoma using graphical gaussian modeling[J]. Genomics, 2013, 101(4): 238-248. DOI: 10.1016/j.ygeno.2013.02.007. |
| [3] | Huang da W, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using david bioinformatics resources[J]. Nature Protocols, 2009, 4(1): 44-57. |
| [4] | Subramanian A, Tamayo P, Mootha VK, et al. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles[J]. PNAS USA, 2005, 102(43): 15545-15550. DOI: 10.1073/pnas.0506580102. |
| [5] | Hosel M, Quasdorff M, Wiegmann K, et al. Not interferon, but interleukin-6 controls early gene expression in hepatitis B virus infection[J]. Hepatology, 2009, 50(6): 1773-1782. DOI: 10.1002/hep.23226. |
| [6] | Idrissi ME, Hachem H, Koering C, et al. Hbx triggers either cellular senescence or cell proliferation depending on cellular phenotype[J]. Journal of Viral Hepatitis, 2016, 23(2): 130-138. DOI: 10.1111/jvh.12450. |
| [7] | Higgs MR, Chouteau P, Lerat H. 'Liver let die': Oxidative DNA damage and hepatotropic viruses[J]. The Journal of General Virology, 2014, 95(Pt 5): 991-1004. |
| [8] | Zhao J, Wu G, Bu F, et al. Epigenetic silence of ankyrin-repeat-containing, sh3-domain-containing, and proline-rich-region-containing protein 1 (aspp1) and aspp2 genes promotes tumor growth in hepatitis b virus-positive hepatocellular carcinoma[J]. Hepatology, 2010, 51(1): 142-153. DOI: 10.1002/hep.23247. |
| [9] | Levrero M, Zucman-Rossi J. Mechanisms of hbv-induced hepatocellular carcinoma[J]. Journal of Hepatology, 2016, 64(1 Suppl): S84-S101. |
| [10] | Miyoshi H, Moriya K, Tsutsumi T, et al. Pathogenesis of lipid metabolism disorder in hepatitis C: Polyunsaturated fatty acids counteract lipid alterations induced by the core protein[J]. Journal of Hepatology, 2011, 54(3): 432-438. DOI: 10.1016/j.jhep.2010.07.039. |
| [11] | Mitchell JK, Lemon SM, McGivern DR. How do persistent infections with hepatitis c virus cause liver cancer?[J]. Current Opinion in Virology, 2014, 14: 101-108. |
 2017, Vol. 38
2017, Vol. 38

