自闭症(Autism)是小儿广泛性发育障碍的一种亚型,主要表现为交流障碍、语言障碍、兴趣范围狭窄和刻板行为[1]。近二十年来,其发病率由0.01%-0.02% 增加到1%-2.6%[2]。 DNA甲基化是一种研究最为广泛的表观遗传标记,可以调控基因的差异性表达、细胞分化、胚胎发育以及基因组印记[3]。研究发现在自闭症患者中存在CpG结合蛋白2(MECP2),神经元特异性烯醇化酶(ENO2),维甲酸相关孤核受体(RORA)等基因启动子区域甲基化异常改变 。这些研究表明DNA甲基化在自闭症患者发病过程中起着重要作用。金属硫蛋白(metallothionein,MT)是一类富含半胱氨酸的短肽,由约60个氨基酸组成,对多种重金属有高度亲和性。MT根据其亚基组成不同分为MT1、MT2、MT3和MT4四种异构体,其中MT3与多种神经退行性疾病相关,在阿兹海默症、唐氏综合征及帕金森病中表达均发生异常改变。MT3不仅在急性脑损伤中表达异常,在大脑慢性变化和先天性异常中也起着重要作用,因此本课题组探讨MT3基因与自闭症的关系。本研究用基质辅助激光解析电离时间飞行质谱仪(MALDI-TOF MS)定量检测MT3基因启动子区域部分CpG位点甲基化,并采用实时荧光定量PCR检测外周血白细胞中MT3基因的表达量,探讨MT3基因与自闭症的关系。
1 资料与方法 1.1 样本收集收集湖北武汉地区2012年1月到2014年10月间自闭症患者33人,其中男童30例,女童3例,年龄2-12岁,平均年龄(4.6±2.2)岁。经医院伦理委员会批准及其监护人知情同意,采集2 ml EDTA-Na抗凝静脉血。并收集同期于武汉大学中南医院就诊和体检的健康儿童39例,其中男童30例,女童9例,年龄2-12岁,平均年龄(5.2±3.2)岁,采集EDTA-Na抗凝静脉血2 ml作为对照。纳入标准:①由两位临床医生独立诊断,入选患者必须符合《精神障碍诊断和统计手册》第四版(DSM-Ⅳ)的诊断;②所有入选患者由临床医师根据儿童孤独症评定量表(CARS)对患儿进行评估,CARS总分大于30的患者纳入研究;③年龄在2-12岁之间。排除标准:①不符合《精神障碍诊断和统计手册》第四版的诊断;② CARS总分小于30分;③年龄大于12岁或小于2岁。③排除患有精神类疾病及Asperger's 综合征,Rett综合征、精神发育迟缓等疾病的儿童。
1.2 研究方法 1.2.1 DNA提取将收集的EDTA-Na抗凝静脉血低温4 ℃离心(2 000 g)10 min,分离血浆,置于-80 ℃冰箱保存备用。取部分血细胞用QIAmp DNA Blood Mini Kit (QIAGEN,Shanghai,China),根据说明书操作提取DNA。提取的DNA采用Nanodrop 2000紫外分光光度计(Thermo,USA)测定DNA的浓度与纯度,选取A260/230的比值在接近1.8,A260/230的比值在2.0附近,DNA浓度大于20×106 ng/L于-20 ℃保存备用。
1.2.2 亚硫酸氢盐处理DNA用EZ DNA 甲基化处理试剂盒(Zymo Research,Orange County,CA)处理DNA,按照试剂盒提供步骤操作。
1.2.3 DNA甲基化定量分析采用EpiDesigner软件(http://www.epidesigner.com)设计覆盖MT3基因启动子区域的引物,上游引物为5′-AGGAAGAGAGGGTAGGGGTAGGTTTTAGGGTT-TAT-3′,下游引物5′-CAGTAATACGACTCACTATAGGGAGAAGGCTTAAACAAATTCTTTC-TACCCCCAAT-3′,扩增片段大小为565 bp。亚硫酸氢盐处理的DNA采用PCR进行扩增,扩增条件为:预变性94 ℃ 4 min、94 ℃ 20 s、56 ℃ 30 s、72 ℃ 60 s,40个循环。扩增后的产物进行虾碱性磷酸酶SAP处理,SAP处理后的产物进行体外转录及T裂解,再进行预处理,最后采用MALDI-TOF-MS定量检测MT3启动子甲基化率。使用MassARRAY Nanodispenser(Sequenom,San Diego)从384孔板裂解反应液中取出22 μl自动装入基质芯片(Spectro CHIPs,Sequenom,San Diego),运用MassARRAY质谱仪(Bruker-Sequenom)收集质谱图,采用Epityper v1.0.5软件(Sequenom,San Diego,CA,USA)进行分析。
1.2.4 实时荧光定量PCR检测用Trizol法提取RNA,提取的RNA用NanoDrop 2 000(Thermo,USA)测定其浓度及纯度。RNA用Takara公司提供的逆转入试剂盒逆转录成cDNA。引物由Primer Premier 5.0软件设计,MT3基因上游引物为5′- TGCCCCTGCCCTTCTGGT 3′,下游引物为5′-GCTTCTGCCTCAGCTGCCTC-3′。以3-磷酸甘油醛脱氢酶(GAPDH)为内参基因,上游引物为5′-GAAGGTGAAGGTCGGAGTC-3′,下游引物为5′-GAAGATGGTGATGGGATTTC-3′。反应体系为20 μl,其中cDNA 1.0 μl、上下游引物各0.6 μl、SYBR Green supermix(Bio-rad,Hercules,CA,USA)10.0 μl以及去离子水7.8 μl。反应条件为:95 ℃ 30 s、62 ℃ 30 s 、72 ℃ 30 s,循环数为40。定量计算公式:2-△Ct,其中△Ct=Ct (MT3)-Ct(GAPDH)。
1.3 统计学方法采用SPSS 20.0软件对数据进行统计学分析,连续性变量采用 x±s 表示。每个CpG位点甲基化率分析采用Mann-Whitney U检验,运用HemI1.0软件进行聚类分析。MT3 mRNA的表达水平采用独立样本t检验。MT3基因甲基化率与mRNA表达的相关性采用Pearson秩相关分析法。P<0.05为差异有统计学意义。
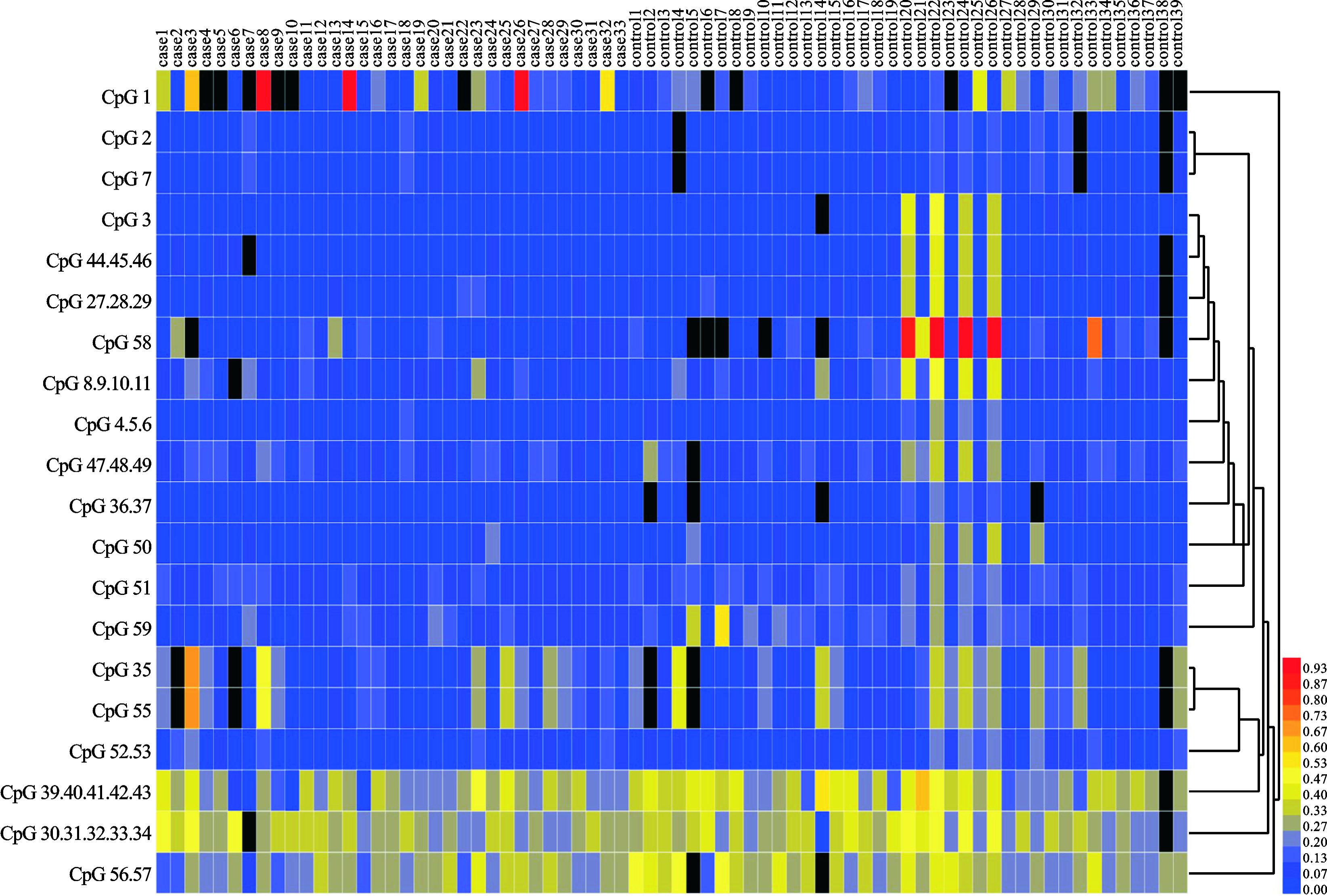
2 结果 2.1 MT3启动子CpG片段甲基化检测结果以MT3基因(转录本ENST00000200691)5′-UTR第一个碱基即转录起始位点为+1,甲基化检测设计的引物覆盖范围为-317-+247 bp。包含42CpG个位点,20个CpG单位(图 1)。通过MassARRY显示MT3基因启动子上有15个CpG 位点甲基化程度与对照组比较差异有统计学意义,其余位点差异无统计学意义,具体结果见表 1。应用聚类分析描绘自闭症患者与对照组MT3基因中20个CpG 单位甲基化水平的分布趋势,从聚类分析图(图 2)中可以看出自闭症患者甲基化水平低于对照组。

|
图 1 检测MT3启动子序列信息 图中序列为反向序列,圆点代表CpG位点,蓝色标记的CpG位点,可以检测到。 |
| 表 1 MT3基因启动子各位点的甲基化率比较(x±s) |
|
图 2 MT3基因启动子甲基化水平聚类分析 图标中纵向代表每个标本,横向代表每一个CpG 单元。其中色彩反映甲基化程度:黑色代表CpG 位点未被分析,红色代表甲基化率为100%,蓝色为甲基化率为0 |
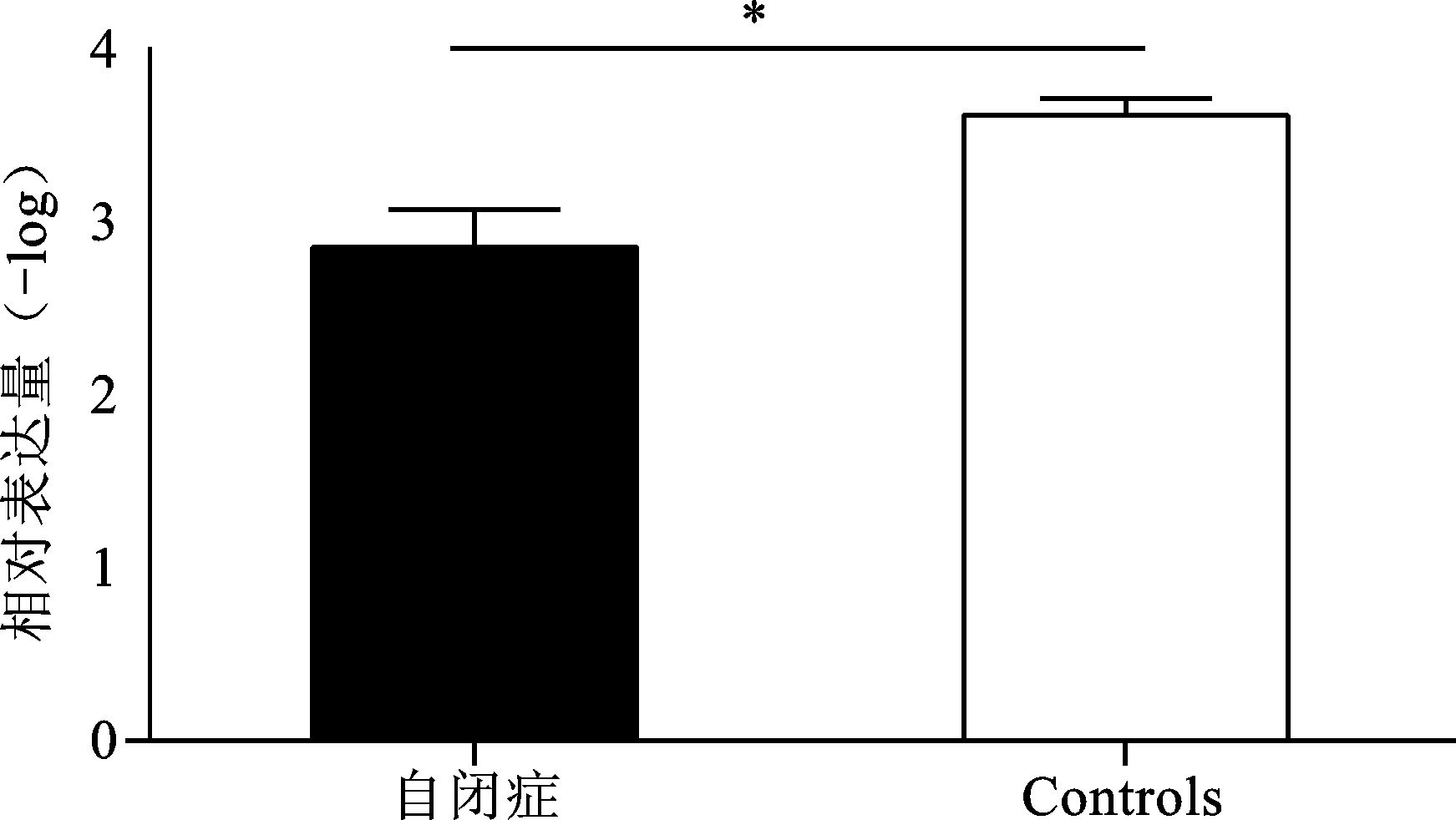
通过实时荧光定量PCR分析发现,MT3 mRNA相对表达水平在病例组和对照组之间有差异(P=0.002)。在自闭症患者中MT3 mRNA表达高于对照组(图 3)。
|
图 3 自闭症与正常对照组中MT3 mRNA表达量比较 *P<0.05 |
对MT3基因甲基化率与其mRNA表达进行相关性分析显示,MT3基因CpG 39.40.41.42.43位点低甲基化与其mRNA相对表达量呈负相关(r=-0.308,P=0.031),其余位点与表达无相关性(P>0.05)。
3 讨论自闭症是一种终身性疾病,症状较轻的患者生活可以自理,较重的患者则需要终身的医疗护理,给社会和家庭带来了极大的负担,因此引起了人们的极大关注。尽管自闭症的病因不明,但是人们普遍认为大脑结构及中枢神经系统受损、遗传因素、免疫功能损伤、心理因素和环境因素与自闭症的发病机制有关。其中遗传因素和环境因素更为重要,且遗传与环境交互作用提示,表观遗传改变可能是环境暴露相关疾病的发病机制之一,因此表观遗传学在自闭症的发病因素中起着重要作用。DNA甲基化是重要的表观遗传学形式之一,与多种疾病的发生发展相关联,在自闭症中表观遗传学起着重要作用。DNA甲基化是指在DNA甲基化转移酶的作用下,在基因组CpG二核苷酸的胞嘧啶5′碳位共价键结合一个甲基基团。大量研究表明可以利用DNA甲基化作为靶点治疗疾病,如5-氮杂胞苷(5-azacytidine)已成为治疗高危骨髓增生异常综合征的一线药物[7]。MT的主要功能是转运体内金属离子,尤其是在脑组织中,MT的功能损害可以引起多种疾病,如自闭症、强迫症等[8]。在自闭症患者中,研究发现MT蛋白及其anti-MT IgG含量增加,也有研究表明增加MT1、MT2的表达是治疗自闭症的一种新的方法[9]。关于MT3与自闭症的关系还没有报道过,本课题组着重探索了一些CpG位点甲基化的变化及MT3基因表达与自闭症的关系。
本研究检测了MT3启动子区20个CpG单位的甲基化水平,发现多个CpG位点甲基化率在自闭症患者中低于对照组。这些结果表明,一些特殊CpG位点低甲基化在自闭症的病因中发挥了更加重要的作用。研究表明DNA 甲基化水平和环境中重金属有密切的关系[10],自闭症患者中体内铅、镉、镍等多种重金属元素含量升高[11]。Takiguchi 等发现用镉处理大鼠TRL1215肝脏细胞,短时间内(1周)可以使基因组DNA 发生了低甲基化[12]。因此,我们推测在自闭症患者中异常升高的重金属元素可以影响MT3基因的甲基化。本课题组前期实验也检测了MT3 mRNA的相对表达量,发现病例组中的表达量高于对照组,但差异没有统计学意义,可能是由于样本量过小,但在本实验中差异具有统计学意义。因此,在今后实验中该加大样本量,进一步探索MT3与自闭症的关系。研究表明,DNA异常甲基化能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达。因此,我们认为MT3启动子的部分CpG位点低甲基化影响了MT3 mRNA的表达,但其具体机制有待进一步研究。
综上所述,本研究结果表明MT3基因启动子区位特定位点异常甲基化与自闭症相关。MT3基因启动子区低甲基化能影响其mRNA的表达,具体作用机制有待进一步研究。
| [1] | Subramanian M, Timmerman CK, Schwartz JL, et al. Characterizing autism spectrum disorders by key biochemical pathways[J]. Front Neurosci, 2015, 9: 313. |
| [2] | Nakai N, Otsuka S, Myung J, et al. Autism spectrum disorder model mice: Focus on copy number variation and epigenetics[J]. Sci China Life Sci, 2015, 58(10): 976-984. DOI: 10.1007/s11427-015-4891-7. |
| [3] |
史玉杰, 李庆贺, 刘晓辉. DNA甲基化与基因表达调控研究进展[J].
中国生物工程杂志, 2013, 33(7): 90-96.
Shi YJ, Li QH, Liu XH. Progress in Studies of DNA Methylation and Gene Expression Regulation[J]. China Biotechnology, 2013, 33(7): 90-96. |
| [4] | Nguyen A, Rauch TA, Pfeifer GP, et al. Global methylation profiling of lymphoblastoid cell lines reveals epigenetic contributions to autism spectrum disorders and a novel autism candidate gene,RORA,whose protein product is reduced in autistic brain[J]. FASEB J, 2010, 24(8): 3036-3051. DOI: 10.1096/fj.10-154484. |
| [5] | Wang Y, Fang Y, Zhang F, et al. Hypermethylation of the enolase gene (ENO2) in autism[J]. Eur J Pediatr, 2014, 173(9): 1233-1244. DOI: 10.1007/s00431-014-2311-9. |
| [6] | Olson CO, Zachariah RM, Ezeonwuka C, et al. Brain region-specific expression of MeCP2 isoforms correlates with DNA methylation within Mecp2 regulatory elements[J]. PLoS One, 2014, 9(3): e90645. DOI: 10.1371/journal.pone.0090645. |
| [7] | Yang X, Lay F, Han H, et al. Targeting DNA methylation for epigenetic therapy[J]. Trends Pharmacol Sci, 2010, 31(11): 536-546. DOI: 10.1016/j.tips.2010.08.001. |
| [8] | Marx G, Gilon C. The Molecular Basis of Memory[J]. ACS Chem Neurosci, 2012, 3(8): 633-642. DOI: 10.1021/cn300097b. |
| [9] | Ghanizadeh A. Gold implants and increased expression of metallothionein-Ⅰ/Ⅱ as a novel hypothesized therapeutic approach for autism[J]. Toxicology, 2011, 283(1): 63-64. DOI: 10.1016/j.tox.2011.02.002. |
| [10] |
曹家雪, 张红平, 杜立新. 环境因素对DNA甲基化的影响[J].
遗传, 2013, 35(7): 839-846.
Cao JX, Zhang HP, Du LX. Influence of environmental factors on DNA methylation[J]. Hereditas, 2013, 35(7): 839-846. |
| [11] | Al-Farsi YM, Waly MI, Al-Sharbati MM, et al. Levels of Heavy Metals and Essential Minerals in Hair Samples of Children with Autism in Oman: a Case-Control Study[J]. Bio Trace Elem Res, 2013, 151(2): 181-186. DOI: 10.1007/s12011-012-9553-z. |
| [12] | Takiguchi M, Achanzar WE, Qu W, et al. Effects of cadmium on DNA-(Cytosine-5) methyltransferase activity and DNA methylation status during cadmium-induced cellular transformation[J]. Exp Cell Res, 2003, 286(2): 355-365. DOI: 10.1016/S0014-4827(03)00062-4. |
 2017, Vol. 38
2017, Vol. 38

