2. 武汉大学药学院/组合生物合成与新药发现教育部重点实验室 湖北 武汉 430071
2. Key Laboratory of Combinatorial Biosynthesis and Drug Discovery, Ministry of Education, & School of Pharmaceutical Sciences, Wuhan University, Wuhan 430071, China
脂肪酸是生物体存在所必需的一种物质,承担了多种重要的生理功能:例如细胞膜的形成必须要有脂肪酸的参与以形成磷脂;脂肪是细胞内一种重要的储能物质;脂肪酸还被认为是脂质信号传导的重要组成部分之一[1]。得益于脂肪酸较高的能量密度和低水溶性,脂肪酸及其衍生物被认为是一类良好的化石燃料替代品;通过改造微生物来生产脂肪酸或脂肪酸衍生物如脂肪醇[2]、脂肪酸酯[3]、脂肪烯烃和脂肪烷烃[4]等作为可再生能源或化工原料也一直是研究热点。此外,脂肪酸还与许多疾病或代谢紊乱的发生与发展息息相关,如肥胖[5]、糖尿病[6]和心血管疾病[7, 8]等。由于脂肪酸在细胞膜合成中的重要作用,在药物研发中脂肪酸代谢途径也被作为靶标来开发新型抗菌疗法[9, 10]。
目前对于脂肪酸合成途径已有较清晰的认识[11]。但由于脂肪酸合成中挂载在酰基载体蛋白(acyl carrier protein, ACP)上的中间产物在胞内含量较低且很不稳定,已有的研究手段难以确定其具体结构,阻碍了对脂肪酸的生物合成途径更深入的了解。为解决这一问题,本研究通过一种硫酯反应来捕获脂肪酸合成中转瞬即逝的中间代谢产物,将中间代谢产物酯解为较为稳定的脂肪酰丁胺,再辅以高分辨质谱检测来获得脂肪酸生物合成中中间代谢物的准确结构信息,为进一步深入了解脂肪酸代谢途径提供帮助。
1 材料与方法 1.1 实验材料试剂包括棕榈酰辅酶A,乙酰辅酶A,丙二酰辅酶A,棕榈酸,叠氮基磷酸二苯酯,三乙胺,二氯甲烷,N-乙酰基半胱胺,NADPH,TCEP,N,O-Bis(trimethylsilyl)acetamide (BSA), n-丁胺购买于Sigma Aldrich公司,其余试剂均为国产或进口分析纯试剂。所用到的10种大肠杆菌来源的脂肪酸合成系统相关蛋白及棕榈酰-SNAC均为本实验室制备并保藏。
实验仪器包括HypersilGOLD高效液相色谱柱(1.9 μm, 150×2.1 mm),LTQ Orbitrap液相色谱高分辨质谱连用系统(美国Thermo Scientific公司)。
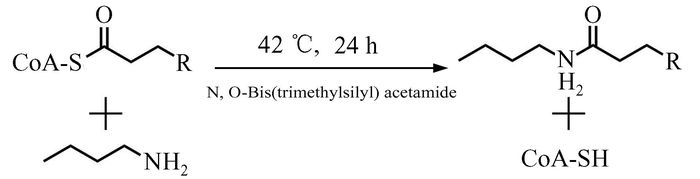
1.2 实验方法 1.2.1 体外硫酯反应的方法将棕榈酰辅酶A溶于水,配制成终浓度为10 mmol/L的溶液。取5 μl上述棕榈酰辅酶A溶液,1 μl BSA和1 μl n-丁胺加入20 μl乙醇中,混合均匀后在42 ℃静置反应24 h。
1.2.2 脂肪酸体外合成体系的建立脂肪酸体外合成体系参考之前报道的方法[12]建立,其反应体系如下:FabA,FabB,FabD,FabF,FabG,FabH,FabI,FabZ,Holo-ACP,TesA蛋白各1 μmol/L,乙酰辅酶A 0.2 mmol/L,丙二酰辅酶A 0.5 mmol/L,NADPH 1 mmol/L,TCEP 1 mmol/L;反应在磷酸盐缓冲液(50 mmol/L,pH=7.6) 中进行。反应温度为37 ℃。
1.2.3 硫酯反应捕获脂肪酸合成的中间代谢产物取上述2 ml反应30 min后的体外反应混合液,加入400 μl BSA,400 μl n-丁胺和7.2 ml乙醇,混合均匀后在42 ℃反应24 h。反应结束后向反应液中加入10 ml乙酸乙酯进行萃取,离心后取有机相浓缩后进行高分辨质谱分析检测硫酯反应捕获的脂肪酸合成中间代谢产物。
1.2.4 高效液相色谱分离方法待测硫酯反应产物首先在流动相中进行梯度洗脱。其中,流动相A为水[含0.1%(V/V)乙酸],B为乙腈[含0.1%(V/V)乙酸];梯度洗脱程序为:0-3 min,80% A;4-15 min,20%-100% B;15-20 min,100% B;20-22 min,20% B。本研究中所有的液相色谱分离均使用上述的分离方法及相同的色谱柱。
1.2.5 硫酯反应产物的质谱检测方法离子源:可加热型电喷雾离子源(HESI);质量分析器:FT Orbitrap;毛细管温度:350 ℃;喷雾电压:3.0 kV;管状透镜电压:80 V;鞘气(N2)压力:2.067×105 Pa;辅助气(N2)流量:200 L/h;扫描采用平行模式进行其扫描质量范围(m/z)为:100-1 000,再对其中响应较强的二级碎片离子进行线性离子阱扫描;归一化能量:35%。
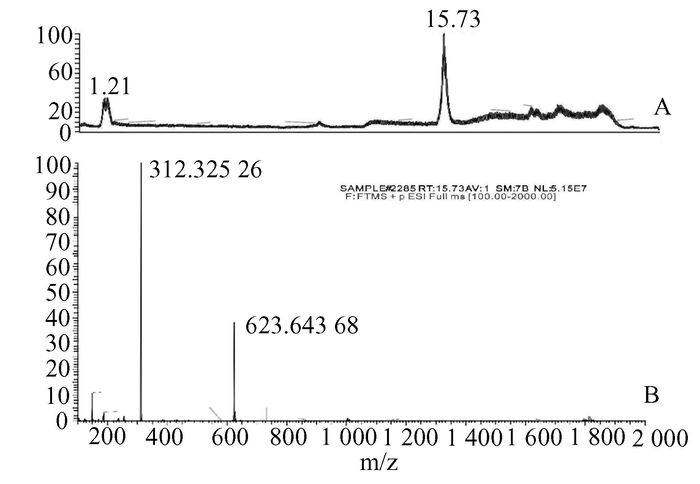
2 结果 2.1 棕榈酰辅酶A的硫酯反应结果为了验证硫酯反应催化天然硫酯类化合物的情况,首先选用了脂肪酸代谢途径和生物体内广泛存在且性质较为活泼的脂酰辅酶A——棕榈酰辅酶A作为底物进行反应。其反应体系和反应示意图如图 1所示。反应24 h后使用高分辨质谱对反应情况进行检测,检测结果如图 2所示,产物的相对分子质量为312.326 09,与理论产物C20H42NO+的相对分子质量312.322 17相符。质谱结果证明该硫酯反应成功将棕榈酰辅酶A转化成为预期产物,且根据总离子流图可以判断在该反应条件下反应较为彻底,底物几乎全部转化为了反应产物。
此外,由于后续生物样品检测中的中间代谢产物积累很少,虽然质谱检测非常灵敏,但也需要确定该反应产物的检测限问题。因此,通过将硫酯反应原料棕榈酰辅酶A的投料量逐级减少的方法确定了目标产物在该条件的检测限,结果显示0.18 μmol/L的棕榈酰辅酶A为该反应的最低检测浓度。
|
图 1 硫酯反应示意图 |
|
图 2 质谱检测以棕榈酰辅酶A为底物的硫酯反应结果 A:总离子流图;B:二级质谱图 |
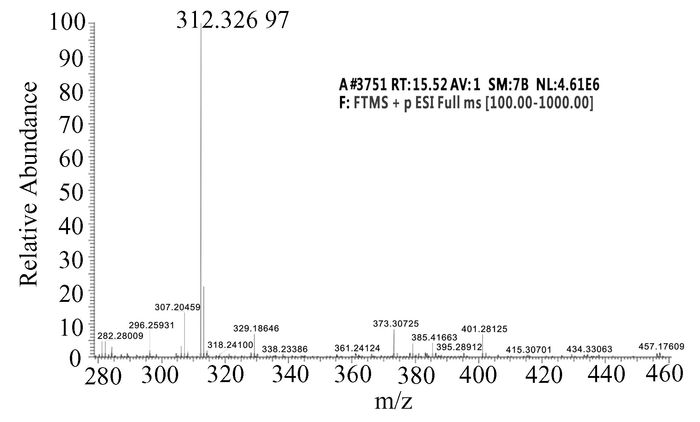
本研究最终目标为酯解产物通过硫酯键连接在ACP上的脂肪酸中间代谢产物,而此类脂酰ACP中间产物性质十分不稳定,较难制备。因此在验证了该硫酯反应对于棕榈酰辅酶A的可行性之后,我们进一步使用了一种更接近于生物体内实际的脂酰ACP的底物类似物即棕榈酰-SNAC作为反应底物进行硫酯反应,以此来验证反应的可行性。结果显示该反应同样能够将棕榈酰-SNAC酯解为预期目标产物,其质谱检测结果如图 3所示。
|
图 3 质谱检测以棕榈酰SNAC为底物的硫酯反应产物二级质谱图 |
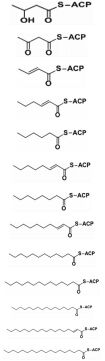
在成功将该反应应用于棕榈酰辅酶A和棕榈酰-SNAC之后,我们进一步将该反应扩展到了实际的脂肪酸生物合成系统当中。基于本课题组之前建立的大肠杆菌脂肪酸合成体外重建系统,我们构建了一个反应速率较为平缓的系统以期通过硫酯反应来捕获更多种类的中间产物。通过高分辨质谱检测,共检测到了13种中间代谢产物,表 1显示了所鉴定到的13种中间代谢产物以及相对应的脂肪酸合成的中间代谢产物结构。
| 表 1 脂肪酸生物合成体系硫酯反应检测到的产物及对应的中间代谢产物 |
目前已确定脂肪酸生物合成体系有两种形式:一种是Ⅰ型脂肪酸合成系统,其多个催化功能单元在一条肽链上,该系统主要存在于真菌及高等动物的细胞质中;另一种为Ⅱ型脂肪酸合成系统,其每个催化功能单元都是一个独立的蛋白,该系统主要存在于细菌、植物以及高等生物的线粒体中。在之前研究中发现某些中间代谢物的积累阻滞了脂肪酸的生物合成产量的提高。因此获得中间代谢物的结构信息不仅可以为脂肪酸生物量提供帮助,还可以使我们对脂肪酸生物合成途径的认识更进一步。
尽管脂肪酸合成相关酶的晶体结构已被解析,并且通过体外重建系统得到了大肠杆菌脂肪酸合成途径的稳态动力学数据[12, 13],但由于脂肪酸合成中挂载在ACP上的中间产物在胞内含量较低、稳定性差,其具体结构仍然难以确定。之前的研究中使用Western Blot或MALDI-TOF质谱的方法来研究脂酰ACP的种类也只能给我们大致的碳链长度信息,而无法区分相同链长脂肪酸中间代谢产物碳链上具体的饱和度及是否有羟基的情况[12]。如何确定脂肪酸合成中间产物的结构信息,是目前仍需解决的难题。
小分子化合物如丙二酰辅酶A类似物等[14, 15],可以作为探针截获如聚酮化合物生物合成中的中间代谢产物,而脂肪酸生物合成途径中的中间产物与聚酮化合物生物合成中的中间代谢物都是挂载在ACP,基于此,本研究开发了一种使用n-丁胺将挂载在ACP上的脂肪酸生物合成中间体酯解的方法来检测脂肪酸合成中的中间代谢产物。最终,我们首次鉴定到在脂肪酸生物合成过程中有一定积累的13种不同的中间代谢产物,并通过分析这些中间代谢物的结构发现其中大部分的化合物的碳链上含有双键,部分中间代谢物碳链上连有羟基。这暗示着我们在脂肪酸的生物合成中催化双键还原的烯脂酰ACP还原酶FabI的催化活性是影响反应速率的一个限制因素,因此才有大量含有双键的脂肪酸中间代谢产物的积累。此外,检测到3-羟基脂酰ACP的积累,可能是催化3-羟基脂酰ACP脱水生成2-烯脂酰ACP的3-羟基脂酰ACP脱水酶FabZ的活性不足导致的。本研究得到的这一中间代谢产物信息很好的印证了大肠杆菌体内实验中所观察到的现象,如朱发银等人通过在大肠杆菌中过表达FabZ,FabG,FabI三个基因结果使大肠杆菌脂肪酸产量相较于对照菌株提升了50%以上[16]。
综上,本研究通过硫酯反应检测出了脂肪酸中间代谢产物大部分含有不饱和双键,部分的碳链上含有羟基,导致这两类中间代谢产物的积累可能与烯脂酰ACP还原酶和3-羟基脂酰ACP还原酶的活性不足有关。这些精确信息可以指导后续的菌株改造,并且为更深入地理解代谢途径提供帮助。
| [1] | Berg J, Tymoczko J, Stryer L. Biochemistry[M]. 7th. New York: W.H. Freeman and Company, 2012. |
| [2] | Liu R, Zhu F, Lu L, et al. Metabolic engineering of fatty acyl-ACP reductase-dependent pathway to improve fatty alcohol production in Escherichia coli[J]. Metab Eng, 2014, 22: 10-21. DOI: 10.1016/j.ymben.2013.12.004. |
| [3] | Tao H, Guo D, Zhang Y, et al. Metabolic engineering of microbes for branched-chain biodiesel production with low-temperature property[J]. Biotechnol Biofuels, 2015, 8: 92. DOI: 10.1186/s13068-015-0270-7. |
| [4] | Liu Q, Wu K, Cheng Y, et al. Engineering an iterative polyketide pathway in Escherichia coli results in single-form alkene and alkane overproduction[J]. Metab Eng, 2015, 28: 82-90. DOI: 10.1016/j.ymben.2014.12.004. |
| [5] | Randle PJ. Regulatory interactions between lipids and carbohydrates: the glucose fatty acid cycle after 35 years[J]. Diabetes/Metabolism Reviews, 1998, 14(4): 263-283. DOI: 10.1002/(ISSN)1099-0895. |
| [6] | Samuel VT, Petersen KF, Shulman GI. Lipid-induced insulin resistance: unravelling the mechanism[J]. The Lancet, 2010, 375(9733): 2267-2277. DOI: 10.1016/S0140-6736(10)60408-4. |
| [7] | Hu FB, Bronner L, Willett WC, et al. FIsh and omega-3 fatty acid intake and risk of coronary heart disease in women[J]. JAMA, 2002, 287(14): 1815-1821. DOI: 10.1001/jama.287.14.1815. |
| [8] | Simopoulos AP. The importance of the omega-6/omega-3 fatty acid ratio in cardiovascular disease and other chronic diseases[J]. Exp Biol Med (Maywood), 2008, 233(6): 674-88. DOI: 10.3181/0711-MR-311. |
| [9] | Campbell JW, John E, Cronan J. Bacterial fatty acid biosynthesis: targets for antibacterial drug discovery[J]. Annual Review of Microbiology, 2001, 55(1): 305-332. DOI: 10.1146/annurev.micro.55.1.305. |
| [10] | Singh SB, Jayasuriya H, Ondeyka JG, et al. Isolation, structure, and absolute stereochemistry of platensimycin, a broad spectrum antibiotic discovered using an antisense differential sensitivity strategy[J]. Journal of the American Chemical Society, 2006, 128(36): 11916-11920. DOI: 10.1021/ja062232p. |
| [11] | Magnuson K, Jackowski S, Rock CO, et al. Regulation of fatty acid biosynthesis in Escherichia coli[J]. Microbiological Reviews, 1993, 57(3): 522-542. |
| [12] | Yu X, Liu T, Zhu F, et al. In vitro reconstitution and steady-state analysis of the fatty acid synthase from Escherichia coli[J]. Proc Natl Acad Sci USA, 2011, 108(46): 18643-18648. DOI: 10.1073/pnas.1110852108. |
| [13] | Liu T, Vora H, Khosla C. Quantitative analysis and engineering of fatty acid biosynthesis in E.coli[J]. Metabolic Engineering, 2010, 12(4): 378-386. DOI: 10.1016/j.ymben.2010.02.003. |
| [14] | Spiteller D, Waterman CL, Spencer JB. A method for trapping intermediates of polyketide biosynthesis with a nonhydrolyzable malonyl-coenzyme a analogue[J]. Angewandte Chemie International Edition, 2005, 44(43): 7079-7082. DOI: 10.1002/(ISSN)1521-3773. |
| [15] | Tosin M, Demydchuk Y, Parascandolo JS, et al. In vivo trapping of polyketide intermediates from an assembly line synthase using malonyl carba(dethia)-N-acetyl cysteamines[J]. Chemical Communications, 2011, 47(12): 3460-3462. DOI: 10.1039/c0cc05077f. |
| [16] |
朱发银. 体外重建体系指导下的微生物代谢工程改造[D]. 武汉: 武汉大学, 2014.
ZHU Fayin. In vitro reconstitution system guide for microbial metabolic engineering[D]. Wuhan: Wuhan University, 2014. |
 2017, Vol. 38
2017, Vol. 38

 ,
,