2. 武汉大学人民医院 消化科 湖北 武汉 430060;
3. 湖北省仙桃市第三人民医院消化科 湖北 仙桃 433000
2. Dept. of Gastroenterology, Renmin Hospital of Wuhan University, Wuhan 430060, China;
3. Dept. of Gastroenterology, The Third Renmin Hospital of Xiantao City, Xiantao 433000 , Hubei, China
宫颈癌在我国发病率高,位居女性生殖道肿瘤之首[1],严重威胁女性健康。其中中晚期宫颈癌患者以放化疗为主。但是在临床中逐渐发现,在临床分期和肿瘤分级同等状况下,其放化疗效果也大不相同,肿瘤对放化疗的敏感性有所不同[2]。说明除了临床病理因素,癌细胞自身的分子生物学特征也是潜在的影响宫颈癌治疗的因素。因此,从分子水平发现放化疗敏感因子,揭示肿瘤对放化疗敏感性的分子机制对于宫颈癌的发病和治疗有非常重要的意义。
长链非编码RNA(long non-coding RNA,lncRNA)近年来成为各国学者研究的热点,lncRNA首次由Okazaki发现[3]。在哺乳动物基因组序列中4%-9%的序列产生的转录本是lncRNA(相应的蛋白编码RNA的比例是1%)[4],lncRNA在生物体发育和基因表达中能够精确调控基因功能,逐渐使人们认识到基因的表达受一个调控网络的控制,虽然关于lncRNA的研究进展迅猛,但是人们目前对绝大部分的lncRNA的功能知之甚少。大量研究发现各种肿瘤细胞如膀胱癌、肺癌和宫颈癌等lncRNA的表达存在差异,lncRNA可以作为生物指标进行疾病诊断和预测[4-7]。
本研究应用高通量lncRNA芯片技术分别检测30例宫颈癌患者放化疗前后宫颈组织中lncRNA表达谱的变异,旨在筛选出与宫颈癌放化疗密切相关的lncRNA和基因,从分子水平预测宫颈癌放化疗敏感性标志物,为进一步阐明宫颈癌的发病机制和为宫颈癌的临床治疗提供实验依据。
1 材料与方法 1.1 标本收集选择2013年6月至2015年6月,在武汉大学人民医院妇科住院接受治疗并且经过放化疗后获手术机会的宫颈癌患者30例,年龄32-65岁,中位年龄45岁。病理类型为鳞癌。每例患者手术前均行2个周期的放化疗,收集每例患者放化疗前宫颈活检组织及手术后肿瘤切除组织。标本采集均经过患者家属签署知情同意书并经本医院伦理委员会审核通过。
1.2 材料提取RNA的Trizol(Invitrogen公司);反转录试剂盒和SYBR-Green Real-time PCR MasterMix试剂盒(ABI公司);real-time PCR引物由TaKaRa公司设计并合成;Human lncRNA Microarrays V3.0的芯片选择、探针设计、图像采集、数据分析等由上海康成生物公司完成。
1.3 芯片的选择利用Arraystar公司所开发出的12×135 k的lncRNA芯片,覆盖目前所有权威数据库NCBI Refseq、UCSC Known Gene、RNAdb,NRED等多个数据库的lncRNA种类。
1.4 探针设计Arraystar lncRNA芯片设计的探针为-60 met的长寡核苷酸,这些长寡核苷酸探针在高严格杂交条件下能够得到高灵敏度以及高特异性的理想实验结果。并且,针对每条序列都设计了多条探针,增加了信号的可靠度。
1.5 组织样本总RNA的提取与检测按照Trizol(Invitrogen)说明书,分别取120 mg的组织样本,按照试剂说明,分别从宫颈癌放化疗组织和宫颈癌组织抽提总RNA。通过紫外分光光度计测定吸光度值260,280,230 nm的吸收值,对总RNA进行计算浓度并评估纯度;同时结合甲醛变性凝胶琼脂电泳检测RNA纯度及完整性。
1.6 cDNA样品合成、标记和杂交利用Invitrogen公司提供的cDNA合成试剂盒进行逆转录,合成双链互补DNA;利用Nimblegen公司的单色DNA标记试剂盒进行双链互补DNA的标记,使用分光光度计检测荧光标记效率,以保证后续芯片实验结果的可靠性;利用NimbleGen提供的杂化系统进行排列杂交,然后利用Nimblegen清洗液试剂盒进行清洗。
1.7 图像采集和数据分析利用Agilent Microarray Scanner(Agilent p/n G2565BA)扫描芯片的荧光强度,然后将扫描图像输入Agilent Feature Extraction软件,以进行网格对齐与表达数据分析。通过分位数标准化以及Agilent Feature Extraction软件将表达数据进行规范,后用Agilent Gene Spring Software软件分析。芯片选择,探针设计,图像采集和数据分析由上海康成生物完成。
1.8 Real-time PCR检测lncRNA变化选差异性变化最大的lncRNA通过Real-time PCR进行验证。采用ABI 7300荧光定量PCR仪,SYBR Green荧光染料(ABI公司)掺入法相对定量,同时以内参GAPDH为参照,计算差异表达的倍数。
2 结果 2.1 lncRNA表达芯片结果将30个组织样本使用Agilent Feature Extraction软件(v11.0.1 .1 )获得芯片图,并读值,得到原始数据。使用GeneSpring GX v12.1软件(Agilent Technologies)对原始数据进行Quantile标准化和随后的数据处理。两组样品间具有统计学意义的差异表达lncRNA或差异表达mRNA通过P-value/FDR筛选。两个样品间差异表达lncRNAs或差异表达mRNAs通过Fold Change筛选。并进行层次聚类和关联分析。
Arraystar人类lncRNA芯片V3.0芯片能够检测30 586个lncRNAs和26 109个蛋白质编码转录本,其中lncRNAs是从权威公共转录组数据库(包括Refseq、UCSC knowngenes、Gencode等)和高影响因子论文中仔细挑选的。这些lncRNA中差异表达的lncRNA共23 530条。其中9 649条表达上调,13 881条表达下调,在这些差异lncRNA中,AC079776.2表达上升628.57倍;RP11-255A11.21下降1 224.73倍。14 037个mRNA表达出现差异性变化,分别为6 056个表达上调和7 981个表达下调。在这些mRNA中,MYH11上升3 425.07倍,而GABRD下降3 293.56倍,为变化最多的mRNA。表 1和表 2分别列出了表达量变化最大的前20个lncRNA和mRNA。
| 表 1 表达量变化最大的前20个lncRNAs |
| 表 2 表达量变化最大的前20个mRNAs |
基因本体论(Gene Ontology,GO)是基因功能国际标准分类体系,分为分子功能(Molecular Function),生物过程(Biological Process)和细胞组分(Cellular Component)三部分。
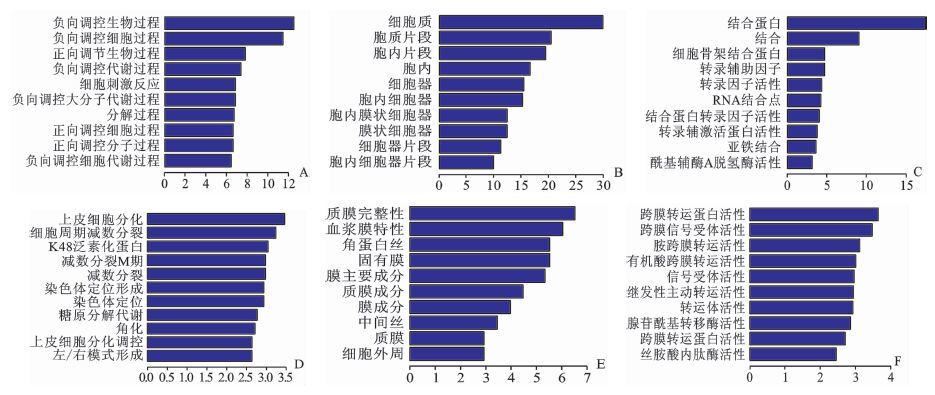
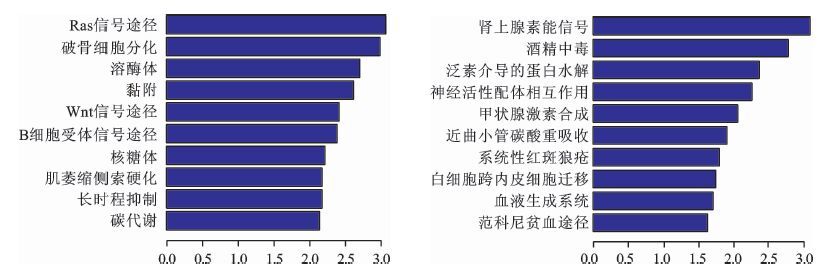
我们通过GO聚类分析发现相对于宫颈癌组织,放化疗后宫颈癌组织中,上调mRNA(Fold>2) 多与细胞膜和细胞器成分相关,具有蛋白结合和细胞骨架蛋白结合等分子功能,参与了负向和正向生物调节、刺激反应、负向大分子代谢等生物过程(图 1A,B,C);下调mRNA(Fold>2) 与细胞膜、细胞器膜和角蛋白纤维成分相关,具有跨膜蛋白激活、受体信号活动、有机胺有机酸膜转移等分子功能,参与了细胞周期、K48泛素化、染色体定位和糖代谢等生物过程(图 1D,E,F)。参考KEGG Pathway,根据分子参与的功能,表达上调的mRNA可能参与了溶酶体、核糖体代谢,细胞贴附和一些信号通路如Ras,Wnt和B细胞受体信号通路等;下调mRNA可能参与激素合成、泛素化蛋白酶解和神经受体配体作用等通路(图 2) 。
|
图 1 mRNA差异表达的Go分析图 A,B和C代表上调mRNA的细胞过程(BP),细胞组分(CC)和分子功能(MF)分析图;D,E和F代表下调mRNA的细胞过程(BP),细胞组分(CC)和分子功能(MF)分析图。 |
|
图 2 上调(左图)和下调(右图)mRNA的通路分析图 |
在lncRNA中,研究得比较深入的是反义(antisense)lncRNA,有超过30%的已注释的人类转录本有相应的反义lncRNA。这些反义lncRNA通过多种机制在转录水平或转录后水平调控相应的正义(sense)mRNA,从而发挥生物学功能。
对此我们分析了变化差异性最明显的20个lncRNA,将差异antisense lncRNAs及其差异sense mRNAs进行整合,以推断lncRNAs的功能(表 3) ,这些lncRNA可能通过影响相应的mRNA来发挥其生物学功能,有可能成为研究宫颈癌放化疗的标志分子。
| 表 3 lncRNA和mRNA共差异表达谱 |
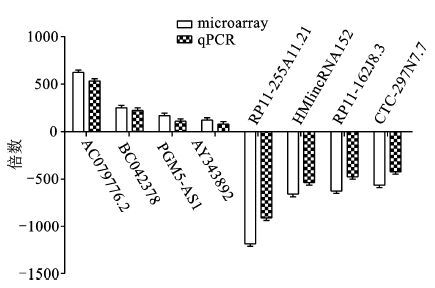
为了验证芯片的结果,我们用GAPDH为内参基因,采用荧光定量PCR法测定各4个变化最明显的上调和下调lncRNA在宫颈癌放化疗组织中表达情况。与宫颈癌组织相比,宫颈癌放化疗组织中基因均出现上调或者下调(图 3) ,其趋势和芯片检测结果一致,证明lncRNA芯片结果可靠。
|
图 3 qRT-PCR检验lncRNA芯片 |
近年来随着测序技术的发展,lncRNA在宫颈癌方面已经有很多研究,一些宫颈癌的标志分子逐渐被提出和发现。如HOTAIR [8, 9],H19[10, 11],P21[12],UCA1[13]和MALAT-1[14]等,这些lncRNA在癌组织中高表达,被认为是癌细胞复发和转移的提示物。我国宫颈癌患者被发现以中晚期为主,而中晚期患者的临床治疗手段主要是手术结合放化疗为主。但是在临床和基础研究中发现即使在患者的病理类型、淋巴结转移、临床分期、治疗时间等临床治疗相同的情况下,个体差异显著,肿瘤对于同步放化疗治疗敏感性不一[15]。
放射肿瘤学认为凋亡也是放射敏感性重要参数[16]。但是一些肿瘤细胞能对射线产生抗性,表现为凋亡率减少,使放疗效果不理想[17],Hong等发现,不同的患者同步放化疗后疗效差异显著,大量患者治疗后出现局部复发和远处转移[18]。因此寻找一些放化疗的标记分子成为今年来各国学者研究的重点。目前应用基因芯片技术在乳腺癌,直肠癌和宫颈癌中筛选与放化疗密切相关基因已有少量报道[19-20],也有一些学者发现一些关键基因如HIF-1等可能与放化疗敏感性有关[22, 23],但是目前尚没有具体的临床和生物学指标特别是lncRNA可以作为放化疗的敏感性指示物。
lncRNA是近年来研究的热点, 本研究中我们借助Arraystar公司的lncRNA芯片, 在临床中收集了30例宫颈癌患者放化疗前后的宫颈癌组织, 分析两组lncRNA表达谱的变化。发现相对宫颈癌组织放化疗组织中差异表达的lncRNA共23 530条。其中9 649条表达上调, 13 881条表达下调。14 037个mRNA表达出现差异性变化, 分别为6 056个表达上调和7 981个表达下调。通过GO分析, 其细胞组分、分子功能和生物过程如下:上调mRNA(Fold>2)多与细胞膜和细胞器成分相关, 具有蛋白结合和细胞骨架蛋白结合等分子功能, 参与了负向和正向生物调节、刺激反应、负向大分子代谢等生物过程(图1A,B,C); 下调mRNA(Fold>2)与细胞膜、细胞器膜和角蛋白纤维成分相关, 具有跨膜蛋白激活、受体信号活动, 有机胺有机酸膜转移等分子功能, 参与了细胞周期、K48泛素化、染色体定位和糖代谢等生物过程(图1D,E,F)。表达上调的mRNA可能参与了溶酶体、核糖体代谢、细胞贴附和一些信号通路如Ras, Wnt和B细胞受体信号通路等; 下调mRNA可能参与激素合成, 泛素化蛋白酶解和神经受体配体作用等通路。并且通过分析变化差异性最明显的20个lncRNA, 发现lncRNA可能通过影响相应的mRNA来发挥其生物学功能, 有可能成为研究宫颈癌放化疗的标志分子。虽然在其他无关通路上的分子我们也检测到存在表达, 但是由于lncRNA芯片在放化疗上目前还没有数据借鉴, 我们后续会核实这些通路是否确实存在。
肿瘤的放化疗敏感性是影响中晚期宫颈癌治疗成败的关键。本研究罗列了与宫颈癌放化疗密切相关的lncRNA及相关转录的分子功能、生物过程和分子生物学通路等,为宫颈癌临床治疗提供数据支持, 为预测宫颈癌放疗敏感性标志物提供分子依据。
| [1] | Brazma A, Hingam PP, Quackenbush J, et al. Minimumin formation about a microarray experiment(MIAME)-toward standards for microarray data[J]. Nat Genet, 2001, 29: 365-371. DOI: 10.1038/ng1201-365. |
| [2] | Boseh FX, Manos MM, Munoz N, et al. Prevalence of human papillomavirus in cervical cancer: a world wide perspective. Intemational Biological Study on Cervical Cancer (IBSCC) Study Group[J]. J Natl Cancer Inst, 1995, 87:796-802. |
| [3] | Okazaki Y, Furuno M, Kasukawa T, et al. Analysis of the mouse transcriptome based on functional annotation of 60770 full-length cDNAs[J]. Nature, 2002, 420(6915): 563-573. DOI: 10.1038/nature01266. |
| [4] | Kung JT, Colognori D, Lee JT. Long non-coding RNAs:past,present and future[J]. Genetics, 2013, 193(3): 651-669. DOI: 10.1534/genetics.112.146704. |
| [5] | Huarte M, Rinn JL. Large non-coding RNAs: missing links in cancer[J]? Hum Mol Gene, 2010, 19(R2): R152-R161. |
| [6] | Tano K, Mizuno R, Okada T, et al. MALAT-1 enhances cell motility of lung adenocarcinoma cells by influencing the expression of motility-related genes[J]. FEBS Lett, 2010, 584(22): 4575-4580. DOI: 10.1016/j.febslet.2010.10.008. |
| [7] | Ozgur E, Mert U, Isin M, et al. Differential expression of long non-coding RNAs during genotoxic stress-induced apoptosis in HeLa and MCF-7 cells[J]. Clin Exp Med, 2013, 13(2): 119-126. DOI: 10.1007/s10238-012-0181-x. |
| [8] | Huang L, Liao LM, Liu AW, et al. Overexpression of long noncoding RNA HOTAIR predicts a poor prognosis in patients with cervical cancer[J]. Arch Gynecol Obstet, 2014, 290(4): 717-723. DOI: 10.1007/s00404-014-3236-2. |
| [9] | Wilson L, Brown JS, Shin GP, et al. Annual direct cost of urinary incontinence[J]. Obstet Gynecol, 2001, 98(3): 398-406. |
| [10] |
王文玲, 高英敏, 高俊丽, 等. 印迹基因H19在宫颈癌中的表达及意义[J].
肿瘤, 2008, 28(1): 89-90.
Wang WL, Gao YM, Gao JL, et al. Expression of H19 imprinting gene in cervica carcinoma and its significance[J]. Tumor, 2008, 28(1): 89-90. |
| [11] | Vidal AC, Henry NM, Murphy SK, et al. PEG1/MEST and IGF2 DNA methylation in CIN and in cervical cancer[J]. Clin Transl Oncol, 2014, 16(3): 266-272. DOI: 10.1007/s12094-013-1067-4. |
| [12] | Farha AK, Dhanya SR, Mangalam SN, et al. Deoxyelephantopin impairs growth of cervical carcinoma SiHa cells and induces apoptosis by targeting multiple molecular signaling pathways[J]. Cell Biol Toxicol, 2014, 30(6): 331-343. DOI: 10.1007/s10565-014-9288-z. |
| [13] | Wang Y, Chen W, Yang C, et al. Long non-coding RNA UCA1a(CUDR) promotes proliferation and tumorigenesis of bladder cancer[J]. Int J Oncol, 2012, 41(1): 276-284. |
| [14] | Lai MC, Yang Z, Zhou L, et al. Long non-coding RNA MALAT-1 overexpression predicts tumor recurrence of hepatocellular carcinoma after liver transplatation[J]. Med Oncol, 2012, 29(3): 1810-1816. DOI: 10.1007/s12032-011-0004-z. |
| [15] | Grigene R, Valuckas KP, Aleknavicius E, et al. The value of prognostic factors for uterine cervical cancer patients treated with irradiation alone[J]. BMC Cancer, 2007, 7: 234. DOI: 10.1186/1471-2407-7-234. |
| [16] | Liu ZZ, Huang WY, Li XS, et al. Prediction value of radiosensitivity of hepatocarcinoma cells for apoptosis and micronucleus assay[J]. World J Gastroenterol, 2005, 11(44): 7036-7039. DOI: 10.3748/wjg.v11.i44.7036. |
| [17] | Otomo T, Hishii M, Arai H, et al. Microarray analysis of temporal gene responses to ionizing radiation in two glioblastoma cell lines: up-regulation of DNA repair genes[J]. J Radiat Res (Tokyo), 2004, 45(1): 53-60. DOI: 10.1269/jrr.45.53. |
| [18] | Hong JH, Tsai CS, Lai CH, et al. Recurrent squamous cell carcinoma of cervix after definitive radiotherapy[J]. International Journal of Radiation Oncology Biology Physics, 2004, 60: 249-257. DOI: 10.1016/j.ijrobp.2004.02.044. |
| [19] | Natowiez R, ncitti R, Horta EG. Prediction of the outcome of preoperative chemotherapy in breast cancer using DNA probes that provide information on both complete and incomplete responses[J]. BMC Bioinformaties, 2008, 9: 149. DOI: 10.1186/1471-2105-9-149. |
| [20] | Schetter AJ, Leung SY, Sohn JJ. MicroRNA expression profiles associated with prognosis and therapeutic outcome in colon adenocarcinoma[J]. JAMA, 2008, 299(4): 425-436. |
| [21] | Grigsby PW, Waston M, Powell MA, et al. Gene expression patterns in advanced human cervical caneer[J]. Int J Gynecol Cancer, 2006, 16(2): 562-556. DOI: 10.1111/ijg.2006.16.issue-2. |
| [22] | Green JA, Kirwan JM, Tiemey JF, et al. Survival and recurrence after concomitant chemotherapy and radio therapy for cancer of the uterine cervix: a systematic review and meta-analysis[J]. Lancet, 2001, 358: 781-786. DOI: 10.1016/S0140-6736(01)05965-7. |
| [23] | Snyder AR, Morgan WF. Gene expression profiling after irradiation: clues to understanding acute and persistent responses[J]?Cancer Metastasis Rev, 2004, 23:259-268. |
 2017, Vol. 38
2017, Vol. 38

