2. 南华大学生命科学院;
3. 湖南师范大学心脏发育研究中心
2. Department of Science Biology, University of South China, Hengyang 421001;
3. Center of Heart Development, College of Life Science, Hunan Normal University, Changsha 410081
TMEM121基因又名Hole,最早在鸡的胚胎心脏中作为心肌特异性高表达因子而被发现[1]。在哺乳动物的胚胎期该基因在多种组织中均有表达,其中在心脏中的表达最高;而在成体期该基因仅在心脏、骨骼肌、肝脏和胰腺中有表达,且心脏仍是表达丰度最高的组织之一。Zhou等进一步利用荧光报告系统对其可能参与的信号通路进行筛查发现,TMEM121能显著的抑制ERK信号通路介导的AP-1与SRE-LUC荧光素酶报告活性[2]。此外同行的研究还发现TMEM121基因内部的PXXP位点的SNP与人类先天性心脏病存在一定的关联:即广东、贵州两地汉族人群中TMEM121基因SNP(rs10569304)位点存在插入/缺失(GCC/---)多态性,其中携带缺失型纯合子基因型的个体患先天性心脏病的风险显著增高[3]。上述研究结果无不暗示TMEM121在心脏功能的调节中发挥重要作用。尽管TMEM121基因作为一个在心脏发育和成体期均有高表达的基因已被发现十余年,但国际同行似乎一直忽略了它在体内的生物学功能,有关TMEM121基因的功能研究迄今仍是一片空白。
近年来生物信息学科的迅猛发展为基因功能研究提供了新的策略。本文利用生物信息学方法对人类TMEM121基因及其蛋白的理化性质及分子结构等进行了分析,并预测了TMEM121在细胞中所参与的功能。生物信息学所获得的TMEM121数据为深入研究该基因的功能提供更多有价值的参考信息。
材料与方法 (一) 材料不同物种TMEM121的编码序列和氨基酸序列的获取
利用NCBI(http://www.ncbi.nlm.nih.gov/)获得人、黑猩猩、猕猴、绿猴、家猫、大鼠、小鼠、家犬、鸡、热带爪蟾等的TMEM121基因的编码序列和氨基酸序列(登录号见表 1)。
| 表 1 不同物种TMEM121的编码和蛋白序列登录号 |
从NCBI-Blastp对比结果中选择几种具有代表性的物种,如人、黑猩猩、猕猴、家猫、家犬等,并下载其氨基酸序列,利用DNAMAN、MEGA 6.0及ClustalX等软件对不同物种进行同源度分析并构建蛋白进化树。
2.2. TMEM121生物信息学分析利用NCBI、ExPASy、ProtFun等公共数据库及在线软件包等分析TMEM121基因的分子结构等,预测其理化性质、信号肽、亲/疏水性、亚细胞定位、激酶磷酸化修饰位点、二级、三级结构及功能等。文中所涉及的数据库及软件如表 2所示。
| 表 2 文中所应用的数据库及软件 |
通过核酸和蛋白质差异来评估不同物种间的亲缘关系,即同源度分析。同源度分值越高表示亲缘关系越近,反之则表示亲缘关系越远。利用NCBI(http://www.ncbi.nlm.nih.gov)获取不同物种TMEM121基因的CDs及蛋白序列,并应用DNAMAN及MEGA软件对不同物种TMEM121蛋白序列进行同源度分析(图 1, A),结果显示人与黑猩猩、猕猴、绿猴、家猫、大鼠、小鼠、家犬、鸡、热带爪蟾的TMEM121蛋白序列同源度分别为99.37%、99.06%、98.12%、97.18%、97.18%、97.18%、96.55%、75.86%和74.92%。选取其中11个不同物种构建蛋白进化树(图 1, B),结果显示TMEM121蛋白在哺乳动物之间具有高度保守性,与鸡、热带爪蟾等的亲缘关系则相对较远, 同源度也高到78%以上。

|
图 1 不同物种间TMEM121蛋白的同源度分析;A、不同物种TMEM121蛋白的序列比对。红色背景内表示其氨基酸残基在各物种间完全一致;蓝色方框内红色字母示氨基酸残基在不同物种间具有相似性。B、不同物种间TMEM121蛋白进化树 |
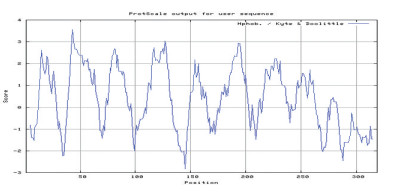
应用EXPASY网站中的ProtParam(http://web.expasy.org/protparam/)在线软件[4]预测分析,得出人类TMEM121蛋白分子式为C1649H2621-N431O418S20,分子量为35813.7 Da,其中亮氨酸含量最高,占序列的15.4%。TMEM121蛋白理论等电点为9.39,其中带负电的酸性氨基酸(Asp和Glu)共24个,带正电荷的碱性氨基酸(Arg和Lys)共34个,因此人类TMEM121蛋白属于碱性蛋白。TMEM121蛋白的网织红细胞半衰期约30h,不稳定系数的阈值设定为40,即小于40表示蛋白质稳定;大于40表示蛋白质不稳定,预测分析得出人TMEM121蛋白的不稳定系数为50.59,因此归类为不稳定蛋白质。Protparam分析得出TMEM121蛋白的脂肪系数为113.04,平均亲水性为0.389。应用EXPASY蛋白分析平台中Protscale在线软件预测人TMEM121蛋白的亲/疏水性, 并绘制亲/疏水性曲线图(见图 2),图中正值表示疏水、负值表示亲水、±0.5之间为两性氨基酸。预测结果显示人TMEM121蛋白最强亲水性位点是第145位的天冬氨酸,亲水性分值为-2.833;最强疏水性位点是位于第43位的异亮氨酸,亲水分值为3.556。由图 2可以看出,TMEM121蛋白序列中疏水氨基酸稍多于亲水性氨基酸,但均值在±0.5之间, 因此为两性氨基酸。
|
图 2 人TMEM121蛋白的亲-疏水性的预测分析 |
应用PSORT Ⅱ(http://www.genscript.com/psort.html)在线软件预测分析,人TMEM121蛋白定位于内质网和线粒体的可能性分别为77.8%和22.2%。
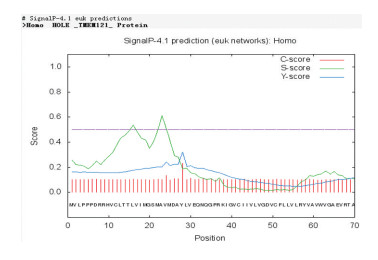
(四) 人TMEM121蛋白的信号肽分析应用SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)在线软件预测分析人TMEM121蛋白有/无信号肽序列[5], 结果(见图 3)。图中C(C-score)代表原始信号肽剪切位点分值,分值越高表示该位点出现剪切位点的可能性越大;S(S-score)代表信号肽分值,分值越高表示该氨基酸位于信号肽区的可能性越大;分值越低,则表示该氨基酸属于成熟蛋白部分或不含信号肽;Y(Y-score)示综合剪切位点分值,是基于C值和S值得出的几何平均值。图中原始剪切位点C值位于第2 8位氨基酸,分值是0.231;信号肽最大S分值为0.611,位于第23位氨基酸;C值和S值的综合值Y值为0.319,位于第28位氨基酸;1-28位氨基酸序列的平均信号肽分值为0.356,cut off值为0.5,因此不足以形成经典的信号肽。
|
图 3 人TMEM121蛋白信号肽分析 |
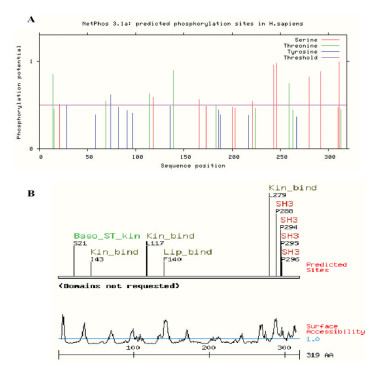
利用Netphos(http://www.cbs.dtu.dk/services/NetPhos/)在线预测分析软件[6],对人TMEM121蛋白的苏氨酸、丝氨酸及酪氨酸的磷酸化位点进行分析,结果显示该蛋白有9个Ser、5个Thr、1个Tyr共15个可能的磷酸化位点(见图 4),但真正的磷酸化位点还需进一步证实。
|
图 4 人TMEM121蛋白磷酸化位点的预测分析; A.人TMEM121蛋白中磷酸化位点预测; B.人TMEM121蛋白中易于被特定蛋白酶磷酸化位点 |
进一步应用Scansite(http://scansite.mit.edu/motifscan_seq.phtml)[7]在线软件分析TMEM121蛋白的motif,发现该蛋白中有如下易被特定蛋白酶磷酸化的位点群,即4个Src同源群3(SH3)、3个激酶结合位点群(ERK结合位点)、1个嗜碱性丝-苏氨酸激酶群(Baso-ST-kin)及1个脂蛋白结合位点群(PIP3结合位点)(见图 3,4B),说明该蛋白活性被改变的可能性大。
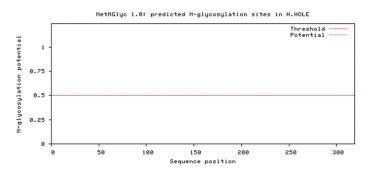
(六) 人TMEM121蛋白糖基化位点分析应用NetNGlyc (http://www.cbs.dtu.dk/services/NetNGlyc/)在线软件[8]对人TMEM121蛋白的糖基化位点进行预测分析,结果显示人TMEM121蛋白中不存在任何糖基化位点(见图 5)。
|
图 5 人TMEM121蛋白序列中糖基化位点分析 |
保守结构域的分析预测可用于判断该蛋白所属类别并为进一步的功能分析提供结构。利用NCBI保守结构域数据库(Conserved Domain Database, CDD)(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)[9, 10]分析人TMEM121序列中的保守结构域,结果TMEM121蛋白中第73-267位氨基酸是典型的CECR6_TMEM121保守结构域,属于CECR6_TMEM121家族成员(见图 6)。CECR6已鉴定为猫眼综合征染色体候选基因6,和TMEM121基因一样,两家族成员的功能迄今仍是未知。

|
图 6 人TMEM121基因保守结构域预测 |
通过SOPHMA程序预测人TMEM121蛋白的二级结构[11, 12](https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.plpage=npsa_sopma.html),发现由155个氨基酸残基构成的α螺旋结构占整个二级结构的48.59%(155/319),是TMEM12二级结构的主要构成原件;无规则卷曲占29.47%(94/319);延伸链占17.8%(57/319)及β转角站4.08%(13/319)(见图 7-A, B)。以上数据说明TMEM121蛋白中大部分氨基酸残基处在一个基本有序的结构中,这有利于执行其应有的功能。应用COILS在线软件对人类TMEM121蛋白进行卷曲螺旋分析,结果显示该蛋白不存在任何卷曲(见图 7-C)。
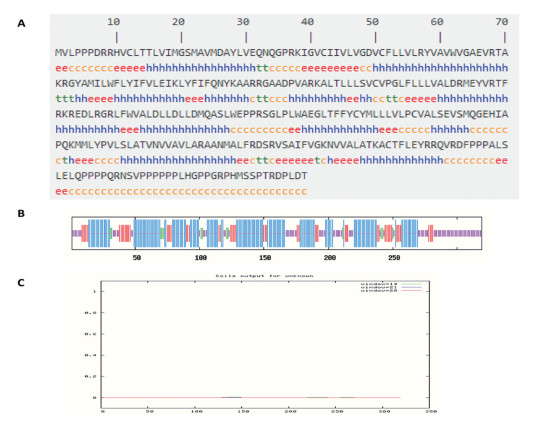
|
图 7 人TMEM121蛋白二级结构预测;A.人TMEM121蛋白二级结构(c:random coil; e:extended strand; h:alpha helix; t:beta turn);B.TMEM121蛋白二级结构简图(蓝色:α-螺旋;红色:β-折叠延伸链;绿色:β-转角;紫色:无规卷曲);C.TMEM121蛋白卷曲螺旋分析 |
Swiss-Model(https://swissmodel.expasy.org/)[13]采用同源建模法可预测蛋白的三级结构及相关参数。因此在Swiss-Model中提交人源TMEM121氨基酸序列,得到1个预测结果(图 8-A),预测结果不太理想,估计与模板覆盖程度不高(仅10.19%)有关。进一步分析人TMEM121蛋白与同源蛋白的相似性波形图(图 8-B),预测值也偏低(不足0.6),因此认为该模型并不太理想。
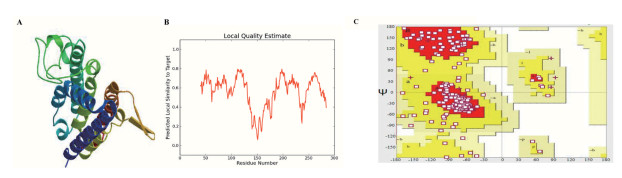
|
图 8 人类TMEM121蛋白三级结构的预测分析; A.人TMEM121蛋白三级结构预测模型; B.TMEM121同源蛋白质相似性波形图;C.人TMEM121蛋白三维模型(A)的拉曼图(二面角)分析 |
进一步应用模拉曼图方法(The Structure Analysis and Verification Server)(http://services.mbi.ucla.edu/SAVES/)[14]分析该模型的空间结构的合理性,得到图 8-C,结果显示预测模型中的大部分氨基酸残基均在拉曼图中的红色核心区内和黄色的合理区域内,说明该模型中的所有氨基酸均形成的二面角均合理可靠,表明该模型的空间结构基本合理。
(十) 人TMEM121的相互作用蛋白分析应用在线STRING数据库(http://string-db.org/)[15]预测人TMEM121的相互作用蛋白,得到TMEM121蛋白相互作用网络(件图 9),预测结果显示TMEM121的互作蛋白主要有参与嗅觉信号途经的嗅觉标记蛋白(olfactory marker protein, OMP)和参与铁代谢和调节的铁蛋白重链多肽1(ferritin heavy chain1, FTH1)(表 3),但2个互作蛋白的分值均不高, 因此参考价值有限。
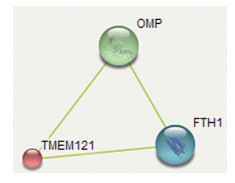
|
图 9 人TMEM121互作蛋白预测 |
| 表 3 人TMEM121蛋白相互作用预测 |
通过Protfun(http://www.cbs.dtu.dk/services/ProtFun/)[16]在线预测人TMEM121编码产物的功能,由分析结果(表 4)可知,人TMEM121蛋白具有转运、受体、转录调控及信号转导功能的可能性分值分别为1.324、0.854、0.521和0.437。
| 表 4 人TMEM121基因编码产物的功能分析 |
作为心肌特异性高表达基因,TMEM121已被克隆十多年,但对其的了解目前仍是知之甚少,对其功能的研究更是一片空白。因此对TMEM121蛋白的结构进行全面的预测分析对研究其功能具有重要的指导意义。
本文利用NCBI公共数据库获取不同物种TMEM121基因的CD序列及编码的氨基酸序列,同源度比对分析发现其与其他哺乳动物的一致性在96%-100%之间,氨基酸序列的一致性也在95%以上,说明人类TMEM121基因与哺乳动物其他物种的同源基因在进化过程中高度保守。在蛋白质相互作用网络中,与人类TMEM121功能相关目前只分析到两个,即嗅觉标记蛋白OMP和铁蛋白重链多肽FTH1。OMP它主要存在于大脑中枢系统的特定部位及嗅球上,是在主要嗅觉通路中可见的唯一标记脑蛋白。[17];FTH1则主要参与铁的代谢与调节,并可调节凋亡相关基因的表达, 从而影响凋亡的进程[18]。另外生物信息学分析结果还发现该蛋白不含信号肽,也无糖基化位点及卷曲螺旋结构等。上述结果提示人类TMEM121蛋白可能并不太活跃,其功能的实现可能主要通过与上述两个蛋白的相互作用来实现。蛋白的磷酸化是通过酶促反应将磷酸基团从一个化合物转移到另一个化合物上,是生物体内普遍存在的一种酶活性调节方式,在细胞的信号传递过程中起着极其重要的作用。本研究发现该蛋白中有4个Src同源群3(SH3)、3个激酶结合位点群(ERK结合位点)、1个嗜碱性丝-苏氨酸激酶群(Baso-ST-kin)及1个脂蛋白结合位点群(PIP3结合位点)等。SH3能够识别富含脯氨酸和疏水残基的特异序列的蛋白质并与之结合, 从而介导蛋白与蛋白相互作用并参与了多种细胞内生化反应,包括信号转导、细胞增殖及细胞运动等。虽然上述预测分析结果还需后续实验加以验证,但初步研究结果提示TMEM121蛋白的这些位点或许可以作为调控其活性的重要靶点。心脏疾病是全球范围内的高发病率疾病,而心源性休克往往在心脏疾病发展过程的终端,预示着心脏疾病的严重程度及死亡率。越来越多的研究报道提示:心脏疾病是一种具有复杂遗传学背景的疾病。特别是对于那些早发型心肌性心脏疾病患者来说,遗传基因是一个主要的危险因素。2005年人类HGP计划完成后,利用全基因组关联分析(GWAS)研究者们发现了大量的疾病易感基因,为精准医学打下了坚实的基础。寻找疾病相关基因依赖于组学研究产生的生物信息数据,也必将促进精准医学的形成和发展。
利用多种生物信息学软件对不同物种TMEM121基因的CDs及氨基酸序列进行了同源度分析,并预测了该基因的编码蛋白的理化性质、分子结构等。研究结果发现:①TMEM121的CDs序列和编码蛋白在哺乳动物间高度同源,同源度高达96%-100%;②对人TMEM121蛋白的磷酸化位点、糖基化位点、疏水性、卷曲螺旋结构及互作蛋白的分析表明,该蛋白具有多个可能的磷酸化位点,但无糖基化位点,亦无卷曲螺旋结构,属两性氨基酸,有2个可能的互作蛋白,推测该蛋白主要通过磷酸化激活的方式作为调控TMEM121功能的靶位点。③亚细胞定位于胞质的可能性比较大,可能具有转运、受体及转录调控的功能等。上述研究结果将为进一步实验研究TMEM121的功能及其作用机制提供了生物信息学参考。
| [1] |
Anna L. Nesset, David M. Bader. Hole is a novel gene product expressed in the developing heart and brain[J]. Mechanisms of Development, 2002, 117(1-2): 347-350. DOI:10.1016/S0925-4773(02)00213-7 |
| [2] |
Zhou JM, Li YQ, Liang P, et al. A novel six-transmembrane protein hhole functions as a suppressor in MAPK signaling pathways[J]. Biochem Biophys Res Commun, 2005, 333(2): 344-352. DOI:10.1016/j.bbrc.2005.05.115 |
| [3] |
Zhang Y, Xu L, Qiu J, et al. Association between SNP rs10569304 on the second expressed region of hHole gene and the congenital heart disease[J]. J Huazhong Univ Sci Technolog Med Sci. 2010 Aug; 30(4): 430-436.
|
| [4] |
Elisabeth G, Christine H, Alexandre G, et al. Protein Identification and Analysis Tools on the ExPASy Server[M]. The Proteomics Protocols Handbook. Humana Press, 2005: 571-607.
|
| [5] |
Petersen TN, Brunak S, von Heijne G, Nielsen H. SignalP 4.0: discriminating signal peptides from transmembrane regions[J]. Nature Methods, 2011, 8(10): 785-786. DOI:10.1038/nmeth.1701 |
| [6] |
Blom N, Gammeltoft S, Brunak S. Sequence and structure-based prediction of eukaryotic protein phosphorylation sites[J]. Journal of Molecular Biology, 1999, 294(5): 1351-1362. DOI:10.1006/jmbi.1999.3310 |
| [7] |
Obenauer JC, Cantley LC, Yaffe MB. Scansite 2. 0: Proteome-wide prediction of cell signaling interactions using short sequence motifs[J]. Nucleic Acids Res. 2003 Jul 1;31(13): 3635-3641.
|
| [8] |
Julenius K., lgaard A. M, Gupta R.. Prediction, conservation analysis and structural characterization of mammalian mucin-type O-glycosylation sites[J]. Glycobiology, 2005, 15: 153-164. DOI:10.1093/glycob/cwh151 |
| [9] |
Marchler-Bauer A, Bryant SH. CD-Search: protein domain annotations on the fly[J]. Nucleic Acids Res. 2004 Jul 1, 32: 327-331.
|
| [10] |
Marchler-Bauer A, Lu S, Anderson JB, et al. CDD: a Conserved Domain Database for the functional annotation of proteins[J]. Nucleic Acids Res. 2011 Jan, 39(D)225-229.
|
| [11] |
Geourjon C, Deléage G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Comput Appl Biosci. 1995 Dec; 11(6): 681-684.
|
| [12] |
Combet C, Blanchet C, Geourjon C, et al. NPS@: network protein sequence analysis[J]. Trends Biochem Sci. 2000 Mar; 25(3): 147-150.
|
| [13] |
Marco Biasini, Stefan Bienert, Andrew Waterhouse, et al. SWISS-MODEL: modelling protein tertiary and quaternary structure using evolutionary information[J]. Nucleic Acids Research. 2014, July, 42(W1): 252-258.
|
| [14] |
Lüthy R, Bowie JU, Eisenberg D. Assessment of protein models with three-dimensional profiles[J]. Nature. 1992 Mar 5;356(6364): 83-85.
|
| [15] |
Szklarczyk D, Franceschini A, Wyder S, et al. STRING v10: protein-protein interaction networks, integrated over the tree of life[J]. Nucleic Acids Res. 2015 Jan; 43: 447-452.
|
| [16] |
Juhl Jensen L., Gupta R., Blo N., et al. Ab initio prediction of human orphan protein function from post-translational modifications and localization features[J]. J. Mol. Biol., 2002, 319: 1257-1265. DOI:10.1016/S0022-2836(02)00379-0 |
| [17] |
高海燕, 诸小侬, 彭诗东, 等. 慢性鼻-鼻窦炎患者嗅觉功能和嗅觉标记蛋白的研究[J]. 中华耳鼻咽喉科杂志, 2004, 39(8): 493-495. |
| [18] |
杜军伟, 王远志, 陈创夫, 等. 铁蛋白重链多肽对RAW264.7细胞中凋亡相关基因表达的影响[J]. 石河子大学学报(自然科学版), 2010, 28(5): 565-568. |
 2018, Vol. 2
2018, Vol. 2

