鸟嘌呤(G)四联体,又称作G-quadruplex或G4等,广泛存在于生物体基因组中。它是由富含G串联重复序列的DNA或RNA折叠形成的一种高级结构。相同或不同平面的G主要是以CGC三联体形式,通过Hoogsteen氢键相互连接形成了一种更加稳定的二级结构[1]。DNA-G4假设模型最早于1958年被提出,但直到1962年才由Gellert等[2]通过X-Ray手段证实了该模型的真实性。由于受到当时研究手段的制约,人们对G4结构及其生物学功能认识非常有限。直到20世纪90年代,一系列用于体内外检测G4位点的物理、生化以及免疫学等相关检测技术的发展和应用,G4结构及其生物学功能重新引起了人们的广泛关注。目前,人和动物G4研究已成为人类疾病治疗和药物基因组学研究的热点之一。
目前已有的研究结果表明,生物体G4具有一系列重要的生物学功能,如稳定染色质状态[3-4]、参与细胞周期[5]、调节离子跨膜运输[6]、参与表观遗传调控[7-11]、调控基因表达[9, 12-13]以及参与DNA损伤和修复[14]等。尤为重要的是,G4现已作为潜在的靶标位点用于人类疾病治疗[15-19]。例如,I期临床试验结果显示,小分子合物CX-5461用于治疗因BRCA1/2缺陷而引起的肿瘤是与它可以稳定体内G4有关[20]。与人及动物G4研究相比,植物G4的鉴定及其生物学功能研究远远滞后。目前,除G4-seq被首次用于在全基因组水平鉴定拟南芥G4位点外,多数植物G4的位点还主要依赖于生物信息学方法,根据形成G4的核心序列在全基因组水平进行预测[21],这种预测的准确性还有待于进一步验证。
根据含有预测G4位点相关的植物基因功能聚类分析结果,植物G4也可能具有一系列重要的生物学功能,如参与基因表达调控[22]、胁迫响应[23-25]和调节植物正常生长发育[26]等。本文系统总结了生物体G4研究方法;重点综述了人和动物G4的生物学功能及其最新研究进展;最后对植物G4研究进行了总结和展望。
1 G4的主要研究方法目前,广泛用于体内外G4研究的方法主要有:物理学、生物化学、免疫学和基因组学等。根据每次检测G4位点数量,这些方法可分为每次检测数量有限的低通量法和在全基因组水平进行研究的高通量法两大类。
1.1 物理学方法研究G4结构的物理学方法主要包括表面离子共振法(Surface plasmon resonance,SPR)、光谱检测法以及毛细管电泳法(Capillary electrophoresis,CE)等。其中,光谱法主要有圆二色谱法(Circular dichroism,CD)、荧光光谱法(Fluorescence spect-rum,FS)、荧光共振能量转移(Fluorescence reso-nance energy transfer,FRET)、核磁共振(Nuclear magnetic resonance,NMR)及X-射线晶体衍射(X-ray diffraction by crystals)等[27]。1962年,Gellert等[2]通过对DNA片段进行高温变性后缓慢复性处理在体外重建了G4结构,并运用X-Ray的方法首次拍摄到G4四链结构。2002年,Cary等[28]通过X-射线晶体衍射证实了染色体端粒的G重复序列也能形成G4结构。由于G4结构在260 nm处有一个明显的吸收峰,Garg等[7, 29]利用圆二色谱手段证实了G4广泛存在于多种植物基因组中。荧光共振能量转移法主要是利用具有类似激发波长的外源竞争物引起荧光标记的G4序列发生荧光偏移来鉴定G4结构。Xu等[20]通过该方法鉴定了CX-5461可作一种有前景的药物用于癌症的治疗;2018年,Chen等[30]利用核磁共振方法证明了DEAH/RHA解旋酶DHX36通过解除G4结构参与调节基因组中G4相关的生物学功能。有报道表明,远红外探针方法也可用于G4结构的鉴定[31]。目前,圆二色光谱法广泛用于验证基因组中预测的或经其它手段鉴定的G4位点[32]。物理学方法检测G4具有快速准确,适用性较大等优点;但该方法需要借助一些特殊或较昂贵的仪器设备,并且每次鉴定的G4数量有限,不适于大批量的鉴定G4位点。因此物理学方法检测G4结构虽可行,但很难在不具备条件的实验室或科研院所全面推广。
1.2 生物化学方法研究G4结构的生物化学法主要有硫酸二甲酯印迹法(Dimethylsulfate,DMS)、凝胶迁移阻滞实验(Electrophoretic mobility shift assay,EMSA)和DNA聚合酶终止法(DNA polymerase stop assay)等[33]。
DMS常与EMSA结合来检测或验证G4,其原理主要是对含有G4的DNA片段进行DMS甲基化处理,DMS可使非G4中的G甲基化,但G4中的G因受Hoogsteen氢键保护而不能被DMS甲基化,然后利用六氢吡啶对DMS处理后的寡核苷酸片段进行差异性切割,寡核苷酸片段中甲基化的鸟嘌呤受六氢吡啶特异性化学切割而发生断裂,而六氢吡啶不能化学切割G4中未被甲基化的G,最后利用变性PAGE分离经六氢吡啶处理后的寡核苷酸片段,根据片段大小推断寡核苷酸片段中形成G4的位置。Armond等[34]应用该方法证实了缺氧诱导因子1R启动子中存在G4结构。
DNA聚合酶终止法的原理:DNA聚合酶在DNA模板链上移动过程中,受G4结构的阻碍从模板上脱落而终止反应,导致所扩增DNA片段长短不一,然后通过PAGE电泳分离目的片段,根据片段大小推断DNA模板中形成G4的位置。反应中加入K、Na等一价金属阳离子及PDS或PEG等小分子化合物可稳定G4结构,以增加反应效果[35-39]。该方法已应用于部分G4位点的鉴定和验证[21]。凝胶迁移实验主要运用含有G4的荧光标记的序列及探针在PAGE电泳中的迁移速度不同,进行目的片段分离,该方法常结合聚合酶终止法鉴定或验证G4位点[40-42]。另外,G4结构的稳定性与其loop的长度有关,正向平行及适当的loop长度将有助于增加G4稳定性[43]。
DMS、EMSA以及DNA聚合酶终止法有着适用性广、结果准确、实验周期短的优点,可适用于多数实验室开展G4相关研究,但每次鉴定G4位点数量有限,属于低通量鉴定方法。
1.3 免疫学方法检测G4的免疫学方法主要是基于D1、1H6、hf2和BG4等特异结合G4抗体的制备及应用[5]。它主要包括免疫荧光检测法和免疫点杂交法两种。2013年Biffi等[5]利用重组BG4抗体鉴定了细胞分裂时期G4位点并观察了其分布。同样,D1与1H6抗体也被应用于细胞水平识别G4结构[44]。例如,Henderson等[45]通过基于1H6抗体的荧光染色方法检测了人和小鼠细胞G4,同时证实了FANCJDNA解旋酶能够清除基因组G4结构。hf2抗体用于检测c-kit原癌基因启动子区平行G4结构[46]。上述几种抗体均有被用于检测G4结构的报道,但是每种抗体识别细胞核内G4结构的特异性方面存在一定的差异。D1抗体对平行性G4结构具有很高的亲和性,但不能用于检测反向平行和杂合G4结构,也不能结合双链DNA、随机单链DNA和DNA发夹结构等[47];1H6抗体对多数G4结构具有广谱亲和性,如对分子内或分子间G4结构均具有相似的结合力,但对分子间RNA G4结构和DNA三链结构亲和力低,不结合由[AGGG(TTAGGG)3]序列组成的分子内G4结构,也不结合任何不能形成G4的单链或双链DNA[45];hf2抗体对DNA G4结构的结合力比对双链DNA的结合力强1 000倍以上,并且对两种分子内相关的平行性G4结构的结合力相差100倍,因此该抗体对不同G4结构具有选择性识别作用[46];BG4抗体对分子内和分子间DNA G4结构具有高的亲和力,包括平行和反平行G4等,但不结合RNA发夹结构以及单链或双链DNA[5]。因此,可以针对每种抗体对G4结构的结合特性,选择性研究细胞核内某一类或几类的G4结构及其生物学功能。目前,只有BG4抗体结合高通量测序被广泛用于全基因组水平G4的鉴定和相关生物学功能研究。其它几种抗体能否用于在全基因组水平鉴定G4结构还有待于进一步验证。
免疫学方法能够识别体内生理状态下G4位点,相对简单易操作,也可在全基因组水平鉴定G4位点,但该方法依赖于抗体的特异性,并且不能分辨G4在基因组内分布的详细信息,相关抗体在不同物种间的通用性还有待于进一步验证。
1.4 G4基因组学方法目前,在全基因组水平鉴定G4位点的高通量方法主要有ChIP-Seq和G4-Seq两种方法。其中,G4-Seq的原理是DNA聚合酶在合成DNA新生链过程中,G4结构的存在可阻止聚合酶的移动并导致新合成DNA链中G4位点处产生错配碱基,结合二代测序数据,在全基因组水平通过鉴定错配碱基来鉴定G4位点[21]。目前,该方法已经用于在全基因组水平鉴定了拟南芥等12个动植物G4位点,与之相类似的方法也可以用于鉴定RNA G4位点[48-49]。通过多物种比较分析发现,G4位点既具有一定的物种特异性,又呈现发育时期特异性[21, 48-50]。另一类基于特异识别G4抗体的ChIP-Seq方法也被广泛用于全基因组水平鉴定G4。与交联ChIP-seq方法相似,该方法主要技术流程包括:首先对受试细胞系或组织材料进行交联处理,再将交联染色质进行片段化处理成大小适宜的染色持片段,随后进行基于抗体如BG4的染色质免疫共沉淀反应,最后将抗体特异结合的G4 DNA进行解交联,纯化经抗体富集的G4 DNA用于高通量测序,经生物信息学分析鉴定全基因组G4位点。目前该方法成功的应用于鉴定人体癌症细胞系的G4位点[32],发现G4位点具有部分调控染色质特性,如与基因组中部分开放性染色质位点(ATAC)共分布,并且G4与核小体松紧程度成正相关[51]。
G4-Seq主要利用G4空间结构所具有特性在DNA水平鉴定G4位点,因此该方法不受物种限制,适用性较广,但该方法主要用于体外鉴定G4,不能真正反应生理状态下的G4位点;基于G4抗体的ChIP-Seq方法具有特异性较强,可以用于体内外鉴定G4位点,但该方法易受抗体特异性影响,并且抗体在不同物种间的通用性还有待于验证。
2 人和动物基因组中G4生物学功能现阶段人及动物的相关研究结果表明,G4主要影响染色质及基因组结构稳定性、调控基因表达、调节离子跨膜运输及参与表观互作等方面(图 1)。
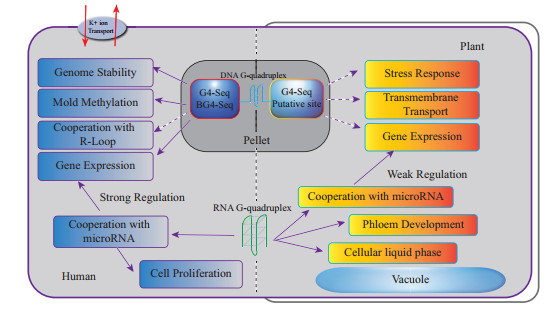
|
| 图 1 G4在人和植物中的主要生物学功能 |
端粒及着丝粒区域的富G序列能够形成G4结构[28, 52]。G4是端粒DNA中富含G序列形成的一种特殊二级结构,稳定G4可以有效阻止端粒酶与端粒DNA相结合从而抑制端粒延伸。应用促进G4的形成或稳定的配合体或小分子合物来抑制端粒酶活性,最终可抑制肿瘤细胞的生长和繁殖以达到抗癌目的,这是基于G4的药物基因组学和癌症治疗的主要关注点之一。因此,端粒G4的形成和稳定导致肿瘤细胞基因组的不稳定性增加。另一方面,由于G4结构具有抵制外切核酸酶对端粒DNA的切割作用,从而有利于维持端粒的稳定性。例如,酵母基因组中因缺失一种端粒加帽蛋白复合体Cdc13导致端粒不稳定性增加,但是G4结构的存在可显著降低这种端粒的不稳定性[53]。另外,G4在正常的DNA双链的某一条单链上出现会造成DNA链的断裂,造成基因组的不稳定[14]。总之,G4对染色质及基因组稳定性具有双重调节功能。
2.2 调控基因表达G4既可以介导全基因组水平基因表达变化,也可以调节单个基因位点表达水平。转录组学分析结果显示,G4稳定性配体TMPyP4通过增加人肿瘤细胞中G4稳定性来改变1 200个基因表达水平[54]。G4可直接参与调控基因表达。在DNA转录时,DNA模板链上G4通过阻断RNA聚合酶移动导致转录停滞甚至终止,从而降低了基因的表达量[55-56];3′ UTR区G4通过阻止RNA聚合酶II移动而及时终止转录,防止了通读现象的发生。mRNA G4可阻碍翻译机器核糖体的结合或移动,从而降低或终止mRNA翻译过程,导致蛋白表达量降低和基因沉默[48-49, 57]。
其次,G4可通过与一些因子互作来间接调控基因表达。PPL3C等有机物质通过与c-MYC和BCL-2启动子区G4结合,降低这两个基因的表达量[7];原癌基因MYC和RAS启动子G4可显著下调基因表达,这主要是由于G4区含有转录因子SP1结合位点,G4形成将不利于转录因子SP1的结合,从而抑制了该转录因子对基因表达的促进作用[58]。白屈菜赤碱通过抑制启动子区G4的形成,降低VEGFA、BCL2和KRAS基因的表达[12],不过串联的G4仍然影响相关的基因表达[13];另外,在全基因组水平,G4可通过影响其附近染色质水平变化来调节相关基因表达[52]。
最后,microRNA前体中G4结构也可间接参与调控基因表达。TmPyP4通过破坏miRNA-149前体中G4结构使癌细胞中成熟的miRNA-149浓度升高,进而降低其靶蛋白蛋白ZBTB2的表达量,最终抑制癌细胞增殖[59]。最新研究结果显示,在染色质三维水平,G4明显富集于拓扑关联区(TAD),并且G4序列富集一些转录因子结合位点,从而暗示G4既可以调控附近基因表达也可以通过染色质成环而远程调控相关基因表达[60]。总之,G4可直接和间接影响基因表达。目前G4对基因表达调控的研究还仍然有限,特别是G4调控基因表达的分子机理还有待于深入研究。
2.3 影响疾病发生目前发现与G4有关的神经性疾病至少有30种[61]。其中解旋酶功能缺陷相关的疾病有9种[62],如解旋酶XPB/XPD作为TFIIH主要组成成份之一,通过与G4结合参与核酸切除修复,其功能缺陷导致色性干皮病(Xeroderma pigmentosum);解旋酶FANCJ通过解除G4结构,它与REV1聚合酶一起参与正常的DNA复制,其功能缺陷导致范可尼贫血症(Fanconi anemia,FA);解旋酶ATRX主要参与G4区域的染色质重排[63],其功能缺陷导致α地中海贫血伴随智力低下症(Alpha thalassemia with mental retardation)等。另外,人FMR1基因中CGG重复序列大量扩增超过200次,扩增后的CGG序列通过形成四联体结构影响FMR1基因表达,从而导致脆性X染色体综合症(Fragile X syndrome)。同样,位于人基因组中第9号染色体上C9ORF72位点的一个基因,其GGGGCC重复序列的大量扩增引起RNA或DNA水平G4结构形成,从而下调该基因表达,导致两种不同的神经退行性疾病的发生,一种是肌萎缩侧索硬化(ALS),另一种是额颞痴呆(FTD)。另外,G4也可以通过影响一些癌症相关基因的表达,它与人类一些癌症的发生密切相关[64-65],但它也可以作为一种潜在的靶标位点用于肿瘤等疾病的诊断和治疗,从而为治疗肿瘤等疾病药物开发提供了契机。
2.4 影响表观修饰人基因组研究结果显示,与非G4序列相比,G4对应的DNA序列是低甲基化,这主要与G4结构是通过抑制DNA甲基化转移酶1(DNMT1)结合或向G4周围序列扩散有关[10]。在染色质水平,G4可以作为基因组中表观调控的靶标位点,说明G4可影响对其附近的染色质水平的表观修饰。例如a-球蛋白基因的数量可变串联重复区(VNTR)富含易形成G4结构的G碱基序列,SWI/SNF染色质重塑家庭的成员的ATRX可特异结合该G4序列,进而招募有利于基因表达的激活性染色质修饰,从而促进a-球蛋白基因的表达[63]。另外,G4可以作为表观遗传调控元件影响基因启动子区附近的组蛋白修饰[66]。这样,G4可以直接或间接影响局部DNA或染色质等表观修饰。
R-Loop即DNA单链及其对应的DNA和RNA杂合链形成的一种三链结构,它普遍于生物体基因组中。已有的研究结果表明,人和植物基因组中存在R-Loop和G4共分布现象[8, 67]。基因启动子区域R-Loop和G4共存显著抑制基因的转录,消除R-loop可降低这种抑制作用,从而说明R-loop可增强G4的作用[8]。在细胞分裂S期时,R-Loop可通过促进G4的形成来抑制基因表达[68];另外,G4通过稳定R-Loop结构参与DNA损伤修复[69]。不过,R-loop与G4间相互作用的模式或机制目前仍不是十分清楚。此外,G4结构也可能参与人体细胞的离子跨膜运输过程[6],DNA复制[70]、免疫球蛋白类别转换和B细胞高频突变[71]等重要的生物学过程。
3 植物G4生物学功能与人、动物和酵母等基因组中G4研究相比,植物G4研究远远滞后。植物G4研究主要集中在基于生物信息学的预测分析上[72]。目前,利用生物信息手段预测了水稻、玉米和拟南芥等16种已测序的单双子叶植物基因组中G4位点,并且利用CD和DNA聚合酶终止实验,在体外初步验证了水稻和拟南芥等4种植物基因组中部分G4序列[73-74]。另外,利用生物信息手段构建了196种植物的G4数据库,从而方便查询相关植物基因组中可能存在的G4位点[72]。序列分析结果显示,基因组中G4的loop长度具有一定亚基因组分布倾向性,如G3L1-3和G3L1-7的G4结构倾向于富集在启动子区。通过对单双子叶植物基因组中含有G4序列的直系同源基因进行功能聚类分析,发现相关基因主要参与了基因表达调控、生殖发育、离子等跨膜运输、能量代谢以及体内外胁迫响应等生物学过程[73-74],说明G4同样参与了植物一些基本生物学过程。Marsico等[21]利用G4-seq在全基因组水平鉴定了拟南芥G4位点,并对其序列特征进行了初步分析,但并未涉及G4相关的生物学功能研究。因此,植物基因组中DNA G4的生物学功能以及相关的作用机制研究还有待于深入开展。
同样,RNA G4具有调控植物基因表达和正常生长发育等生物学功能。例如,SMXL4/5基因mRNA未形成G4时,SMXL4/5蛋白能够正常表达参与了韧皮部细胞的分化与生长,其mRNA G4可抑制该基因表达并影响韧皮部细胞的分化[26]。
SHR mRNA及其突变体相关的研究表明,SHR mRNA G4结构能够调节植物根细胞液-液分离[75],说明RNA G4可能在植物生理调节方面起到了重要的作用。光对G4结构的建成与消失具有一定的催化作用,推测G4也可能参与了植物光合作用,但需要进一步研究提供相关证据[76-77]。与动物不同,目前未发现植物microRNA前体上的G4具有调控其靶基因表达的作用[78]。
4 总结与展望G4是普遍存在于生物体基因组中一种高级结构。它广泛参与了动植物体生长发育、基因组结构稳定性、基因表达调控和DNA损伤及修复等一系列重要的生物学过程。随着全基因组水平鉴定G4方法的发展和应用,将大力推进动植物体G4生物学功能研究。特别是G4在动植物体作用的分子机制还有待于深入研究,例如,G4是如何通过招募或排斥一些反式作用因子来实现其生物学功能;G4是如何通过影响表观修饰或与表观修饰因子互作来参与一些生物学过程的调控等。
深入解析G4对人类疾病发生的机制将有助于加深人们对癌症发病机理的认识。目前,G4作为靶标位点用于抗癌药物设计[79],癌症和神经性疾病的诊断和治疗[16-18],从而加速了现代医学的发展。例如,肝癌细胞中G4分布增加,因此G4可用于肝癌早期辅助诊断[64]。G4作为潜在的药物靶标用于治疗精神类疾病等[19]。
同样,阐明植物G4生物学功能及其作用机制,将为基于基因表达调控的作物表观遗传育种提供理论参考。逆境胁迫特异的G4可作为分子标记用于作物抗逆分子育种,加速其育种进程。总之,随着研究的深入,G4在人类疾病诊断和治疗,农用物分子育种等方面将具有更加广阔的应用前景。
| [1] |
谢君, 刘次全, 屈良鸽. 以Hoogsteen氢键形成的pNA(T)· nNA(AT)三螺旋更稳定[J]. 科学通报, 2013, 20(48): 2141-2144. |
| [2] |
Gellert M, Lipsett MN, Davies DR. Helix formation by Guanylic Acid[J]. Proc Natl Acad Sci USA, 1962, 48: 2013-2018. DOI:10.1073/pnas.48.12.2013 |
| [3] |
Henderson E, Kang SG. Identification of non-telomeric G4-DNA binding proteins in human, E. coli, yeast, and Arabidopsis[J]. Mol Cells, 2002, 14: 404-410. |
| [4] |
Robert B, Clarke MM, Dean WL, et al. Polyethylene glycol binding alters human telomere G-quadruplex structure by conformational selection[J]. Nucleic Acids Res, 2013, 41(16): 7934-7946. DOI:10.1093/nar/gkt440 |
| [5] |
Biffi G, Tannahill D, Mccafferty J, et al. Quantitative visualization of DNA G-quadruplex structures in human cells[J]. Nat Chem, 2013, 5(3): 182-186. DOI:10.1038/nchem.1548 |
| [6] |
Debnath M, Chakraborty S, Kumar YP, et al. Ionophore constructed from non-covalent assembly of a G-quadruplex and liponucleoside transports K+-ion across biological membranes[J]. Nat Commun, 2020, 11(1): 469. |
| [7] |
Micheli E, Altieri A, Cianni L, et al. Perylene and coronene derivatives binding to G-rich promoter oncogene sequences efficiently reduce their expression in cancer cells[J]. Biochimie, 2016, 125: 223-231. DOI:10.1016/j.biochi.2016.04.008 |
| [8] |
Belotserkovskii BP, Shin JHS, Hanawalt PC. Strong transcription blockage mediated by R-loop formation within a G-rich homopurine-homopyrimidine sequence localized in the vicinity of the promoter[J]. Nucleic Acids Res, 2017, 45(11): 6589-6599. DOI:10.1093/nar/gkx403 |
| [9] |
Sngar A, Vandana JJ, Chambers VS, et al. Structure of a(3+1)hybrid G-quadruplex in the PARP1 promoter[J]. Nucleic Acids Res, 2019, 47(3): 1564-1572. DOI:10.1093/nar/gky1179 |
| [10] |
Mao SQ, Ghanbarian AT, Spiegel J, et al. DNA G-quadruplex structures mold the DNA methylome[J]. Nat Struct & Mol Biol, 2018, 25: 951-957. |
| [11] |
Mukherjee AK, Sharma S, Chowdhury S. Non-duplex G-Quadruplex structures emerge as mediators of epigenetic modifications[J]. Trends Genet, 2019, 35(2): 129-144. |
| [12] |
Jana J, Mondal S, Bhattacharjee P, et al. Chelerythrine down regulates expression of VEGFA, BCL2 and KRAS by arresting G-Quadruplex structures at their promoter regions[J]. Sci Rep, 2017, 7: 40706. DOI:10.1038/srep40706 |
| [13] |
Rigo R, Sissi C. Characterization of G4-G4 crosstalk in the c-KIT promoter region[J]. Biochemistry, 2017, 56(33): 4309-4312. DOI:10.1021/acs.biochem.7b00660 |
| [14] |
Hänsel-Hertsch R, Antonin MD, Balasubramanian S. DNA G-quadruplexes in the human genome:detection, functions and therapeutic potential[J]. Nat Rev Mol Cell Biol, 2017, 18(5): 279-284. DOI:10.1038/nrm.2017.3 |
| [15] |
Reed AJ, Connelly RP, Williams A, et al. Label-free pathogen detection by a deoxyribozyme cascade with visual signal readout[J]. Sensor Actuat B-Chem, 2019, 282: 945-951. DOI:10.1016/j.snb.2018.11.147 |
| [16] |
Han Y, Zhang F, Gong H, et al. Double G-quadruplexes in a copper nanoparticle based fluorescent probe for determination of HIV genes[J]. Mikrochim Acta, 2018, 186(1): 30. |
| [17] |
Moruno-Manchon JF, Koellhoffer EC, Gopakumar J, et al. The G-quadruplex DNA stabilizing drug pyridostatin promotes DNA damage and downregulates transcription of Brca1 in neurons[J]. Aging, 2017, 9(9): 1957-1970. DOI:10.18632/aging.101282 |
| [18] |
Zuffo M, Stucchi A, Campos-Salinas J, et al. Carbohydrate-naphthalene diimide conjugates as potential antiparasitic drugs:Synthesis, evaluation and structure-activity studies[J]. Eur J Med Chem, 2019, 163: 54-66. DOI:10.1016/j.ejmech.2018.11.043 |
| [19] |
Asamitsu S, Yabuki Y, Ikenoshita S, et al. Pharmacological prospects of G-quadruplexes for neurological diseases using porphyrins[J]. Biochem Biophys Res Commun, 2020. |
| [20] |
Xu H, Di Antonio M, McKinney S, et al. CX-5461 is a DNA G-quadruplex stabilizer with selective lethality in BRCA1/2 deficient tumours[J]. Nat Commun, 2017, 8: 14432. DOI:10.1038/ncomms14432 |
| [21] |
Marsico G, Chambers VS, Sahakyan AB, et al. Whole genome experimental maps of DNA G-quadruplexes in multiple species[J]. Nucleic Acids Res, 2019, 47(8): 3862-3874. DOI:10.1093/nar/gkz179 |
| [22] |
Griffin BD, Bass HW. Review:Plant G-quadruplex(G4)motifs in DNA and RNA; abundant, intriguing sequences of unknown function[J]. Plant Sci, 2018, 269: 143-147. DOI:10.1016/j.plantsci.2018.01.011 |
| [23] |
Yadav V, Hemansi, Kim N, et al. G quadruplex in plants:A ubiquitous regulatory element and its biological relevance[J]. Front Plant Sci, 2017, 8: 1163. DOI:10.3389/fpls.2017.01163 |
| [24] |
Shakiba E, Edwards JD, Jodari F, et al. Genetic architecture of cold tolerance in rice(Oryza sativa)determined through high resolution genome-wide analysis[J]. PLoS One, 2017, 12(3): e0172133. DOI:10.1371/journal.pone.0172133 |
| [25] |
Andorf CM, Kopylov M, Dobbs D, et al. G-quadruplex(G4)motifs in the maize(Zea mays L.)genome are enriched at specific locations in thousands of genes coupled to energy status, hypoxia, low sugar, and nutrient deprivation[J]. J Genet Genomics, 2014, 41(12): 627-647. DOI:10.1016/j.jgg.2014.10.004 |
| [26] |
Cho H, Cho HS, Nam H, et al. Translational control of phloem development by RNA G-quadruplex-JULGI determines plant sink strength[J]. Nat Plants, 2018, 4(6): 376-390. DOI:10.1038/s41477-018-0157-2 |
| [27] |
Sun D, Hurley LH. The importance of negative superhelicity in inducing the formation of G-quadruplex and i-Motif structures in the c-Myc promoter:Implications for drug targeting and control of gene expression[J]. J Med Chem, 2009, 52(9): 2863-2874. DOI:10.1021/jm900055s |
| [28] |
Parkinson GN, Lee MP, Neidle S. Crystal structure of parallel quadruplexes from human telomeric DNA[J]. Nature, 2002, 417(6891): 876-880. DOI:10.1038/nature755 |
| [29] |
Garg R, Aggarwal J, Thakkar B. Genome-wide discovery of G-quadruplex forming sequences and their functional relevance in plants[J]. Sci Rep, 2016, 6: 28211. DOI:10.1038/srep28211 |
| [30] |
Chen MC, Tippana R, Demeshkina NA, et al. Structural basis of G-quadruplex unfolding by the DEAH/RHA helicase DHX36[J]. Nature, 2018, 558(7710): 465-469. DOI:10.1038/s41586-018-0209-9 |
| [31] |
Suseela YV, Narayanaswamy N, Pratihar S, et al. Far-red fluorescent probes for canonical and non-canonical nucleic acid structures:current progress and future implications[J]. Chem Soc Rev, 2018, 47(3): 1098-1131. DOI:10.1039/C7CS00774D |
| [32] |
Hänsel-Hertsch R, Spiegel J, Marsico G, et al. Genome-wide mapping of endogenous G-quadruplex DNA structures by chromatin immunoprecipitation and high-throughput sequencing[J]. Nat Protoc, 2018, 13(3): 551-564. DOI:10.1038/nprot.2017.150 |
| [33] |
Sun D, Hurley LH. Biochemical techniques for the characterization of G-quadruplex structures:EMSA, DMS footprinting, and DNA polymerase stop assay[J]. Methods Mol Biol, 2010, 608: 65-79. |
| [34] |
Armond RD, Wood S, Sun D, et al. Evidence for the presence of a guanine quadruplex forming region within a polypurine tract of the hypoxia inducible factor 1R promoter[J]. Biochemistry, 2005, 44: 16341-16350. |
| [35] |
Sen D, Gilbert W. A sodium-potassium switch in the formation of four-stranded G4-DNA[J]. Nature, 1990, 344: 410-414. DOI:10.1038/344410a0 |
| [36] |
Behmand B, Balanikas E, Martinez-Fernandez L, et al. Potassium ions enhance guanine radical generation upon absorption of low energy photons by G-quadruplexes and modify their reactivity[J]. J Phys Chem Lett, 2020, 11(4): 1305-1309. DOI:10.1021/acs.jpclett.9b03667 |
| [37] |
Zhang ML, Xu YP, Kumar A, et al. Studying the potassium-induced G-quadruplex DNA folding process using microscale thermophoresis[J]. Biochemistry, 2019, 58(38): 3955-3959. DOI:10.1021/acs.biochem.9b00447 |
| [38] |
Ma G, Yu Z, Zhou W, et al. Investigation of Na+ and K+ competitively binding with a G-quadruplex and discovery of a stable K+-Na+-quadruplex[J]. J Phys Chem B, 2019, 123(26): 5405-5411. DOI:10.1021/acs.jpcb.9b02823 |
| [39] |
Kan ZY, Lin Y, Wang F, et al. G-quadruplex formation in human telomeric(TTAGGG)4 sequence with complementary strand in close vicinity under molecularly crowded condition[J]. Nucleic Acids Res, 2007, 35(11): 3646-3653. DOI:10.1093/nar/gkm203 |
| [40] |
Qin Y, Rezler EM, Gokhale V, et al. Characterization of the G-quadruplexes in the duplex nuclease hypersensitive element of the PDGF-A promoter and modulation of PDGF-A promoter activity by TMPyP4[J]. Nucleic Acids Res, 2007, 35(22): 7698-7713. DOI:10.1093/nar/gkm538 |
| [41] |
Dexheimer TS, Sun D, Hurley LH. Deconvoluting the structural and drug-recognition complexity of the G-quadruplex-forming region upstream of the bcl-2 P1 Promoter[J]. J Am Chem Soc, 2006, 128(16): 5404-5415. DOI:10.1021/ja0563861 |
| [42] |
Guo K, Pourpak A, Beetz-Rogers K, et al. Formation of pseudosym-metrical G-quadruplex and i-Motif structures in the proximal promoter region of the RET oncogene[J]. J Am Chem Soc, 2007, 129(33): 10220-10228. DOI:10.1021/ja072185g |
| [43] |
Cheng M, Cheng Y, Hao J, et al. Loop permutation affects the topology and stability of G-quadruplexes[J]. Nucleic Acids Res, 2018, 46(18): 9264-9275. DOI:10.1093/nar/gky757 |
| [44] |
Huang ZL, Dai J, Luo WH, et al. Identification of G-quadruplex-binding protein from the exploration of RGG motif/G-quadruplex interactions[J]. J Am Chem Soc, 2018, 140(51): 17945-17955. DOI:10.1021/jacs.8b09329 |
| [45] |
Henderson A, Wu Y, Huang YC, et al. Detection of G-quadruplex DNA in mammalian cells[J]. Nucleic Acids Res, 2014, 42(2): 860-869. DOI:10.1093/nar/gkt957 |
| [46] |
Fernando H, Rodriguez R, Balasubramanian S. Selective recognition of a DNA G-quadruplex by an engineered antibody[J]. Biochemistry, 2008, 47(36): 9365-9371. DOI:10.1021/bi800983u |
| [47] |
Liu HY, Zhao Q, Zhang TP, et al. Conformation selective antibody enables genome profiling and leads to discovery of parallel G-quadruplex in human telomeres[J]. Cell Chem Biol, 2016, 23(10): 1261-1270. DOI:10.1016/j.chembiol.2016.08.013 |
| [48] |
Kwok CK, Marsico G, Balasubramanian. Detecting RNA G-quadruplexes(rG4s)in the transcriptome[J]. CSH Perspect Biol, 2018, 10(7): a032284. |
| [49] |
Guo JU, Bartel DP. RNA G-quadruplexes are globally unfolded in eukaryotic cells and depleted in bacteria[J]. Science, 2016, 353(6306): 5371-5371. DOI:10.1126/science.aaf5371 |
| [50] |
Chambers VS, Marsico G, Boutell JM, et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome[J]. Nat Biotechnol, 2015, 33(8): 877-881. DOI:10.1038/nbt.3295 |
| [51] |
Hänsel-Hertsch R, Beraldi D, Lensing SV, et al. G-quadruplex structures mark human regulatory chromatin[J]. Nat Genet, 2016, 48(10): 1267-1272. DOI:10.1038/ng.3662 |
| [52] |
Smith FW, Feigon J. Quadruplex structure of Oxytricha telomeric DNA oligonucleotides[J]. Nature, 1992, 356(6365): 164-168. DOI:10.1038/356164a0 |
| [53] |
Smith JS, Chen Q, Yatsunyk LA, et al. Rudimentary G-quadruplex-based telomere capping in Saccharomyces cerevisiae[J]. Nat Struct Mol Biol, 2011, 18(4): 478-485. DOI:10.1038/nsmb.2033 |
| [54] |
Verma A, Yadav VK, Basundra R, et al. Evidence of genome-wide G4 DNA-mediated gene expression in human cancer cells[J]. Nucleic Acids Res, 2009, 37(13): 4194-4204. DOI:10.1093/nar/gkn1076 |
| [55] |
Rodriguez R, Miller KM. Unravelling the genomic targets of small molecules using high-throughput sequencing[J]. Nat Rev Genet, 2014, 15(12): 783-796. DOI:10.1038/nrg3796 |
| [56] |
Nayun Kim. The interplay between G-quadruplex and Transcription[J]. Curr Med Chem, 2019, 26(16): 2898-2917. DOI:10.2174/0929867325666171229132619 |
| [57] |
Bugaut A, Balasubramanian S. 5′-UTR RNA G-quadruplexes:translation regulation and targeting[J]. Nucleic Acids Res, 2012, 40(11): 4727-4741. DOI:10.1093/nar/gks068 |
| [58] |
Raiber EA, Kranaster R, Lam E, et al. A non-canonical DNA structure is a binding motif for the transcription factor SP1 in vitro[J]. Nucleic Acids Res, 2012, 40(4): 1499-1508. DOI:10.1093/nar/gkr882 |
| [59] |
Ghosh A, Ekka MK, Tawani A, et al. Restoration of miRNA-149 expression by TmPyP4 induced unfolding of quadruplex within its precursor[J]. Biochemistry, 2019, 58(6): 514-525. DOI:10.1021/acs.biochem.8b00880 |
| [60] |
Hou Y, Li F, Zhang R, et al. Integrative characterization of G-Quadruplexes in the three-dimensional chromatin structure[J]. Epigenetics, 2019, 14(9): 894-911. DOI:10.1080/15592294.2019.1621140 |
| [61] |
Mirkin SM. Expandable DNA repeats and human disease[J]. Nature, 2007, 447(7147): 932-940. DOI:10.1038/nature05977 |
| [62] |
Maizels N. G4-associated human diseases[J]. EMBO Rep, 2015, 16(8): 910-922. DOI:10.15252/embr.201540607 |
| [63] |
Law MJ, Lower KM, Voon HP, et al. ATR-X syndrome protein targets tandem repeats and influences allele-specific expression in a size-dependent manner[J]. Cell, 2010, 143(3): 367-378. DOI:10.1016/j.cell.2010.09.023 |
| [64] |
Biffi G, Tannahill D, Miller J, et al. Elevated levels of G-Quadruplex formation in human stomach and liver cancer tissues[J]. PLoS One, 2014, 9(7): e102711. DOI:10.1371/journal.pone.0102711 |
| [65] |
Tomar JS. In-silico modeling studies of G-quadruplex with soy isoflavones having anticancerous activity[J]. J Mol Model, 2015, 21(8): 193. DOI:10.1007/s00894-015-2723-0 |
| [66] |
Schiavone D, Guilbaud G, Murat P, et al. Determinants of G quadruplex-induced epigenetic instability in REV1-deficient cells[J]. EMBO J, 2014, 33(21): 2507-2520. DOI:10.15252/embj.201488398 |
| [67] |
Fang Y, Chen LF, Lin KD, et al. Characterization of functional relationships of R-loops with gene transcription and epigenetic modifications in rice[J]. Genome Res, 2019, 29(8): 1287-1297. DOI:10.1101/gr.246009.118 |
| [68] |
Šviković S, Crisp A, Tan-Wong SM, 等. R-loop formation during S phase is restricted by PrimPol-mediated repriming[J]. EMBO J, 2018, 38(3): e99793. |
| [69] |
Tan J, Wang X, Phoon L, et al. Resolution of ROS-induced G-quadruplexes and R-loops at transcriptionally active sites is dependent on BLM helicase[J]. FEBS Lett, 2020. |
| [70] |
Paeschke K, Capra JA, Zakian VA. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase[J]. Cell, 2011, 145(5): 678-691. DOI:10.1016/j.cell.2011.04.015 |
| [71] |
Duquette ML, Huber MD, Maizels N. G-rich proto-oncogenes are targeted for genomic instability in B-cell lymphomas[J]. Cancer Res, 2007, 67(6): 2586-2594. DOI:10.1158/0008-5472.CAN-06-2419 |
| [72] |
Ge F, Wang Y, Li H, et al. Plant-GQ:An integrative database of G-Quadruplex in plant[J]. J Comput Biol, 2019, 26(9): 1013-1019. DOI:10.1089/cmb.2019.0010 |
| [73] |
Garg R, Aggarwal J, Thakkar B. Genome-wide discovery of G-quadruplex forming sequences and their functional relevance in plants[J]. Sci Rep, 2016, 6: 28211. DOI:10.1038/srep28211 |
| [74] |
Yadav V, Hemansi, Kim N, et al. G quadruplex in plants:A ubiquitous regulatory element and its biological relevance[J]. Front Plant Sci, 2017, 8: 1163. DOI:10.3389/fpls.2017.01163 |
| [75] |
Zhang Y, Yang M, Duncan S, et al. G-quadruplex structures trigger RNA phase separation[J]. Nucleic Acids Res, 2019, 47(22): 11746-11754. |
| [76] |
McBrayer D, Schoonover M, Long KJ, et al. N-methylmesoporphyrin IX exhibits G-quadruplex specific photocleavage activity[J]. Chem Bio Chem, 2018, 20(15): 1924-1927. |
| [77] |
Wang S, Yue L, Li ZY, et al. Light-induced reversible reconfiguration of DNA-based constitutional dynamic networks:Application to switchable catalysis[J]. Angew Chem Int Ed Engl, 2018, 57(27): 8105-8109. DOI:10.1002/anie.201803371 |
| [78] |
Jackowiak P, Hojka-Osinska A, Gasiorek K, et al. Effects of G-quadruplex topology on translational inhibition by tRNA fragments in mammalian and plant systems in vitro[J]. Int J Biochem Cell Biol, 2017, 92: 148-154. DOI:10.1016/j.biocel.2017.10.001 |
| [79] |
Wu G, Chen L, Liu W, et al. Molecular recognition of the hybrid-type G-quadruplexes in human telomeres[J]. Molecules, 2019, 24(8): E1578. DOI:10.3390/molecules24081578 |




