2. 浙江大学生物质化工教育部重点实验室, 浙江大学 化学工程与生物工程学院, 浙江 杭州 310027
2. Key Laboratory of Biomass Chemical Engineering of Ministry of Education, College of Chemical and Biological Engineering, Zhejiang University, Hangzhou 310027, China
葡萄糖胺-6-磷酸合成酶(GFA,EC 2.6.1.16)是氨基己糖代谢通路中第一个关键酶[1-2],也是含糖胺生物大分子(如:几丁质)代谢调控过程中一个关键的节点[3]。其能够利用L-谷氨酰胺,不可逆地催化D-果糖-6-磷酸转化成D-葡萄糖胺-6-磷酸(GlcN6P)。GFA是酰胺转移酶超家族中一员,其结构由两个行使不同功能的结构域组成:转氨酶结构域和异构酶结构域[4-5]。通过抑制葡萄糖胺-6-磷酸合成酶的活性,能够导致相关糖蛋白及几丁质的生物合成受阻,致使致病菌无法合成细胞壁而死亡[6]。但在哺乳动物中,该酶被抑制短时间内不会对个体造成严重的伤害,其所造成的结果因组织不同而异。因此,GFA也被认为将成为抗细菌和抗真菌新药研发中极具前景的潜在靶点[7-8]。
研究表明:无论在胞内还是在胞外,GFA的存在形式(活性的二聚体与无活性的六聚体)都处于一种平衡状态[9]。同时研究发现,随着蛋白浓度的增加及产物(GlcN6P)浓度的提高,GFA更倾向于以六聚体形式存在,造成其比活力将随之降低[10]。这必将大大增加GFA的生产及应用方面的成本,严重影响其应用潜力。此外,来源不同的GFA往往在异源表达的过程中存在表达量少、提取困难等问题,因此迫切需要通过合适的手段,建立一条高效的生产工艺。本研究中,来源于真菌致病菌(白色念珠菌)中的GFA在大肠杆菌BL21(DE3) Codon plus中成功实现异源表达,并对其酶学性质进行了表征,大大提高了其生产效率。在此基础上,验证其作为抗真菌靶点的可行性,为其进一步在抗真菌新药研发中的应用提供基础。
2 实验部分 2.1 试剂与仪器(1) 本实验使用的菌种主要有:实验室保藏的大肠杆菌BL21(DE3) Codon plus,大肠杆菌DH5α白色念珠菌(Candida albicans ATCC 64548)。
(2) 主要试剂有:1 mmol·L-1的GLUPA(γ-谷氨酰对硝基苯胺);pH 7.2的磷酸盐(PBS)缓冲液;LB(Luria-Bertani)培养基(pH 7.2,胰化蛋白胨10 g·L-1、酵母提取物5 g·L-1和NaCl 10 g·L-1);对硝基苯胺(天津大茂化学试剂厂);Bacilysin(实验室保存)。质粒提取试剂盒(Solarbio,D1100),琼脂糖凝胶回收试剂盒(Omega,D2500-01)。
2.2 实验过程 2.2.1 对硝基苯胺标准曲线的绘制用pH为7.2的PBS缓冲液分别配制终浓度为10、20、30、40、50 mmol·L-1的对硝基苯胺溶液,溶解后,使用可见光分光光度计,以pH为7.2的PBS缓冲液为空白对照,测量吸光度A420,并绘制标准曲线。
2.2.2 Gfa基因制备与表达载体的构建从NCBI基因文库调取白色念珠菌细胞GFA的基因序列(Gfa,GenBank:NC-032091.1),设计上游引物5'-CGCGGATCCATGTGTGGTATTTTTGGTTACGTCAATT-3'、下游引物5'-CCGCTCGAGTTACTCAACAGTAACTGATTTAGCCAAGTT-3' (下划线部分分别为BamH I、Xho I酶切位点)。以白色念珠菌基因组为模板进行PCR扩增。PCR克隆该基因过程中在该序列的上下游分别引入限制性内切酶BamH I / Xho I的酶切位点,将PCR扩增产物用BamH I/Xho I双酶切后并克隆至载体pET28(a)。经酶切和PCR验证后,对所获得的阳性克隆进一步测序验证,将测序正确的阳性质粒命名为pET28(a)-G。
PCR反应条件:98 ℃预变性2 min,30次扩增循环(98 ℃、30 s;55 ℃、30 s;72 ℃、3 min),72 ℃延伸10 min。
2.2.3 重组质粒pET28(a)-G的转化与GFA的诱导表达将pET28(a)-G重组质粒通过CaCl2法转化大肠杆菌BL21(DE3)-Condon Plus感受态细胞,挑取单菌落接种到5 mL LB培养液中,37 ℃培养过夜,次日以2% 的接种量转至10 mL LB培养液中,在菌体浓度OD600达到0.6后,加入IPTG(异丙基硫代半乳糖苷,终浓度为0.5 mmol·L-1),16 ℃诱导过夜,取1 mL菌液,12 000 r·min-1离心2 min,收集菌体细胞,超声破碎,5 000 r·min-1离心10 min,用聚丙烯酰胺凝胶电泳(SDS-PAGE)分别定性检测沉淀和上清中重组GFA的表达量,并分析重组蛋白的可溶性。
2.2.4 SDS-PAGE蛋白电泳将发酵液于10 000~12 000 r·min-1离心5 min,取上清液进行SDS-PAGE电泳[11]。离心后所得沉淀用相同浓度的缓冲液溶解后,取样进行蛋白电泳。采用的聚丙烯酰胺浓度为12%,染色液为0.1% 的考马斯亮蓝R-250,脱色液配方中甲醇: 水: 乙酸(体积比)为2:7:1。
2.2.5 GFA酶活力测定将1 mL酶液加入2 mL的反应体系中。反应体系如下(终浓度):
1 mmol·L-1底物(GLUPA),1 mmol·L-1 EDTA(乙二胺四乙酸),1 mmol·L-1 DTT(二硫苏糖醇),及0.5 mmol·L-1 PMSF(苯甲基磺酰氟) 溶解于20 mmol·L-1 HEPES(4-羟乙基哌嗪乙磺酸) 缓冲液中,在pH为7.5,37 ℃下反应10 min。反应结束后,100 ℃煮沸5 min终止反应,冷却至室温,将反应体系置于420 nm下,利用分光光度计测量产物的吸光值度A420,以未加底物的反应体系为空白对照。
测定酶活时,将单位时间(10 min)催化产生1 μmol对硝基苯胺的酶量(mL)定义为一个酶活单位。
2.2.6 GFA异源表达条件的优化1) 诱导时间的优化
诱导时间分别为10、12、14、16、18、20 h,诱导结束后取酶液分别测定不同诱导时间条件下的GFA活力,确定最佳诱导时间。酶活测定条件同2.2.5节。
2) GFA最佳诱导温度的确定
诱导温度分别设定为15、20、25、30、35 ℃,诱导结束后取酶液分别测定不同诱导时间条件下的GFA活力,确定最佳诱导时间。酶活测定条件同2.2.5节。
3) GFA最佳诱导剂IPTG浓度的确定
在GFA诱导过程中,分别在每个培养基中加入终浓度为0.06、0.125、0.25、0.50、1.0、2.0 mmol·L-1的IPTG,诱导结束后取酶液分别测定不同诱导时间条件下的GFA活力,确定最佳诱导时间。酶活测定条件同2.2.5节。
4) GFA最适反应pH值的确定
分别配制不同pH的PBS缓冲液:pH值为5.6、5.8、6.0、6.3、6.5、7.0、8.0,取酶液分别测定不同pH条件下的GFA活力。其他条件同2.2.5节。
5) GFA最适反应温度的确定
将反应温度设为30、35、40、45、50、55、60、65和70 ℃,测定试样在pH 7.0时的GFA活力。
2.2.7 应用双倒数作图法确定葡萄糖胺-6-磷酸合成酶的Vmax(最大反应速度)和KM(米氏常数)分别配制浓度为0.1、0.2、0.5、1.0、2.0 mmol·L-1的底物,取5支试管,每支试管分别放入不同浓度的底物和2 mL的酶液,37 ℃水浴反应30 min。以4 mL的水为空白对照。测量反应体系的吸光度(A420),计算出对硝基苯胺的浓度,然后以对硝基苯胺的生成速度倒数与底物浓度的倒数作图,求出KM及Vmax。
2.2.8 Bacilysin对GFA的抑制作用研究取5支试管,每支试管中加入1 mL的酶液,1 mL底物,1 mL 30 mg·mL-1的抑制剂,使抑制剂的终浓度为10 mg·mL-1。37 ℃水浴加热,每隔20 min取一次样,以纯水为空白对照,测量A420,然后计算出酶活。
取5支试管,每个试管中加入2 mL的酶液和2 mL的底物,放在37 ℃水浴锅中加热,仍是每隔20 min取一次样,以纯水为空白对照,测量A420,然后计算出酶活。
3 结果与讨论 3.1 葡萄糖胺-6-磷酸合成酶表达优化的结果与分析 3.1.1 葡萄糖胺-6-磷酸合成酶的异源表达图 1中标记的条带即为过表达的葡萄糖胺-6-磷酸合成酶,相对分子质量约为80×103,与文献报道的结果吻合(约78.4×103)[12-13]。由上图可以明显发现,在大肠杆菌BL21(DE3)Codon plus中过表达的GFA,基本处于可溶状态,包涵体的成分较少,说明来源于致病菌细胞内的葡萄糖胺-6-磷酸合成酶,可以在该宿主中正常表达。同时,通过后续酶活的测定(见3.2节)表明,本研究中所得到的重组GFA蛋白基本为活性状态的可溶性蛋白。
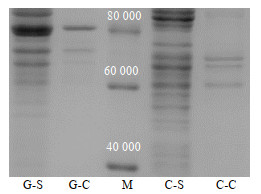
|
图 1 SDS-聚丙烯酰胺凝胶电泳结果 Fig.1 SDS-PAGE analysis of heterologously expressed GFA (M: Marker G-S: Supernatant of the bacteria lysate G-C: precipitation of the bacteria lysate C-S: supernatant of the bacteria lysate (control) C-C: precipitation of the bacteria lysate (control)) |
如图 2所示,加入IPTG对GFA的进行诱导表达后,酶活随诱导时间延长而升高,这是因为IPTG不会被大肠杆菌代谢,从而产生持久的诱导效果;当诱导时间为15 h时酶活达到最高,约为14.4 U·mL-1;继续增加诱导时间,会造成酶活降低。这主要是由于IPTG对宿主细胞具有一定的毒性(抑制)作用,会造成细胞的裂解[14]、宿主细胞内蛋白的降解[15],从而影响外源蛋白的表达。随着诱导时间的延长这种抑制作用越明显[16]。因此,考虑到GFA的催化活性与生产效率,其最佳的诱导时间确定为15 h。
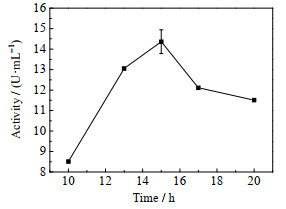
|
图 2 诱导时间对酶活的影响 Fig.2 Effects of inducing time on extracellular enzyme activity of GFA |
据报道,IPTG的摄取进入细胞需要利用细胞膜表面的质子泵,这会大大增加细胞生长的负担,对细胞造成“毒性”,从而抑制菌体的生长及外源蛋白的表达[17-18]。因此,IPTG终浓度是影响外源蛋白表达的重要因素之一。由图 3可知,随着诱导剂浓度增加,酶活显著提高。当诱导剂的终浓度为0.1 mmol·L-1时,酶活最高,达到19.5 U·mL-1;但继续增加诱导剂浓度,酶活又会表现出显著的降低。因此,综合各方面的考虑,在GFA的诱导表达过程中,诱导剂(IPTG)的浓度确定为0.1 mmol·L-1。
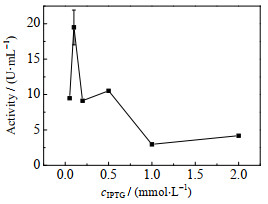
|
图 3 诱导剂浓度对酶活的影响 Fig.3 Effects of concentrations of IPTG on extracellular enzyme activity of GFA |
由图 4可知,诱导温度对异源表达的GFA活性具有十分明显的影响。随着诱导温度的增加(15~30 ℃),酶活逐渐增加,当30 ℃进行诱导时,此时酶活达到最高约为18 U·mL-1。当诱导温度处于30~40 ℃时,酶活又逐渐降低。这是因为随着温度的升高,大肠杆菌繁殖速度加快,导致蛋白表达量急剧增加,从而增加其空间结构折叠错误的概率,形成无活性的包涵体,导致酶活降低[19]。虽然大肠杆菌的最适生长温度为37~38 ℃,但利用其作为宿主进行外源蛋白的异源表达时,考虑到重组GFA蛋白的可溶性及其生化活性的维持,因此在进行异源表达时采用相对较低的诱导温度,约为28 ℃。
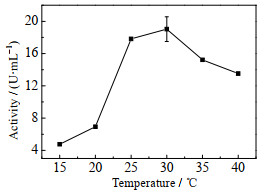
|
图 4 诱导温度与酶活的关系 Fig.4 Effects of induction temperatures on extracellular enzyme activity of GFA |
如图 5所示,诱导时机的不同将显著影响外源蛋白的活力。随着菌体浓度的增加,其经过诱导所生产的酶,活力也随之增加。当OD600为1.0左右时,酶活最高,约34.1 U·mL-1;继续培养,当OD600超过1.0时,酶活会有所降低。这一方面可能是因为菌体浓度太高,瞬间表达的蛋白浓度较高,从而形成非正常折叠、无定型的包涵体,使酶活降低。另一方面,研究表明[20],当在宿主细胞内进行蛋白的异源表达时,不同的诱导时机对宿主所造成的负担不尽相同:指数期前期进行诱导,对宿主所造成的负担要远高于在指数期后期所造成的负担。这不仅会造成外源蛋白表达量的下降,同时也会造成宿主细胞中一些非必须蛋白的生物合成,从而影响宿主细胞的生长速率。Dykhuizen等[21]指出,造成这种结果的原因主要涉及蛋白的转录与翻译过程,与总蛋白浓度无关。因此,选择一个恰当的诱导时机(OD600为1.0左右),无论对于GFA的表达量还是宿主细胞的生长速率都有重要的意义。
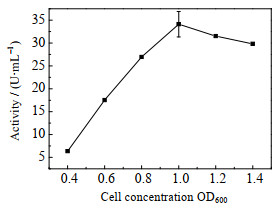
|
图 5 菌体浓度对酶活的影响 Fig.5 Effects of cell concentration on extracellular enzyme activity of GFA |
由图 6可知,在pH为7.2的时候,酶活最高,表明葡萄糖胺-6-磷酸合成酶的最适反应pH为中性,偏酸或偏碱都会影响其结构的稳定性,造成酶活降低。
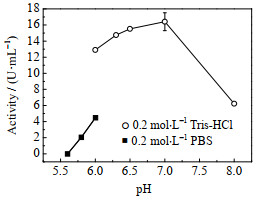
|
图 6 反应体系pH与缓冲液种类对酶活的关系 Fig.6 Effects of pH and buffer solution on the catalytic activity of GFA |
由图 7可知,葡萄糖胺-6-磷酸合成酶酶活最高点所对应的温度是37 ℃,在37 ℃之前,酶活随着温度升高而增加,这是因为在一定温度范围内,温度升高会使分子热运动加速,从而提高化学反应速率;但酶的本质是蛋白质,温度过高会破坏其空间结构。所以随着温度的持续升高,酶活会显著降低。

|
图 7 反应温度与酶活的关系 Fig.7 Effects of reaction temperature on the catalytic activity of GFA |
通过对硝基苯胺的生成速度倒数与底物浓度的倒数作图(Lineweaver-Burk双倒数作图),如图 8所示,得到双倒数方程(double-reciprocal plot):y=0187 9x+0.040 32(R2=0.992 59),图中,V为反应速度,ρsubstrate为底物质量浓度,其中横轴截距为-1/KM,纵轴截距为1/Vmax。由双倒数字方程可获得最大反应速度Vmax和反应速率KM,当x=0时,y=1/Vmax,即Vmax=24.80 mg·(mL·min)-1;当y=0时,x=-1/KM,KM=0.47 mg·mL-1,即当反应速率达到1/2 Vmax时,ρsubstrate为0.47 mg·mL-1。

|
图 8 双倒数作图分析GFA酶学性质 Fig.8 Lineweaver−Burk plots of GFA |
前期研究发现,bacilysin是一种新型的、高效的抗真菌化合物[22]。由于bacilysin在结构上属于谷氨酰胺的结构类似物,因而探索其对谷氨酰胺类化合物的代谢过程调控具有重要的意义。本研究中,当在GFA的反应体系中加入bacilysin后,其转氨酶活性明显降低,20 min内其残余酶活仅剩45.2%,且随着时间的延长,该酶的催化活力剧烈下降(图 9)。这与作者之前的研究结果吻合,其最低抑菌浓度达到1.8 μg·mL-1[23]。这主要是由于抑制N-乙酰-D-葡糖胺的生成,造成白色念珠菌的细胞壁无法正常合成[24],实现高效的抗真菌作用。
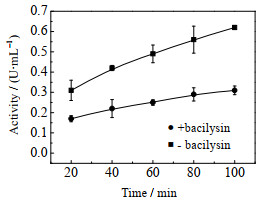
|
图 9 Bacilysin对GFA酶活的抑制作用 Fig.9 Inhibitory effects of bacilysin on the catalytic activity of GFA |
通过本研究,在大肠杆菌BL21(DE3) Codon plus中成功表达来源于致病菌白色假丝酵母(Candida albicans ATCC 64548)中、具有活性的葡萄糖-6-磷酸合成酶。通过诱导条件优化,得到GFA的最佳异源表达条件为:诱导剂浓度为0.1 mmol·L-1;诱导温度为28 ℃;诱导时间为15 h;诱导时机为OD600达到1.0。
确定了最佳的反应条件:反应温度为37 ℃;反应pH为7.2,反应时间为20 min,通过双倒数方程确定米氏常数KM为0.47 mg·mL-1。在此基础上探索其在抗真菌领域的应用,当反应体系中加入抑制剂(bacilysin)时,GFA转氨酶活性会显著降低54.8 %,且随着时间的增加抑制作用越明显。
本研究对于进一步提高葡萄糖胺-6-磷酸合成酶的应用潜力,加快新型抗真菌药物的研发具有重要意义。
| [1] |
MILEWSKI S. Glucosamine-6-phosphate synthase-the multi——facets enzyme[J]. Biochimica Et Biophysica Acta, 2002, 1597(2): 173-192. DOI:10.1016/S0167-4838(02)00318-7 |
| [2] |
DURAND P, GOLINELLI-PIPANEAU B, MOUILLERON S, et al. Highlights of glucosamine-6P synthase catalysis[J]. Archives of Biochemistry & Biophysics, 2008, 474(2): 302-317. |
| [3] |
MOUILLERON S, BADET-DENISOT M A, BADET B, et al. Dynamics of glucosamine-6-phosphate synthase catalysis[J]. Archives of Biochemistry & Biophysics, 2011, 505(1): 1-12. |
| [4] |
WANG S, LI P, SU J, et al. Characterization and expression of glucosamine-6-phosphate synthase from Saccharomyces cerevisiae in Pichia pastoris[J]. Biotechnology Letters, 2014, 36(10): 2023-2028. DOI:10.1007/s10529-014-1561-y |
| [5] |
MISZKIEL A, WOJCIECHOWSKI M. Long range molecular dynamics study of interactions of the eukaryotic glucosamine -6- phosphate synthase with fructose-6-phosphate and UDP-GlcNAc[J]. Journal of Molecular Graphics & Modelling, 2017, 78: 14-25. |
| [6] |
KATO N, DASGUPTA R, SMARTT C T, et al. Glucosamine: fructose-6-phosphate aminotransferase: gene characterization, chitin biosynthesis and peritrophic matrix formation in Aedes aegypti[J]. Insect Molecular Biology, 2010, 11(3): 207-216. |
| [7] |
FLOQUET N, RICHEZ C, DURAND P, et al. Discovering new inhibitors of bacterial glucosamine-6P synthase (GlmS) by docking simulations[J]. Bioorganic & Medicinal Chemistry Letters, 2007, 17(7): 1966-1970. |
| [8] |
RAMAKRISHNAN J, RATHORE S, RAMAN S. Review on fungal enzyme inhibitors-potential drug targets to manage human fungal infections[J]. RSC Advances, 2016, 6(48): 42387-42401. DOI:10.1039/C6RA01577H |
| [9] |
MOUILLERON S, BADET-DENISOT M A, PECQUEUR L, et al. Structural basis for morpheein-type allosteric regulation of Escherichia coli glucosamine-6-phosphate synthase: equilibrium between inactive hexamer and active dimer[J]. Journal of Biological Chemistry, 2012, 287(41): 34533-34546. DOI:10.1074/jbc.M112.380378 |
| [10] |
BENEITO-CAMBRA M, GAREIL P, BADET B, et al. First investigations for the characterization of glucosamine-6-phosphate synthase by capillary electrophoresis[J]. Journal of Chromatography B, 2017, 1072: 130-135. |
| [11] |
夏颖, 赵杰, 夏黎明. 一种新型内切-β-葡聚糖酶基因在里氏木霉中的重组与表达[J]. 高校化学工程学报, 2016, 30(3): 626-632. XIA Y, ZHAO J, XIA L M. Recombination and expression of a novel endo-β-glucanase gene from Penicillium Echinulatum in Trichoderma Reesei[J]. Journal of Chemical Engineering of Chinese Universities, 2016, 30(3): 626-632. |
| [12] |
RACZNSKA J, OLCHOWY J, KONARIEV P V, et al. The crystal and solution studies of glucosamine-6-phosphate synthase from Candida albicans[J]. Journal of Molecular Biology, 2007, 372(3): 672-688. DOI:10.1016/j.jmb.2007.07.002 |
| [13] |
HUANG X, TSUJI N, MIYOSHI T, et al. Characterization of glutamine: Fructose-6-phosphate aminotransferase from the ixodid tick, Haemaphysalis longicornis, and its critical role in host blood feeding[J]. International Journal for Parasitology, 2007, 37(3/4): 383-92. |
| [14] |
WANG Y H, JING C F, YANG B, et al. Production of a new sea anemone neurotoxin by recombinant Escherichia coli: Optimization of culture conditions using response surface methodology[J]. Process Biochemistry, 2005, 40(8): 2721-2728. DOI:10.1016/j.procbio.2004.12.024 |
| [15] |
MURBY M, UHLEN M, STAHL S. Upstream strategies to minimize proteolytic degradation upon recombinant production in Escherichia coli[J]. Protein Expression & Purification, 1996, 7(2): 129-136. |
| [16] |
BEIGI L, KARBALAEI-HEIDARI H R, KHARRATI-KOPAEI M. Optimization of an extracellular zinc-metalloprotease (SVP2) expression in Escherichia coli BL21(DE3) using response surface methodology[J]. Protein Expression & Purification, 2012, 84(1): 161-166. |
| [17] |
KRZEWINSKI F, BRASSART C, GAVINI F, et al. Characterization of the lactose transport system in the strain Bifidobacterium bifidum DSM 20082[J]. Current Microbiology, 1996, 32(6): 301-307. DOI:10.1007/s002849900054 |
| [18] |
MALAKAR P, VENKATESH K V. Effect of substrate and IPTG concentrations on the burden to growth of Escherichia coli on glycerol due to the expression of lac proteins[J]. Applied Microbiology and Biotechnology, 2012, 93(6): 2543-2549. DOI:10.1007/s00253-011-3642-3 |
| [19] |
VILLAVERDE A, CARRIO M M. Protein aggregation in recombinant bacteria: Biological role of inclusion bodies[J]. Biotechnology Letters, 2003, 25(17): 1385-1395. DOI:10.1023/A:1025024104862 |
| [20] |
SHACHRAI I, ZASLAVER A, ALON U, et al. Cost of unneeded proteins in E. coli is reduced after several generations in exponential growth[J]. Molecular Cell, 2010, 38(5): 758-767. DOI:10.1016/j.molcel.2010.04.015 |
| [21] |
STOEBEL D M, DEAN A M, DYKHUIZEN D E. The cost of expression of Escherichia coli lac operon proteins is in the process, not in the products[J]. Genetics, 2008, 178(3): 1653-1660. DOI:10.1534/genetics.107.085399 |
| [22] |
WANG T, LIU X H, WU M B, et al. Molecular insights into the antifungal mechanism of bacilysin[J]. Journal of Molecular Modeling, 2018, 24(5): 118-128. DOI:10.1007/s00894-018-3645-4 |
| [23] |
WANG T, WU M B, CHEN Z J, et al. Separation, determination and antifungal activity test of the products from a new Bacillus amyloliquefaciens[J]. Natural Product Research, 2016, 30(10): 1215-1218. DOI:10.1080/14786419.2015.1048246 |
| [24] |
WEI W L, MONARD M, GAULD J. Computational insights into substrate binding and catalytic mechanism of the glutaminase domain of glucosamine-6-phosphate synthase (GlmS)[J]. Rsc Advances, 2017, 7(47): 29626-29638. DOI:10.1039/C7RA04906D |




