扩展功能
文章信息
- 王鸿宇, 阎云基, 郭天雪, 潘亚文
- WANG Hong-Yu, YAN Yun-Ji, GUO Tian-Xue, PAN Ya-Wen
- CircRNA对胶质瘤生物学功能作用机制的研究进展
- Research progress in the biological function and mechanism of circRNA in glioma
- 国际神经病学神经外科学杂志, 2020, 47(3): 320-324
- Journal of International Neurology and Neurosurgery, 2020, 47(3): 320-324
-
文章历史
收稿日期: 2019-11-29
修回日期: 2020-05-12
2. 兰州大学第二医院神经外科, 甘肃 兰州 730030;
3. 兰州大学神经病学研究所, 甘肃 兰州 730030
2. Department of Neurosurgery, Second Hospital of Lanzhou University, Lanzhou, Gansu 730030, China;
3. Institute of Neurology, Lanzhou University, Lanzhou, Gansu 730030, China
神经胶质瘤是中枢神经系统中最常见的恶性肿瘤,占全部恶性脑肿瘤的80%以上,具有高发病率和高死亡率的特征[1, 2]。在过去的几十年里,神经胶质瘤总发病率和病死率在全球范围内的显著增加[3]。由于胶质瘤的高侵袭性, 尽管在胶质瘤治疗中, 手术治疗联合放疗或化疗取得进展,但是患者复发率较高,预后较差[4]。根据中国胶质瘤基因组图谱(CGGA)统计,神经胶质瘤的总体中位生存时间(OS)大约为37.6个月,其中高级别胶质瘤(间变性胶质瘤,多形性胶质母细胞瘤等)OS为14.4个月,低级别胶质瘤(WHOⅡ级)OS为78.1个月[5]。因此,探索胶质瘤发生发展的分子机制,寻找有效的治疗靶点,对改善胶质瘤患者的预后至关重要。
环状RNA(CircRNA)是通过不同的机制产生的一类内源性具有重要生物学功能的非编码RNA,它广泛存在于病毒、原核及真核生物的细胞结构中[6-7]。CircRNA在过去的数十年里被误认为是RNA转录后的剪接错误,表现为不具有蛋白质编码能力的封闭环状RNA。越来越多的研究表明,环状RNA通过充当microRNA (miRNA)海绵和RNA结合蛋白的调节子,参与真核细胞的基本生物学过程[8]。Li等[9]发现CircRNA的异常表达可调节肿瘤的发生和发展,并有希望作为肿瘤诊断和预后的生物标志物。此外,越来越多的CircRNA被证明在结直肠癌、胃癌、乳腺癌和神经胶质瘤都充当是miRNA海绵[10-12]。
1 CircRNA的生成及调控CircRNA在过去被认为是一类不具有蛋白质编码能力的封闭CircRNA。由于CircRNA具有非线性构象,且缺乏聚腺苷酸[poly(A)]的尾巴,因此,在新一代RNA测序(RNA-seq)分析中很少见到CircRNA,只有使用non-poly(A) RNAs或使用RNase R(一种优先于消化线性RNA的酶)对CircRNA进行分析,才能发现环状RNA在外显子的前体mRNA(pre-mRNA)反向剪接过程中广泛表达[7]。尽管CircRNA的表现形式不同,但当前大多数研究发现CircRNA是由外显子的前体mRNA(pre-mRNA)反向剪接产生的,其中正向5'剪接位点(ss)与反向3'剪接位点(ss)连接在一起,并在连接位点通过3'-5'磷酸二酯键连接产生的RNA环[13]。CircRNA的形成取决于顺式调控元件和反式作用因子,其一般通过调控控制pre-mRNA的转录产生circRNA的Pol Ⅱ,影响反向剪接的效率,调节CircRNA转换对于细胞中稳态CircRNA表达水平进行调节[7]。Pol Ⅱ转录和mRNA的前处理紧密耦合,并且转录延伸率是决定剪接结果的重要因素[14]。CircRNA形成外显子的反向剪接可能在转录前和转录后同时发生[7]。
2 CircRNA调节肿瘤细胞生物学特性的机制Conn等[15]发现数百个CircRNA在人类上皮-间质转化(EMT)期间受到调控,表明某些CircRNA可能影响人类EMT相关的细胞功能,例如迁移,侵袭和肿瘤转移。在神经元分化过程中,CircRNA表达的增加通常与线性宿主转录物的上调相关,CircRNA可能被神经元末端和分子突触后平台用作突触标志物来保持分子记忆,由于其稳定性,CircRNA可以充当拓扑复杂的平台来组装核糖核蛋白(ribonucleoprotein,RNP)颗粒、转运蛋白或RNA[16]。表明CircRNA与肿瘤的发生发展密切相关。CircRNA调节肿瘤细胞生物学特性的机制见图 1。
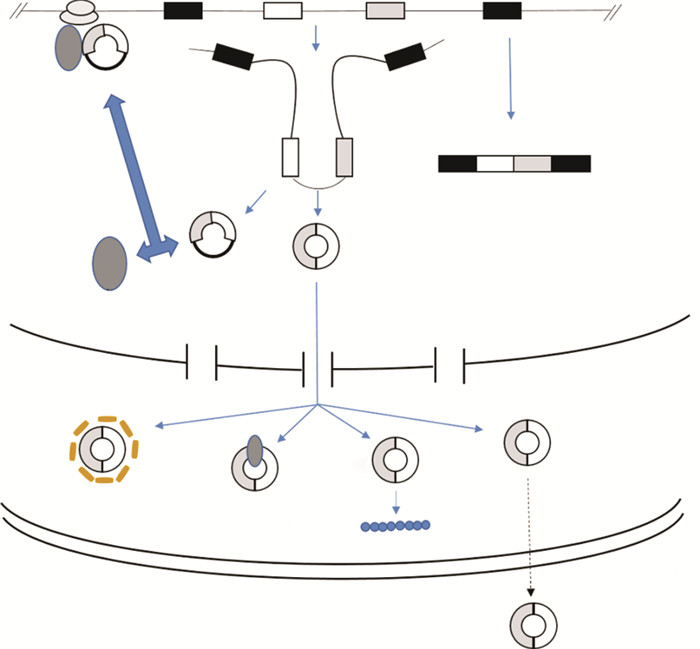
|
| (a)CircRNA的加工影响其线性mRNA对应物的剪接;(b)CircRNA可以调节其亲本基因的转录;(c)CircRNA可以充当miRNA海绵;(d)CircRNA可以与蛋白质相互作用并影响其作用方式;(e)CircRNA可以翻译; (f)CircRNA可以作为检验的分子标志物。 图 1 CircRNA调节肿瘤细胞生物学特性的机制 |
由于大多数CircRNA均来自蛋白质编码基因的中间外显子,CircRNA的加工会影响其前体转录物的剪接,从而导致基因表达结果发生变化[17]。尽管反向剪接效率不如正常剪接,但已证明在CircRNA生物发生中使用5'和3'ss可与mRNA剪接竞争,从而导致具有外显子包涵体的线性mRNA水平降低[17-18]。
2.2 CircRNA可以调节其亲本基因的转录保留内含子的CircRNA(EIcircRNA)可以与U1小核糖核蛋白(U1snRNPs)相互作用,EIcircRNA-U1 snRNP复合物在其亲本基因的启动子上与Pol Ⅱ结合,从而增强基因表达,阻断此类RNA-RNA相互作用会破坏EIcircRNA与Pol Ⅱ的相互作用,从而降低其亲本基因的转录[19]。
2.3 CircRNA可以充当miRNA海绵CircRNA分子海绵机制是指主要分布在恶性肿瘤的细胞质中的CircRNA具有分子海绵作用,有与miRNA相互作用的可能性,从而影响相应的miRNA调控的靶向转录物的释放[20]。其中CircRNA的分子海绵机制被证实在神经胶质瘤、胃癌、乳腺癌等多种恶性肿瘤中对肿瘤细胞的生长、增殖、侵袭、迁移还有自噬等细胞生理功能具有明确的调节作用[10-12]。例如,CircRNA U2AF1可以通过上调NOVA2和吸收miR-7-5p来促进神经胶质瘤的进展[9]。CircRNA NFIX通过海绵miR-34a-5p促进胶质瘤进展并调节notch信号通路[21]。
2.4 CircRNA可以与蛋白质相互作用并影响其作用方式CircRNA可以与不同的蛋白质相互作用形成特定的CircRNP,这些核糖核酸还原酶(RNR)随后会影响相关蛋白质的作用方式[7]。Ashwal-fluss等[22]发现多功能蛋白MBL可以促进由同一基因位点产生的circMbl的生物学功能,还发现MBL蛋白与CircMbl相关,因此,他们推测在MBL和CircMbl产生之间存在反馈回路,当蛋白质过量时,它会通过促进circMbl产生而降低自身mRNA的产生,这种CircRNA也可以通过结合多余的MBL蛋白来消除它。
2.5 CircRNA可以翻译Salzman等[23]发现反向剪接产生的绝大多数有意义的CircRNA主要位于细胞质中,因此,他们提出了一个有趣的问题,即它们是否可翻译。Legnini等[24]发现Circ-ZNF609在肌肉分化中的功能导致特异性控制成肌细胞增殖与重链多核糖体相关,并以剪接依赖性和帽依赖性的方式翻译成蛋白质,从而提供了真核生物中编码蛋白质CircRNA的一个例子。
2.6 CircRNA可以作为检验的分子标志物固有的圆环形特征使CircRNA在细胞内和细胞外血浆(包括血液和唾液)中异常稳定[25]。因此,CircRNA可能可以作为某些检验的分子标志物。
3 CircRNA在胶质瘤中的研究Rybak-wolf等[16]分析了数千种人和小鼠的神经元CircRNA,发现CircRNA在哺乳动物大脑中异常丰富,且CircRNA在神经元分化过程中总体上调,与其mRNA同工型相比通呈现差异表达。比较人类额叶皮层、甲状腺、肝脏和肌肉,发现神经系统中CircRNA表达总体富集,并且神经细胞和神经胶质细胞均可诱导CircRNA产生,circRNA虽然在神经元之间中分布不均,但在突触中高度富集,揭示大脑的不同部位都产生特定的高表达CircRNA,神经元分化过程中CircRNA表达均增加[16]。
3.1 CircRNA调节胶质瘤细胞的生长、增殖、侵袭、凋亡关于CircRNA调节胶质瘤细胞的生长、增殖、侵袭、凋亡方面的研究较多。Peng等[26]发现circCPA4(hsa_circ_0082374)在神经胶质瘤组织呈现高表达,在敲除circCPA4后发现其可抑制神经胶质瘤中的细胞增殖和转移,且circCPA4与let-7相互作用并充当let-7的海绵,通过竞争性内源性RNA(ceRNA)机制,circCPA4海绵通过let-7调节CPA4的表达和神经胶质瘤的进展。Chen等[27]发现circ-EZH2在神经胶质瘤中过表达,且circ-EZH2的异位表达充当miR-1265的miRNA海绵,通过下调胶质瘤细胞中的miR-1265释放其对DDAH1和CBX3起抑制作用,显著促进细胞生长,迁移和侵袭,但抑制细胞凋亡。Chen等[28]还发现circ_0074026的高表达和与患者的肿瘤大小、WHO分级、无进展生存期和总生存期有关。Zhang等[33]发现了CircRNAs可能通过由长基因间非蛋白编码RNA p53诱导产生多肽转录物(LINC-PINT),该肽可直接与聚合酶相关因子复合物(PAF1c)相互作用,并抑制多个癌基因的转录延伸。
3.2 CircRNA调节胶质瘤细胞的血管生成转录因子SP1可以与血管内皮生长因子A(VEGFA)的启动子区域结合以增加其转录水平。Meng等[29]研究发现circSCAF11在神经胶质瘤细胞中上调,敲除circSCAF11可抑制肿瘤细胞增殖、侵袭和生长并诱导G0/G1期停滞。CircSCAF11通过使miR-421海绵化而正调控SP1的表达,转录因子SP1与血管内皮细胞生长因子A(VEGFA)启动子的结合,激活VEGFA的转录,从而在神经胶质瘤的发展中构建了CircSCAF11/ miR-421 / SP1 / VEGFA轴正向调控,支持神经胶质瘤的血管生成和肿瘤发展[29]。
3.3 CircRNA调节胶质瘤细胞对化疗药物的敏感性Hua等[30]发现CircRNA CEP128在神经胶质瘤组织和细胞系中上调,CircRNA CEP128的抑制可通过调节miR-145-5p抑制胶质瘤细胞中的细胞增殖并提高替莫唑胺(TMZ)的细胞毒性。此外,Meng等[29]在进行CircRNA调节胶质瘤血管生成方面研究时也发现CircSCAF11通过CircSCAF11/ miR-421 / SP1 / VEGFA轴正向调控,可增强细胞对替莫唑胺的抗性。
3.4 CircRNA调节胶质瘤BTB渗透性血液-肿瘤屏障(BTB)的存在严重限制了抗肿瘤药物向脑神经胶质瘤组织的有效跨屏障传递。Wu等[31]研究发现KHDRBS3蛋白可以通过CircRNA-cDENND4C/miR-577轴调节BTB的通透性,其机制为CircRNA-cDENND4C充当miR-577的分子海绵,该分子海绵与miR-577结合并抑制了其对靶基因ZO-1,occludin和claudin-1的负调控,从而调节了BTB的通透性,可有效促进抗肿瘤药多柔比星跨BTB传递从而诱导胶质瘤的凋亡,但在该研究中CircRNA并非最上游调控序列,KHDRBS3蛋白可结合到CircRNA-cDENND4C,并增加了其稳定性,抑制CircRNA-cDENND4C下调miR-577,从而提高了BTB的通透性。
3.5 CircRNA调节胶质瘤细胞抗辐射作用Zhao等[32]发现从U251细胞中分离出的细胞外囊泡(ev)和从抗辐射U251细胞中分离出的ev之间的CircRNA,利用RNA测序共检测到1 235个CircRNA,与U251细胞的细胞外囊泡(Nor ev)相比,抗辐射U251细胞中的细胞外囊泡(RR ev)中circATP8B4明显上调,通过实验验证RR EVs中circATP8B4作为U251 miR 766海绵并起到促进细胞耐辐射作用,并且来自RR evs的circATP8B4可以转移到正常胶质瘤U251细胞上发挥相同的作用。
4 总结胶质瘤是人类颅脑肿瘤中发病率最高,危害最大的恶性肿瘤,在过去十余年的研究中,尽管在胶质瘤治疗中手术治疗联合放疗或化疗取得进展,但胶质瘤的预后仍然不佳。CircRNA作为一种早已被发现却早期对其认识不足的非编码RNA分子,现已有多种CircRNA被证实可以通过分子海绵机制对胶质瘤的发生发展产生影响,随着CircRNA对胶质瘤作用机制的进一步研究,在不久的将来,可能会给我们在肿瘤疾病的治疗中提供更多的思路,也许这些机制可能指导我们在胶质瘤的治疗中取得突破性的进展。
| [1] |
Ricard D, Idbaih A, Ducray F, et al. Primary brain tumours in adults[J]. Lancet (London, England), 2012, 379(9830): 1984-1996. DOI:10.1016/S0140-6736(11)61346-9 |
| [2] |
Jiang T, Mao Y, Ma WB, et al. CGCG clinical practice guidelines for the management of adult diffuse gliomas[J]. Cancer letters, 2016, 375(2): 263-273. DOI:10.1016/j.canlet.2016.01.024 |
| [3] |
Milano MT, Johnson MD, Sul J, et al. Primary spinal cord glioma:a Surveillance, Epidemiology, and End Results database study[J]. J Neurooncol, 2010, 98(1): 83-92. DOI:10.1007/s11060-009-0054-7 |
| [4] |
Zheng SQ, Qi Y, Wu J, et al. CircPCMTD1 Acts as the Sponge of miR-224-5p to Promote Glioma Progression[J]. Front Oncol, 2019, 9: 398. DOI:10.3389/fonc.2019.00398 |
| [5] |
Patel C K, Vemaraju R, Glasbey J, et al. Trends in peri-operative performance status following resection of high grade glioma and brain metastases:The impact on survival[J]. Clinical neurology and neurosurgery, 2018, 164(67-71). |
| [6] |
Greene J, Baird AM, Brady L, et al. Circular RNAs:Biogenesis, Function and Role in Human Diseases[J]. Frontiers in molecular biosciences, 2017, 4: 38. DOI:10.3389/fmolb.2017.00038 |
| [7] |
Li X, Yang L, Chen LL. The Biogenesis, Functions, and Challenges of Circular RNAs[J]. Molecular cell, 2018, 71(3): 428-442. DOI:10.1016/j.molcel.2018.06.034 |
| [8] |
Sun HD, Xi PC, Sun ZQ, et al. Circ-SFMBT2 promotes the proliferation of gastric cancer cells through sponging miR-182-5p to enhance CREB1 expression[J]. Cancer manag Res, 2018, 10: 5725-5734. DOI:10.2147/CMAR.S172592 |
| [9] |
Li GX, Huang M, Cai YQ, et al. Circ-U2AF1 promotes human glioma via derepressing neuro-oncological ventral antigen 2 by sponging hsa-miR-7-5p[J]. J Cell Physiol, 2019, 234(6): 9144-9155. DOI:10.1002/jcp.27591 |
| [10] |
Xie HJ, Ren XL, Xin SN, et al. Emerging roles of circRNA_001569 targeting miR-145 in the proliferation and invasion of colorectal cancer[J]. Oncotarget, 2016, 7(18): 26680-26691. DOI:10.18632/oncotarget.8589 |
| [11] |
Kulcheski FR, Christoff AP, Margis R. Circular RNAs are miRNA sponges and can be used as a new class of biomarker[J]. J Biotechnol, 2016, 238: 42-51. DOI:10.1016/j.jbiotec.2016.09.011 |
| [12] |
Li PF, Chen SC, Chen HL, et al. Using circular RNA as a novel type of biomarker in the screening of gastric cancer[J]. Clinica chimica acta; international journal of clinical chemistry, 2015, 444: 132-136. DOI:10.1016/j.cca.2015.02.018 |
| [13] |
Chen LL. The biogenesis and emerging roles of circular RNAs[J]. Nature reviews Molecular cell biology, 2016, 17(4): 205-211. |
| [14] |
Bentley DL. Coupling mRNA processing with transcription in time and space[J]. Nature reviews Genetics, 2014, 15(3): 163-175. DOI:10.1038/nrg3662 |
| [15] |
Conn SJ, Pillman KA, Toubia J, et al. The RNA binding protein quaking regulates formation of circRNAs[J]. Cell, 2015, 160(6): 1125-1134. DOI:10.1016/j.cell.2015.02.014 |
| [16] |
Rybak-Wolf A, Stottmeister C, Glazar P, et al. Circular RNAs in the Mammalian Brain Are Highly Abundant, Conserved, and Dynamically Expressed[J]. Molecular cell, 2015, 58(5): 870-885. DOI:10.1016/j.molcel.2015.03.027 |
| [17] |
Zhang XO, Wang HB, Zhang Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 159(1): 134-147. |
| [18] |
Kelly S, Greenman C, Cook P R, et al. Exon Skipping Is Correlated with Exon Circularization[J]. Journal of molecular biology, 2015, 427(15): 2414-2417. DOI:10.1016/j.jmb.2015.02.018 |
| [19] |
Li ZY, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nature structural & molecular biology, 2015, 22(3): 256-264. |
| [20] |
Hansen TB, Jensen TI, Clausen BH, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384-388. DOI:10.1038/nature11993 |
| [21] |
Xu HY, Zhang Y, Qi L, et al. NFIX Circular RNA Promotes Glioma Progression by Regulating miR-34a-5p via Notch Signaling Pathway[J]. Front Mol Neurosci, 2018, 11: 225. DOI:10.3389/fnmol.2018.00225 |
| [22] |
Ashwal-Fluss R, Meyer M, Pamudurti NR, et al. circRNA biogenesis competes with pre-mRNA splicing[J]. Molecular cell, 2014, 56(1): 55-66. |
| [23] |
Salzman J, Gawad C, Wang PL, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J]. PloS one, 2012, 7(2). |
| [24] |
Legnini I, Di Timoteo G, Rossi F, et al. Circ-ZNF609 Is a Circular RNA that Can Be Translated and Functions in Myogenesis[J]. Molecular cell, 2017, 66(1): 22-37. |
| [25] |
Memczak S, Papavasileiou P, Peters O, et al. Identification and Characterization of Circular RNAs As a New Class of Putative Biomarkers in Human Blood[J]. PloS one, 2015, 10(10): e0141214. DOI:10.1371/journal.pone.0141214 |
| [26] |
Peng H, Qin CY, Zhang C, et al. circCPA4 acts as a prognostic factor and regulates the proliferation and metastasis of glioma[J]. Journal of cellular and molecular medicine, 2019, 23(10): 6658-6665. DOI:10.1111/jcmm.14541 |
| [27] |
Gao FQ, Du Y, Zhang Y, et al. Circ-EZH2 Knockdown Reverses DDAH1 and CBX3-Mediated Cell Growth and Invasion in Glioma through miR-1265 Sponge Activity[J]. Gene, 2020, Feb5: 144196. |
| [28] |
Chen MH, Liu XJ, Xie P, et al. Circular RNA circ_0074026 indicates unfavorable prognosis for patients with glioma and facilitates oncogenesis of tumor cells by targeting miR-1304 to modulate ERBB4 expression[J]. J Cell Physiol, 2020, 235(5): 4688-4697. DOI:10.1002/jcp.29347 |
| [29] |
Meng Q, Li S, Liu Y, et al. Circular RNA circSCAF11 Accelerates the Glioma Tumorigenesis through the miR-421/SP1/VEGFA Axis[J]. Mol Ther Nucleic Acids, 2019, 17: 669-677. DOI:10.1016/j.omtn.2019.06.022 |
| [30] |
Hua L, Huang L, Zhang X, et al. Knockdown of circular RNA CEP128 suppresses proliferation and improves cytotoxic efficacy of temozolomide in glioma cells by regulating miR-145-5p[J]. Neuroreport, 2019, 30(18): 1231-1238. DOI:10.1097/WNR.0000000000001326 |
| [31] |
Wu P, Gao Y, Shen SY, et al. KHDRBS3 regulates the permeability of blood-tumor barrier via cDENND4C/miR-577 axis[J]. Cell death Dis, 2019, 10(7): 536. DOI:10.1038/s41419-019-1771-2 |
| [32] |
Zhao MJ, Xu JJ, Zhong SL, et al. Expression profiles and potential functions of circular RNAs in extracellular vesicles isolated from radioresistant glioma cells[J]. Oncol Rep, 2019, 41(3): 1893-1900. |
| [33] |
Zhang ML, Zhao K, Xu XP, et al. A peptide encoded by circular form of LINC-PINT suppresses oncogenic transcriptional elongation in glioblastoma[J]. Nat Commun, 2018, 9(1): 4475. |
 2020, Vol. 47
2020, Vol. 47