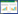扩展功能
文章信息
- 谭颖, 管宇宙, 朱立, 崔丽英
- TAN Ying, GUAN Yu-Zhou, ZHU Li, CUI Li-Ying
- 乙酰胆碱受体抗体阳性重症肌无力外周血单个核细胞miRNA表达谱
- Expression profile of microRNAs in peripheral blood mononuclear cells of patients with myasthenia gravis with acetylcholine receptor antibodies
- 国际神经病学神经外科学杂志, 2019, 46(6): 589-595
- Journal of International Neurology and Neurosurgery, 2019, 46(6): 589-595
-
文章历史
收稿日期: 2019-09-21
修回日期: 2019-12-03
2. 中国医学科学院北京协和医院核医学科, 北京市 100010;
3. 中国医学科学院神经科学中心, 北京市 100010
乙酰胆碱受体(anti-acetylcholine receptor, AchR)抗体阳性的重症肌无力(myasthenia gravis, MG)患者占总体85%~90%,其发病被认为与体液免疫相关,胸腺中的异常生发中心伴B细胞浸润与AchR抗体滴度有关[1],但抗体和基因对临床疗效分层的价值目前仍不明晰[2]。本病涉及到许多复杂的机制,如何理解各临床型之间的转化、如何更好地监测病情波动等,仍是临床研究面临的问题。
miRNA一般由22个核苷酸组成,可以特异性结合到目标mRNA的3’端,导致mRNA降解或抑制下游蛋白的表达,调控目标基因的转录和表达。在MG中差异表达的miRNA参与的生物学过程涵盖了淋巴细胞功能、细胞因子产生等。Cron等[3]发现早发型MG的miR-150上调。Cao等[4]发现miR-653通过干扰TRIM9的功能影响淋巴细胞的增生和凋亡。Bao等[5]筛选了126种与MG相关的miRNA,并确定了上述miRNA的富集通路。这些研究为miRNA在重症肌无力中的作用奠定了理论证据。本实验将对比未经免疫治疗的AchR-MG患者和正常人外周血单个核细胞的miRNA,并采用高通量测序,以提高实验的敏感性。通过生物富集分析,预测对AchR-MG发病可能产生影响的通路,为进一步的研究打下理论基础。
1 对象与方法 1.1 研究对象本研究采用病例对照研究方法,从北京协和医院门诊入组4例AchR-MG,所有患者为首次诊断,尚未接受任何免疫性治疗。纳入标准:①有重症肌无力典型的临床和电生理特征;②放射免疫沉淀法检测外周血血清中乙酰胆碱受体抗体≥0.4 nmol/L。同期入组了4例年龄、性别匹配的正常对照组,正常对照组排除标准:①任何已知的自身免疫性疾病病史;②任何已知的肿瘤病史;③合并高血压、糖尿病。研究对象基本资料见表 1。本研究获得了北京协和医院伦理委员会审批,所有入组患者均签署了知情同意书。研究方案获得了北京协和医院研究委员会审核,许可号JS-1685。
| 编号 | 性别 | 年龄 | 病程(年) | MGFA评分 | MGFA分型 | AchR-Ab(nmol/L) | 其他自身抗体 | 免疫药物 |
| 1 | 女 | 28 | 1 | 4 | IIA | 5.83 | 无 | 无 |
| 2 | 女 | 58 | 1 | 8 | IIA | 3.41 | ANA1:80 | 无 |
| 3 | 男 | 40 | 3 | 6 | IIA | 1.28 | AMA-M2弱阳性 | 无 |
| 4 | 男 | 68 | 1.5 | 12 | IIB | 10.47 | 无 | 无 |
| 注:MGFA:美国重症肌无力协会;AchR-Ab:乙酰胆碱受体抗体滴度;ANA:抗核抗体;AMA-M2:抗线粒体抗体M2型 | ||||||||
在获得研究对象书面同意后采取血样8 ml,混入PBS,转入淋巴细胞分离液,离心后清洗、回收;溶解于TRIzol后,放入-80℃冰箱保存。用TRIzol(Invitrogen, Carlsbad, CA, USA)试剂法提取收集RNA,Agilent 2100 Bioanalyzer检测样品纯度。
1.2.2 提取miRNA和构建cDNA文库采用15%聚丙烯酰胺胶对总RNA进行分离,选择miRNA(18~30 nt)。根据TruSeq Small RNA Sample Preparation Kit(Illumina)流程进行文库制备。使用Qubit 2.0对文库进行初步定量至浓度为1 ng/μl,用Agilent 2100对文库的插入片段大小进行检测,再通过Q-PCR使文库有效浓度>2 nM。
1.2.3 高通量测序将miRNA和外源人工合成的miR-168内参稀释至10nM后,进行测序(Illumina, Santiago, CA, USA),用测序软件进行实时分析,后续用ACGT 101-miR软件(LC Sciences, Houston, TX, USA)进行分析。
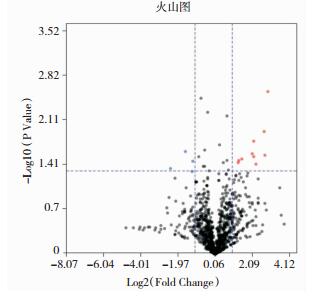
1.2.4 差异miRNA筛选用主成分分析法(Principal Component Analysis)分析两组内各样本的生物学重复性,差异miRNA用DEseq程序分析,差异定义为P < 0.05(P值经过多重矫正)。按照差异miRNA的P值从小到大进行排序,如表 2和表 3。分别绘制火山图(图 1)和热图(图 2)。
| miRNA | log2(fold change) | t值 | P值 |
| mmu-miR-3968 | 0.47 | 7.59 | 3.05×10-4 |
| miR-4785 | 2.90 | 5.37 | 2.66×10-3 |
| miR-2277-5p_R-1 | 0.66 | 4.19 | 6.50×10-3 |
| miR-1273h-5p | 2.67 | 3.44 | 1.13×10-2 |
| miR-1264_R-1 | 2.12 | 3.47 | 1.64×10-2 |
| miR-130b-5p_R+1 | 0.22 | 3.20 | 1.85×10-2 |
| miR-7977_R+3_1ss6AG | 0.38 | 4.49 | 2.07×10-2 |
| hsa-miR-1-3p_1ss21AG | 2.04 | 4.09 | 2.58×10-2 |
| hsa-miR-1273c_R-1 | 2.70 | 3.10 | 2.69×10-2 |
| hsa-miR-3677-3p | 2.10 | 2.99 | 2.90×10-2 |
| eca-miR-191b_1ss2AG | 1.45 | 2.91 | 3.14×10-2 |
| miR-3140-5p | 1.31 | 3.01 | 3.26×10-2 |
| miR-1296-5p | 0.66 | 3.48 | 3.28×10-2 |
| miR-1285-3p | 1.24 | 2.73 | 3.54×10-2 |
| miR-532-5p | 0.45 | 2.70 | 3.56×10-2 |
| ssc-miR-339 | 0.22 | 2.84 | 3.73×10-2 |
| miR-21-3p | 0.73 | 2.67 | 4.62×10-2 |
| miRNA | log2(fold change) | t值 | P值 |
| miR-210-3p | -0.77 | -5.08 | 3.43×10-3 |
| miR-664a-3p | -0.43 | -4.35 | 5.73×10-3 |
| miR-101-3p_R+1 | -0.56 | -3.05 | 2.27×10-2 |
| bta-miR-2484 | -1.63 | -3.04 | 2.35×10-2 |
| miR-548ak_R-1 | -0.88 | -3.06 | 2.89×10-2 |
| miR-19b-3p | -1.23 | -2.77 | 3.33×10-2 |
| miR-20a-5p | -0.66 | -3.12 | 4.02×10-2 |
| miR-20b-5p | -0.58 | -2.70 | 4.12×10-2 |
| PC-5p-18860_70 | -0.44 | -2.63 | 4.47×10-2 |
| hsa-miR-26b-5p_R+1 | -0.31 | -2.50 | 4.75×10-2 |
| hsa-miR-19a-3p | -1.28 | -2.53 | 4.97×10-2 |
|
| 注:以log2(fold change)为横坐标,-log10(P值)为纵坐标,对差异表达分析中所有的miRNA绘制火山图。其中散点代表miRNA在不同样本中差异表达倍数变化。阈值|log2(Foldchange)| > 1,P < 0.05。红色代表上调miRNA,蓝色代表下调miRNA,灰色的点未达到上述差异标准的miRNA。 图 1 差异miRNA的火山图 |
|
| 注:纵坐标代表不同样本,实验组为MG1、MG2、MG3和MG4,对照组为HC1、HC2、HC3和HC4。不同色块表示miRNA在不同样品中表达变化。表达量越大越接近红色,表达量相对较小接近蓝色。图上方的树形图对不同样本的聚类分析可以看到,实验组的总体miRNA表达与对照组存在较大差异。左侧树状图代表来自不同样本不同基因的聚类结果。 图 2 差异miRNA的热图 |
采用TargetScan和miRanda软件对差异miRNA分子进行靶基因预测,将TargetScan算法中靶基因得分小于50的舍去,同时将miRanda算法中最大自由能大于-10的去除。
1.2.6 基因条目和京都基因与基因组百科全书富集分析采用基因条目(Gene Ontology, GO)/京都基因与基因组百科全书(Kyoto encyclopaedia of Genes and Genomes, KEGG)的显著富集分析,应用超几何检验方式,获得差异基因在整个基因背景中的富集通路。
1.3 统计学分析对高通量测序获得的miRNA数据,采用t检验。用Benjamini-Hochberg方法矫正GO和KEGG富集分析获得的P值。P < 0.05为差异具有统计学意义。上述分析采用SPSS软件19.0版本。
2 结果 2.1 临床症状4例AchR-MG患者中有2例分别合并抗核抗体(ANA)1 : 80和抗线粒体抗体(AMA-M2)弱阳性,均无其他临床症状,经风湿免疫科会诊排除了自身免疫性疾病。
2.2 RNA质量2100峰图基线平稳,RNA 28S和18S条带清晰并很少见到降解的5S条带。所有样本均达到RIN≥11.0,28S/18S>1.2。
2.3 两组差异miRNA筛选和靶基因预测如表 2、表 3,共筛选出差异性miRNA共28种,其中上调17种,下调11种。以两组差异较大的miR-4785为例,预测到METTL22、TMEM38A、ZNF324、ITGB4、CDC34等395种靶基因。miR-210-3p也是一种较为重要的miRNA,用同样方法预测到包括CREB3L3、PDZD4在内的537种。
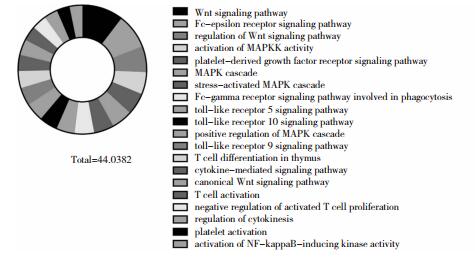
2.4 GO富集分析识别了319条GO(图 3)。按与免疫相关进行筛选,找到21条GO,按照重要性排序如图 4所示,依次为Wnt signal pathway、Fc-epsilon receptor signaling pathway、regulation of Wnt signaling pathway等。
|
| 注:采用ggplot2对GO富集分析结果以散点图展示,Rich factor表示位于该GO的差异基因个数/位于该GO的总基因数。P值代表富集显著程度,P值越小,GO富集程度越高;P值越小,散点颜色接近红色。 图 3 AchR-MG与健康对照组的GO散点图 |
|
| 注:以-log10(P值)为扇面面积,以不同颜色代表不同通路,绘制了在GO富集分析中P值小于0.05的免疫相关通路的饼图。其中面积越大,代表该通路的P值越小,该通路越有意义。 图 4 AchR-MG与免疫相关的重要GO |
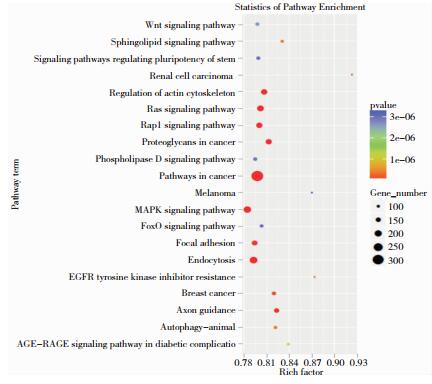
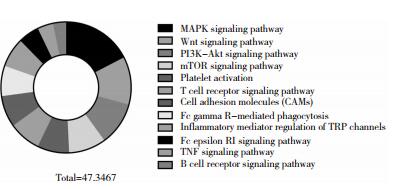
共获得了119个风险通路(图 5)。按照KEGG数据库信息进一步筛选了与免疫相关的通路,按照重要性排序如图 6所示,分别为Platelet activation、T cell receptor signaling pathway等。
|
| 注:采用ggplot2对KEGG富集分析结果以散点图展示,Rich factor表示位于该KEGG的差异基因个数/位于该KEGG的总基因数,P值越小,KEGG富集程度越高,富集显著性越高;P值越小,散点颜色接近红色。 图 5 AchR-MG的KEGG散点图 |
|
| 注:以-log10(P值)为扇面面积,以不同颜色代表不同通路,绘制了在KEGG富集分析中P值小于0.05的免疫相关通路的饼图。其中面积越大,代表该通路的P值越小,该通路越有意义。 图 6 AchR-MG与免疫有关的KEGG |
MG涉及到多个生理机制和途径,例如Toll样受体表达增多[6, 7];I型干扰素增高[8];Treg细胞免疫失衡;异位生发中心的B细胞浸润、浆细胞增多[9]等。研究证实,miRNA调控与帕金森、痴呆等疾病具有一定关联[10-13]。在AchR-MG中,已发现下调表达的有:miR-7-5p[9]、miR-320a[14]、miR-15b、miR-122、miR-140-3p、miR-185、miR-192、miR-20b、miR-885-5p[15]、miR-27a-3p[16]、miR-612[17]。上调表达的有:miR-150-5p、miR-21-5p、miR-3654、miR-3651[16-17]。在骨骼肌特异性受体酪氨酸激酶抗体(muscle-specific tyrosine kinase, MuSK)MG中下调的有miR-210-3p、miR-324-3p[18],上调的有miR-151a-3p、let-7、miR-155[19, 20]等。MG患者胸腺瘤中的miR-125a-3p异常表达[21],MG实验动物中的miR-145低水平表达[22]。
本研究发现,AchR-MG的外周血单个核细胞中miRNA表达差异的共28种,其中上调17种,下调11种,这表明AchR抗体致病过程中上调占优势。其中miR-21(上调)、miR-20b(下调)均与文献报道相符[23, 24]。MiR-21-3p预测到的靶基因众多,其中包括CD80基因(gene ID 941),该基因编码CD80又称为B7-1,一般存在于活化的B细胞、T细胞、树突状细胞、IFN-γ活化的单核细胞表面。CD80可正负两个方向协同调控T细胞活化和细胞因子的产生[25]。此外,CD80还与结肠癌组织的异常甲基化[26]、CD4阳性T细胞行为相关[27]。miR-21-3p是否通过CD80在MG中起到致病作用,有待在今后进行进一步验证。
本研究发现了以Wnt signaling pathway、Fc receptor signaling pathway、T cell signal pathway为代表的重要GO。Wnt信号在细胞增殖等多个面发挥着重要作用[28],如系统性红斑狼疮[29],该信号有调控T细胞增殖和分化的重要作用[30, 31]。Fc受体信号通路与MG有关,重组多价Fc分子有治疗MG的潜力[32],高加索人的FcγRIIA单核苷酸多态性与MG相关[33]。TCR信号通路上的PTPN2基因多态性与早发型重症肌无力患病风险相关[34],一些经抗T淋巴细胞相关抗原4(cytotoxic T lymphocyte-associated antigen-4, CTLA-4)治疗后的个体出现了MG、多发性肌炎等免疫性疾病[35, 36]。Wnt signaling pathway、Fc receptor signaling pathway和T cell signal pathway是否参与到MG中,有待进一步研究。
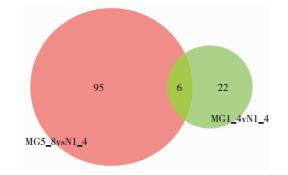
在我们的另一项研究中,用同样的方法观察了MuSK-MG和正常对照组外周血单个核白细胞miRNA的差异,并将AchR-MG的差异miRNA和MuSK-MG得到差异miRNA进行了对比,绘制韦恩图(图 7)。取P < 0.05为标准,在本研究中28个差异表达miRNA中,有6个同时也是MuSK-MG特异性表达的,其余22个差异miRNA是AchR特有的。这6个miRNA为:miR-548ak_R-1、miR-20b-5p、miR-19a-3p、miR-19b-3p、miR-101-3p_R+1、bta-miR-2484。
|
| 注:红色圆形内代表MuSK-MG特异表达的miRNA,共101个,绿色圆形内代表AchR-MG特异表达的miRNA,共28个。两圆交集内有6个miRNA,代表共同差异表达的miRNA有6个。 图 7 AchR-MG和MuSK-MG差异miRNA的韦恩图 |
本研究还发现了28种AchR-MG特异性表达的miRNA,找到了多种重要通路,但上述结果仅为初步探索所见,需要在今后的研究中进一步验证。
本研究也存在一些不足,如对照组仅通过病史排除了自身免疫性疾病和肿瘤,但并未进行自身抗体、全身影像学的全面筛查,不能排除潜在的疾病风险。未同时对比双阴性MG,很难确定这28种miRNA是否为AchR-MG独有的表达谱。此外,筛选试验入组例数较少也带来了一些偏倚。为解决这些缺陷,需要在以后纳入更多的亚组、扩大样本量,并尽量采取多种手段对结果进行重复验证,找到有潜力提示病情进展的miRNA,对真正可能参与发病的靶基因和相关蛋白进行功能验证。
| [1] |
Cron MA, Maillard S, Villegas J, et al. Thymus involvement in early-onset myasthenia gravis[J]. Ann N Y Acad Sci, 2018, 1412(1): 137-145. DOI:10.1111/nyas.13519 |
| [2] |
李海峰. 重症肌无力个体化治疗研究方法学的几个问题[J]. 国际神经病学神经外科学杂志, 2018, 45(3): 215-220. |
| [3] |
Cron MA, Maillard S, Truffault F, et al. Causes and Consequences of miR-150-5p Dysregulation in Myasthenia Gravis[J]. Front Immunol, 2019, 10(3): 1-14. |
| [4] |
Cao YL, Wang J, Zhang H, et al. MicroRNA-653 Inhibits Thymocyte Proliferation and Induces Thymocyte Apoptosis in Mice with Autoimmune Myasthenia Gravis by Downregulating TRIM9[J]. Neuroimmunomodulation, 2019, 26(1): 7-18. DOI:10.1159/000494802 |
| [5] |
Bo C, Wang J, Zhang H, et al. Global pathway view analysis of microRNA clusters in myasthenia gravis[J]. Mol Med Rep, 2019, 19(3): 2350-2360. |
| [6] |
Cavalcante P, Barzago C, Baggi F, et al. Toll-like receptors 7 and 9 in myasthenia gravis thymus:amplifiers of autoimmunity?[J]. Ann N Y Acad Sci, 2018, 1413(1): 11-24. DOI:10.1111/nyas.13534 |
| [7] |
Cordiglieri C, Marolda R, Franzi S, et al. Innate immunity in myasthenia gravis thymus:pathogenic effects of Toll-like receptor 4 signaling on autoimmunity[J]. J Autoimmun, 2014, 52(3): 74-89. |
| [8] |
Cufi P, Dragin N, Ruhlmann N, et al. Central role of interferon-beta in thymic events leading to myasthenia gravis[J]. J Autoimmun, 2014, 52(10): 44-52. |
| [9] |
Barzago C, Nogales-Gadea G, de Baets M, et al. A novel infection-and inflammation-associated molecular signature in peripheral blood of myasthenia gravis patients[J]. Immunobiology, 2016, 221(11): 1227-1236. DOI:10.1016/j.imbio.2016.06.012 |
| [10] |
van Kralingen JC, McFall A, Ord ENJ, et al. Altered Extracellular Vesicle MicroRNA Expression in Ischemic Stroke and Small Vessel Disease[J]. Transl Stroke Res, 2019, 10(5): 495-508. DOI:10.1007/s12975-018-0682-3 |
| [11] |
Funamizu N, Lacy CR, Kamada M. MicroRNA-200b and -301 are associated with gemcitabine response as biomarkers in pancreatic carcinoma cells[J]. Int J Oncol, 2019, 54(3): 991-1000. |
| [12] |
Starhof C, Hejl AM, Heegaard NHH, et al. The biomarker potential of cell-free microRNA from cerebrospinal fluid in Parkinsonian Syndromes[J]. Mov Disord, 2019, 34(2): 246-254. DOI:10.1002/mds.27542 |
| [13] |
Maldonado-Lasuncion I, Atienza M, Sanchez-Espinosa MP, et al. Aging-related changes in cognition and cortical integrity are associated with serum expression of candidate microRNAs for Alzheimer Disease[J]. Cereb Cortex, 2018, 1(5): 1-12. |
| [14] |
Cheng Z, Qiu S, Jiang L, et al. MiR-320a is downregulated in patients with myasthenia gravis and modulates inflammatory cytokines production by targeting mitogen-activated protein kinase 1[J]. J Clin Immunol, 2013, 33(3): 567-576. DOI:10.1007/s10875-012-9834-5 |
| [15] |
Nogales-Gadea G, Ramos-Fransi A, Suarez-Calvet X, et al. Analysis of serum miRNA profiles of myasthenia gravis patients[J]. PLoS One, 2014, 9(3): e91927. DOI:10.1371/journal.pone.0091927 |
| [16] |
Punga T, Le Panse R, Andersson M, et al. Circulating miRNAs in myasthenia gravis:miR-150-5p as a new potential biomarker[J]. Ann Clin Transl Neurol, 2014, 1(1): 49-58. |
| [17] |
Punga AR, Andersson M, Alimohammadi M, et al. Disease specific signature of circulating miR-150-5p and miR-21-5p in myasthenia gravis patients[J]. J Neurol Sci, 2015, 356(1): 90-96. |
| [18] |
Punga AR, Punga T. Circulating microRNAs as potential biomarkers in myasthenia gravis patients[J]. Ann N Y Acad Sci, 2018, 1412(1): 33-40. DOI:10.1111/nyas.13510 |
| [19] |
Punga T, Bartoccioni E, Lewandowska M, et al. Disease specific enrichment of circulating let-7 family microRNA in MuSK+myasthenia gravis[J]. J Neuroimmunol, 2016, 292(3): 21-26. |
| [20] |
Sabre L, Guptill JT, Russo M, et al. Circulating microRNA plasma profile in MuSK+ myasthenia gravis[J]. J Neuroimmunol, 2018, 325(12): 87-91. |
| [21] |
Li J, Qiu D, Chen Z, et al. Altered expression of miR-125a-5p in thymoma-associated myasthenia gravis and its down-regulation of foxp3 expression in Jurkat cells[J]. Immunol Lett, 2016, 172(1): 47-55. |
| [22] |
Wang J, Zheng S, Xin N, et al. Identification of novel MicroRNA signatures linked to experimental autoimmune myasthenia gravis pathogenesis:down-regulated miR-145 promotes pathogenetic Th17 cell response[J]. J Neuroimmune Pharmacol, 2013, 8(5): 1287-1302. DOI:10.1007/s11481-013-9498-9 |
| [23] |
Cron MA, Maillard S, Delisle F, et al. Analysis of microRNA expression in the thymus of Myasthenia Gravis patients opens new research avenues[J]. Autoimmun Rev, 2018, 17(6): 588-600. DOI:10.1016/j.autrev.2018.01.008 |
| [24] |
Chunjie N, Huijuan N, Zhao Y, et al. Disease-specific signature of serum miR-20b and its targets IL-8 and IL-25, in myasthenia gravis patients[J]. Eur Cytokine Netw, 2015, 26(3): 61-66. DOI:10.1684/ecn.2015.0367 |
| [25] |
Bengsch F, Knoblock DM, Liu A, et al. CTLA-4/CD80 pathway regulates T cell infiltration into pancreatic cancer[J]. Cancer Immunol Immunother, 2017, 66(12): 1609-1617. DOI:10.1007/s00262-017-2053-4 |
| [26] |
Scarpa M, Castagliuolo I, Erroi F, et al. CD80 down-regulation is associated to aberrant DNA methylation in non-inflammatory colon carcinogenesis[J]. BMC Cancer, 2016, 16(1): 388. DOI:10.1186/s12885-016-2405-z |
| [27] |
Ville S, Poirier N, Blancho G, et al. Co-stimulatory blockade of the CD28/CD80-86/CTLA-4 balance in transplantation:impact on memory T cells?[J]. Front Immunol, 2015, 6(411): 1-10. |
| [28] |
Nusse R. Cell signalling:Disarming Wnt[J]. Nature, 2015, 519(7542): 163-164. DOI:10.1038/nature14208 |
| [29] |
Koutsokeras T, Healy T. Systemic lupus erythematosus and lupus nephritis[J]. Nat Rev Drug Discov, 2014, 13(3): 173-174. DOI:10.1038/nrd4227 |
| [30] |
Deng YJ, Huang ZX, Zhou CJ, et al. Gene profiling involved in immature CD4+ T lymphocyte responsible for systemic lupus erythematosus[J]. Mol Immunol, 2006, 43(9): 1497-1507. DOI:10.1016/j.molimm.2005.07.039 |
| [31] |
Ma J, Wang R., Fang X, et al. Beta-catenin/TCF-1 pathway in T cell development and differentiation[J]. J Neuroimmune Pharmacol, 2012, 7(4): 750-762. DOI:10.1007/s11481-012-9367-y |
| [32] |
Thiruppathi M, Sheng JR, Li L, et al. Recombinant IgG2a Fc (M045) multimers effectively suppress experimental autoimmune myasthenia gravis[J]. J Autoimmun, 2014, 52(1): 64-73. |
| [33] |
van der Pol WL, Jansen MD, Kuks JB, et al. Association of the Fc gamma receptor IIA-R/R131 genotype with myasthenia gravis in Dutch patients[J]. J Neuroimmunol, 2003, 144(1): 143-147. |
| [34] |
Xiong X, Xiang M, Cheng X, et al. PTPN22 R620W Polymorphism is Associated with Myasthenia Gravis Risk:A Systematic Review and Meta-Analysis[J]. Med Sci Monit, 2015, 21(4): 2567-2571. |
| [35] |
Kang KH, Grubb W, Sawlani K, et al. Immune checkpoint-mediated myositis and myasthenia gravis:A case report and review of evaluation and management[J]. Am J Otolaryngol, 2018, 39(5): 642-645. DOI:10.1016/j.amjoto.2018.06.003 |
| [36] |
Makarious D, Horwood K, Coward J. Myasthenia gravis:An emerging toxicity of immune checkpoint inhibitors[J]. Eur J Cancer, 2017, 82(1): 128-136. |
 2019, Vol. 46
2019, Vol. 46