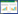扩展功能
文章信息
- 张金灿, 廖勇仕, 周敏, 刘小飞, 廖凌骁
- ZHANG Jin-Can, LIAO Yong-Shi, ZHOU Min, LIU Xiao-Fei, LIAO Ling-Xiao
- miR-23c靶向调控MTDH抑制胶质瘤U87细胞增殖迁移与侵袭
- miR-23c inhibits the proliferation, migration, and invasion of glioma U87 cells via targeted regulation of MTDH
- 国际神经病学神经外科学杂志, 2019, 46(4): 395-400
- Journal of International Neurology and Neurosurgery, 2019, 46(4): 395-400
-
文章历史
收稿日期: 2019-06-08
修回日期: 2019-08-13
MicroRNAs (miRs)是一组内源的约为22个(20~24)核苷酸的小非编码RNA,近年来研究发现,miRs对细胞内各种生理学过程均发挥重要的调节作用[1]。miRs对靶基因的调节常常是一种网络调节方式即一个miRNA可调控一个或多个靶基因,同时一个靶基因也可能受一个或多个miRNA调控。miRs可能调控人类约1/3的基因表达,且miRs在不同物种中其序列和位置都存在高度的保守性。miRs发挥重要功能与其位置和序列的高度保守性密切相关。近年来,在很多人类的癌症中发现有特异性的miRs表达失调[2]。这些miRs通过负性调控原癌基因或抑癌基因等表达参与癌症的进展和侵袭[3-5]。在糖尿病肾病时miR-23c能够通过调控ELAV样RNA结合蛋白1抑制肾小管上皮细胞的坏死[6]。miR-23c最早发现结直肠癌组织中miR-23c高表达,Steviol抑制胃肠道肿瘤细胞的增殖与其调节miR-23c有关[7]。miR-23c可通过调节ERBB2IP抑制肝细胞癌的细胞增殖并促进细胞凋亡[8]。有研究发现异粘蛋白(metadherin,MTDH)在许多肿瘤中高表达,如食管鳞状细胞癌、胃癌等,并且参与肿瘤的发生、发展[9, 10]。但是miR-23c与MTDH在胶质瘤中的作用研究尚少。本研究旨在探讨miR-23c对胶质瘤U87细胞增殖、迁移和侵袭的影响及其可能的初步机制。
1 材料与方法 1.1 材料主要材料Lip2000、miR-23c mimics、miR-无关序列、MTDH质粒、MTT粉以及Transwell小室均购自美国Sigma公司,RNA抽提试剂盒购于Applied Biosystems公司,DMEM培养基和小牛血清购自Gibco公司子,MTDH 3’UTR的各种报告基因(Wild-MTDH-3’UTR vector,Mut-MTDH-3’UTR vector)由复能基因公司构建,双报告基因载体从复能基因购买。双荧光素酶活性检测试剂盒购自Promega公司。MTDH抗体和β-actin抗体购自CST公司。
1.2 方法 1.2.1 qRT-PCR检测MTDH表达水平qRT-PCR将miR-23c mimics或miR-无关序列转染于U87细胞,分为miR-23c组与Control组。48h后提取用RNA提取试剂盒提取细胞总RNA,逆转录合成cDNA。PCR扩增反应为20μl体系,包括:Taq DNA polymerase 5U/μl)0.2μl,2×SYBR Mix 10μl,miRNA RT product 2.0μl,MiR-PCR primers(5μM)0.4μl,灭菌蒸馏水7.4μl。循环体系为: 95℃ 3 min, 95℃ 12s. 62℃ 35s, 72℃ 30s, 35个循环。以GAPDH为内参,所测定的miR-23c的相对表达量采用2-ΔΔCT法分析。
1.2.2 Western blot检测MTDH蛋白表达Western blot细胞转染与分组同qRT-PCR。48h后提取细胞总蛋白,BCA法测定蛋白浓度。各组取等量样本,进行SDS-PAGE凝胶电泳后将蛋白转移至PVDF膜上,5%脱脂牛奶封闭,加入MTDH抗体或β-actin抗体,4℃过夜。TBST洗膜30 min,加入二抗室温孵育1h,TBST洗膜30min,然后加ECL发光剂,X片曝光、显影、定影。
1.2.3 荧光素酶活性检测将荧光素酶报告载体与miR-23c mimics或miR-无关序列共转染于U87细胞。转染48h后,收集细胞。按照Promega公司双荧光素酶活性检测试剂盒说明书操作,在单光子检测仪检测细胞荧光素酶的活性。计算相对荧光素酶活性=萤火虫荧光素酶活性值/海肾荧光素酶活性值。
1.2.4 MTT法检测细胞增殖活性将miR-23c mimics或miR-无关序列以及在转染miR-23c的同时也转染MTDH质粒时,即分为Control组,miR-23c组和miR-23c+MTDH组。各组U87细胞培养24 h后,用胰酶(0.25%)常规消化,尽量制成单细胞悬液,离心收集。用含胎牛血清(10%)的DMEM培基(含50U/ml青霉素,50mg/ml链霉素)稀释细胞悬液,取(1-2)×104个细胞接种于孔板(96孔)中,并常规每组设置复孔6个。置于5%的CO2孵箱中于37℃培养24h或48h。每孔加入20μl灭菌的5mg/ml的MTT液。选择波长,在酶标仪上570nm处检测96孔板中各孔的吸光值,记录统计结果。
1.2.5 细胞划痕实验细胞分组同MTT法,将各组细胞采用胰酶常规消化后制成浓度为4~5×106的单细胞悬液。当细胞生长的融合率达到70%~80%时用10μl的Tip头做一划痕,然后用PBS冲洗3~4次以洗掉已经掉落的细胞,继续培养24 h或48 h后进行拍照,测量多个不同点的划痕的宽度,计算出划痕愈合率的平均值。统计分析各组平均划痕愈合率的差异。
1.2.6 Transwell侵袭实验细胞分组同MTT法,先将小室铺上一层基质胶,准备好细胞悬液(2×105/ml),然后将300μL细胞悬液加入到上室,并加入300μL含10%胎牛血清的DMEM培基到下室。在37℃条件下培养24 h或48 h后,用PBS冲洗下室2~3遍,然后将下室用90%酒精固定20 min,弃掉固定液,PBS冲洗2~3次后,用苏木素对酯膜进行染色,封片观察。将transwell下室底部的膜切下,倒置显微镜下随机观察并拍照计数膜底部的细胞个数(每个视野)(10个视野),以穿膜细胞数目(10个视野以上的平均值)评价肿瘤细胞的侵袭能力。
1.3 统计学分析统计学处理用SPSS 19.0软件统计处理所有数据,计量资料采取均数±标准差(x±s)的形式表示,两组间比较采用t检验,当P < 0.05时,为差异有统计学意义。
2 结果 2.1 miR-23c对胶质瘤U87细胞增殖的影响MTT结果显示,在48h时miR-23c组U87细胞的OD值为(0.668±0.032),明显低于Control组的(1.031±0.060),差异有统计学意义(P < 0.01),见图 1。提示外源高表达miR-23c能抑制胶质瘤U87细胞的增殖。
|
| 注:与Control组相比,** P < 0.01 图 1 MTT法检测miR-23c对U87细胞增殖能力的影响 |
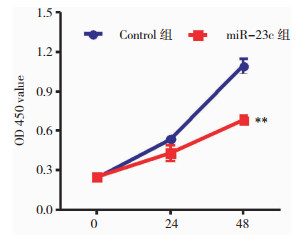
细胞划痕48 h后结果显示,miR-23c组细胞划痕愈合率(0.35±0.02)明显低于对照组的(0.59±0.03),差异有统计学意义(P < 0.01),见图 2。提示外源高表达miR-23c能抑制胶质瘤U87细胞的迁移能力。
|
| 注:与Control组相比,** P < 0.01。 图 2 划痕实验检测miR-23c对U87细胞迁移能力的影响 |
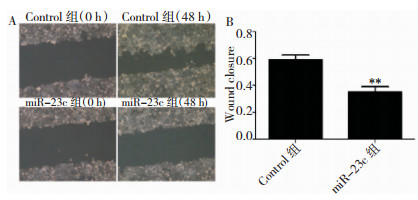
Transwell结果显示,miR-23c组U87细胞穿过基质胶的细胞数量为(153.2±8.30),与对照组(348.4±12.12)相比,差异有统计学意义(P < 0.01),见图 3。提示外源高表达miR-23c能抑制胶质瘤U87细胞的侵袭能力。
|
| 注:与Control组相比,**P < 0.01。 图 3 Transwell侵袭实验检测miR-23c对U87细胞侵袭能力的影响 |
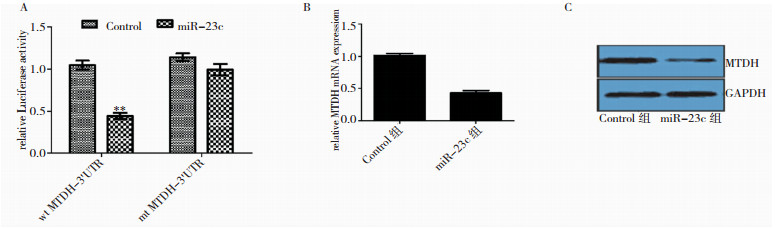
生物信息学结果显示miR-23c与MTDH有共同的结合位点。萤光素酶报告基因结果显示,miR-23c mimics对突变型Mut -miR-23c /MTDH质粒组荧光素酶活性强度无明显影响,但对野生型Wt-miR-23c /MTDH报告质粒组荧光素酶活性强度下降了约57%。差异有统计学意义(P < 0.01)(图 4A)。qRT-PCR实验结果显示,miR-23c组MTDH mRNA的相对表达量为(0.435±0.034)明显低Control组(1.013±0.032),差异有统计学意义(P < 0.01)(图 4B)。蛋白质印迹试验结果显示,miR-23c组MTDH蛋白表达水平较Control组明显降低(图 4C)。结果提示,MTDH是miR-23c直接调控的靶基因。
|
| A:荧光素酶报告载系统检测miR-23c对抑制MTDH 3’UTR的荧光酶活性的影响,与Control组相比,** P < 0.01;B:qRT-PCR检测miR-23c对MTDH mRNA的影响,与Control组相比,** P < 0.01;C:Western blot检测miR-23c对MTDH蛋白的影响 图 4 MTDH是miR-23c直接调控的靶基因 |
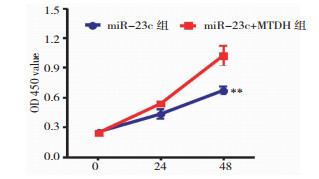
前面的实验结果已经证实miR-23c能抑制MTDH表达,但是miR-23c抑制MTDH表达是否对U87细胞的生物学行为起作用尚不清楚。为了验证这一点,接下来的实验在转染miR-23c的同时也转染MTDH质粒即分为两个组:miR-23c组和miR-23c+MTDH组。MTT结果显示,miR-23c+MTDH组U87细胞的OD值为(1.025±0.059),明显高于miR-23c组的(0.672±0.024),差异有统计学意义(P < 0.01),见图 5。
|
| 注:与miR-23c组相比,** P < 0.01 图 5 MTT法检测miR-23c+MTDH对U87细胞增殖能力的影响 |
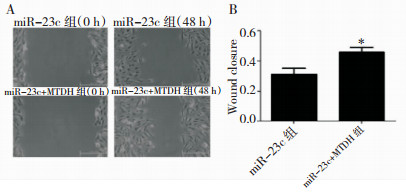
细胞划痕结果显示,细胞划痕48h后miR-23c+MTDH组细胞划痕愈合率为(0.45±0.04),明显高于miR-23c组细胞划痕愈合率(0.31±0.03),差异有统计学意义(P < 0.05)。见图 6。
|
| 注:与miR-23c组相比,*P < 0.05。 图 6 划痕实验检测miR-23c+MTDH对U87细胞迁移能力的影响 |
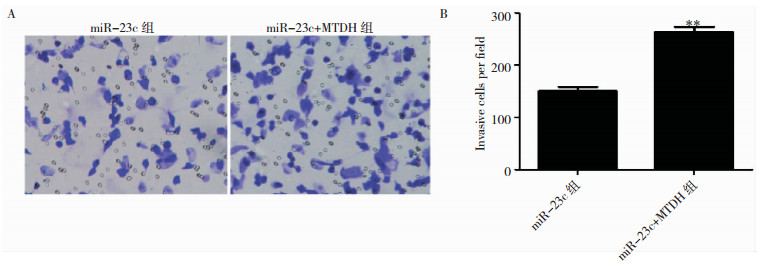
Transwell结果显示,miR-23c +MTDH组U87细胞穿过基质胶的细胞数量为(260.9±10.23),明显高于miR-23c组(148.4±9.4),差异有统计学意义(P < 0.01),见图 7。
|
| 与miR-23c组相比,**P < 0.01。 图 7 Transwell侵袭实验检测miR-23c+MTDH对U87细胞侵袭能力的影响 |
胶质瘤是严重危害人类生命健康最常见的中枢神经系统肿瘤之一。胶质瘤的发病率高约占颅内肿瘤发病率的30%,常与周围组织分界不清,呈侵润型生长方式,手术难以全切效果不佳,且术后在脊髓或颅内其他部位容易发生复发转移,且胶质瘤的病程进展迅速,一年生存率低于50%[11]。因此,胶质瘤的分子发病机制及其防治方法一直都是神经外科学领域的热点和难点。目前基因诊断和治疗已经越来越受到关注,寻找肿瘤发病的特异性靶点成为了目前研究的热点,特异性激活或抑制其表达,可能会达到促进肿瘤细胞凋亡抑制肿瘤增殖迁移以及侵袭的目的。
近年来随着对肿瘤分子机制研究的逐渐深入,学者们认识到肿瘤的形成和进展与细胞周期调控失衡密切相关。miRs参与了细胞增殖分化以及凋亡的调控,其功能与肿瘤形成和进展密切相关。已有研究证实miR-23c在人类癌症中起重要作用[12-14]。但是miR-23c在胶质瘤中的作用研究尚少。本次研究通过MTT实验和细胞划痕以及Transwells实验分别检测了细胞的增殖与迁移以及侵袭能力,发现过表达miR-23c可抑制胶质瘤细胞U87的增殖、迁移以及侵袭。本实验结果与Zhang等人在肝细胞癌中的实验结果相吻合[8]。但miR-23c抑制胶质瘤细胞U87增殖、迁移以及侵袭的机制尚不明确。随后进一步采用TargetScanVert寻找miR-23c可能的作用靶基因,结果显示MTDH可能是miR-23c调控的下游靶基因。
MTDH第一次被发现在2002年暴露于HIV-1的原发性人类胎儿星形胶质细胞中,2005年MTDH的cDNA被成功克隆[15]。一些研究人员已经证实发现MTDH在许多癌症组织和细胞系中存在高表达,MTDH参与调节与肿瘤形成与进展有关的信号通路,调节肿瘤相关基因的表达。MTDH还能调节肿瘤的血管生成、侵袭性、转移和化疗耐药性。对MTDH的信号通路、药物或分子靶标的研究表明MTDH在恶性肿瘤中具有潜在的治疗作用。据报道,MTDH的异位表达增强了许多类型癌细胞的增殖,包括乳腺癌细胞,前列腺癌细胞,神经母细胞瘤细胞,食管鳞状细胞癌细胞,HCC细胞,神经胶质瘤细胞和胃癌细胞[9, 10, 16, 17]。相反,MTDH的削弱降低了它们的增殖能力。我们的实验结果证实miR-23c可直接抑制MTDH的表达,且miR-23c抑制胶质瘤细胞U87增殖、细胞迁移以及细胞侵袭的机制部分与其下调MTDH表达有关。表明miR-23c可能可作为一个潜在的治疗靶点用于胶质瘤的治疗。但是本实验对象仅为U87细胞系,miR-23c在其它细胞系与动物学水平中的作用尚不清楚。因此,进一步在动物学水平探讨miR-23c及其调控MTDH表达在神经胶质瘤中的作用及机制可为胶质瘤的防治提供重要的实验依据。
| [1] |
Yang F, Ning Z, Ma L, et al. Exosomal mirnas and mirna dysregulation in cancer-associated fibroblasts[J]. Mol Cancer, 2017, 16(1): 148. DOI:10.1186/s12943-017-0718-4 |
| [2] |
Acunzo M, Romano G, Wernicke D, et al. Microrna and cancer-a brief overview[J]. Adv Biol Regul, 2015, 57: 1-9. DOI:10.1016/j.jbior.2014.09.013 |
| [3] |
袁洁, 费智敏. miRNA-21-治疗神经胶质瘤的新靶标?[J]. 国际神经病学神经外科学杂志, 2018, 45(2): 211-214. |
| [4] |
Xie Y, Murray-Stewart T, Wang Y, et al. Self-immolative nanoparticles for simultaneous delivery of microrna and targeting of polyamine metabolism in combination cancer therapy[J]. J Control Release, 2017, 246: 110-119. DOI:10.1016/j.jconrel.2016.12.017 |
| [5] |
Momtazi AA, Shahabipour F, Khatibi S, et al. Curcumin as a microrna regulator in cancer:A review[J]. Rev Physiol, Biochemi Pharmacol, 2016, 171: 1-38. |
| [6] |
Li X, Zeng L, Cao C, et al. Long noncoding rna malat1 regulates renal tubular epithelial pyroptosis by modulated mir-23c targeting of elavl1 in diabetic nephropathy[J]. Exp Cell Res, 2017, 350(2): 327-335. DOI:10.1016/j.yexcr.2016.12.006 |
| [7] |
Yamazaki N, Koga Y, Taniguchi H, et al. High expression of mir-181c as a predictive marker of recurrence in stage ii colorectal cancer[J]. Oncotarget, 2017, 8: 6970-6983. |
| [8] |
Zhang L, Wang Y, Wang L, et al. Mir-23c suppresses tumor growth of human hepatocellular carcinoma by attenuating erbb2ip[J]. Biomed Pharmacother, 2018, 107: 424-432. DOI:10.1016/j.biopha.2018.07.155 |
| [9] |
Yu C, Chen K, Zheng H, et al. Overexpression of astrocyte elevated gene-1(AEG-1) is associated with esophageal squamous cell carcinoma (ESCC) progression and pathogenesis[J]. Carcinogenesis, 2009, 30(5): 894-901. DOI:10.1093/carcin/bgp064 |
| [10] |
Jian-bo X, Hui W, Yu-long H, et al. Astrocyte-elevated gene-1 overexpression is associated with poor prognosis in gastric cancer[J]. Med Oncol, 2011, 28(2): 455-462. DOI:10.1007/s12032-010-9475-6 |
| [11] |
刘志强, 方薪淇, 宋涛, 等. 92例高级别胶质瘤患者术后生存分析[J]. 国际神经病学神经外科学杂志, 2018, 45(6): 577-581. |
| [12] |
Lee SG, Su ZZ, Emdad L, et al. Astrocyte elevated gene-1 activates cell survival pathways through pi3k-akt signaling[J]. Oncogene, 2008, 27(8): 1114-1121. DOI:10.1038/sj.onc.1210713 |
| [13] |
Ash SC, Yang DQ, Britt DE. Lyric/aeg-1 overexpression modulates bccipalpha protein levels in prostate tumor cells[J]. Biochem Biophysi Res Communi, 2008, 371(2): 333-338. DOI:10.1016/j.bbrc.2008.04.084 |
| [14] |
Chen J, Xia Y, Sui X, et al. Steviol, a natural product inhibits proliferation of the gastrointestinal cancer cells intensively[J]. Oncotarget, 2018, 9(41): 26299-26308. |
| [15] |
Thirkettle HJ, Girling J, Warren AY, et al. Lyric/AGE-1 is targeted to different subcellular compartments by ubiquitinylation and intrinsic nuclear localization signals[J]. Cancer Res, 2009, 15(9): 3003-3013. |
| [16] |
Xia Z, Zhang N, Jin H, et al. Clinical significance of astrocyte elevated gene-1 expression in human oligodendrogliomas[J]. Clin Neurol Neurosurgery, 2010, 112(5): 413-419. DOI:10.1016/j.clineuro.2010.02.007 |
| [17] |
Song H, Li C, Li R, et al. Prognostic significance of aeg-1 expression in colorectal carcinoma[J]. Int J colorectal Dis, 2010, 25(10): 1201-1209. DOI:10.1007/s00384-010-1009-3 |
 2019, Vol. 46
2019, Vol. 46