扩展功能
文章信息
- 邹涵, 王苟思义, 叶宁荣, 李闫文, 黄琦, 刘宏伟, 熊祖剑, 李学军
- ZOU Han, WANGGOU Si-Yi, YE Ning-Rong, LI Yan-Wen, HUANG Qi, LIU Hong-Wei, XIONG Zu-Jian, LI Xue-Jun
- 胶质瘤患者的生存风险预测模型
- Survival risk prediction model for patients with glioma
- 国际神经病学神经外科学杂志, 2019, 46(1): 1-6
- Journal of International Neurology and Neurosurgery, 2019, 46(1): 1-6
-
文章历史
收稿日期: 2018-12-10
修回日期: 2019-02-01
2. 中南大学湘雅医院神经外科, 湖南 长沙 410008
2. Department of Neurosurgery, Xiangya Hospital, Central South University, Changsha 410008, China
胶质瘤是中枢神经系统中最常见的恶性肿瘤,不同患者其生存时间差异很大。通常,胶质瘤患者的5年生存率不超过35%[1]。虽然治疗方法多种多样,但没有哪一种能够完全治愈胶质瘤,所以找到胶质瘤发生、发展以及导致低生存率的致病因素对于认识和治疗胶质瘤都有重要的意义[2-6]。现在认为,其低生存率是由于异常的基因表达量引起的,而且基因表达量也常被用于对各种肿瘤的预后建模[7]。Lasso回归适用于自变量众多,但样本量有限的数据分析和模型构建[8]。所以,我们对TCGA数据库中胶质瘤病例的基因表达量做单因素生存分析,使用筛选得到的上万个预后相关的基因做Lasso回归建模,以求能够根据少数基因的表达量精确地预测出患者的生存时间。
1 材料与方法 1.1 数据收集653个患者的基因表达量和临床信息均从TCGA(https://cancergenome.nih.gov/)获取,获取和使用此数据时遵循TCGA的相关政策。对获取的基因表达量,依据GENCODE(https://www.gencodegenes.org)第28版注释探针,得到相应的基因名。
1.2 鉴定预后相关的基因通过使用R/Bioconductor的“survival”包和“survminer”包,对60483个基因表达量一对一地做单因素生存分析,选取其中P值小于0.05的基因表达量用于进一步构建模型。
1.3 Lasso回归因为基因之间的相互作用会形成共线性基因群,所以使用R/Bioconductor中的“lars”包和“glmnet”包做Lasso回归,以减少共线性的影响,从而提高模型的准确性和可解释程度。在众多模型中,再使用交叉验证以确定相应的参数,得到合适的模型。利用所得到的模型,我们计算了每一个患者的风险分数,从而对总生存期风险定量。风险分数=
之后,为了探索模型的预测效力,我们绘制了受试者工作特征曲线,并计算了曲线下面积;依据风险分数把患者分为高风险组和低风险组,分组界值由R/Bioconductor的“survival”包和“survminer”包计算得出,对所得两组分别绘制Kaplan-Meier曲线;再使用R/Bioconductor的“gplots”包绘制每一个患者在模型中所涉及到的基因表达量的热图。
同时,为了验证模型的有效性和可重复性,使用R随机将患者以6 : 4的比例分为训练组和验证组。在训练组中,依据风险分数把患者分为高风险组和低风险组,分组界值由R/Bioconductor的“survival”包和“survminer”包计算得出,对所得两组分别绘制Kaplan-Meier曲线。在验证组中,依据训练组所得到的分组界值将患者分为高风险组和低风险组,同样对所得两组绘制Kaplan-Meier曲线。
1.5 Cox回归为了检测模型是否受临床特征的影响,并比较两者对预后的影响,我们使用R/Bioconductor的“survival”包先对风险分数和各个临床特征(年龄、性别、组织学分类和胶质瘤级别)分别做单因素Cox回归,再对P小于0.05的变量做多因素Cox回归。
所有的统计学分析均使用了3.5.1版本的R软件和相应的软件包。图片由R软件绘制,部分图片经过Adobe Illustrator CS6的后期处理。
2 结果 2.1 患者的临床特征患者的临床特征统计结果见表 1。从表中可见,胶质瘤患者主要集中在31~60岁的中年人群,没有10岁以下儿童患者,提示临床上遇到10岁以下颅内肿瘤的儿童不应首先考虑拟诊为胶质瘤;也没有91岁以上老年人患者,提示91岁以上的老年人患胶质瘤的概率较小。四种组织学类型数量差不多,提示组织学类型不会造成较大的数据偏倚。病理级别主要集中在G2级和G3级,G4级较少。男女比约3 : 2,符合认知。发病后患者的生存时间差别很大,提示不同患者的预后差别很大,同时也反映了本模型的临床意义,即相对准确地预测每一位患者的生存状况。
| 特点 | 结果 | |
| 年龄(年) | 1~10 | 0 |
| 11~20 | 9 | |
| 21~30 | 81 | |
| 31~40 | 142 | |
| 41~50 | 110 | |
| 51~60 | 122 | |
| 61~70 | 88 | |
| 71~80 | 38 | |
| 81~90 | 8 | |
| 91~100 | 0 | |
| 组织学类型 | 少突胶质细胞瘤 | 172 |
| 少突-星形细胞瘤 | 112 | |
| 星形细胞瘤 | 166 | |
| 胶质母细胞瘤 | 148 | |
| 级别 | G2 | 213 |
| G3 | 237 | |
| G4 | 148 | |
| 性别 | 男 | 350 |
| 女 | 248 | |
| 生存状况 | 存活 | 419 |
| 死亡 | 234 | |
| 平均生存时间(天) | 833.3±887.8 |
首先,使用Cox比例风险回归模型对每一个基因的表达量做单因素生存分析,选择P值小于0.05的基因,共有31641个,并对各种基因类型进行统计,结果见图 1。可见,传统认为对生命活动具有重要影响的mRNA占比确实较大,达到44%,提示其本身对胶质瘤患者的预后具有重要作用。同时,最近被逐渐重视的非编码RNA也占有相当大的比例,说明非编码RNA的作用,如对mRNA表达的调节等,也对预后有很重要的作用。
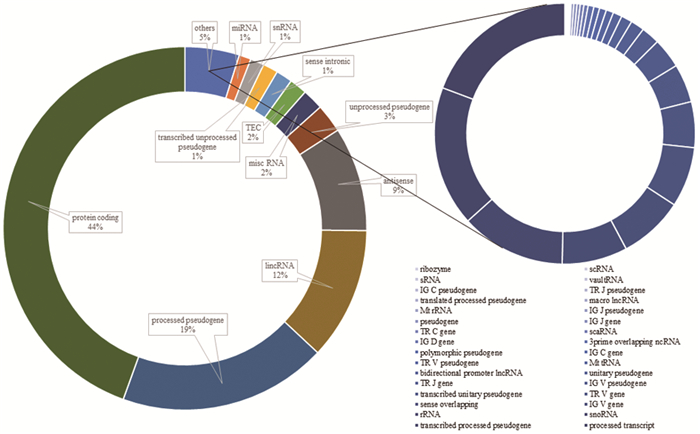
|
| 图 1 总生存率相关的基因类型 |
利用Lasso回归和筛选出的与预后相关的31641个基因表达量建立预后模型(图 2A),并用交叉验证确立最合适的模型(图 2B),最终得出的模型含有40个基因表达量,模型所包含的变量、相应的系数和生物学类型见表 2。从表中可见,占比最多的基因类型是mRNA(protein coding),除了mRNA,其次为假基因RNA,这也说明了二者对患者预后具有重要的影响。
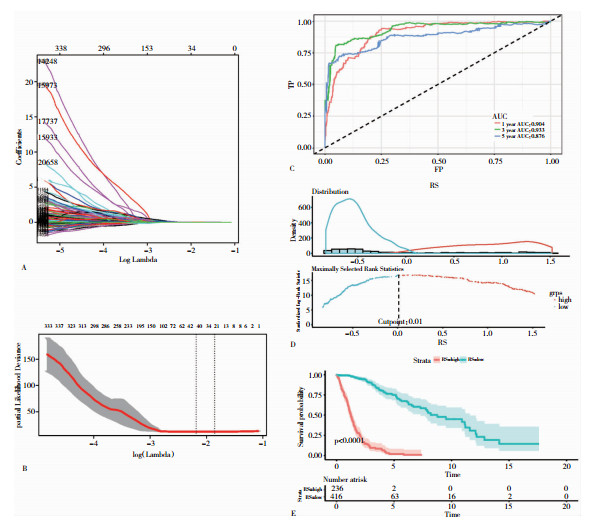
|
| 图 2 进一步的基因筛选和模型构建 A.各系数随着系数总和的收缩而收缩;B.交叉验证以确定模型;C.模型的受试者工作特征曲线和曲线下面积;D.高低风险分数的分界值;E.两组Kaplan-Meier曲线图。 |
| 基因 | 系数 | 生物学类型 |
| RP11-182J1.18 | -2.11E-03 | 反义基因antisense |
| AC006445.6 | -1.49E-03 | 已加工假基因processed pseudogene |
| FRA10AC1 | -1.99E-04 | 蛋白质编码基因protein coding |
| C10orf85 | -5.03E-05 | lincRNA |
| NTNG2 | -3.61E-05 | 蛋白质编码基因protein coding |
| C1orf233 | -2.42E-05 | 蛋白质编码基因protein coding |
| SLITRK5 | -2.08E-05 | 蛋白质编码基因protein coding |
| DESI1 | -1.51E-05 | 蛋白质编码基因protein coding |
| GNL1 | -5.55E-06 | 蛋白质编码基因protein coding |
| CUEDC2 | -4.87E-06 | 蛋白质编码基因protein coding |
| WEE1 | 4.59E-08 | 蛋白质编码基因protein coding |
| EMP3 | 1.87E-07 | 蛋白质编码基因protein coding |
| TGIF1 | 5.35E-06 | 蛋白质编码基因protein coding |
| TNFRSF12A | 8.45E-06 | 蛋白质编码基因protein coding |
| GLMP | 1.03E-05 | 蛋白质编码基因protein coding |
| HOXD11 | 4.21E-05 | 蛋白质编码基因protein coding |
| EN1 | 5.87E-05 | 蛋白质编码基因protein coding |
| EFEMP2 | 6.97E-05 | 蛋白质编码基因protein coding |
| CPQ | 7.01E-05 | 蛋白质编码基因protein coding |
| LSP1 | 8.15E-05 | 蛋白质编码基因protein coding |
| CRNDE | 1.32E-04 | lincRNA |
| GAS2L3 | 1.45E-04 | 蛋白质编码基因protein coding |
| RP11-396A22.4 | 1.51E-04 | 已加工假基因processed pseudogene |
| POLR2J4 | 2.38E-04 | 转录未加工假基因transcribed unprocessed pseudogene |
| RAB42 | 2.39E-04 | 蛋白质编码基因protein coding |
| RP11-473M20.16 | 2.95E-04 | lincRNA |
| RP5-865M20.1 | 4.67E-04 | 已加工假基因processed pseudogene |
| HOXD12 | 7.33E-04 | 蛋白质编码基因protein coding |
| AC009302.2 | 9.62E-04 | 已加工假基因processed pseudogene |
| RP11-435F17.3 | 2.45E-03 | 已加工假基因processed pseudogene |
| CLEC18C | 3.21E-03 | 蛋白质编码基因protein coding |
| RP11-386G11.10 | 3.26E-03 | 反义基因antisense |
| FTHL1 | 3.81E-03 | 已加工假基因processed pseudogene |
| RP11-713M6.1 | 4.13E-03 | 已加工假基因processed pseudogene |
| RP11-522B15.6 | 5.07E-03 | 已加工假基因processed pseudogene |
| RP11-100E13.2 | 5.32E-03 | 已加工假基因processed pseudogene |
| RP11-374M1.5 | 6.16E-03 | lincRNA |
| RP11-671J11.5 | 1.32E-02 | TEC (To be Experimentally Confirmed) |
| RPL12P2 | 1.34E-02 | 已加工假基因processed pseudogene |
| RP11-306I1.1 | 1.83E-02 | 已加工假基因processed pseudogene |
然后,依据模型计算的结果,绘制时间依赖的1年、3年和5年生存情况的受试者工作特征曲线(图 2C),其曲线下面积很高(1年达0.904,3年达0.933,5年达0.876),说明了模型中包含的基因表达量能很好地预测患者的生存情况。
为进一步探究模型所得每个患者的风险分数与每个患者生存状况的关系,将患者依据风险分数的高低分为高风险组和低风险组两组(图 2D),分别绘制Kaplan-Meier曲线,并分别计算高低风险组两组时序检验(log-rank test),结果见图 2E。由图可见,高风险分数组的患者预后明显差于低风险分数组的患者(P < 0.0001),这也验证了模型的有效性。
模型中含有的40个基因即为进一步筛选得到的基因,它们在这653个患者中的表达情况见图 3。图中可见,保护性因素在一部分患者中低表达(图 3,左侧),而在其他患者则高表达(图 3,右侧);危险性因素正好相反。进一步分析发现,左侧的患者多为风险分数高的患者,其组织学类型多为G4级胶质母细胞瘤;右侧的患者多为风险分数低的患者,其组织学类型多为G2、G3级的胶质瘤。
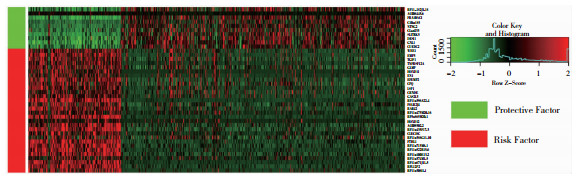
|
| 图 3 模型中40个基因表达量的热图 |
为了验证模型的有效性和可重复性,把所有的患者以6 : 4的比例随机分为训练组和验证组。同样地,在训练组中将患者依据风险分数的高低分为两组,分组界值见图 4A;分别绘制高低风险组两组的Kaplan-Meier曲线,并计算相应的时序检验(log-rank test),结果见图 4B。由图可见,高风险分数组的患者预后明显差于低风险分数组的患者(P < 0.000 1),这也验证了模型的有效性。
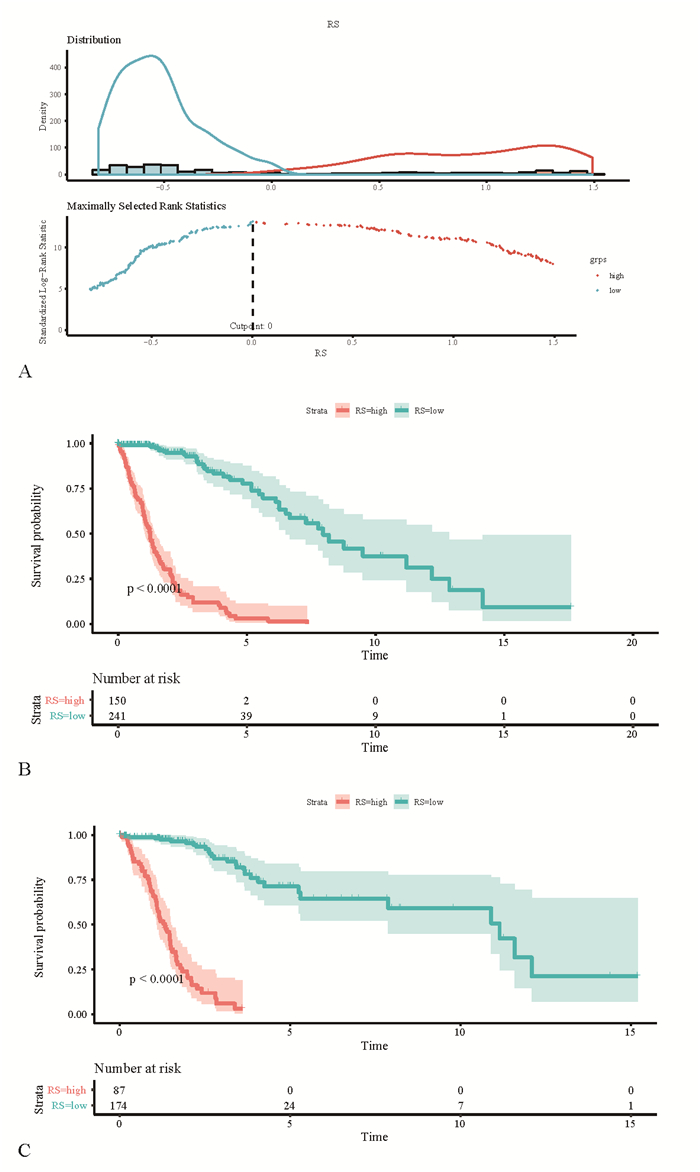
|
| 图 4 验证模型 以6 : 4的比例随机将患者分为训练组和验证组。A.训练组的风险分数界值;B.训练组的Kaplan-Meier曲线图;C.验证组的Kaplan-Meier曲线图。 |
再用训练组得到的风险分数的分组界值,将验证组依据风险分数的高低分为两组,绘制Kaplan-Meier曲线,并计算时序检验(log-rank test),结果见图 4C。由图可见,高风险分数组的患者预后同样明显差于低风险分数组的患者(P < 0.000 1),这也验证了模型的有效性和可重复性。
2.5 模型独立于临床特征为了评价模型是否能独立于患者的临床特征,即不受临床特征的影响,我们对每一个患者由模型所得的风险分数、年龄、性别、胶质瘤级别和组织学类型分别做单因素的Cox回归分析。如表 3所示,在单因素分析中,除了性别,风险分数和其他所有的临床特征均与预后相关(P < 0.05)。
| 变量 | 分类 | 单因素分析 | 多因素分析 | |||||
| 风险比(95%可信区间) | 系数 | P值(Wald test) | 风险比(95%可信区间) | 系数 | P值(Wald test) | 多因素分析P值 | ||
| 年龄 | 年龄 < 50, 0; 年龄 > 50, 1 |
1.068 (1.057-1.078) | 0.0654 | < 2e-16 | 1.0198 (1.0075-1.032) | 0.01963 | 0.0015 | < 2e-16 |
| 性别 | 男, 1;女, 0 | 1.242 (0.949-1.627) | 0.217 | 0.115 | ||||
| 级别 | Ⅰ, 1; Ⅱ, 2; Ⅲ, 3; Ⅳ, 4 |
4.906 (3.966-6.068) | 1.5904 | < 2e-16 | 1.0974 (0.7473-1.612) | 0.09298 | 0.6354 | |
| 组织学类型 | 少突细胞瘤, 1; 少突-星形细 胞瘤, 2; 星形细胞瘤, 3; 胶质母细胞瘤,4 |
2.579 (2.217-3.001) | 0.9475 | < 2e-16 | 0.8853 (0.7104-1.103) | -0.12178 | 0.27832 | |
| 风险分数 | 4.953 (4.203-5.838) | 1.6 | < 2e-16 | 4.3767 (3.2887-5.825) | 1.47628 | < 2e-16 | ||
接着,为了探究风险分数和除了患者性别之外的各个临床特征之间是否因相互作用,产生对预后有影响的假象,我们对每一个患者的风险分数和除了患者性别之外的各个临床特征做多因素的Cox回归分析。如表 3,风险分数仍具有统计学意义,这说明它可以独立于临床特征;胶质瘤级别和组织学类型不再成为有统计学意义的指标,剩余的年龄和风险分数二者相比较,风险分数的风险比和权重均高于年龄,这说明风险分数对预后的影响要比年龄更重要。以上结果均证明并突出了本实验所构建模型的有效性和重要性。
3 讨论在本实验中,我们利用Lasso回归挑选了40个与胶质瘤患者预后相关的基因表达量,构建了能有效预测患者预后的模型。
在图 3中可见,这40个基因的表达情况在两个人群中明显不同,从临床的角度看,对于G4级的胶质母细胞瘤患者,其胶质瘤的恶性程度最高、预后最差,患者的生存时间很短,这其中有些患者甚至丧失了手术的机会。在热图中,这些患者的保护性基因的表达量明显低于其他患者,而危险性基因的表达量则明显高于其他患者,这表明,正是由于以这些基因为代表的基因群组在这两个不同人群中的差异表达导致了他们的预后不同。
在模型中所涉及的40个基因中,只有少数几个被发现与肿瘤患者的预后相关,应该对这40个基因进一步研究,找到它们的作用机制。在这40个基因中,有近一半的基因并非编码mRNA的基因,应当对这些基因给予足够的重视,研究它们与mRNA的互作关系。现在,针对胶质瘤患者的一些非编码RNA的治疗方法渐渐成为研究热点[9, 10]。我们希望通过对这些基因的进一步研究,开发出新的药物,靶向增加保护性基因的表达、减少危险性基因的表达,从而延长胶质瘤患者的生存时间。
相比于常规分子病理检测,该模型能够精准和个体化地为患者服务。虽然常规分子病理检测(如IDH是否有突变、1p/19q是否缺失)确实可以对不同分子亚型患者的预后做出大致判断(如IDH野生型和1p/19q无缺失的患者群体预后差),但分子病理检测的结果代表的是不同亚型群体的整体间的预后差异情况,这种预测并不准确,不能达到个体化诊疗的标准[11]。首先,对不同分子亚型的患者,在整体预后好的亚型中有的患者预后差,更类似于整体预后差的亚型,反之亦然;而且,在同一种分子亚型患者中,不同患者的生存时间仍有很大的差异。而我们的模型可以将生存风险具体到每一个患者,能更为精确地预测患者预后,能够更好地为患者个体化服务。
为了获得最准确的预测结果,每位患者均需要检测40个风险基因的表达量,在临床实际应用上确实存在一定缺陷,对于一些患者而言费用将较高昂,所以我们将致力于在确保精度的情况下缩小需要检测的基因数目,尽量减少患者的花费,并考虑对科研成果的转化,如研制相应的检测平板。
虽然本实验得出很多可观的结论,但仍有很多局限性。首先,我们没有在其他数据库中验证这一模型是否适用于其他数据库的患者,因为验证的前提是需要满足两个数据库的测序方法一致、数据库有完善的预后信息和足够多的验证患者数量,而现在还没有能够同时满足这些条件的数据库[12]。其次,我们也没有在最开始就将TCGA的数据库分成训练组和验证组,因为我们担心如果挑选其中一部分患者构建模型,会导致所选择的基因不能代表整个数据库的情况,丧失某些重要的基因。我们希望随着技术的进步和时代的发展,能够有这样一个数据库出现,以用于验证我们的模型。
| [1] |
Behin A, Hoang-Xuan K, Carpentier AF, et al. Primary brain tumours in adults[J]. Lancet, 2003, 361(9354): 323-331. DOI:10.1016/S0140-6736(03)12328-8 |
| [2] |
刘志雄. 关于颅底外科的几点思考[J]. 中国耳鼻咽喉颅底外科杂志, 2017, 23(4): 295-298. |
| [3] |
Huang J, Zhao D, Liu Z, et al. Repurposing psychiatric drugs as anti-cancer agents[J]. Cancer Lett, 2018, 419: 257-265. DOI:10.1016/j.canlet.2018.01.058 |
| [4] |
Jiang T, Mao Y, Ma W, et al. CGCG clinical practice guidelines for the management of adult diffuse gliomas[J]. Cancer Lett, 2016, 375(2): 263-273. DOI:10.1016/j.canlet.2016.01.024 |
| [5] |
Sang S, Wanggou S, Wang Z, et al. Clinical Long-Term Follow-Up Evaluation of Functional Neuronavigation in Adult Cerebral Gliomas[J]. World Neurosurg, 2018, 119: e262-e271. DOI:10.1016/j.wneu.2018.07.127 |
| [6] |
李小煜, 沈庆煜, 彭英. 胶质母细胞瘤非手术治疗研究进展[J]. 国际神经病学神经外科学杂志, 2018, 45(5): 515-519. |
| [7] |
Quackenbush J. Microarray analysis and tumor classification[J]. N Engl J Med, 2006, 354(23): 2463-2472. DOI:10.1056/NEJMra042342 |
| [8] |
Lee S, Seo MH, Shin Y. The lasso for high dimensional regression with a possible change point[J]. J R Stat Soc Series B Stat Methodol, 2016, 78(1): 193-210. DOI:10.1111/rssb.12108 |
| [9] |
Katsushima K, Natsume A, Ohka F, et al. Targeting the Notch-regulated non-coding RNA TUG1 for glioma treatment[J]. Nat Commun, 2016, 7: 13616. DOI:10.1038/ncomms13616 |
| [10] |
Floyd D, Purow B. Micro-masters of glioblastoma biology and therapy:increasingly recognized roles for microRNAs[J]. Neuro Oncol, 2014, 16(5): 622-627. DOI:10.1093/neuonc/nou049 |
| [11] |
韩硕, 张晓华. 基于胶质瘤分子分型的靶向治疗[J]. 国际神经病学神经外科学杂志, 2018, 45(1): 83-86. |
| [12] |
王非一凡, 李学军. 医疗大数据时代的脑胶质瘤研究[J]. 国际神经病学神经外科学杂志, 2016, 43(5): 456-459. |
 2019, Vol. 46
2019, Vol. 46