扩展功能
文章信息
- 董发达, 马晓虎
- DONG Fa-da, MA Xiao-hu
- MicroRNA相关基因Dicer rs1057035多态性与脑胶质瘤的相关性研究
- Association of the microRNA-related gene Dicer rs1057035 polymorphism and glioma
- 国际神经病学神经外科学杂志, 2018, 45(2): 151-154
- Journal of International Neurology and Neurosurgery, 2018, 45(2): 151-154
-
文章历史
收稿日期: 2017-12-22
修回日期: 2018-03-05
MicroRNA(miRNA)是一类广泛存在于真核生物中的长度约22~24个核苷酸的非编码小RNA,目前研究认为其参与人体约60%蛋白质合成的调控,在胚胎发育,细胞分化、增殖、凋亡等诸多病理生理过程中均起到重要调节作用[1]。近年来,还研究发现miRNA参与包括胶质瘤在内的多种肿瘤性疾病的发生发展过程[2-4]。Dicer蛋白是一种细胞质核糖核酸内切酶,属于RNase Ⅲ家族中的一员,可将pre-miRNA剪切为成熟miRNA,是miRNA生成的关键酶之一。越来越多研究结果显示Dicer与恶性肿瘤的发生、发展密切相关[5, 6]。有荟萃分析结果显示Dicer基因rs1057035多态性与恶性肿瘤易感性密切相关[7],然而目前研究所涉及的恶性肿瘤主要包括膀胱癌、宫颈癌、乳腺癌、结直肠癌及肝细胞癌等,国内外关于Dicer基因rs1057035多态性与脑胶质瘤的相关性研究目前鲜有报道。因此,本研究推测rs1057035位点基因多态性与脑胶质瘤遗传易感性相关,通过比较200例脑胶质瘤患者和184例非肿瘤对照组患者rs1057035位点基因型和等位基因分布频率,探讨rs1057035基因多态性与脑胶质瘤的关系,为临床工作提供参考。
1 对象与方法 1.1 研究对象选择2015年1月至2017年1月于我科住院治疗的脑胶质瘤患者作为病例组,所有脑胶质瘤患者诊断均经病理检查证实,且样本采集前未经过放化疗治疗,病例组共纳入200例患者,其中男性111例,女性89例,平均年龄(46.1±9.8)岁。随机选择同期在我科住院治疗的非肿瘤患者作为对照组,各项检查均排除合并有肿瘤性疾病可能,且无肿瘤家族史,对照组共纳入184例患者,其中男性106例,女性78例,平均年龄(45.8±9.3)岁。所有入组患者均为陕西汉族人,且彼此无血缘关系。本研究通过本院伦理委员会审核批准,所有入组成员均充分知情同意。
1.2 主要仪器和试剂DNA提取试剂盒购自北京天根生化科技有限公司,引物为上海化工生物技术服务有限公司提供,PCR仪及高速低温离心机均购自德国Eppendorf公司,垂直电泳槽购自北京六一仪器厂,紫外透射仪购自无锡君达仪器有限公司。
1.3 方法 1.3.1 临床资料收集采集内容主要包括患者年龄、性别、吸烟及酗酒史,放射线职业暴露史、肿瘤性疾病家族史等基本信息,病例组患者还需记录肿瘤WHO病理分级结果。本研究将吸烟定义为每天≥1支香烟,持续≥3个月;酗酒定义为每周至少1次饮乙醇≥60g,或每天饮乙醇≥20g,持续≥3个月;放射线职业暴露史定义为从事接触放射线的相关工作≥1年;肿瘤家族史定义为一级直系亲属中存在恶性肿瘤患者。
1.3.2 基因多态性检测采集清晨空腹时外周静脉血用于实验室检查,留取部分样本采用DNA提取试剂盒提取全血基因组DNA。采用聚合酶链反应-限制性内切酶分析法检测Dicer基因rs1057035位点单核苷酸多态性。PCR反应体系(25μL)包括:上、下游引物各1μL(上游:5’-AGTTAGGACTGCGGAAAG-3’;下游:5’-GCAATGTGAGACCGAATG-3’),模板DNA 1μL,dNTP混合物2μL,TaqDNA聚合酶0.25μL,10×Buffer 2.5μL,其余部分由灭菌蒸馏水补足。反应条件:首先95℃预变性5min,然后按照95℃变性30s、58℃退火30s、72℃延长30s的顺序循环34周期,最后72℃延长7min。取0.5μL延伸产物使用限制性内切酶HhaⅠ进行酶切,酶切体系(20μL)包括:PCR产物17μL,10×Buffer 2μL和内切酶1μL,反应条件为37℃ 16h,然后65℃下酶切10min终止反应。取酶切产物进行电泳,紫外灯下观察并摄片。
1.4 统计学处理采用SPSS 19.0统计软件分析处理,本研究计量资料均符合正态分布及方差齐性的计量资料,结果以x±s表示,计数资料结果以构成百分比(%)表示,组间比较采用χ2检验。基因型分布采用Hardy-Weinberg平衡定律检验。采用比值比(Odd Ratio,OR)及其95%可信区间(Confidence Interval,CI)表示相对风险度。p < 0.05为差异有统计学意义。
2 结果 2.1 基线资料比较如表 1,病例组与对照组比较,两组在年龄,性别,吸烟,酗酒,放射线职业暴露史和肿瘤性疾病家族史等多项指标差异均无统计学意义(均P>0.05)。
| 组别 | 例数 | 年龄(岁) | 男性[n(%)] | 吸烟[n(%)] | 酗酒[n(%)] | 放射线职业暴露史[n(%)] | 肿瘤性疾病家族史[n(%)] |
| 病例组 | 200 | 46.1±9.8 | 111(55.5) | 68(34.0) | 27(13.5) | 3(1.5) | 20(10.0) |
| 对照组 | 184 | 45.8±9.3 | 106(57.6) | 76(41.3) | 19(10.3) | 2(1.1) | 16(8.7) |
| t/χ2 | 0.328 | 0.173 | 2.182 | 0.916 | - | 0.192 | |
| P | 0.747 | 0.677 | 0.140 | 0.339 | 1.000* | 0.661 | |
| *Fisher精确检验 | |||||||
如图 1,Dicer基因rs1057035位点基因型包括TT,TC和CC三种,本研究中两组患者rs1057035位点基因型分布经检验均符合Hardy-Weinberg遗传平衡(P>0.05),具有群体代表性。
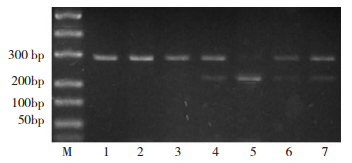
|
| 图 1 Dicer基因rs1057035位点PCR-RFLP电泳图(1, 2, 3:TT;4, 6, 7:TC,5:CC;M:maker) |
如表 2,对照组与病例组比较,Dicer基因rs1057035位点等位基因分布频率差异均具有统计学意义(χ2=5.137,P=0.023),但基因型分布频率差异无统计学意义(χ2=5.133,P=0.077)。
| 病例组(n=200) | 对照组(n=184) | χ2 | P | |
| TT | 161(80.5) | 130(70.7) | 5.133 | 0.077 |
| TC | 36(18.0) | 49(26.6) | ||
| CC | 3(1.5) | 5(2.7) | ||
| T | 358(89.5) | 309(84.0) | 5.137 | 0.023 |
| C | 42(10.5) | 59(16.0) |
如表 3,按照年龄、性别、吸烟分层,各分层中对照组与病例组在显性基因模型和隐性基因模型下差异均无统计学意义(均P>0.05);按照是否酗酒分层,携带突变等位基因C可降低酗酒者胶质瘤发病风险(OR=0.253,95%CI 0.067~0.949,P=0.036);按照有无肿瘤性疾病家族史分层,携带突变等位基因C可降低无肿瘤性疾病家族史患者胶质瘤发病风险(OR=0.561,95%CI 0.339~0.930,P=0.024);按照胶质瘤WHO分型分层,携带突变等位基因C可降低Ⅲ型、Ⅳ型胶质瘤发病风险(OR=0.525,95%CI 0.284~0.969,P=0.037)。
| 项目 | 病例组 | 对照组 | 显性基因模型(TT vs TC+CC) | 隐性基因模型(TT+TC vs CC) | |||||||||
| TT | TC | CC | TT | TC | CC | OR (95%CI) | P | OR (95%CI) | P | ||||
| 年龄 | |||||||||||||
| ≤45岁 | 87 | 19 | 0 | 53 | 19 | 2 | 0.551(0.271~1.119) | 0.097 | 0.973(0.937~1.011) | 0.168 | |||
| >45岁 | 74 | 17 | 3 | 67 | 30 | 3 | 0.549(0.288~1.047) | 0.067 | 1.066(0.210~5.417) | 0.939 | |||
| 性别 | |||||||||||||
| 男 | 90 | 19 | 2 | 74 | 30 | 2 | 0.540(0.287~1.014) | 0.053 | 0.954(0.132~6.899) | 0.963 | |||
| 女 | 71 | 17 | 1 | 56 | 19 | 3 | 0.645(0.316~1.319) | 0.228 | 0.284(0.029~2.789) | 0.251 | |||
| 吸烟 | |||||||||||||
| 是 | 52 | 15 | 1 | 51 | 24 | 1 | 0.628(0.300~1.311) | 0.214 | 1.119(0.069~18.249) | 0.937 | |||
| 否 | 109 | 21 | 2 | 79 | 25 | 4 | 0.575(0.309~1.068) | 0.078 | 0.400(0.072~2.227) | 0.280 | |||
| 酗酒 | |||||||||||||
| 是 | 22 | 5 | 0 | 10 | 8 | 1 | 0.253(0.067~0.949) | 0.036 | 0.947(0.852~1.053) | 0.228 | |||
| 否 | 139 | 31 | 3 | 120 | 41 | 4 | 0.652(0.392~1.084) | 0.098 | 0.710(0.157~3.223) | 0.656 | |||
| 肿瘤性疾病家族史 | |||||||||||||
| 有 | 14 | 5 | 1 | 10 | 6 | 0 | 0.714(0.177~2.875) | 0.635 | 1.053(0.952~1.164) | 0.364 | |||
| 无 | 147 | 31 | 2 | 120 | 43 | 5 | 0.561(0.339~0.930) | 0.024 | 0.366(0.070~1.914) | 0.216 | |||
| WHO分型 | |||||||||||||
| Ⅰ型+Ⅱ型 | 83 | 20 | 2 | 130 | 49 | 5 | 0.638(0.362~1.125) | 0.119 | 0.695(0.132~3.647) | 0.666 | |||
| Ⅲ型+Ⅳ型 | 78 | 16 | 1 | 0.525(0.284~0.969) | 0.037 | 0.381(0.044~3.307) | 0.364 | ||||||
| 注:放射线职业暴露史患者例数较少,分层分析未列出。 | |||||||||||||
MiRNA本身不编码蛋白质,却可通过与靶mRNA3’端非编码区结合,直接降解靶mRNA,或抑制靶mRNA翻译过程,从而参与蛋白质合成的调控,有研究估计体内约60%蛋白质合成过程与miRNA相关[1]。近年来相继有研究发现miRNA-221、miRNA-218、miRNA-16等多种miRNA与胶质瘤遗传易感性相关[2-4, 8]。miRNA相关单核苷酸多态性(single nucleotide polymorphism,SNP)是指一系列与miRNA生成、成熟及发挥作用相关的SNPs,包括miRNA核酸序列SNP,miRNA生物合成酶SNP和miRNA结合位点SNP三大类别[9, 10],Dicer基因SNPs均属于miRNA生物合成酶SNP。
Dicer酶是miRNA成熟的关键酶之一,Dicer酶表达异常可直接影响miRNA生理功能发挥。既往多数研究均认为Dicer是一种抑癌因子,多种恶性肿瘤患者被发现存在Dicer表达下降,然而有研究发现前列腺癌和Burkitt’s淋巴瘤患者Dicer基因表达上调[11, 12]。同样,在研究Dicer与恶性肿瘤预后之间关系时也得到相似结论,黑色素瘤和结直肠癌患者预后不佳可能与Dicer高表达相关,而乳腺癌患者预后不佳可能与Dicer低表达相关[13, 14]。笔者分析认为这些研究结果提示针对不同部位肿瘤性疾病与Dicer关系有必要分别进行探讨。
Dicer基因rs1057035位点突变广泛存在亚洲人群中,既往研究发现该位点多态性与多种恶性肿瘤易感性相关,Yu等[7]一项纳入12675例患者的荟萃分析结果显示rs1057035多态性与恶性肿瘤易感性密切相关,携带Dicer基因rs1057035位点等位基因C可降低肿瘤性疾病发病风险,然而目前尚缺乏rs1057035多态性与胶质瘤相关性的研究报告。本研究发现Dicer基因rs1057035位点等位基因C在对照组人群中分布频率为16%,与既往研究相似。本研究结果还显示病例组与对照组比较,rs1057035位点等位基因分布频率差异均具有统计学意义,携带突变等位基因C可降低胶质瘤患病风险。本研究进一步按照年龄,性别,吸烟,酗酒,肿瘤性疾病家族史和胶质瘤WHO分型进行分层,分别探讨与胶质瘤易感性的关系,结果提示携带突变等位基因C可降低酗酒者、无肿瘤性疾病家族史患者胶质瘤发病风险,也可降低发生Ⅲ型、Ⅳ型胶质瘤发病风险,但其中具体机制目前尚不清楚,可能在于本研究纳入患者例数较少,期待后期大样本研究加以验证。
综上所述,Dicer基因rs1057035多态性可能与胶质瘤易感性密切相关,等位基因C可降低胶质瘤发病风险,但本研究纳入对象有限,需要更多相关研究结果验证。另外,关于Dicer在肿瘤性疾病中发挥功能的具体机制,rs1057035位点与其他SNPs相互作用等问题也期待后续进一步研究。
| [1] |
Lin S, Gregory RI. MicroRNA biogenesis pathways in cancer[J]. Nat Rev Cancer, 2015, 15(6): 321-333. DOI:10.1038/nrc3932 |
| [2] |
Tu Y, Gao X, Li G, et al. MicroRNA-218 inhibits glioma invasion, migration, proliferation, and cancer stem-like cell self-renewal by targeting the polycomb group gene Bmi1[J]. Cancer Res, 2013, 73(19): 6046-6055. DOI:10.1158/0008-5472.CAN-13-0358 |
| [3] |
Yang TQ, Lu XJ, Wu TF, et al. MicroRNA-16 inhibits glioma cell growth and invasion through suppression of BCL2 and the nuclear factor-κB1/MMP9 signaling pathway[J]. Cancer Sci, 2014, 105(3): 265-271. DOI:10.1111/cas.12351 |
| [4] |
Palumbo S, Miracco C, Pirtoli L, et al. Emerging roles of microRNA in modulating cell-death processes in malignant glioma[J]. J Cell Physiol, 2014, 229(3): 277-286. DOI:10.1002/jcp.24446 |
| [5] |
Heravi-Moussavi A, Anglesio MS, Cheng SW, et al. Recurrent somatic DICER1 mutations in nonepithelial ovarian cancers[J]. N Engl J Med, 2012, 366(3): 234-242. DOI:10.1056/NEJMoa1102903 |
| [6] |
Anglesio MS, Wang Y, Yang W, et al. Cancer-associated somatic DICER1 hotspot mutations cause defective miRNA processing and reverse-strand expression bias to predominantly mature 3p strands through loss of 5p strand cleavage[J]. J Pathol, 2013, 229(3): 400-409. DOI:10.1002/path.4135 |
| [7] |
Yu YY, Kuang D, Yin XX. Association between the DICER rs1057035 polymorphism and cancer risk:evidence from a meta-analysis of 1, 2675 individuals.[J]. Asian Pac J Cancer Prev, 2015, 16(1): 119-124. DOI:10.7314/APJCP.2015.16.1.119 |
| [8] |
付胜伟. 微小分子RNA在胶质瘤中的研究进展[J]. 国际神经病学神经外科学杂志, 2012, 39(4): 373-376. |
| [9] |
刘于海, 牛朝诗. MicroRNA与胶质母细胞瘤的相关性研究[J]. 国际神经病学神经外科学杂志, 2009, 36(5): 412-416. |
| [10] |
Rah H, Jeon YJ, Lee BE, et al. Association of polymorphisms in microRNA machinery genes (DROSHA, DICER1, RAN, and XPO5) with risk of idiopathic primary ovarian insufficiency in Korean women[J]. Menopause, 2013, 20(10): 1067-1073. DOI:10.1097/GME.0b013e3182883907 |
| [11] |
Kurzynskakokorniak A, Koralewska N, Pokornowska M, et al. The many faces of Dicer:the complexity of the mechanisms regulating Dicer gene expression and enzyme activities[J]. Nucleic Acids Res, 2015, 43(9): 4365-4380. DOI:10.1093/nar/gkv328 |
| [12] |
Chiosea S, Jelezcova E, Chandran U, et al. Overexpression of Dicer in precursor lesions of lung adenocarcinoma[J]. Cancer Res, 2007, 67(5): 2345-2350. DOI:10.1158/0008-5472.CAN-06-3533 |
| [13] |
Jafarnejad SM, Ardekani GS, Ghaffari M, et al. Sox4 Mediated Dicer Expression is Critical for Suppression of Melanoma Cell Invasion[J]. Oncogene, 2013, 32(17): 2131-2139. DOI:10.1038/onc.2012.239 |
| [14] |
Michael S, Marina S, Daniel S, et al. Comparative microarray analysis of microRNA expression profiles in primary cutaneous malignant melanoma, cutaneous malignant melanoma metastases, and benign melanocytic nevi[J]. Cell Tissue Res, 2013, 351(1): 85-98. DOI:10.1007/s00441-012-1514-5 |
 2018, Vol. 45
2018, Vol. 45