扩展功能
文章信息
- 龙泓羽, 肖文彪, 吴龑昊, 罗朝辉, 肖波
- LONG Hong-Yu, XIAO Wen-Biao, WU Yan-Hao, LUO Zhao-Hui, XIAO Bo
- 颞叶癫痫microRNA基因甲基化模式分析及机制探究
- Genome-wide DNA methylation patterns of miRNAs in temporal lobe epilepsy patients and its underlying mechanism
- 国际神经病学神经外科学杂志, 2017, 44(4): 352-356
- Journal of International Neurology and Neurosurgery, 2017, 44(4): 352-356
-
文章历史
收稿日期: 2016-11-16
修回日期: 2017-07-26
癫痫是一组由于脑神经元异常过度放电所致的短暂、反复发作的神经系统功能紊乱为特征的慢性病和综合征,是最常见的神经疾病之一,全世界约1%的人患有癫痫。颞叶癫痫(temporal lobe epilepsy, TLE)是成人部分性癫痫的最常见形式[1]。虽然过去的研究中,TLE的神经病理学方面取得了巨大进展,但其潜在发病和耐药机制仍未阐明[2]。microRNA(miRNA)是内源性小非编码RNA(约22个核苷酸),主要通过特异性结合mRNA转录本3’UTR序列,使mRNA翻译抑制或mRNA去腺苷化而降解,从而调节转录后基因表达[3]。在癫痫动物模型和癫痫患者一系列研究中发现超过100种不同的miRNA水平发生变化[4]。研究表明miRNA失调在癫痫的发生和发展中起重要作用[5]。
基因启动区DNA甲基化对基因抑制至关重要[6]。最近的研究表明DNA甲基化不仅调节蛋白编码基因的表达,而且影响miRNA的表达[7]。例如在乳腺癌研究中李永生等[8]观察到,在miRNAs基因的启动子中甲基化变化比在蛋白质编码基因中更频繁。此外,通过比较分析表达和启动子甲基化,Miller-Delaney等[9]确定了TLE中一组差异甲基化miRNAs,并提出miRNA可能对DNA甲基化更为敏感。然而,关于TLE中通过DNA甲基化调控miRNA的机制了解甚少。
本研究使用450 K甲基化芯片定量分析TLE患者和健康对照外周血DNA中485577个CpG位点的甲基化。通过重新注释DNA甲基化芯片获得miRNA基因启动子甲基化水平,随后对差异甲基化miRNA进行功能富集分析。本研究可能有助于了解TLE中miRNAs基因表达调节机制,及其在癫痫发生以及耐药机制中的作用。
1 对象与方法 1.1 样本研究方案由中南大学湘雅医院伦理委员会批准,获得参与研究者的知情同意。从30个TLE患者和30个性别和年龄匹配的健康对照者中收集外周血样品。所有患者符合国际抗癫痫联盟(ILAE)TLE的诊断标准,耐药组纳入标准为应用足量足疗程的两种或两种以上合理的抗癫痫药物治疗,癫痫发作仍未能完全控制的患者。敏感组纳入标准为合理应用抗癫痫药物治疗,依据最近12个月内癫痫发作的最长间隔,3倍最长发作间隔(≥12月)无癫痫发作患者[10]。所有患者于2010年至2014年在湘雅医院神经内科招募,均接受全面临床检查,包括病史、查体、脑磁共振(MRI)和脑电图(EEG),详见表 1。
| 观察指标 | TLE组(n=30) | 对照组(n=30) |
| 性别 | ||
| 男 | 18 | 18 |
| 女 | 12 | 12 |
| 年龄 | ||
| 男(x±s, n) | 25.28±11.75 | 28.17±13.96 |
| 女(x±s, n) | 33.33±14.28 | 35.83±11.19 |
| EEG | ||
| 左侧颞叶[n(%)] | 10 (33.33) | NA |
| 右侧颞叶[n(%)] | 11 (36.67) | NA |
| 双侧颞叶[n(%)] | 9 (30.00) | NA |
| MRI | ||
| 正常[n(%)] | 14 (46.67) | NA |
| 海马硬化[n(%)] | 9 (30.00) | NA |
| 其他病变[n(%)] | 7(23.33) | NA |
| 药物反应 | ||
| 耐药 | 10(33.33) | NA |
| 敏感 | 20(66.67) | NA |
根据操作说明使用试剂盒分离全血DNA。NanoDrop分光光度计测定DNA的含量,并计算OD260/OD280比值。对于全基因组甲基化分析,使用illumina人甲基化450 K芯片(illumina, Inc.美国)在485577个CpG位点测量DNA甲基化水平。DNA样品根据illumina推荐的方案进行亚硫酸氢钠转化并与芯片杂交。
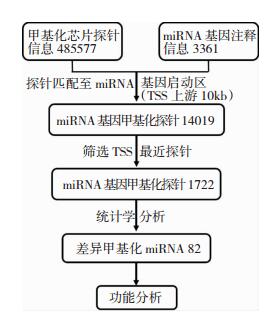
1.3 miRNA甲基化重注释从miRBase下载人类miRNA的注释信息文件。miRNA启动子被定义为每个pri-miRNA的转录起始位点(TSS)上游10 kb。根据探针位置及miRNA基因转录起始终止位点信息,用bedtools软件将全基因组甲基化探针匹配到miRNA基因启动子区域,构建miRNA甲基化谱。与蛋白质编码基因一样,最靠近miRNA启动子的DNA甲基化与转录沉默紧密相关[11],本研究只使用最靠近TSS的探针来决定miRNA启动子的DNA甲基化状态。见图 1。
|
| 图 1 miRNA基因DNA甲基化重注释结果图 |
MiRNA靶基因数据来源于miRGator,miRGator整合了9个(3个实验验证,6个预测算法)miRNA靶基因预测数据库[12]。在预测数据库4/9个中都为阳性的被认为是miRNA靶基因。使用DAVID对差异甲基化miRNA的靶基因进行基因本体论(GO)和京都基因和基因组百科全书(KEGG)通路注释[13]。将校正的P(Benjamini) < 0.05认为是GO和KEGG通路显著性的阈值。
1.5 差异甲基化的统计学分析我们使用student-t检验来比较TLE患者与健康对照以获得未调整的P值,并使用错误发现率(false discovery rate, FDR)多重检验来校正,FDR小于5%的甲基化位点认为是显著的。而对比临床亚组的student-t检验以确定未校正的P值,P < 0.01的miRNA甲基化位点认为是显著的。所有统计学计算用R软件完成。
2 结果 2.1 miRNA甲基化重注释及差异甲基化miRNA筛选基于TLE全基因组甲基化数据及miRNA注释信息,采用计算策略将探针数据重新匹配到miRNA相关的启动子区域(TSS上游10 kb)。总共有14019个探针序列对应于1722个miRNA启动子区,本研究保留最接近TSS的单个探针以表示miRNA启动子的DNA甲基化状态(即1722个)。为了进一步研究差异甲基化miRNA可能的生物学意义,我们对miRNA启动子的近端探针子集进行统计学分析。在TLE和健康对照之间,miRNA启动子中分析的CpG位点中4.8%(82/1722)(FDR < 5%)存在显著差异,其中70(85%)位点高甲基化和12(15%)位点低甲基化。在82个差异甲基化miRNA中有37个新发现的miRNA(尚未正式命名);有45个已知的miRNA,其中包括MIR23B、MIR27B和LET7A等。
2.2 差异甲基化miRNA功能分析45个已知的差异甲基化miRNA中共有2222个靶基因,经DAVID数据库分析后按照某些共同属性富集在MAPK信号通路、神经营养信号通路、GnRH信号通路等不同的“信号通路”,以及神经元投射发育、神经元分化、轴突导向、突触、神经元投射、转录调节活性等“生物学过程”、“细胞成分”和“分子功能”子集。见表 2。
| 名称 | Benjamini (校正P值) |
|
| GOTERM_BP | GO:0045449~转录调节 | 1.40E-13 |
| GO:0010628~基因表达正性调节 | 2.71E-08 | |
| GO:0031175~神经元投射发育 | 1.03E-05 | |
| GO:0048812~神经元投射形态形成 | 2.83E-05 | |
| GO:0030182~神经元分化 | 5.67E-05 | |
| GO:0048666~神经元发育 | 9.17E-05 | |
| GO:0030334~细胞迁移调节 | 1.25E-04 | |
| GO:0007409~轴突形成 | 3.46E-04 | |
| GO:0001568~血管发育 | 0.0021 | |
| GO:0007411~轴突导向 | 0.0044 | |
| GOTERM_MF | GO:0030528~转录调节活性 | 3.33E-10 |
| GO:0003700~转录因子活性 | 4.21E-10 | |
| GO:0003677~DNA结合 | 2.78E-07 | |
| GO:0043565~序列特异性DNA结合 | 6.75E-06 | |
| GO:0016563~转录激活活性 | 1.07E-04 | |
| GO:0004672~蛋白激酶活性 | 7.55E-04 | |
| GO:0016564~转录抑制活性 | 9.78E-04 | |
| GO:0004674~蛋白丝氨酸/苏氨酸激酶活性 | 0.0031 | |
| GO:0003682~染色质结合 | 0.0034 | |
| GOTERM_CC | GO:0031981~细胞核腔 | 5.03E-07 |
| GO:0005654~细胞核浆 | 3.12E-06 | |
| GO:0044451~核质部分 | 4.95E-06 | |
| GO:0005794~高尔基体 | 9.88E-05 | |
| GO:0043233~细胞器腔 | 9.77E-05 | |
| GO:0031974~膜封闭腔 | 9.38E-05 | |
| GO:0070013~细胞内细胞器腔 | 1.04E-04 | |
| GO:0045202~突触 | 1.15E-04 | |
| GO:0043005~神经元投射 | 4.47E-04 | |
| GO:0005667~转录因子复合物 | 4.67E-04 | |
| KEGG通路 | hsa04010:MAPK信号通路 | 3.89E-04 |
| hsa05200:恶性肿瘤信号通路 | 5.46E-04 | |
| hsa04722:神经营养信号通路 | 0.0083 | |
| hsa05215:前列腺癌 | 0.0437 | |
| hsa05210:结直肠癌 | 0.0407 | |
| hsa04912:GnRH信号通路 | 0.0450 |
对TLE患者临床资料分析进行多层次分组:根据药物治疗情况可分为药物敏感组和耐药组,根据磁共振结果分为海马硬化和MRI阴性组。组间进行两样本t检验,找出差异甲基化miRNA基因(P < 0.01)。药物治疗组差异甲基化miRNA基因有23个,包括MIR30A、MIR128和MIR219等;磁共振组差异甲基化miRNA基因有3个,包括MIR219和MIR5581等。见表 3。
| 临床分组 | 基因名称 | 基因ID号 | P值 |
| 药物耐药VS药物敏感 | MIR1295A | ENSG00000221390 | 0.0016 |
| MIR514A1 | ENSG00000207868 | 0.0095 | |
| MIR514A2 | ENSG00000207866 | 0.0095 | |
| MIR1289-2 | ENSG00000221287 | 0.0035 | |
| MIR302D | ENSG00000199145 | 0.0022 | |
| MIR203 | ENSG00000207568 | 0.0009 | |
| AC105052.1 | ENSG00000238997 | 0.0026 | |
| AL160269.1 | ENSG00000265367 | 0.0089 | |
| AL356479.1 | ENSG00000216037 | 0.0015 | |
| AC044860.1 | ENSG00000221266 | 0.0009 | |
| AL392111.1 | ENSG00000263626 | 0.0053 | |
| AC097470.1 | ENSG00000265720 | 0.0083 | |
| MIR30A | ENSG00000207827 | 0.0004 | |
| AP002490.1 | ENSG00000212089 | 0.0011 | |
| MIR4641 | ENSG00000266494 | 0.0018 | |
| MIR128-2 | ENSG00000207625 | 0.0003 | |
| MIR219-1 | ENSG00000199036 | 0.0087 | |
| AL139821.1 | ENSG00000266069 | 0.0025 | |
| AC092846.2 | ENSG00000265001 | 0.0100 | |
| MIR4660 | ENSG00000263407 | 0.0030 | |
| AC027319.1 | ENSG00000221535 | 0.0005 | |
| MIR3177 | ENSG00000265820 | 0.0052 | |
| MIR4745 | ENSG00000265767 | 0.0013 | |
| MRI阴性VS海马硬化 | MIR5581 | ENSG00000263675 | 0.0020 |
| MIR219-1 | ENSG00000199036 | 0.0059 | |
| AL121761.1 | ENSG00000239120 | 0.0083 |
本研究是TLE患者外周血miRNA基因的首次全基因组甲基化分析。研究结果显示,在TLE和健康对照组以及不同的临床亚组中存在miRNA基因甲基化改变。85%(70/82) miRNA基因高甲基化是TLE患者和健康对照之间的主要改变,这与TLE患者和TLE啮齿动物模型的海马中基因高甲基化为主要效应的发现相吻合[9, 14]。此外,有研究表明在TLE患者中两种DNA甲基转移酶(DNMT1和DNMT3a)表达上调[15]。
通过DAVID数据库分析,差异甲基化miRNA基因参与MAPK信号通路、神经营养信号通路等多条生物学通路。神经营养信号通路在神经发育和其他高阶活动如学习和记忆起重要作用[16]。Wang等[17]在癫痫患者循环miRNA表达谱分析中也报道了神经营养信号通路与癫痫相关。另一种失调的通路MAPK信号通路控制基本细胞过程如生长、增殖、分化、迁移和凋亡[18]。在神经系统中,p38 MAPK信号传导具有控制细胞死亡和存活的多种功能,并且可以调控神经元功能,例如突触可塑性[19]。此外p38 MAPK信号通路通过调节P-糖蛋白表达参与难治性癫痫耐药机制的形成[20]。
我们的研究显示在MAPK信号通路中大多数基因是异常甲基化miRNA的靶基因。在这些基因中,钙通道基因和肌细胞增强子结合因子2c(MEF2C)涉及癫痫或癫痫表型。在癫痫患者和癫痫动物模型中已经鉴定了几种钙通道基因的功能获得性突变,说明了钙通道在癫痫病理生理学中的作用[21]。例如,CACNA1A,电压依赖性钙通道家族的成员,其点突变导致脑钙通道CaV2.1的损害,可能在癫痫的发病机制中发挥重要作用[22]。钙通道基因可以是几种异常甲基化miRNA的靶标,例如let-7a、miR-27b、miR-23b和miR-190b。丙戊酸(VPA)和加巴喷丁是常用的抗癫痫药物,其抗癫痫性质已部分归因于抑制离子通道包括电压依赖性钙通道。异常甲基化miRNA(miR-27b和let-7a等)可能靶向电压依赖性钙通道,进而改变钙通道阻断剂的抗癫痫作用。MEF2C,异常甲基化miR-23b的可能靶标,是早期神经发生、神经元分化和迁移所必需的,MEF2C的点突变或缺失可导致严重的精神发育迟滞和癫痫发作[23]。
同时,我们分析发现在药物治疗组和磁共振组差异甲基化miRNA基因中都存在MIR219。MIR219在恒河猴和大鼠呈现脑特异性表达并随年龄梯度逐渐升高的表达模式,提示MIR219参与调控神经系统发育过程的重要环节[24]。MIR219靶基因预测及功能分析提示其参与突触可塑性相关的生物过程,包括钙离子转运、神经递质转运、谷氨酸分泌、长时程增强和长时程抑制等[25]。癫痫动物模型研究显示MIR219通过靶向CaMKIIγ负调节NMDA受体的功能,从而抑制癫痫形成[26]。而颞叶癫痫患者无颗粒细胞层病理改变组与颗粒细胞层分散组比较也存在MIR219差异性表达[27]。肿瘤研究中发现甲基化可调控MIR219表达,进而调节ERK1/2相关的信号转导通路参与胃癌的发生发展[28]。因此我们推测差异甲基化MIR219可能参与TLE病理学进展及耐药机制形成。
DNA甲基化是动态稳定的过程,并与环境因素密切相关。DNA甲基化通过调控miRNA,从而影响信号转导通路中基因表达调控,导致通路功能失调进而影响疾病的发生发展。DNA甲基化与miRNA机制研究不仅能够加深对TLE疾病机制的理解,而且有助于进一步找到TLE临床表型相关的生物标记物,对TLE的诊断和治疗提供新的靶点和导向。
| [1] |
Tellez-Zenteno JF, Hernandez-Ronquillo L. A review of the epidemiology of temporal lobe epilepsy[J]. Epilepsy Res Treat, 2012, 2012: 630853. |
| [2] |
Blumcke I, Coras R, Miyata H, et al. Defining clinico-neuropathological subtypes of mesial temporal lobe epilepsy with hippocampal sclerosis[J]. Brain Pathol, 2012, 22(3): 402-411. DOI:10.1111/bpa.2012.22.issue-3 |
| [3] |
Djuranovic S, Nahvi A, Green R. miRNA-mediated gene silencing by translational repression followed by mRNA deadenylation and decay[J]. Science, 2012, 336(6078): 237-240. DOI:10.1126/science.1215691 |
| [4] |
Alsharafi WA, Xiao B, Abuhamed M M, et al. miRNAs:biological and clinical determinants in epilepsy[J]. Front Mol Neurosci, 2015, 8: 59. |
| [5] |
Jimenez-Mateos EM, Henshall DC. Epilepsy and microRNA[J]. Neuroscience, 2013, 238: 218-229. DOI:10.1016/j.neuroscience.2013.02.027 |
| [6] |
Jones PA. Functions of DNA methylation:islands, start sites, gene bodies and beyond[J]. Nat Rev Genet, 2012, 13(7): 484-492. DOI:10.1038/nrg3230 |
| [7] |
Sato F, Tsuchiya S, Meltzer SJ, et al. MicroRNAs and epigenetics[J]. FEBS J, 2011, 278(10): 1598-1609. DOI:10.1111/j.1742-4658.2011.08089.x |
| [8] |
Li Y, Zhang Y, Li S, et al. Genome-wide DNA methylome analysis reveals epigenetically dysregulated non-coding RNAs in human breast cancer[J]. Sci Rep, 2015, 5: 8790. DOI:10.1038/srep08790 |
| [9] |
Miller-Delaney SF, Bryan K, Das S, et al. Differential DNA methylation profiles of coding and non-coding genes define hippocampal sclerosis in human temporal lobe epilepsy[J]. Brain, 2015, 138(Pt 3): 616-631. |
| [10] |
Kwan P, Arzimanoglou A, Berg AT, et al. Definition of drug resistant epilepsy:consensus proposal by the ad hoc Task Force of the ILAE Commission on Therapeutic Strategies[J]. Epilepsia, 2010, 51(6): 1069-1077. |
| [11] |
Morita S, Takahashi RU, Yamashita R, et al. Genome-wide analysis of DNA methylation and expression of microRNAs in breast cancer cells[J]. Int J Mol Sci, 2012, 13(7): 8259-8272. |
| [12] |
Cho S, Jang I, Jun Y, et al. MiRGator v3.0:a microRNA portal for deep sequencing, expression profiling and mRNA targeting[J]. Nucleic Acids Res, 2013, 41(Database issue): D252-D257. |
| [13] |
Huang DW, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44-57. |
| [14] |
Kobow K, Kaspi A, Harikrishnan KN, et al. Deep sequencing reveals increased DNA methylation in chronic rat epilepsy[J]. Acta Neuropathol, 2013, 126(5): 741-756. DOI:10.1007/s00401-013-1168-8 |
| [15] |
Zhu Q, Wang L, Zhang Y, et al. Increased expression of DNA methyltransferase 1 and 3a in human temporal lobe epilepsy[J]. J Mol Neurosci, 2012, 46(2): 420-426. DOI:10.1007/s12031-011-9602-7 |
| [16] |
Reichardt LF. Neurotrophin-regulated signalling pathways[J]. Philos Trans R Soc Lond B Biol Sci, 2006, 361(1473): 1545-1564. DOI:10.1098/rstb.2006.1894 |
| [17] |
Wang J, Yu JT, Tan L, et al. Genome-wide circulating microRNA expression profiling indicates biomarkers for epilepsy[J]. Sci Rep, 2015, 5: 9522. DOI:10.1038/srep09522 |
| [18] |
Dhillon AS, Hagan S, Rath O, et al. MAP kinase signalling pathways in cancer[J]. Oncogene, 2007, 26(22): 3279-3290. DOI:10.1038/sj.onc.1210421 |
| [19] |
Correa SA, Eales KL. The Role of p38 MAPK and Its Substrates in Neuronal Plasticity and Neurodegenerative Disease[J]. J Signal Transduct, 2012, 2012: 649079. |
| [20] |
Shao Y, Wang C, Hong Z, et al. Inhibition of p38 mitogen-activated protein kinase signaling reduces multidrug transporter activity and anti-epileptic drug resistance in refractory epileptic rats[J]. J Neurochem, 2016, 136(5): 1096-1105. DOI:10.1111/jnc.13498 |
| [21] |
Gambardella A, Labate A. The role of calcium channel mutations in human epilepsy[J]. Prog Brain Res, 2014, 213: 87-96. DOI:10.1016/B978-0-444-63326-2.00004-1 |
| [22] |
Imbrici P, Jaffe SL, Eunson LH, et al. Dysfunction of the brain calcium channel CaV2.1 in absence epilepsy and episodic ataxia[J]. Brain, 2004, 127(Pt 12): 2682-2692. |
| [23] |
Nowakowska BA, Obersztyn E, Szymanska K, et al. Severe mental retardation, seizures, and hypotonia due to deletions of MEF2C[J]. Am J Med Genet B Neuropsychiatr Genet, 2010, 153B(5): 1042-1051. |
| [24] |
张满仓. miR-219时空表达特征及功能研究[D]. 华东师范大学, 2008. http://cdmd.cnki.com.cn/Article/CDMD-10269-2008146683.htm
|
| [25] |
熊璐. miR-219调控NMDAR信号通路在微波辐射致突触可塑性损伤中的作用研究[D]. 中国人民解放军军事医学科学院, 2014.
|
| [26] |
Zheng H, Tang R, Yao Y, et al. MiR-219 Protects Against Seizure in the Kainic Acid Model of Epilepsy[J]. Mol Neurobiol, 2016, 53(1): 1-7. DOI:10.1007/s12035-014-8981-5 |
| [27] |
Zucchini S, Marucci G, Paradiso B, et al. Identification of miRNAs differentially expressed in human epilepsy with or without granule cell pathology[J]. PLoS One, 2014, 9(8): e105521. DOI:10.1371/journal.pone.0105521 |
| [28] |
雷荟仔. miR-219-2-3p在胃癌中的作用及其DNA甲基化调控[D]. 重庆医科大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10631-1014109734.htm
|
 2017, Vol. 44
2017, Vol. 44