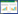扩展功能
文章信息
- 王莹, 李曦, 张隆伯, 伍军, 宋涛, 霍雷, 方加胜
- Wang Ying, Li Xi, Zhang Longbo, Wu Jun, Song Tao, Huo Lei, Fang Jiasheng
- BCL3基因表达:一个新的胶质瘤预后预测因子
- B-cell CLL/lymphoma 3 gene expression:a new prognostic factor for glioma
- 国际神经病学神经外科学杂志, 2017, 44(2): 159-164
- Journal of International Neurology and Neurosurgery, 2017, 44(2): 159-164
-
文章历史
收稿日期: 2017-02-15
修回日期: 2017-04-20
2. 中南大学临床药理研究所, 湖南 长沙 410078
2. Institute of Clinical Pharmacology, Central South University, Changsha, Hunan, 410078
胶质瘤起源于神经间质细胞,在恶性脑肿瘤中约占50%~60%,是中枢神经系统中最为常见的恶性肿瘤[1]。随着核磁共振成像技术的普及,胶质瘤的检出率大大提高,同期胶质瘤的发病率也逐年增长,年增长率达1.2%。胶质瘤恶性程度高,复发率高,预后不佳。个体化治疗是根据患者的临床特点,结合胶质瘤的分子病理学特点和突变基因,制定治疗方案,是一种新型治疗方式。如果能找到更多的可以预测胶质瘤预后的因子,将有助于实现胶质瘤的个体化治疗,改善胶质瘤的预后。
BCL3基因是一个原癌基因,最早发现于慢性B淋巴细胞白血病中,其编码的BCL3蛋白与核因子κB (Nuclear Factor κB, NFκB) 通路关系紧密,是核因子κB抑制蛋白 (Inhibitor of NFκB, IκB) 家族的成员之一, 而NFκB具有明显的抑制细胞凋亡的功能,与肿瘤的发生、生长和转移等多个过程密切相关。目前在胶质瘤研究领域,关于BCL3的报道还很少。仅有Mansour等[2]研究发现诱饵受体1(Decoy Receptor 1,DcR1) 的产生依赖于BCL3/p50,并且可以减弱替莫唑胺在胶质瘤细胞中的作用。Wu等报道BCL3在胶质瘤中的表达水平升高,可以通过干扰RNA的转录影响信号转导及转录激活因子3(Signal transducer and activator of transcription 3, STAT3) 的作用[3],通过多种细胞因子和生长因子调节胶质瘤细胞的增殖、凋亡、侵袭和血管生成。但是BCL3基因表达与胶质瘤发生发展和预后的关系仍不明确。本研究下载了癌症基因组图谱计划 (TCGA) 和中国脑胶质瘤基因组图谱计划 (CGGA) 中的临床样本数据进行数据分析,以探究BCL3基因表达与脑胶质瘤预后的关系。
1 材料和方法 1.1 样本本研究下载了TCGA和CGGA两个数据库中四个数据集的BCL3基因表达数据及其临床预后数据。这四个数据集分别为TCGA数据库中的低级别胶质瘤 (LGG) 数据集和胶质母细胞瘤 (GBM) 数据集以及CGGA数据库中的胶质瘤数据集1(Batch 1) 和胶质瘤数据集2(Batch 2)。LGG数据集包含530个样本,所有的样本均采用RNA测序的方法进行了全基因组表达分析[4]。GBM数据集包含528个样本,所有样本均采用Affymetrix Human Genome U 133A Array基因表达芯片进行全基因组表达分析[5]。CGGA数据库中的Batch 1和Batch 2分别包含220个和85个弥漫性胶质瘤样本,所有样本均采用Agilent Whole Human Genome Oligo Microarray (4×44K) 基因组表达芯片进行全基因组表达分析[6, 7]。所有样本的基本信息见表 1。
| 数据集名称 | LGG | GBM | Batch 1 | Batch 2 |
| 来源 | TCGA | TCGA | CGGA | CGGA |
| 样本类型 | 低级别胶质瘤 | 胶质母细胞瘤 | 弥漫性胶质瘤 | 弥漫性胶质瘤 |
| 样本量 | 530 | 528 | 220 | 85 |
| 年龄范围 (岁) | 14~87 | 10~89 | 12~70 | 23~66 |
| 表达检测手段 |
基于Illumina二代测序平台的RNA测序 | Affymetrix 133A基因表达芯片 | Agilent Whole Human Genome Oligo Microarray基因表达芯片 | Agilent Whole Human Genome Oligo Microarray基因表达芯片 |
本研究中所有的基因表达数据和临床数据均来源于公用数据库。TCGA数据库的LGG和GBM数据集中BCL3基因的表达数据和样本总生存期数据均来源于cBioPortal数据库 (http://www.cbioportal.org/index.do)。其中RNA测序数据归一化的方法为RSEM[8],Affymetrix基因芯片数据归一化的方法为RMA[9]。CGGA数据库的两个数据集 (Batch 1和Batch 2) 中BCL3基因的表达数据和样本总生存期数据来源于CGGA数据库 (http://www.cgga.org.cn/)。下载的数据采用GeneSpring GX 11.0软件进行归一化[6, 7]。
本研究使用Cox回归分析和kaplan-meier (KM) 分析来确定BCL3基因表达与胶质瘤患者生存期之间的关系。在Cox回归分析中我们校正了年龄,而在KM生存分析中,我们将样本BCL3基因表达水平的中位数作为分界点,把BCL3表达水平大于中位数的样本归为BCL3高表达,BCL3表达水平小于中位数的归为BCL3低表达。在该研究中使用KM方法作生存曲线图,所有统计分析均使用SPSS 20.0软件完成。
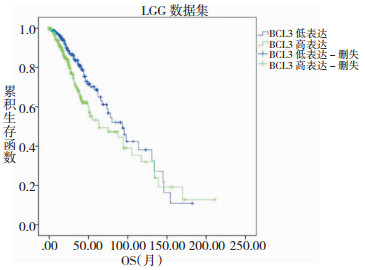
2 结果在LGG数据集中有524个样本具有总生存期 (OS) 数据,生存范围为0.03~211月,中位生存期为23.19月;有488个样本具有无病生存期 (DFS) 数据,无病生存期的范围为0.03~172.63月,中位无病生存期为18.53月。Cox回归分析显示,BCL3的表达与LGG患者的OS和DFS均显著相关,P值分别为1.98×10-10和4.27×10-4。将LGG患者分为BCL3高表达组和低表达组并进一步分析显示,高表达组的OS和DFS均显著差于低表达组 (图 1,图 2),高表达组的死亡风险为低表达组的1.631倍,疾病复发风险为低表达组的1.241倍。详细信息请查看表 2。
|
| 图 1 LGG数据集中BCL3表达与总生存期OS (月) 的关系 |
|
| 图 2 LGG数据集中BCL3表达与无进展生存期DFS (月) 关系 |
| 数据集名称 | LGG | GBM | Batch 1 | Batch 2 |
| Cox OS P1* | 1.98×10-10 | 0.034 | 1.13×10-5 | 2.13×10-3 |
| Cox OS P2* | 5.52×10-3 | 0.037 | 8.40×10-5 | 7.49×10-3 |
| HR OS (95%CI)& | 1.631(1.154~2.304) | 1.222(1.012~1.475) | 2.590(1.612~4.161) | 2.566(1.286~5.120) |
| Cox DFS P1* | 4.27×10-4 | 2.91×10-4 | NA | NA |
| Cox DFS P2* | 0.153 | 1.31×10-3 | NA | NA |
| HR DFS (95%CI)& | 1.241(0.923~1.670) | 1.435(1.151~1.788) | NA | NA |
| BCL3高表达组中位OS (月) | 30.19 | 11.73 | 18.84 | 10.16 |
| BCL3低表达组中位OS (月) | 39.75 | 13.63 | 25.48 | 34.65 |
| BCL3高表达组中位DFS (月) | 17.28 | 6.01 | NA | NA |
| BCL3低表达组中位DFS (月) | 18.13 | 7.62 | NA | NA |
| 注:*为BCL3表达值Cox回归分析P值;#为按照BCL3表达值中位数分组后Cox回归分析P值; & HR为分组后Cox回归分析计算。 | ||||
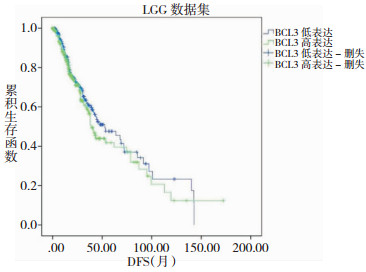
在GBM数据集中有515个个样本具有OS数据,OS的范围为0.1~127.5月,中位OS为12.42月;有380个样本具有DFS数据,DFS的范围为0.1~82.69月,中位DFS为6.7月。Cox回归分析显示,BCL3的表达与GBM患者的OS和DFS均显著相关,P值分别为0.034和2.91×10-4。GBM患者中BCL3高表达组的OS和DFS均显著差于低表达组 (图 3,图 4),高表达组的死亡风险为低表达组的1.222倍,疾病复发风险为低表达组的1.435倍。详细信息请查看表 2。
|
| 图 3 GBM数据集中BCL3表达与总生存期OS (月) 的关系 |
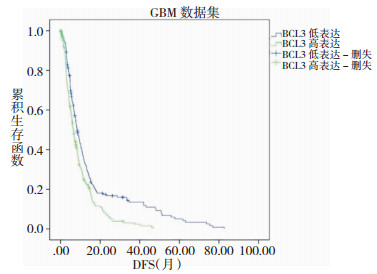
|
| 图 4 GBM数据集中BCL3表达与无进展生存期 (月) 的关系 |
CGGA数据库仅包含OS数据,其中Batch1的OS范围为0.87~53.48月,中位OS为21.32月;Batch2的OS范围为0.68~68.84月,中位OS为16.68月。BCL3的表达在Batch1和Batch2数据集中均与患者的OS显著相关,P值分别为1.13×10-5和2.13×10-3。Batch1和Batch2数据集中BCL3高表达组的OS均显著差于低表达组 (图 5,图 6)。在Batch1数据集中高表达组的死亡风险为低表达组的2.590倍,而在Batch2数据集中高表达组的死亡风险为低表达组的2.566倍。详细信息请查看表 2。
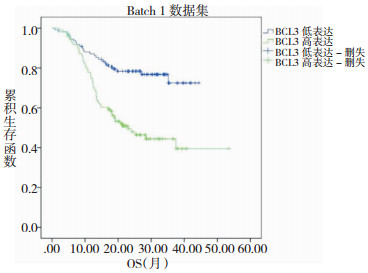
|
| 图 5 Batch1数据集中BCL3表达与总生存期OS (月) 的关系 |
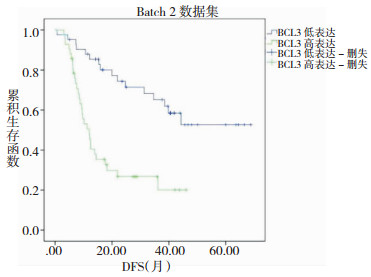
|
| 图 6 Batch1数据集中BCL3表达与总生存期OS (月) 的关系 |
本研究通过对TCGA和CGGA两个公用数据库中的胶质瘤患者临床数据进行分析,首次系统地分析了BCL3基因表达与胶质瘤患者生存时间的关系。研究发现BCL3基因的表达水平与胶质瘤患者的总生存期及无病生存期显著均相关,将BCL3基因表达水平分为高表达组和低表达组,通过生存分析发现BCL3基因表达水平越高,胶质瘤患者的总生存期和无病生存期越短,患者的预后更差。BCL3基因位于19号染色体,其编码的蛋白BCL3在细胞中的分布位置与诱导因子类型相关。尽管IL-6、IL-15、IL-21、TNF-α和IGF1均可以诱导骨髓瘤细胞中BCL3的表达[10],促进骨髓瘤细胞的增殖,但具体的机制存在差异。BCL3基因的高表达可能与细胞因子信号通路激活有关,同时可能由细胞捕获BCL3基因或BCL3基因扩增引起。雌激素对泌乳素细胞的调控,可通过NFκB家族相关的几个基因实现。有研究表明BCL3稍高表达就可以阻断雌二醇的抗细胞增殖作用,而NFκB2/p52即使充分过表达仍难以阻断雌二醇对抗细胞增殖的作用,体现了BCL3的高表达可以促进细胞增殖的作用[11]。在转移性乳腺癌的研究中发现,敲除BCL3会抑制肿瘤细胞的活动性和移动性。BCL3在抗细胞凋亡及促进细胞增殖的过程中,提高了细胞生存的几率,导致突变累积,为肿瘤的形成奠定了基础[12]。
近年来,虽然在胶质瘤的发病机制、诊断及治疗方面取得了很多进展,但是患者的预后仍比较差。有学者[13-15]报告,采取替莫唑胺常规化疗、密集化疗及电磁治疗,新诊断的胶质母细胞瘤患者的中位生存期可以由12.1个月提升到19.4个月,但患者的预后仍存在很大的个体差异。随着分子生物学的发展,EGFR (表皮生长因子受体) 扩增、MGMT (06-甲基鸟嘌呤-DNA-甲基转移酶) 启动子甲基化、IDH1/IDH2(异柠檬酸脱氢酶) 突变、1p19q联合缺失、TP53基因突变等诊断方法逐渐应用于临床,为预测患者的预后和个体化治疗提供了依据,仍不能满足临床需求。IFN-a、IFN-b、TNF-a、IL-2、G-CSF、GM-CSF是目前已被批准用于临床肿瘤治疗的几种细胞因子,而且被证实与NF-κB信号通路有关。以NF-κB为靶点,使用抗氧化剂抑制NF-κB活性以及针对p65和p50设计小干扰RNA (Small interfering RNA, SiRNA) 抑制NF-κB合成是常见的癌症治疗策略。小分子RNA (Micro-RNA, miRNA) 是一种由21-23个核苷酸构成的非编码RNA, 可以负向调节转录后基因的表达。在胃癌的研究中发现,正常情况下miR-19b可以通过抑制BCL3的作用抑制肿瘤细胞的增殖和肿瘤进展,而miR-19b在胃癌中表达水平下降,敲除BCL3基因可以产生与敲除miR-19b基因相同的效应[16]。在卵巢癌中miR-125b可以与BCL3基因的信使RNA负向作用抑制BCL3基因的表达,产生抑制卵巢癌细胞生长的效应[17]。在脑胶质瘤中miR-21的表达水平明显升高,是由于RAS信号通路激活后, 磷酸化作用使得激活剂蛋白-1(AP-1) 表达增强并诱导miR-21转录引起的,而miR-21又可以通过抑制下游靶点影响AP-1的表达,形成一个自我调节的环路[18]。经典的NF-κB途径在脑肿瘤细胞的增殖和侵袭过程中异常活化。Bmi-1可以通过激活NF-κB信号通路促进胶质瘤的血管生成,提高肿瘤的侵袭性[19],而抑制NF-κB通路可以抑制胶质瘤细胞的侵袭能力[20]。被NF-κB激活的miR221/222可以抑制PUMA、PTEN等抑癌基因的表达, 并与NF-κB形成正反馈环路, 不断增强自身的作用[21]。与正常脑组织相比,BCL3基因在胶质瘤中表达水平升高,BCL3可以与NF-κB亚单位结合,抑制RNA的转录,同时可以与Jab1, Pirin, Tip60及Bard1多种转录共调节因子作用[22],影响AP1[23]和STAT3[24, 25]等多个通路的信号传导,可能与胶质瘤细胞的增殖、凋亡及侵袭性有关。以BCL3为靶点抑制NFκB信号通路,有可能成为胶质瘤治疗的新方向。
本研究发现BCL3基因表达水平越高,患者预后越差。BCL3基因表达可以作为一个新的胶质瘤预后预测因子,但是BCL3在胶质瘤中具体的作用机制、与其他分子标记物间的关系及临床应用,仍需进一步的研究。
| [1] | Wen PY, Reardon DA. Neurooncology in 2015:progress in glioma diagnosis, classification and treatment[J]. Nature Rev Neurol, 2016, 12(2): 69–70. DOI:10.1038/nrneurol.2015.242 |
| [2] | Mansour NM, Bernal GM, Wu L, et al. Decoy receptor DcR1 is induced in a p50/Bcl-dependent manner and attenuates the efficacy of temozolomide[J]. Cancer Res, 2015, 75(10): 2039–2048. DOI:10.1158/0008-5472.CAN-14-2144 |
| [3] | Wu J, Li J, Jiang G, et al. B-cell CLL/Lymphoma 3 promotes glioma cell proliferation and inhibits apoptosis through the oncogenic STAT3 pathway[J]. Int J Oncol, 2016, 49(6): 2471–2479. |
| [4] | The cancer genome atlas research network, Brat DJ, Verhaak RG, et al. Comprehensive, integrative genomic analysis of diffuse lower-grade gliomas[J]. N Engl J Med. 2015, 372(26):2481-2498. |
| [5] | The cancer genome atlas research network. Comprhhensive genomic characterization defines human glioblastoma genes and core pathways[J]. Nature, 2008, 455(7216): 1061–1068. DOI:10.1038/nature07385 |
| [6] | Yan W, Zhang W, You G, et al. Moleciular classification of gliomas based on whole genome gene expression:a systematic report of 225 samples from the chinese glioma cooperative group[J]. Neuro-Oncology, 2012, 14(12): 1432–1440. DOI:10.1093/neuonc/nos263 |
| [7] | Sun Y, Zhang W, Chen D, et al. A glioma classification scheme based on coexpression modules of EGFR and PDGFRA[J]. Proc Natl Acad Sci USA, 2014, 111(9): 3538–3543. DOI:10.1073/pnas.1313814111 |
| [8] | Bo Li, Dewey CN. RSEM:accurate transcript quantification from RNA-Seq with or without a reference genome[J]. BMC Bioinformatics, 2011, 12: 323. DOI:10.1186/1471-2105-12-323 |
| [9] | Irizarry RA, Hobbs B, Collin F, et al. Exploration, normalization, and summaries of high density oligonucleotide array probe level data[J]. Biostatistics, 2003, 4(2): 249–264. DOI:10.1093/biostatistics/4.2.249 |
| [10] | Brenne AT, Fagerli UM, Shaughnessy JD. Jr, et al. high expression of BCL3 in human myeloma cells is associated with increased proliferation and inferior prognosis[J]. Eur J Haematol, 2009, 82(5): 354–363. DOI:10.1111/ejh.2009.82.issue-5 |
| [11] | Mitsui T, Ishida M, Izawa M, et al. inhibition of bcl3 gene expression mediates the anti-proliferation action of estrogen in pituitary lactotrophs in primary culture[J]. Mol Cell Endocrinol, 2011, 345(1-2): 68–78. DOI:10.1016/j.mce.2011.07.021 |
| [12] | Maldonado V, Melendez-Zajgla J. Role of BCL-3 in solid tumors[J]. Mol Cancer, 2011, 10: 152. DOI:10.1186/1476-4598-10-152 |
| [13] | Oike T, Suzuki Y, Sugawara K, et al. Radiotherapy plus concomitant adjuvant temozolomide for glioblastoma[J]. Plos One, 2013, 8(11): e78943. DOI:10.1371/journal.pone.0078943 |
| [14] | Gilbert MR, Wang M, Aldape KD, et al. Dose-dense temozolomide for newly diagnosed glioblastoma:a randomized Phase Ⅲ clinical trial[J]. J Clin Oncol, 2013, 31(32): 4085–4091. DOI:10.1200/JCO.2013.49.6968 |
| [15] | Stupp R, Taillibert S, Kanner AA, et al. Maintenance Therapy With Tumor? Treating Fields Plus Temozolomide vs Temozolomide Alone for Glioblastoma:A Randomized Clinical Trial[J]. JAMA, 2015, 314(23): 2535–2543. DOI:10.1001/jama.2015.16669 |
| [16] | Wang H, Xiong M, Hu Y, et al. MicroRNA-19b inhibits proliferation of gastric cancer cells by targeting B-cell CLL/lymphoma 3[J]. Oncol Rep, 2016, 36(4): 2079–2086. |
| [17] | Guan Y, Yao H, Zheng Z, et al. MiR-125b targets BCL3 and suppresses ovarian cancer proliferation[J]. Int J Cancer, 2011, 128(10): 2274–2283. DOI:10.1002/ijc.25575 |
| [18] | 朱潇鹏, 徐庆福, 吕胜青. miR-21与AP-1构成的自我调节环路在脑胶质瘤中的研究进展[J]. 国际神经病学神经外科学杂志, 2013, 40(4): 387–390. |
| [19] | Jiang L, Song L, Wu J, et al. Bmi-1 promotes glioma angiogenesis by activating NF-κB signaling[J]. PLoS One, 2013, 8(1): e55527. DOI:10.1371/journal.pone.0055527 |
| [20] | Song L, Liu L, Wu ZQ, et al. Knockdown of stomatin-like protein 2(STOML2) reduces the invasive ablity of glioma cells through inhibition of the NF-κB/MMP-9 pathway[J]. J Pathol, 2012, 226(3): 534–543. DOI:10.1002/path.v226.3 |
| [21] | 王非一凡, 李学军. miR221/222家族在胶质瘤中的研究进展[J]. 国际神经病学神经外科学杂志, 2014, 41(6): 565–570. |
| [22] | Andrews N, Helliwell T, Walker C, et al. Differing expression of bax and bcl-3 may influence the different cure rates in mouth and orophayrngeal cancer[J]. Clin Otolaryngol, 2000, 25(6): 570–576. |
| [23] | Rebollo A, Dumoutier L, Renauld JC, et al. Bcl-3 expression promotes cell survival following interleukin-4 deprivation and is controlled by AP1 and AP1-like transcription factors[J]. Mol Cell Biol, 2000, 20(10): 3407–3416. DOI:10.1128/MCB.20.10.3407-3416.2000 |
| [24] | Zhao H, Wang W, Zhao Q, et al. BCL3 exerts an oncogenic function by regulating STAT3 in human cervical cancer[J]. Onco Targets Ther, 2016, 9: 6617–6629. |
| [25] | Brocke-Heidrich K, Ge B, Cvijic H, et al. BCL3 is induced by IL-6 via Stat3 binding to intronic enhancer HS4 and represses its own transcription[J]. Oncogene, 2006, 25(55): 7297–7304. DOI:10.1038/sj.onc.1209711 |
 2017, Vol. 44
2017, Vol. 44