扩展功能
文章信息
- 王非一凡, 李学军
- 医疗大数据时代的脑胶质瘤研究
- 国际神经病学神经外科学杂志, 2016, 43(5): 456-459
-
文章历史
收稿日期: 2016-08-10
修回日期: 2016-10-08
在神经胶质瘤的诊疗领域,无论是肿瘤的流行病学调查、发生机制研究、临床诊断与治疗、预防与监测,还是临床试验研究、新药的研发,都贯穿着对数据的收集、管理和分析。对临床、科研数据的深度挖掘,为胶质瘤研究提供了新思路、新方法,不断推动着胶质瘤研究的快速发展。
大数据(Big Data)是指无法在一定时间范围内用常规软件工具进行捕捉、管理和处理的数据集合,是需要新处理模式才能具有更强的决策力、洞察发现力和流程优化能力来适应海量、高增长率和多样化的信息资产[1, 2]。当前,大数据在生物、医学、金融、电子商务、能源和交通等领域都得到广泛应用。而胶质瘤的发生发展过程中包含着复杂的遗传学分子机制,受到内在基因与外在环境的交互影响。包括胶质瘤在内的肿瘤学基础研究,需要从细胞分子层面对基因组学、转录组学、表观组学、蛋白组学以及代谢组学等各方面的数据进行整合,正是这一特点为大数据技术带来了施展拳脚的舞台。
1 大数据带来胶质瘤研究观念转变2013年3月,美国临床肿瘤学会 (ASCO) 公布了一个利用大数据协助癌症治疗的项目CancerLinQ[3]。CancerLinQ是一个“快速学习系统”,允许研究人员进入、访问和分析匿名癌症患者的病历。该项目旨在收集全球肿瘤患者的诊疗数据,用于改进肿瘤临床诊疗模式,以提高患者治疗质量,评估诊疗方法的利弊,促进临床研究的开展。而由多个制药公司参与的非营利性组织:癌症生命科学协会CEO圆桌会(The CEO Roundtable on Cancer),在2014年宣布推出了PDS计划(Project Data Sphere)[4, 5]。该计划打造了一个第三阶段癌症临床试验数据共享和分析平台,初始数据集由阿斯利康、拜耳、新基医药、纪念斯隆-凯特琳癌症中心、辉瑞、赛诺菲等机构共同提供。而这些海量的数据已去除患者的个人信息,并进行了统一编号,供生命科学公司、医院、医疗机构以及独立研究者免费使用。科研人员可以访问平台内置的分析工具或将数据插入到自主研发的分析软件中。
在数据智能化技术开发领域,2014年8月IBM公司通过Watson人工智能系统与纪念斯隆-凯特琳癌症中心合作,提取大量临床数据,筛选数百万条记录文本、期刊文章以及临床试验报告,通过整合这些信息,可为临床医生提供规范化临床路径及个体化治疗建议,这是大数据技术结合人工智能技术在医学领域的重大突破[6]。与传统科学研究相比,基于临床信息的生物大数据具有更可靠的临床资料与样本积累,通过海量信息和数据的采集与分析,能够更准确地进行分子分类。
2 大数据推动胶质瘤分子机制研究肿瘤研究中的基因组学、转录组学、表观组学、蛋白组学以及代谢组学可统称为高通量组学(图 1)。高通量组学研究产生的大数据一般具有“4V”特点:数据量巨大(Volume)、数据种类繁(Variety)、价值有待深入挖掘(value)和检索响应速度快(velocity)。而利用大数据技术对这些高通量、大样本数据进行分析,有助于我们发现规律性的肿瘤分子靶点。
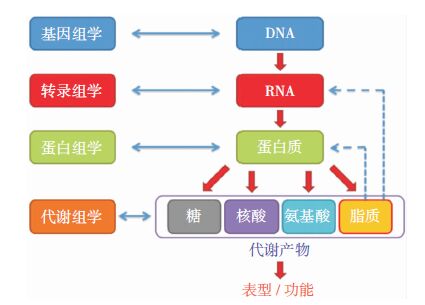
|
| 图 1 多组学多层次的肿瘤学研究 |
Oncomine(www.oncomine.org)则是该类癌症整合数据挖掘平台的代表之一,旨在促进来自全基因组表达谱分析的发现,挖掘癌症基因信息[7]。到目前为止(2016年4月),该数据库已经收集了775个基因表达数据集、86733 个癌症组织和正常组织的样本数据[8]。Oncomine拥有最全面的突变谱、基因表达数据和肿瘤生物标志物,对这一类大数据价值的充分挖掘,将为肿瘤分子靶点的探寻和靶向药物开发提供机遇。
肿瘤基因组图谱(The Cancer Genome Atlas,TCGA)计划,由美国国家癌症和肿瘤研究所(NCI)及国家人类基因组研究所(NHGRI)联合发起,最初的研究目标便是面向胶质母细胞瘤基因组图谱,目前已经完成了多种肿瘤的基因组测序研究。研究人员可以在多组学研究的数据基础上,结合Oncomine资源,对数据进行深入挖掘,不断探索胶质瘤相关的分子靶点(图 2)。Thirumurthi等人[9]基于GeneBank数据库对人Sift6基因调控区序列进行分析,发现MDM2通过介导SIRT6磷酸化来调节AKT1信号通路,从而促进恶性胶质瘤的发生。明确胶质瘤发生的基因突变控制机制,通过检测和评估肿瘤驱动基因与基因组库信息匹配,为预测肿瘤发生风险提供可行的实践方法,这将是未来恶性肿瘤预测的发展趋势。

|
| 图 2 数据挖掘流程 |
与TCGA相对应,由天坛医院江涛教授研究团队牵头的中国人脑胶质瘤基因组学数据库(Chinese Glioma Genome Atlas,CGGA),建立了首个针对中国人群脑胶质瘤的高通量NGS测序大数据网络[10],通过绘制脑胶质瘤融合基因全景图,发现了一系列新的胶质瘤分子病理学标记物。该项目首次在继发胶质母细胞瘤中通过全转录组测序构建了包括214个融合基因的全级别脑胶质瘤融合基因谱,发现了重复出现的PTPRZ1-MET融合基因及其四种不同的融合方式[11]。江涛教授的研究表明,PTPRZ1-MET融合基因是胶质瘤恶性进展的关键驱动因子,可导致患者的中位生存期由8个月缩短至4个月,并筛选出可靶向抑制该融合基因激酶活性的小分子化合物PLB-1001。以上一系列国内外工作在探究胶质瘤发病分子机制的同时,也为胶质瘤精准医疗的提供了潜在治疗靶点。
3 大数据指导胶质瘤精准医学治疗大数据技术应用的飞速发展极大地推动了恶性肿瘤的个体化治疗。利用现代医学平台全面地汇集患者的数据资料(基本临床信息、多模态影像数据和多组学数据),分析并利用这些数据,能够帮助临床医生针对患者个体制定出最佳治疗方案以及进行最优的药物选择,从而实现精准医疗。精准医疗是面向每一位患者的“定制”医疗模式,在这种模式下,医疗的决策、实施等都是针对每一个病人个体特征而制定的,疾病的诊断和治疗是在合理选择病人个体化遗传、分子或细胞学信息的基础上进行的。在胶质瘤的精准治疗方面中,基于大数据挖掘基础上的个体化治疗为脑胶质瘤治疗开辟了全新的治疗天地。
在恶性胶质瘤中VEGF呈过度表达,胶质瘤特征之一是瘤体内能形成新的血管,而 VEGF 能促进内皮细胞增殖,迁移和管腔形成,对瘤体内新血管的形成起关键作用。胶质瘤细胞分泌VEGF与相邻上皮细胞高水平表达的VEGFR-2相结合,促进瘤体内血管发生。VEGER-2选择性强抑制剂PTK787(Vatalanib)能干扰VEGF和PDGF介导的相关血管形成,PTK787与放疗联用能显著抑制P53基因缺乏,并对放疗耐受的移植瘤的生长进行抑制。MD Anderson 癌症研究中心与Dukes大学医学中心对PTK787进行的IB期临床试验中,PTK787与替莫唑胺联用,在11例病例中有5例病情稳定[12],这也代表了大数据精准医学的临床研发新模式:早期即筛选获益优势人群,临床研究分期更加模糊,有效性观察更趋提前,临床研发周期逐步缩短。
端粒逆转录酶(TERT)基因编码是高度特异的逆转录酶,可以协同其它端粒酶复合体延伸染色体3’末端[13]。Labussière等人通过将TERT突变与其他遗传变异进行关联,对395例胶质母细胞瘤(Glioblastoma,GBM)患者资料进行大数据分析,发现联合TERT突变联合表皮生长因子受体(EGFR)扩增及异柠檬酸脱氢酶(IDH)突变能重新定义胶质母细胞瘤的预后:当出现EGFR扩增或者TERT启动子突变存在时,预后较差,中间生存期约12~16个月;当EGFR未发生改变时,中间生存期得以延长,IDH野生型约超过2年,IDH突变型超过3年[14]。Gilbert等人[15]在RTOG 0825的临床试验中随机选取了637名患者,在这些胶质瘤患者的标准放化疗和替莫唑胺维持治疗中加入贝伐单抗,并对其疗效进行评估[15]。该项研究中的大数据包括了测量基因富集程度的分子分层,这些基因在胶质瘤细胞入侵和新血液供应的构建中发挥了重要作用,而贝伐单抗则能够阻止这些基因发挥以上作用。最终结果显示较低的间叶细胞标记与患者服用贝伐单抗后拥有较高存活率具有显著关联。根据这种关联和43种基因的检查结果,研究者创建了一种新型基因表达预测器的模型,可对使用贝伐单抗患者的结局进行预测。
随着对大数据样本量与分子病理的广泛应用,以上这些针对胶质瘤细胞受体,关键基因和调控分子的分子靶向治疗已成为肿瘤治疗研究中的热点,为实施胶质瘤的精准医学治疗提供了独特视角与有力工具。
4 大数据时代胶质瘤诊疗的挑战目前医疗大数据的特点是复杂化、碎片化、不兼容和非完整性,这就导致了临床医生及科研人员很难访问和使用,所以医疗数据标准化、规范化的收集成了一大难题。一例胶质瘤患者的医疗数据可以来自于不同的医院和科室、报告和记录,这些数据往往是非结构化的。与此同时,来源于不同地区、研究机构、人员、方法、仪器的测序数据,都包含不同程度的系统性差异。毋庸置疑,所有的精准模型都是建立在海量病人数据之上。越早建立起成熟的大数据质控机制,不断用更新的病人信息修正预测模型,成为目前大数据应用于胶质瘤诊疗的当务之急。
为此,美国FDA牵头了RNA测序质量控制(RNA-seq metrics for quality control,SEQC)项目[16, 17],通过对比多个试验室RNA-seq数据的可比性,评估了不同测序平台和数据分析法的表现,并将它们与DNA芯片进行比较。这一研究检测了30个RNA测序实验室的现有技术和主要方法。结果显示,在发现接头区域和分析差异性基因表达时,使用合适的生物信息学方法,不同研究组就可以获得可靠的可重复结果。SEQC项目代表了大规模测序走向临床应用的第一步,涉及12个国家的150名研究者,华大基因也是该项目的主要参与者之一。这类研究不仅能帮助人们更加全面地理解测序大数据,还能催生更多策略来增强临床大数据的可重复性。除此之外,进一步加强各学科间的交流与合作,进行标准化的多中心海量数据的收集(包括基础生物医学信息与临床信息),研究新型的数据处理工具和方法,包括数据分析构架、软件系统等,提高数据资源的利用效率,对医疗大数据的标准化、规范化收集均具有重要意义。
5 结语综上,大数据时代的来临对传统的胶质瘤研究模式提出了挑战,在面临数据碎片化、隐私保护等问题的同时,也为胶质瘤的诊疗带来了重大机遇。它将影响到我们防病治病的方式,重塑医院临床管理构建和科研投入模式。大数据的发展使人们对胶质瘤的认知从细胞水平深入到分子水平,这为临床医生对胶质瘤进行“精准”预测与诊断、寻找新的基因靶点、开发及使用新的靶向药物、进行个体化治疗,以及对胶质瘤的实时监测等提供了全新的机遇。而胶质瘤的治疗原则也将从传统以手术切除为主转变为分子生物学综合诊疗,胶质瘤的预防、检测、诊断、治疗和康复都将因大数据时代的到来而面目一新。
| [1] | Howe D, Costanzo M, Fey P, et al. Big data:The future of biocuration. Nature , 2008, 455 (7209) : 47–50. DOI:10.1038/455047a |
| [2] | Swan M. The quantified self:Fundamental disruption in big data science and biological discovery. Big Data , 2013, 1 (2) : 85–99. DOI:10.1089/big.2012.0002 |
| [3] | Schilsky RL, Michels DL, Kearbey A H, et al. Building a rapid learning health care system for oncology:The regulatory framework of CancerLinQ. Journal of Clinical Oncology , 2014, 32 (22) : 2373–2379. DOI:10.1200/JCO.2014.56.2124 |
| [4] | Hede K. Project data sphere to make cancer clinical trial data publicly available. Journal of the National Cancer Institute , 2013, 105 (16) : 1159–1160. DOI:10.1093/jnci/djt232 |
| [5] | Green AK, Reeder-Hayes KE, Corty RW, et al. The project data sphere initiative:accelerating cancer research by sharing data. The oncologist , 2015, 20 (5) : 464. DOI:10.1634/theoncologist.2014-0431 |
| [6] | Murdoch TB, Detsky AS. The inevitable application of big data to health care. Jama , 2013, 309 (13) : 1351–1352. DOI:10.1001/jama.2013.393 |
| [7] | Rhodes DR, Yu J, Shanker K, et al. ONCOMINE:a cancer microarray database and integrated data-mining platform. Neoplasia , 2004, 6 (1) : 1–6. DOI:10.1016/S1476-5586(04)80047-2 |
| [8] | Rhodes DR, Kalyana-Sundaram S, Mahavisno V, et al. Oncomine 3.0:genes, pathways, and networks in a collection of 18,000 cancer gene expression profiles.. Neoplasia , 2007, 9 (2) : 166–180. DOI:10.1593/neo.07112 |
| [9] | Thirumurthi U, Shen J, Xia W, et al. MDM2-mediated degradation of SIRT6 phosphorylated by AKT1 promotes tumorigenesis and trastuzumab resistance in breast cancer. Science signaling , 2014, 7 (336) : ra71. DOI:10.1126/scisignal.2005076 |
| [10] | Jiang T, Mao Y, Ma W, et al. CGCG clinical practice guidelines for the management of adult diffuse gliomas. Cancer letters , 2016, 375 (2) : 263–273. DOI:10.1016/j.canlet.2016.01.024 |
| [11] | Bao ZS, Chen HM, Yang MY, et al. RNA-seq of 272 gliomas revealed a novel, recurrent PTPRZ1-MET fusion transcript in secondary glioblastomas. Genome research , 2014, 24 (11) : 1765–1773. DOI:10.1101/gr.165126.113 |
| [12] | Conrad C, Friedman H, Reardon D, et al. A phase I/II trial of single-agent PTK 787/ZK 222584(PTK/ZK), a novel, oral angiogenesis inhibitor, in patients with recurrent glioblastoma multiforme (GBM)[C]//ASCO Annual Meeting Proceedings. 2004, 22(14_suppl):1512. |
| [13] | 荆尧, 陈世文. 胶质母细胞瘤分子标志物的研究进展. 国际神经病学神经外科学杂志 , 2014, 41 (2) : 185–188. |
| [14] | Labussière M, Boisselier B, Mokhtari K, et al. Combined analysis of TERT, EGFR, and IDH status defines distinct prognostic glioblastoma classes. Neurology , 2014, 83 (13) : 1200–1206. DOI:10.1212/WNL.0000000000000814 |
| [15] | Gilbert MR, Dignam J, Won M, et al. RTOG 0825:phase III double-blind placebo-controlled trial evaluating bevacizumab (Bev) in patients (Pts) with newly diagnosed glioblastoma (GBM)[C]//ASCO Annual Meeting Proceedings. 2013, 31(18_suppl):1. |
| [16] | Xu J, Gong B, Wu L, et al. Comprehensive Assessments of RNA-seq by the SEQC Consortium:FDA-Led Efforts Advance Precision Medicine. Pharmaceutics , 2016, 8 (1) : 8. DOI:10.3390/pharmaceutics8010008 |
| [17] | Seqc/Maqc-Iii Consortium. A comprehensive assessment of RNA-seq accuracy, reproducibility and information content by the Sequencing Quality Control Consortium. Nature biotechnology , 2014, 32 (9) : 903–914. DOI:10.1038/nbt.2957 |
 2016, Vol. 43
2016, Vol. 43