扩展功能
文章信息
- 陈纯, 叶钦勇
- 线粒体转录因子A与神经系统疾病关系的研究进展
- 国际神经病学神经外科学杂志, 2014, 41(3): 297-300
- Disease Surveillance, 2014, 41(3): 297-300
-
文章历史
- 收稿日期:2014-03-17
- 修回日期:2014-05-24
2. 福建医科大学附属协和医院神经内科/福建医科大学脑血管病研究所, 福建省福州市 350001
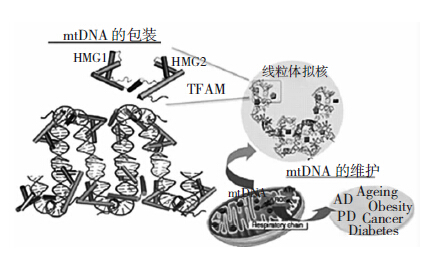
线粒体是真核细胞中重要的细胞器,其代谢活动对细胞中ATP生成至关重要。线粒体拥有自身的线粒体DNA(mtDNA),包含37个无内含子的基因,编码氧化应激途径中的各个亚单位及自身蛋白表达所需的tRNA和rRNA[1]。细胞正常活动所需的大多数ATP来自线粒体呼吸链氧化磷酸化,故维护正常的mtDNA是健康的必要条件。线粒体转录因子A(mitochondrial transcription factor A,TFAM or mTFA),是由核基因编码的高迁移率族蛋白,作为线粒体DNA的重要转录因子之一,通过参与线粒体DNA转录启动、DNA复制,在维持胞内mtDNA功能稳定中扮演重要角色[2]。 1 TFAM的结构和功能 1.1 线粒体拟核
人类mtDNA呈双链环状,分为重链H和轻链L,均为编码链。mtDNA的调节因子主要集中在非编码区,包括三个转录启动子,轻链启动子(light-strand promoter,LSP),重链启动子1(high-strand promoter 1,HSP1)和重链启动子2(heavy-strand promoter 2,HSP2)。在启动子的参与下,mRNA进行转录和转录后加工。单个或多个mtDNA的拷贝,与调节蛋白,伴侣蛋白及介导代谢的酶一起共同形成的结构称为线粒体拟核。在每个线粒体中都存在几个这样的遗传单位[3]。 1.2 TFAM结构和转录
线粒体转录调节机制包括线粒体RNA聚合酶(mtRNAP)、转录因子B2(TFBM2)以及不可缺少的转录因子A(TFAM)[4]。TFAM由核基因编码,属HMG(high mobility group)蛋白超家族成员,由串联的两个HMG结构域HMG1和HMG2及含有25个氨基酸的羧基末端构成,具有较高的迁移率和DNA亲和力。其中,HMG结构域与LSP结合并大幅度弯曲DNA,形成U型结构[5],羧基末端则结合TFBM(见图 1)。
TFAM识别HSP1或LSP启动子(-35到-15)上游约15 bp的位点,募集mtRNAP和TFBM并形成复合物,迅速与转录起始位点下游结合。若除去TFBM,仅有mtRNAP存在的情况下,TFAM激活启动子非依赖性的非特异性转录[7]。Huu等[8]学者在研究mtDNA的U型结构时发现,若DNA链不是U型弯曲的,TFAM的羧基端将背离转录起始位点,使转录不能顺利启动。故推断,TFAM折叠DNA的目的之一是为羧基端参与转录过程提供必要条件,而TFAM的羧基段亦被认为在TFAM与DNA的特异性结合及转录启动中起关键作用。 1.3 TFAM与线粒体拟核
在研究线粒体D环结构中发现,大约每个mtDNA基因组共含有1000个TFAM分子,足以覆盖mtDNA全部区域[9]。裸露的DNA和游离的TFAM在线粒体中都是极不稳定而易被降解。新的研究表明,存在于线粒体基质中的ATP依赖Lon蛋白能降解游离的TFAM[10]。高等真核生物的mtDNA几乎均被TFAM包裹,而人类TFAM分子与mtDNA-蛋白复合体紧密相连,且似乎成为线粒体拟核结构的主要成分,并不仅作为一个转录因子。
研究表明,过表达或抑制TFAM基因,将相应地引起mtDNA水平升高或致命的减少,提示TFAM是mtDNA一个剂量依赖性的影响因子。过多的TFAM抑制转录和mtDNA复制,可能与TFAM非特异性的黏附增加mtDNA致密性有关[11]。因哺乳动物mtDNA转录与复制过程相耦联,TFAM致命性的降低,使LSP的转录活动减少,复制提前终止,并失去TFAM在稳定、维护、组装mtDNA的保护作用。而适当增加TFAM能平衡HSP1和LSP的转录,调节LSP依赖的mtDNA复制,使拟核结构更为致密[12]。因此,TFAM和mtDNA通过相互作用来保持动态平衡,故mtDNA在线粒体内只能以拟核的形式存在。 1.4 TFAM与mtDNA的相互作用
近年研究提示,TFAM单体以被动扩散的形式沿着DNA链快速进行滑动,直至形成TFAM集落,与mtDNA充分交联。TFAM饱和的DNA呈现出更高的灵活性而非更高的密度,因TFAM蛋白具有灵活的空间结构,其与DNA交联部位存在几个bp的解旋,既不影响蛋白间相互作用,又能稳定地解旋和压缩,形成弹性的弯曲而不僵硬[13]。而mtDNA的转录调节、复制及损伤修复必须依靠此种TFAM的滑动和解链。 2 TFAM与神经系统疾病 2.1 TFAM与帕金森病
线粒体功能障碍是包括帕金森病(Parkinson’s disease,PD)在内的多种神经变性病的特点之一。PD相关的几种常见基因,如PARKIN、DJ-1、LRRK2和Pink1,均与线粒体功能密切相关。其中DJ-1基因能抑制活性氧簇(reactive oxygen species,ROS)生成而起保护作用,PARKIN则通过提高TFAM的表达增强线粒体再生,这两种基因的突变导致胞内ROS水平增加,TFAM结合DNA的能力减弱,影响线粒体的再生[14]。一方面,TFAM基因敲除小鼠出现帕金森病样的运动障碍,且该症状能被左旋多巴缓解,提示TFAM功能障碍与PD有关。研究亦证实,诱导TFAM的过表达能逆转1-甲基-4-苯基-四氢吡啶离子(MPP+)对多巴胺能神经元细胞造成的损伤,包括呼吸链复合体Ⅰ的活力,mtDNA的表达,ATP含量和ROS水平[15]。因此可将TFAM视为未来治疗PD的靶点之一。另一方面,学者对部分PD患者和正常人群进行线粒体单核苷酸多态性(single nucleotide polymorphism,SNP)位点分析,并未发现常见的几种TFAM的SNP与PD之间存在联系,受试PD患者中亦没有发现TFAM基因突变[16]。不过,新的研究[17]提示其有可能是帕金森病痴呆的危险因素。与对照组相比,男性受试者中,rs23066604基因型频率存在较大的差异,前额叶皮质线粒体的拷贝数亦大量减少。但这种变化在路易体痴呆(dementia with lewy bodies,DLB)患者体内并未发现。
近期Kent等[18]发现,在PD的转基因小鼠模型及MPTP小鼠模型中,中脑组织的TFAM水平下降,TFAM和ERK1/2的磷酸化水平均升高。细胞外调节蛋白ERK(extracellular regulated protein kinases)是胞内信号转导的关键。TFAM是ERK2的直接目标,具有潜在的磷酸化位点,其HMG1结构域的磷酸化将削弱TFAM结合mtDNA的能力,而易被降解,影响mtDNA的转录。认为,TFAM的磷酸化具有重大的生物学意义,明确TFAM表达的调控机制可能有助于阐明与疾病相关的线粒体自稳和细胞生存。
值得关注的是,据统计,50%~80%的PD患者出现代谢综合征和胰岛素抵抗。线粒体损伤和胰岛素信号转导之间的紧密联系已得到了证实,例如胰岛素分泌障碍的胰岛β细胞中发现TFAM的缺失,故认为PD患者TFAM的波动并非局限于黑质中,似乎累及多系统,符合线粒体疾病的特点,具体机制尚不清楚。 2.2 TFAM与阿尔茨海默病
TFAM过表达能抑制β-淀粉样蛋白(β-amyloid,Aβ)引起的呼吸链活性降低,ATP、细胞色素C氧化酶含量减少及ROS水平升高,保护mtDNA免受Aβ的毒性作用。人TFAM编码基因定位于染色体区10q21.1,与晚发型阿尔茨海默病(late-onset Alzheimer’s disease,LOAD)位点相同。目前,已证实存在两个TFAM的SNP在mtDNA主要序列的变异与LOAD相关。其中,外显子1即rs1937(+35G/C)是唯一的错义突变(Ser12Thr),带有该基因型的患者和正常个体均出现了极高频率的GG纯合子,被视为LOAD的中度危险因子[19]。内含子4即4rs23606604(+113G/A),其等位基因A能增加AD发生的风险,也被证实为LOAD的另一个中度危险因子。 2.3 TFAM与亨廷顿病
在亨廷顿病(Huntington disease,HD)发病机制中,转录辅助活化因子1ɑ(PGC-1α)受损引起的线粒体功能障碍[20]受到重视。PGC-1α基因敲除的小鼠脑中观察到纹状体的海绵状变性,与HD临床表现相符[21]。TFAM是PGC-1α通过下游调控因子核呼吸因子1(NRF-1)调节线粒体功能最重要的靶点之一。在HD转基因小鼠肌肉、纹状体和HD病人肌肉活检中均观察到明显的PGC-1α表达降低及相应的NRF-1和TFAM表达下降[22]。而Elahe等[23]亦认为,TFAM在调控HD发病年龄中发挥重要作用。已证实TFAM的2个SNP能够影响HD患者运动症状的发病时间,分别为3’端的rs1049432和rs11006132,均表现出中度的连锁不平衡性,后者更为重要。不仅如此,相关的SNP间还存在复杂的联合作用和相互关系,可能为未来阐明线粒体损伤与HD的关系提供新的基因学证据。 2.4 TFAM与线粒体脑肌病
Tess等[24]首次报道了TFAM数量异常与线粒体功能障碍相关的病例,在一个活检证实含破碎红纤维的患者肌肉组织中发现,mtDNA存在4100个碱基对缺失及高水平的TFAM和一个额外的TFAM产物。有研究证实TFAM涉及MELAS等线粒体病,在患者肌肉中检测到PGC-1α和TFAM水平增加提示线粒体再生或通过某种潜在的代偿机制激发[25],但该机制可能广泛存在于多种病理变化中,故近年来更专注于另一个突变位点酪氨酸-核糖体RNA合成酶(YARS)表型变异对MELAS特异的致病性[26]。 3 结语
TFAM作为线粒体重要的转录因子,在维持mtDNA稳定中起关键作用,其缺失和突变导致mtDNA结构和功能的紊乱,引起呼吸链和线粒体的损伤,这一变化存在于多种神经系统疾病中。TFAM的SNP突变参与到痴呆、亨廷顿等疾病的演变过程中。适当的TFAM水平升高能够逆转神经元损伤,视为一个潜在的药物靶点,未来可能参与到相关疾病的防治当中。深入了解TFAM的作用及调控机制对阐明疾病的发生发展有重要意义,TFAM的磷酸化及其具体的调控途径是近年研究热点。
| [1] | Kang D, Hamasaki N. Maintenance of mitochondrial DNA integrity: repair and degradation. Curr Genet, 2002, 41: 311-322. |
| [2] | Larsson NG, Wang J, Wilhelmsson H, et al. Mitochondrial transcription factor A is necessary for mtDNA maintenance and embryogenesis in mice. Nat Genet, 1998, 18: 231-236. |
| [3] | Brown TA, Tkachuk AN, Shtengel G, et al. Superresolution fluorescence imaging of mitochondrial nucleoids reveals their spatial range, limits, and membrane interaction. Mol Cell Biol, 2011, 31: 4994-5010. |
| [4] | Shi Y, Dierckx A, Wanrooij PH, et al. Mammalian transcription factor A is a core component of the mitochondrial transcription machinery. Proc Natl Acad Sci U.S.A, 2012, 109: 16510-16515. |
| [5] | Sologub M, Litonin D, Anikin M, et al. TFB2 is a transient component of the catalytic site of the human mitochondrial RNA polymerase. Cell, 2009, 139: 934-944. |
| [6] | Rubio-Cosials A, Sola M. U-turn DNA bending by human mitochondrial transcription factor A. Curr Opin Struct Biol, 2013, 23(1): 6-24. |
| [7] | McCulloch V, Shadel GS. Human mitochondrial transcription factor B1 interacts with the C-terminal activation region of h-mtTFA and stimulates transcription independently of its RNA methyltransferase activity. Mol Cell Biol, 2003, 23: 5816-5824. |
| [8] | Ngo HB, Kaiser JT, Chan DC. Tfam, a mitochondrial transcription and packaging factor, imposes a U-turn on mitochondrial DNA. Nat Struct Mol Biol, 2011, 18: 1290-1296. |
| [9] | Takamatsu C, Umeda S. Ohsato T, et al. Regulation of mitochondrial D-loops by transcription factor A and single-stranded DNA-binding protein. EMBO Rep, 2002, 3(5): 451-456. |
| [10] | Lu B, Lee J, Nie XB, et al. Phosphorylation of human TFAM in mitochondria impairs DNA binding and promotes degradation by the AAA+ Lon protease. Mol Cell, 2013, 49(1): 121-132. |
| [11] | Furukawa R, Yamada Y, Matsushima Y, et al. The manner in which DNA is packaged with TFAM has an impact on transcription activation and inhibition. FEBS Open Bio, 2012, 2: 145-150. |
| [12] | Campbell CT, Kolesar JE, Kaufman BA. Mitochondrial transcription factor A regulates mitochondrial transcription initiation, DNA packaging, and genome copy number. Biochim Biophys Acta, 2012, 1819: 921-929. |
| [13] | Farge G, Laurens N, Broekmans OD, et al. Protein sliding and DNA denaturation are essential for DNA organization by human mitochondrial transcription factor A. Nat Commun, 2012, 3: 1013. |
| [14] | Thomas KJ, McCoy MK, Blackinton J, et al. Dj-1 acts in parallel to the PINK1/parkin pathway to control mitochondrial function and autophagy. Human Molecular Gentics, 2011, 20: 40-50. |
| [15] | Piao Y, Kim HG, Oh MS, et al. Overexpression of TFAM, NRF-1 and myr-AKT protects the MPP(+)-induced mitochondrial dysfunctions in neuronal cells. Biochim Biophys Acta, 2012, 1820(5): 577-585. |
| [16] | Alvarez V, Corao AI, Sánchez-Ferrero E, et al. Mitochondrial transcription factor A (TFAM) gene variation in Parkinson's disease. Neurosci Lett, 2008, 432: 79-82. |
| [17] | Gatt AP, Jones EL, Francis PT, et al. Association of a polymorphism in mitochondrial transcription factor A (TFAM) with Parkinson's disease dementia but not dementia with Lewy bodies. Neurosci Lett, 2013, 12: 557. |
| [18] | Wang KZ, Zhu JH, Dagda R, et al. Mitochondrial Dysfunction Accompanied by ERK-Dependent Phosphorylation of TFAM in a Chronic MPP+ Model. Biophysical J, 2013, 1: 104. |
| [19] | Alvarez V, Corao AI, Alonso-Montes C, et al. Mitochondrial transcription factor A (TFAM) gene variation and risk of late-onset Alzheimer's disease. J Alzheimers Dis, 2008, 13(3): 275-280. |
| [20] | Chaturvedi RK, Adhihetty P, Shukla S, et al. Impaired PGC-1alpha function in muscle in Huntington's disease. Hum Mol Genet, 2009, 18: 3048-3065. |
| [21] | Lin J, Wu PH, Tarr PT, et al. Defects in adaptive energy metabolism with CNS-linked hyperactivity in PGC-1alpha null mice. Cell, 2004, 119: 121-135. |
| [22] | Cui L, Jeong H, Borovecki F, et al. Transcriptional repression of PGC-1alpha by mutant huntingtin leads to mitochondrial dysfunction and neurodegeneration. Cell, 2006, 127: 59-69. |
| [23] | Taherzadeh-Fard E, Saft C, Akkad DA, et al. PGC-1alpha downstream transcription factors NRF-1 and TFAM are genetic modifiers of Huntington disease. Mol Neurodegener, 2011, 6(1): 32. |
| [24] | Tessa A, Manca ML, Mancuso M, et al. Abnormal H-Tfam in a single mtDNA deletion. Funct Neurol, 2000, 15(4): 211-214. |
| [25] | Joseph AM, Runqi AA, Robinson BH, et al. Compensatory responses of potein import and transcription factor expression in mitochondrial DNA defects. Am J Physiol Cell Physiol, 2004, 286(4): C867-C875. |
| [26] | Riley LG, Menezes MJ, Rudinger-Thirion J, et al. Phenotypic variability and identification of novel YARS2 mutations in YARS2 mitochondrial myopathy, lactic acidosis and sideroblastic anaemia. Orphanet J Rare Dis, 2013, 17(8): 193. |
 2014, Vol. 41
2014, Vol. 41