2. 澳门理工大学 应用科学学院, 中国 澳门 999078
2. Faculty of Applied Sciences, Macao Polytechnic University, Macao 999078, China
2022年,中国有超过
在众多医学影像中,结构磁共振成像(structural Magnetic Resonance Imaging, sMRI)能够很好地表征脑组织的解剖结构,专注于识别微妙的大脑结构变化,具有很高的空间分辨率,其获得技术相对便捷,且是非侵入性的,不使用辐射,对人体没有伤害,在AD诊断与MCI进展预测中显示出优秀的临床实践价值[2],因此sMRI已经成为诊断预测AD的主要辅助工具。现有的阿尔茨海默病诊断方法一般需要医生视觉观察受试者sMRI影像以寻找与AD相关的局部脑萎缩区域。然而,这需要大量具备相关经验的影像科医生才能做出正确的诊断。
近年来,有许多研究将结构磁共振成像与机器学习技术相结合用于AD诊断[3-8],这些方法的出现一定程度上可以减轻医生的判读工作量。当前比较流行的机器学习方法如利用SVM构建提取判别性特征的模型,对sMRI图像进行特征分类[4-8]。但是发现随着样本量的增加,这些方法在推理效率以及准确率上无法达到诊断要求。因此,有研究者则利用深度学习模型诊断阿尔兹海默症sMRI图像,以此提高模型构建特征空间的鲁棒性以及推理速度的高效性[9-18],这些模型多基于卷积神经网络(Convolutional Neural Network, CNN) 实现AD分类预测。其中,Liu M等[9]提出一种多实例卷积神经网络LDMIL,自动学习sMRI图像中与疾病相关的标记及周围图像块从而更好识别疾病。Lian C等[10]提出一种分层卷积神经网络H-FCN来学习sMRI中判别性局部斑块和区域并联合多尺度特征来实现分层分类。但是由于卷积核感受野的局部性,CNN很难建模全局依赖关系。而且很多基于卷积神经网络的模型多将3D sMRI切片处理进行训练,这不能很好地学习三维空间的信息和捕捉到更全面的特征。于是,Zhu W等[11]提出一种具有双重注意力的多实例图像块卷积神经网络DA-MIDL,用于捕捉sMRI中细微的局部和全局特征,实现了更优的分类性能。Zhang X等[17]则提出残差注意力网络3D ResAttNet来捕获sMRI图像中的局部、全局和空间信息来提升分类性能,并提出可解释的基于梯度的定位类激活映射图用于可视化模型预测解释。此后,Zhang X等[18]又提出sMRI-PatchNet包含一个可解释图像块定位与选择机制来挑选与AD相关的病理斑块位置,该机制的实现基于从大量医学图像学习的知识,然后通过带有全局空间信息和局部图像信息模块的PatchNet学习图像块之间和图像块内的局部信息,从而提升分类效率。上述方法都是基于卷积神经网络或者结合注意力机制,近年来,基于自注意力机制的Transformer[19]在全局建模上表现出优异的性能,但主要应用在自然语言处理任务中,随着Vision Transformer (ViT)[20]的出现,提出将图像分块,展平成序列输入 Transformer 编码,最后接入一个全连接层执行图像分类任务,将Transformer引入了图像领域。Swin Transformer[21]为了提高计算速度,引入了滑动窗口自注意力机制,实现了并行计算,而且层级式的窗口设计可以捕获图像中不同区域的长范围依赖,提高空间建模能力,此外,Swin Transformer更加灵活,可以增加模型大小和深度来提高性能。因此,想要将Swin Transformer应用到AD的分类预测任务上,但由于基础Swin Transformer是基于2D图像,而AD的sMRI图像为3D,所以需要将Swin Transformer进行维度调整,改进为3D Swin Transformer实现AD分类。
此外,由于深度学习模型在输出数据方面十分严苛,需要手工设计一种合理的输出尺寸使得模型的准确率达到平稳的状态,但是这种设计方式对于实际临床来说是不合理的,因为每个患者sMRI样本的尺寸都是可变,而且每个样本中发生萎缩的区域对尺寸的变换同样敏感。此外,当前这些深度学习模型对于结果可解释性不强,即在病灶区域定位方面能力不强,无法对既定的病理位置如海马体区域或前额区域进行特定的响应输出,从而无法解释该模型对大脑的记忆力区域或者情感区域的关注度是否达到既定标准,无法给予医生一种较直观的模型解释。因此基于上述挑战,本文提出自适应特征提取模块(Adaptive Feature Extraction Module,AFEM)更加精准定位脑萎缩区域,通过调整Transformer模块堆叠数量与特征维度来修改模型结构,建立AFEM-Transformer-Base深度学习模型,相较于基础Transformer模型,AFEM-Transformer-Base模型的灵敏度和准确率得到了大幅提升,再评估该模型在基于sMRI的多项阿尔兹海默症诊断任务中的诊断价值与应用前景。本文的主要贡献如下:
(1) 提出AFEM-Transformer模型,以3D Swin Transformer为主干,融合AFEM模块,实现基于3D sMRI的AD病情诊断和预测。
(2) 提出自适应特征提取模块AFEM,以实现精准脑萎缩区域的定位和病理特征提取。
(3) 实验数据表明本文的模型比卷积神经网络模型和基础Transformer模型,在预测和分类性能上较大幅度提升,验证了本文方法的有效性,对AD病情的自动化辅助预测和诊断更加准确。
1 相关工作 1.1 自适应特征提取计算机辅助医疗诊断领域的关键挑战之一便是让模型自适应地学习病灶区域比例、形状的几何变化。为了定位提取特征和自适应地匹配区域的几何变换,可变形卷积(Deformable ConvNets) 首先被提出,其允许传统卷积中的采样格发生任意形式的变形[22]。近年来,越来越多的方法将可变形的思想融入到Transformer方法中[23-25],其中最具代表的便是Deformable Patch-based Transformer (DPT) [24]。DPT提出的DePatch模块可以根据输入的2D图像自动调整每个被选择的patch的大小和位置,虽然DePatch模块可以根据sMRI扫描或输入特征图自动微调每个选定补丁的比例和位置,但它没有将可变形操作集成到Transformer。相反,Deformable Attention Transformer(DAT) [25]通过引入一组可变形键和值,将可变形操作集成到Transformer主干中。然而,它的可变形注意力缺乏连贯性,因为不同注意力层之间的可变形键和值是在没有结合先前可变形参数的情况下单独学习的。为解决上述问题,本文结合DePatch模块和DPT的特点进行改进,使其支持3D影像输入,并命名为自适应特征提取模块(AFEM)。自适应特征提取模块可以更加精准定位脑萎缩区域。
1.2 基于Transformer的预测本文提出的AFEM-Transformer模型的骨干网络为Swin Transformer,它是由Liu Z等[21]在2021年提出的。Swin Transformer通过移动窗口对分层 Transformer的表征进行计算,并通过将自注意力计算限制在不重叠的局部窗口,在不同的层中允许跨窗口连接使模型可以学习到跨窗口的信息。同时采用下采样的层级设计,逐渐增大感受野使模型可以处理超分辨率的图片且获取到全局和局部的特征,这种分层结构可以灵活地在不同尺度上建模,并使得计算复杂度和图像大小线性相关。这些良好的特性非常适合于医学图像,尤其是超分辨率的MRI图像,图像尺寸大,数据参数量远高于一般图像,因此尽可能降低模型的复杂度是非常有必要的,但是Swin Transformer是针对2D图像设计的,而本文研究使用的sMRI图像是3D影像,因此不能够直接照搬原Swin Transformer结构。于是,根据本文研究的3D sMRI影像的特性和实际情况,对Swin Transformer进行了相应的修改和优化,使它适应3D图像的维度特性,同时保留该模型原有的良好特性,即改进为以3D Swin Transformer为主干网络,加入AFEM。基于此,本文还研究了不同Transformer层深度与特征维度对实验结果的影响。实验结果表明,随着深度和维度的增加,模型的准确率、特异性、敏感性、AUC指标都会随之提高,最终经过对比研究,设计了性能最优的AFEM-Transformer-Base模型。
2 方法 2.1 整体架构本文提出的模型AFEM-Transformer整体架构如图1所示,由3D Swin Transformer骨干网络、自适应特征提取模块和Transformer全局特征提取块组成。AFEM-Transformer的具体实现过程:首先输入大小为
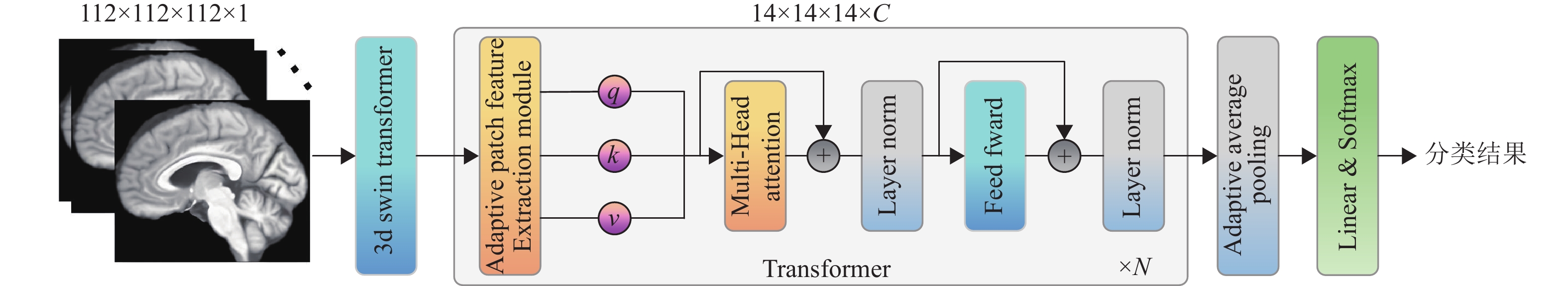
|
图 1 AFEM-Transformer 网络架构 Figure 1 AFEM-Transformer network architecture |
其中,Transformer块中带有
| $ q = x{{\boldsymbol{W}}_q},k = x{{\boldsymbol{W}}_k},v = x{{\boldsymbol{W}}_v} $ | (1) |
| $ {z^{(m) }} = \sigma ({q^{(m) }}{k^{{{(m) }^{\text{T}}}}}/\sqrt d ) {v^{(m) }},m = 1,2,\cdots,M $ | (2) |
| $ z = {\text{Concat}}({z^{(1) }},\cdots,{z^{(M) }}) {W_o} $ | (3) |
式中:
本文整体模型主要是将自适应特征提取模块(AFEM)替换嵌入到Transformer中,建立对应的深度学习模型。本文通过修改骨干网络结构、Transformer堆叠数量N与特征维度C分别建立了AFEM-Transformer-Tiny、AFEM-Transformer-Small、AFEM-Transformer-Base三种不同深度的深度学习模型。其中AFEM-Transformer-Base采用3D Swin-B作为骨干网络,共堆叠了9层Transformer,特征维度为512。而AFEM-Transformer-Tiny、AFEM-Transformer-Small的Transformer层数和特征维度分别为3、6和192、384,分别用3D Swin-T、3D Swin-S作为骨干网络,整体设计见表1。
| 表 1 各模型的基本设置 Table 1 Basic settings of different models |
自适应特征提取模块的结构如图2所示,其主要功能是可变形定位特征块,进行特征提取,核心是将输入特征图中的每个选定块生成位置偏移因子
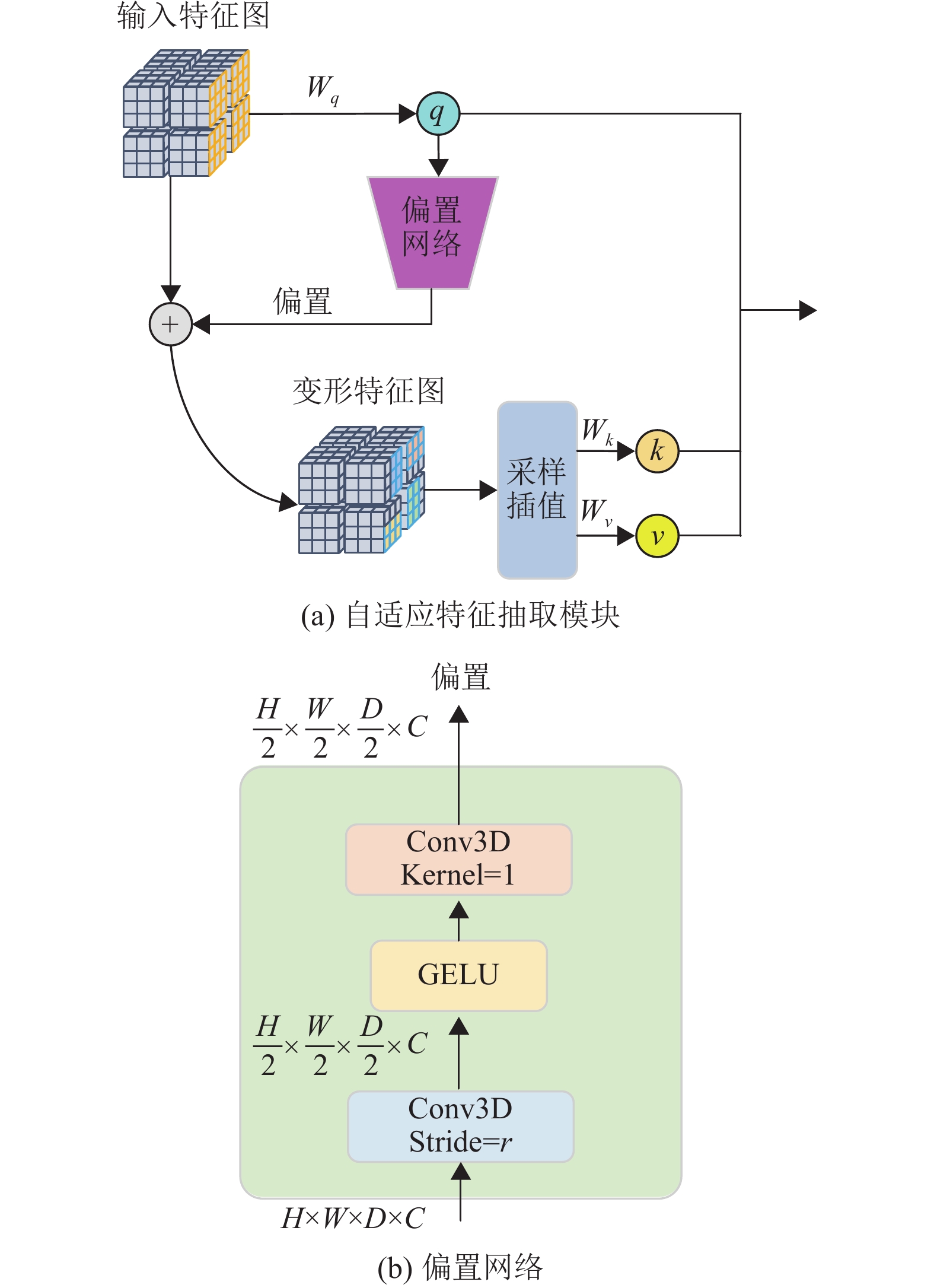
|
图 2 自适应特征提取模块总览图 Figure 2 The overview of AFEM |
| $ q = x{{\boldsymbol{W}}_q},\tilde k = {x'}{{\boldsymbol{W}}_k},\tilde v = {x'}{{\boldsymbol{W}}_v} $ | (4) |
| $ {x'} = \varphi (x;(\Delta l,\Delta s) ) $ | (5) |
式中:
偏置网络
| $ (\Delta l,\Delta s) = ({r_l},{r_s}) {\text{tanh}}({\phi _o}(q) ) $ | (6) |
本文选取2004~2019年之间由阿尔茨海默症神经影像学计划[26]收集并共享的
| 表 2 ADNI数据集的人口学和临床细节1) Table 2 Demographic and clinical details of the ADNI dataset |
本文提出的深度学习模型基于Python3.9和Pytorch1.10实现。所有实验均在单张NVIDIA RTX3090显卡上进行。本文建立的AFEM-Transformer深度学习模型与多种机器模型将在训练集和验证集上完成模型的迭代训练,之后分别在测试集上进行分类结果的预测以测试分类性能。采用准确率(Accuracy, ACC)、敏感性(Sensitivity, SEN)、特异性(Specificity, SPE) 和试者工作特征曲线下面积(AUC) 4个指标来评价模型分类性能。对于二分类问题,可以将模型预测的结果和样本的真实类别的组合划分为真正例(True Positive, TP)、真反例(True Negative, TN)、假正例(False Positive, FP)、假反例(False Negative, FN),其中TP表示模型预测结果为患病且该样本真实患病的被试个体,TN表示模型预测结果为健康且真实健康的被试个体,FP表示模型预测结果为患病但实际健康的被试个体,FN表示模型预测结果为健康但实际患病的被试个体。
Accuracy是正确诊断的受试者数除以所有受试者数量,表示所有样本中预测结果与实际状态相同的样本占总样本的百分比,计算公式为
| $ {\text{Accuracy = }}\frac{{{\text{TP + TN}}}}{{{\text{TP + TN + FP + FN}}}} $ | (7) |
Sensitivity表示所有正例中被诊断为正例的比例,即所有病人中被预测正确的比例用于评估模型对正例的分类性能,体现的是分类模型对患者的识别能力的强弱,计算公式为
| $ {\text{Sensitivity = }}\frac{{{\text{TP}}}}{{{\text{TP + FN}}}} $ | (8) |
Specificity表示的是在所有负例中被诊断为负例的比例,用于评估模型对负例的分类性能,即计算所有健康人中预测正确的所占比例,是衡量模型对正常对照的识别能力,计算公式为
| $ {\text{Specificity = }}\frac{{{\text{TN}}}}{{{\text{TN + FN}}}} $ | (9) |
AUC(Area Under Curve),即ROC曲线下的面积,AUC值越大,代表模型诊断准确性越高。
3.3 AD诊断任务实验结果在AD诊断任务(AD vs NC) 中,将本文提出的AFEM-Transformer模型与3个卷积分类模型(LD-MIL[9], H-FCN[10], DA-MIDL[11]) 和基础Transformer模型从不同深度进行对照,实验结果见表3。其中在相同维度AFEM-Transformer其各项指标分数相比基础的Transformer模型均有着较大幅提升,体现了AFEM-Transformer模型的有效性。此外,值得注意的是,AFEM-Transformer-Base模型比卷积分类模型分类性能好且在AFEM-Transformer中表现最好,这意味着增加模型深度和特征维度后的大型深度学习模型的性能更好,可以更加准确地识别正例,即阿尔茨海默患者。此外,本文还将AFEM-Transformer-Base模型与3D ResAttNet[18]和sMRI-PatchNet[19]进行了简要的AD分类结果比较,见表4,从指标结果可以看出,即使本文使用的实例最多,但是并没有出现过拟合,泛化性能稳定,分类性能在各指标上都表现更好。
| 表 3 不同机器模型在AD诊断任务(AD vs NC) 中的分类性能比较 Table 3 Performance comparison of different models for AD classification (AD vs NC) |
| 表 4 不同机器模型在ADNI数据集AD诊断任务(AD vs NC) 和MCI进展预测任务(pMCI vs sMCI)中的分类性能比较 Table 4 A brief performance comparison of different models for AD classification MCI conversion prediction (pMCI vs sMCI) on the ADNI dataset |
在MCI进展预测任务(pMCI vs sMCI) 中,本文同样将提出的AFEM-Transformer模型同3个卷积分类模型和基础Transformer模型从不同深度进行对照,实验结果见表5。在相同维度上,AFEM-Transformer的实验指标都高于基础Transformer模型,值得注意的是,Transformer-Tiny的指标结果都高于基础Transformer模型所有维度上的结果,体现了AFEM-Transformer的有效性和优越性。此外,AFEM-Transformer-Base模型的准确率为0.823、特异性为0.796、敏感性为0.844、AUC为0.855,指标最高,优于AFEM-Transformer-Tiny和AFEM-Transformer-Small模型,由此可见,随着Backbone和Transformer堆叠层的加深,模型性能也逐渐提高。从与卷积分类模型对比结果可知,AFEM-Transformer-Base分类效果更好,进一步验证了AFEM-Transformer模型的有效性。此外,本文也将AFEM-Transformer-Base模型与3D ResAttNet[18]和sMRI-PatchNet[19]进行了简要的MCI进展预测结果比较,见表4,可以从指标看出,虽然并没有每个指标都达到最优,就准确率和敏感性而言,位于上游水平,可见本文的模型对脑萎缩的微观结构变化捕捉更敏感。
| 表 5 不同机器模型在MCI进展预测任务(pMCI vs sMCI)中的分类性能比较 Table 5 Performance comparison of different models for MCI conversion prediction (pMCI vs sMCI) |
本文的AD诊断和MCI进展预测结果证实加入自适应特征提取模块(AFEM) 的AFEM-Transformer深度学习模型对比基础Transformer模型在同规模的模型深度上,各项性能指标均有大幅提升,见表3、表5。为了进一步验证自适应特征提取模块定位脑萎缩区域的能力,将自适应特征提取模块定位的病灶区域通过不同视图可视化投射到sMRI影像中,见图3,用于可视化验证AFEM的有效性。其中不同颜色的颗粒代表不同病灶,同一颜色颗粒聚集在一块区域,用于区分萎缩范围,定位到不同区域的病灶区域,并聚焦注意力,可以辅助模型更好地提取有用特征,实现更精准的分类预测,一定程度上也体现了Transformer在临床医学应用中的可解释性问题。

|
图 3 自适应特征提取模块定位的病灶区域情况 Figure 3 Location of the lesion areas by using adaptive feature extraction module |
针对AD患者的sMRI样本的尺寸都是可变的,而且每个样本中发生萎缩的区域对尺寸的变换同样敏感,但当前这些深度学习模型对于结果的可解释性不强,即在病灶区域定位方面能力不强的问题,本文提出自适应特征提取模块AFEM用于自适应地学习病灶区域比例、形状的几何变化,精准定位脑萎缩区域实现自适应特征提取,同时将AFEM模块融入3D Swin Transformer模型,设计AFEM-Transformer模型用于实现基于3D sMRI 影像的AD分类和病情进展预测。实验结果表明,相较卷积神经网络模型,AFEM-Transformer-Base展现出良好优越性,相较于同层次基础Transformer模型,本文的AFEM-Transformer模型在多项阿尔兹海默症诊断中都展现出更优秀的性能,验证了AFEM-Transformer模型的有效性,对比实验确定最优性能AFEM-Transformer-Base模型。AFEM-Transformer模型可以用于初步定位出sMRI影像中的病灶区域以辅助医生做出临床诊,具有重要的诊断价值与应用前景。
本研究的不足主要在于:(1) 随着模型深度的加深,该深度学习模型的参数量也随之上升,对于计算机计算资源的要求也进一步提高,使得该方法难以大范围普及。未来的研究可以考虑引入重参数化等轻量级结构[28],在保证模型精度不下降的前提下,进一步减少模型参数量,降低计算资源的消耗。(2) 除了sMRI之外,正电子断层扫描(Positron Emission Computed Tomography, PET) 和弥散张量成像(Diffusion Tensor Imaging, DTI) 也是AD诊断和MCI预测中的重要临床依据[29-30]。后续研究可以联合sMRI、PET和DTI等影像技术,实现基于多模态的阿尔兹海默症诊断。
| [1] |
SCHELTENS P, DE STROOPER B, KIVIPELTO M, et al. Alzheimer's disease[J].
Lancet, 2021, 397(10284): 1577-1590.
DOI: 10.1016/S0140-6736(20)32205-4. |
| [2] |
LEMPRIÈRE S. New tools could improve Alzheimer disease diagnosis from structural MRI[J].
Nature Reviews Neurology, 2020, 16(6): 297-297.
DOI: 10.1038/s41582-020-0357-z. |
| [3] |
ANTONAKAKIS M, DIMITRIADIS S I, ZERVAKIS M, et al. Aberrant whole-brain transitions and dynamics of spontaneous network microstates in mild traumatic brain injury[J].
Frontiers in Computational Neuroscience, 2019, 13: 90-90.
|
| [4] |
SUDHARSAN M, THAILAMBAL G. Alzheimer's disease prediction using machine learning techniques and principal component analysis (PCA)[J].
Materials Today: Proceedings, 2023, 81: 182-190.
DOI: 10.1016/j.matpr.2021.03.061. |
| [5] |
DOU X, YAO H, FENG F, et al. Characterizing white matter connectivity in Alzheimer's disease and mild cognitive impairment: an automated fiber quantification analysis with two independent datasets[J].
Cortex, 2020, 129: 390-405.
DOI: 10.1016/j.cortex.2020.03.032. |
| [6] |
罗刚, 泮思林, 乔思波, 等. 深度学习技术在胎儿超声心动图图像自动识别中的应用[J].
实用医学杂志, 2022, 38(14): 1830-1833.
LUO G, PAN S L, QIAO S B, et al. Deep learning technology for automatic recognition of fetal echocardiography images[J]. The Journal of Practical Medicine, 2022, 38(14): 1830-1833. DOI: 10.3969/j.issn.1006-5725.2022.14.022. |
| [7] |
杨磊, 唐灿. 人工智能在乳腺癌超声诊断的应用价值[J].
实用医学杂志, 2022, 38(1): 106-110.
YANG L, TANG C. CAD machine diagnosis system in ultrasonic diagnosis of breast cancer[J]. The Journal of Practical Medicine, 2022, 38(1): 106-110. DOI: 10.3969/j.issn.1006-5725.2022.01.020. |
| [8] |
廖淑婷, 于向荣. 能谱CT和人工智能在甲状腺癌诊断中的应用[J].
实用医学杂志, 2022, 38(2): 129-133.
LIAO S T, YU X R. Application of spectral CT and artificial intelligence in the diagnosis of thyroid cancer[J]. The Journal of Practical Medicine, 2022, 38(2): 129-133. DOI: 10.3969/j.issn.1006⁃5725.2022.02.001. |
| [9] |
LIU M, ZHANG J, ADELI E, et al. Landmark-based deep multi-instance learning for brain disease diagnosis[J].
Medical Image Analysis, 2018, 43: 157-168.
DOI: 10.1016/j.media.2017.10.005. |
| [10] |
LIAN C, LIU M, ZHANG J, et al. Hierarchical fully convolutional network for joint atrophy localization and Alzheimer’s disease diagnosis using structural mri[J].
IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 42(4): 880-893.
|
| [11] |
ZHU W, SUN L, HUANG J, et al. Dual attention multi-instance deep learning for Alzheimer’s disease diagnosis with structural MRI[J].
IEEE Transactions on Medical Imaging, 2021, 40(9): 2354-2366.
DOI: 10.1109/TMI.2021.3077079. |
| [12] |
曾安, 黄殷, 潘丹, 等. 基于卷积循环神经网络的阿尔茨海默症早期诊断[J].
生物医学工程研究, 2020, 39(3): 249-255.
ZENG A, HUANG Y, PAN D, et al. Early diagnosis of Alzheimer′s disease based on convolutional recurrent neural network[J]. Journal of Biomedical Engineering Research, 2020, 39(3): 249-255. |
| [13] |
王聪, 袁榕澳, 李川. 基于3D卷积神经网络的阿尔茨海默症及轻度认知功能障碍诊断[J].
现代计算机, 2021(11): 120-123.
WANG C, YUAN R A, LI C. Analysis of Alzheimer’s disease medical images based on deep machine learning[J]. Modern Computer, 2021(11): 120-123. DOI: 10.3969/j.issn.1007-1423.2021.11.024. |
| [14] |
QIN Z, LIU Z, GUO Q, et al. 3D convolutional neural networks with hybrid attention mechanism for early diagnosis of Alzheimer’s disease[J].
Biomedical Signal Processing and Control, 2022, 77: 103828.
DOI: 10.1016/j.bspc.2022.103828. |
| [15] |
ZHANG J, ZHOU L, WANG L, et al. Diffusion kernel attention network for brain disorder classification[J].
IEEE Transactions on Medical Imaging, 2022, 41(10): 2814-2827.
DOI: 10.1109/TMI.2022.3170701. |
| [16] |
石磊, 彭少康, 张亚萌, 等. 基于特征增强金字塔网络的阿尔茨海默症早期诊断研究[J].
数据采集与处理, 2022, 37(4): 727-735.
SHI L, PENG S K, ZHANG Y M, et al. Early diagnosis of Alzheimer’s disease based on feature enhanced pyramid network[J]. Journal of Data Acquisition and Processing, 2022, 37(4): 727-735. |
| [17] |
ZHANG X, HAN L, ZHU W, et al. An explainable 3D residual self-attention deep neural network for joint atrophy localization and Alzheimer’s disease diagnosis using structural MRI[J].
IEEE Journal of Biomedical and Health Informatics, 2021, 26(11): 5289-5297.
|
| [18] |
ZHANG X, HAN L, HAN L, et al. sMRI-PatchNet: A novel efficient explainable patch-based deep learning network for Alzheimer’s disease diagnosis with structural MRI[J].
IEEE Access, 2023, 11: 108603-108616.
DOI: 10.1109/ACCESS.2023.3321220. |
| [19] |
VASWANI A, SHAZEER N, PARMAR N, et al. Attention is all you need[C]//Proceedings of the 31st International Conference on Neural Information Processing Systems (NeurIPS). New York: ACM, 2017: 6000-6010.
|
| [20] |
DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16×16 words: transformers for image recognition at scale[EB/OL]. arXiv: 2010.11929 (2021-01-03) [2024-01-25]. https://doi.org/10.48550/arXiv.2010.11929
|
| [21] |
LIU Z, LIN Y, CAO Y, et al. Swin transformer: hierarchical vision transformer using shifted windows[C]//Proceedings of the IEEE/CVF International Conference on Computer Vision (ICCV) . New York: IEEE, 2021: 9992-10002.
|
| [22] |
ZHU X, HU H, LIN S, et al. Deformable ConvNets V2: more deformable, better results[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR) . New York: IEEE, 2019: 9300-9308.
|
| [23] |
ZHU X, SU W, LU L, et al. Deformable detr: deformable transformers for end-to-end object detection[EB/OL]. arXiv: 2010.04159 (2021-03-28) [2024-01-25]. https://doi.org/10.48550/arXiv.2010. 04159
|
| [24] |
CHEN Z, ZHU Y, ZHAO C, et al. DPT: deformable patch-based transformer for visual recognition[C]//Proceedings of the 29th ACM International Conference on Multimedia. New York: Association for Computing Machinery, 2021: 2899-2907.
|
| [25] |
XIA Z, PAN X, SONG S, et al. Vision transformer with deformable attention[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Los Alamitos: IEEE, 2022: 4784-4793.
|
| [26] |
JACK J C R, BERNSTEIN M A, FOX N C, et al. The Alzheimer's disease neuroimaging initiative (ADNI) : MRI methods[J].
Journal of Magnetic Resonance Imaging, 2008, 27(4): 685-691.
|
| [27] |
PENNY W D, FRISTON K J, ASHBURNER J T, et al. Statistical parametric mapping: the analysis of functional brain images[M]. Amsterdam: Academic Press, 2007: 1-647.
|
| [28] |
DING X, ZHANG X, MA N, et al. RepVGG: Making VGG-style convnets great again[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Nashville: IEEE, 2021: 13728-13737.
|
| [29] |
PAN X, PHAN T L, ADEL M, et al. Multi-view separable pyramid network for AD prediction at MCI stage by 18 F-FDG brain PET imaging[J].
IEEE Transactions on Medical Imaging, 2020, 40(1): 81-92.
|
| [30] |
HUANG J, ZHOU L, WANG L, et al. Attention-diffusion-bilinear neural network for brain network analysis[J].
IEEE Transactions on Medical Imaging, 2020, 39(7): 2541-2552.
DOI: 10.1109/TMI.2020.2973650. |
 2025, Vol. 42
2025, Vol. 42

