2. 广东工业大学 信息工程学院, 广东 广州 510006
2. School of Information Engineering, Guangdong University of Technology, Guangzhou 510006, China
肺癌是当前我国乃至全球范围内发病率和死亡率最高的恶性肿瘤之一[1-2]。非小细胞肺癌(Non-small Cell Lung Cancer, NSCLC) 约占所有肺癌病例的85%,主要包括肺腺癌(Lung Adenocarcinoma, LUAD) 和肺鳞状细胞癌(Lung Squamous Cell Carcinoma, LUSC) 两大组织病理亚型[3-4]。不同亚型在临床上对应不同的治疗方案,对不同亚型的精确鉴别对指导患者的靶向治疗和预后评估意义重大。对活检组织的全视野数字病理图像(Whole Slide Images, WSIs) 的组织病理形态分析是临床上诊断肺癌的“金标准”。然而,在超高像素的WSIs上进行人工阅片和诊断是一项极其耗时、耗力的工作[5]。
近年来,基于深度学习的计算机辅助诊断技术(Computer Aided Diagnosis, CAD) 得到了广泛的研究,通过自动化的WSIs分析可以辅助病理医生提高诊断的准确性和效率。最近的研究提出结合多实例学习(Multiple Instance Learning, MIL) 可以仅在诊断级别的注释监督下处理WSIs。传统的MIL方法多基于单一分辨率的切片图像,但是考虑到NSCLC的肿瘤异质性,单一尺度的信息往往难以充分代表病理切片的细节,可能导致判断结果偏差。并且现有特征提取方法大都使用卷积神经网络(Convolutional Neural Networks, CNNs),其卷积运算的局部感受野限制了网络对远端像素之间的远程依赖关系进行建模的能力,这对于具有巨大异质性的癌症类型鉴别至关重要。
为了提高网络对WSIs整体病理特征的建模能力,有工作开始关注整合多尺度信息的端到端框架以及新型的特征提取网络Transformer[6]。具体来说,一些工作[7-8]采用了多分辨率的切片图像作为模型输入,得到表达细胞形态或组织结构等不同粒度信息的特征向量。然而,这类方法通常对不同尺度特征进行简单的拼接或者叠加,缺乏对不同尺度特征之间语义关联和依赖关系的建模,没有充分挖掘和利用多尺度信息的潜力。另外,Transformer 最近在自然语言处理领域取得了巨大成功,它在扩大感受野和捕捉序列数据中的长期依赖关系方面展现出了卓越的能力[6]。研究表明,将图像分割成小图像块作为输入序列数据,可以完全取代CNNs中的标准卷积,即Vision Transformer (ViT) [9],其在WSIs分析中展示了巨大的潜力。通过扩大感受野,可以捕捉更大范围内的上下文信息,覆盖不同区域的形态学变化,有助于减小类内差异;而建立像素之间的长程依赖关系,则可以挖掘不同区域之间的潜在关联和互补性,突出整体的语义特征,进而拉大类间差异。
尽管如此,由于NSCLC的肿瘤异质性,现有方法亚型分类性能仍然难以满足临床应用需求。LUAD和LUSC这两种亚型虽然各有差异,但它们同属肺癌,所以形态上有一定的相似度,如图1(a) 左1和图1(b) 右1所示,容易产生混淆,产生病理误判。两种亚型的细胞形态学特征可能比较接近,导致区分困难。同时,每种亚型内部也存在较大的异质性,不同区域或细胞亚群之间的形态学和分子特征可能有所差异,临床上可能会增加误诊风险,延误患者的最佳治疗时机[10]。这些因素的共同作用,使得LUAD和LUSC的准确区分成为一个富有挑战性的任务。临床上,病理学家需要综合考虑多种形态学特征和辅助检查结果,才能做出恰当的诊断。而对于计算机辅助诊断系统来说,如何有效地提取和融合多尺度、多模态的特征信息,构建鲁棒和可解释的分类模型,是目前亟待解决的关键问题。
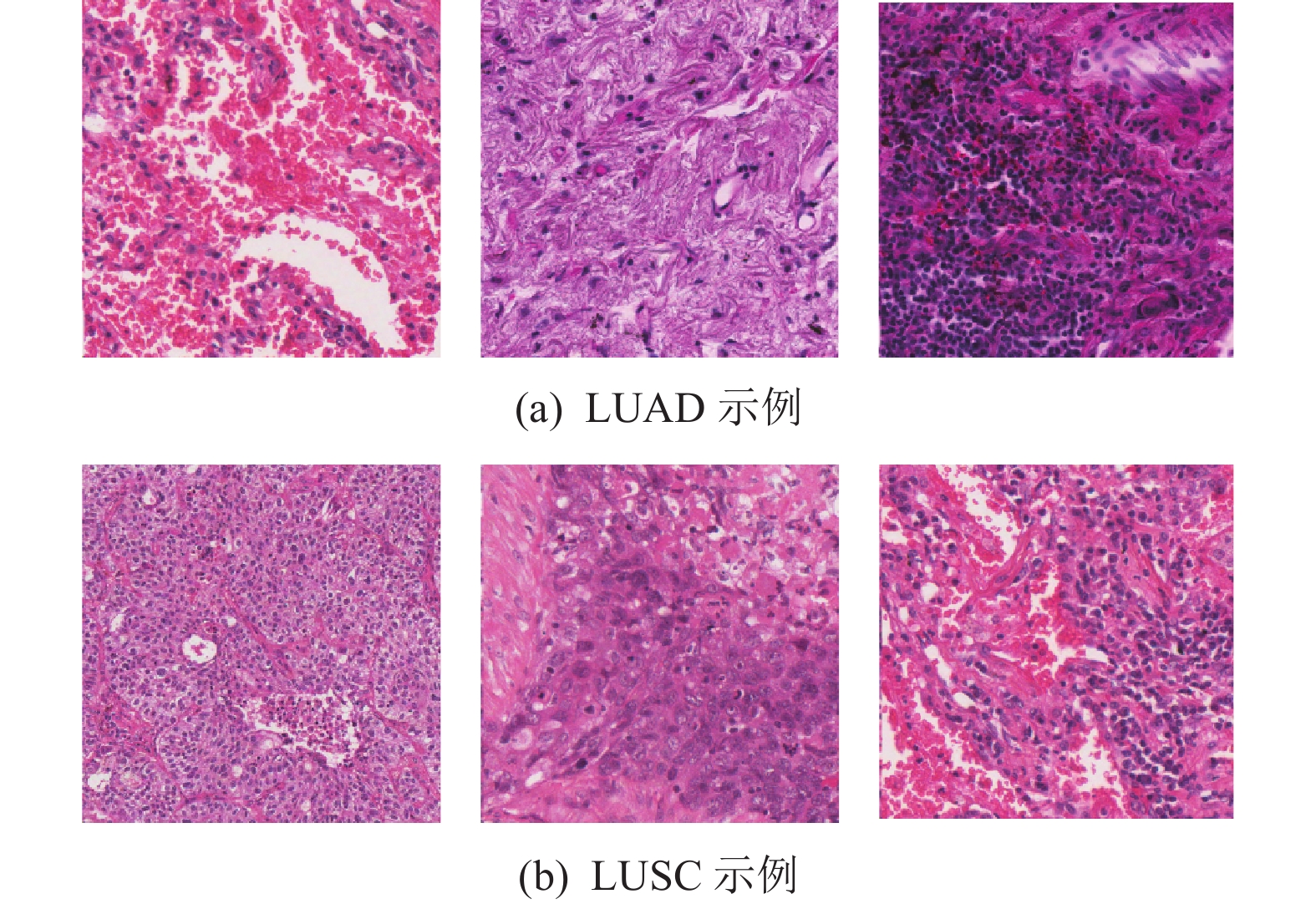
|
图 1 非小细胞肺癌亚型在20倍率下的组织病理结构示例 Figure 1 Examples of histopathological structures of the NSCLC subtypes at the 20 magnification |
为了解决上述问题,本文提出了一个结合了多尺度特征和ViT的多实例学习框架(MCBAT-MIL),用于对NSCLC两种主要亚型LUAD和LUSC进行端到端的分类。具体来说:
(1) MCBAT-MIL在多实例学习框架下利用ViT的自注意力机制对各自尺度下提取到的不同实例之间的关联性信息进行建模,提取更具鉴别性的特征,以应对NSCLC的肿瘤异质性。
(2) 为了捕捉不同尺度之间的内在关联,本文还设计了一个基于ViT的多尺度特征融合模块,模拟病理学家以不同的倍率放大WSIs以检查可疑区域和病理学细节的工作流程,进一步提升模型对NSCLC亚型的分类与识别能力。
(3) 最后,本文在包级损失的基础上增加了基于注意力的实例级损失,为模型提供除包标签外的额外监督信号,并显式地引导模型学习更有区分性的实例特征表示,使模型更好地识别这些关键实例,从而提高整体分类效果。
1 相关工作NSCLC亚型分类的计算机辅助诊断方法可以分为三类:基于人工设计特征的传统方法、基于像素级精确注释的强监督方法和只需要切片级标签的弱监督方法。
(1) 传统方法:传统机器学习方法经常结合计算机视觉和图像处理技术,从组织病理学图像中提取经过人工设计的特征,如形态、纹理或颜色特征等,进而应用这些特征来执行NSCLC亚型分类。例如文献[11]使用EfficientNet和灰度共生矩阵分别提取NSCLC的深度特征和纹理特征,然后融合在一起完成非小细胞肺癌的分型。文献[12]从肺癌 WSIs 中提取
(2) 强监督方法:强监督信号下的NSCLC亚型分类方法通常使用基于深度学习的技术,不仅可以自动从图像中提取抽象特征,还可以更有效地捕获复杂的图像模式,为精确分类提供了新的可能性。例如,文献[3]使用Inception V3训练肺癌WSI,实现对LUAD、LUSC和正常肺组织的自动分类。文献[13]中使用了ResNet34、ResNet50、DenseNet161等6种不同的分类模型完成肺鳞癌、肺腺癌以及小细胞癌等3种肺癌亚型分类工作。尽管上述基于 CNN 的方法已经取得了令人满意的性能,但这些方法通常需要对病理图像进行细粒度的像素级注释,而像素级注释耗时耗力,成本昂贵,限制了其在临床上的应用。
(3) 弱监督方法:为了减轻注释负担,最新的研究利用弱监督多实例学习方法,仅需要切片级标签训练模型。例如,文献[14]提出了AB-MIL,在多实例学习的实例池化步骤中引入了注意力机制,通过加权特征聚合来突出WSI分类中最具辨别力的图块。文献[15]在文献[14]的基础上加入了额外的聚类损失以提高性能,提出了CLAM系列方法。然而,由于NSCLC组织的异质性,MIL模型捕捉亚型之间的鉴别性差异具有挑战性。文献[16]利用RNN对实例特征进行聚合,提出了RNNMIL方法。文献[17]将Transformer引入MIL框架,通过自注意力机制建模实例之间的关系,提出了TransMIL方法。此外,一些研究探索了多尺度特征在MIL中的应用。例如文献[7]提出了DSMIL方法,通过融合不同尺度下提取的特征来增强对肿瘤异质性的建模能力。文献[18]提出了多尺度图Transformer模型MG-Trans,通过在输入和特征空间中减少冗余,并充分建模实例之间的空间关系。
然而,由于NSCLC组织的异质性,现有的MIL模型在捕捉亚型之间的鉴别性差异方面仍然具有挑战性。本文提出的MCBAT-MIL方法旨在解决这一问题,主要有以下改进:(1) 引入了多尺度特征表示,通过融合不同尺度下的细粒度和全局特征,更好地建模肿瘤异质性;(2) 设计了多尺度瓶颈注意力Transformer模块,通过跨尺度的自注意力机制,充分挖掘不同尺度特征之间的互补性和关联性,实现更有效的多尺度特征融合;(3) 在标准的MIL分类损失的基础上,引入了实例级别的对比学习损失,提供额外的细粒度监督信号,进一步提升模型的区分能力。综合以上改进,MCBAT-MIL可以更好地应对NSCLC亚型分类中的异质性挑战,取得了优于现有方法的性能。
2 MCBAT-MIL鉴于WSIs的超高像素,精细的像素级注释难以获得,因此本文使用多实例学习框架,仅使用切片诊断级别的标签即可完成端到端的亚型分类。如图2所示,病理切片实际上是以金字塔形式组织和保存的,包含有多分辨率的不同级别特征,病理学家在实际诊断过程中会从组织器官到细胞到分子多层次地观察病变,对病理诊断意义重大。因此,本文考虑使用5倍和20倍下的多尺度特征,更充分地利用从细胞到组织不同层次的信息,提升判断的准确性和鲁棒性。
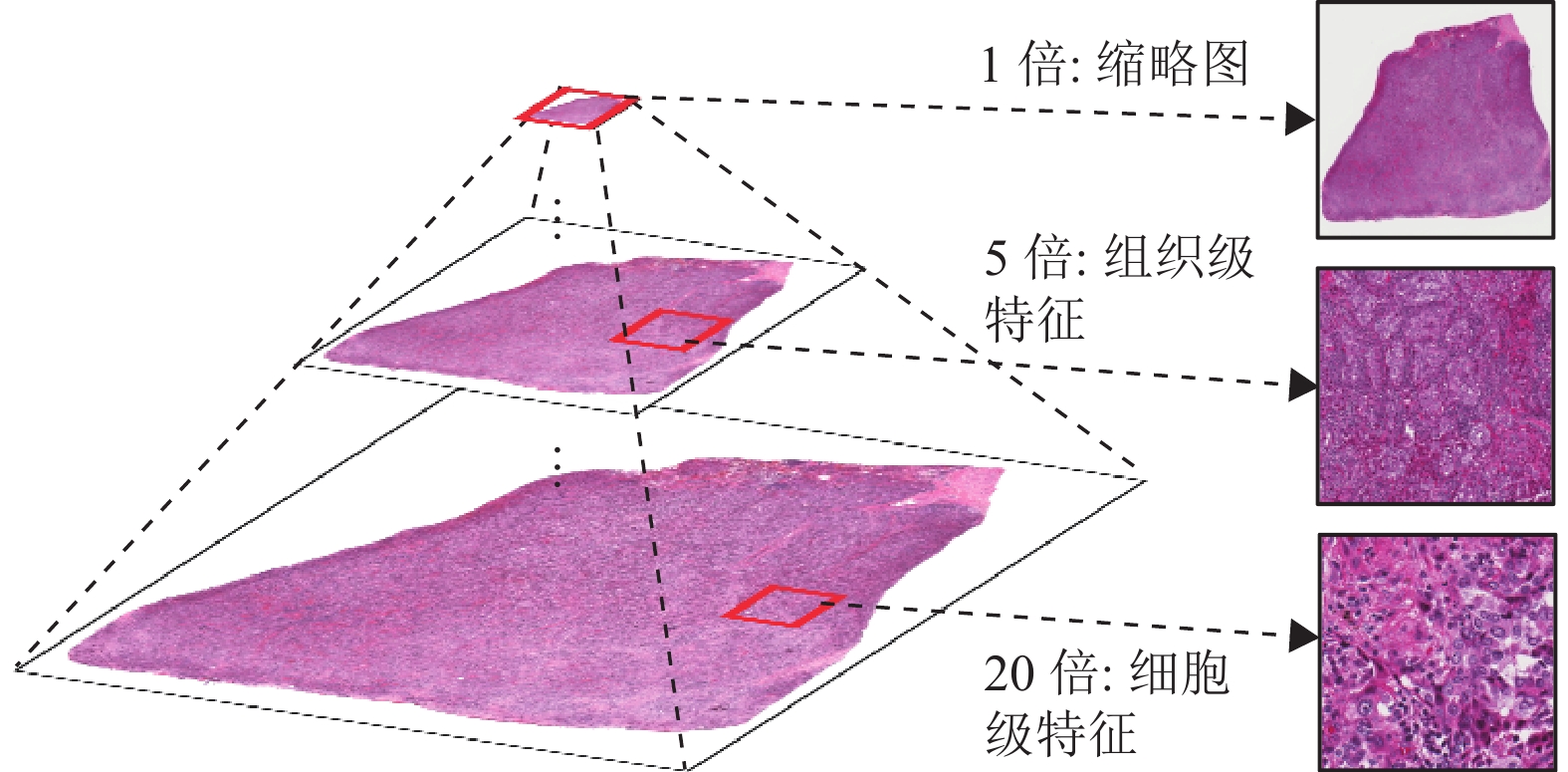
|
图 2 WSI的金字塔型存储模式 Figure 2 Pyramid storage mode of the WSI |
另外,ViT 中的自注意力支持在序列中较远的位置之间建立远程依赖关系,这有利于更好地理解全局上下文线索,例如WSIs的背景和结构。并且ViT 架构的灵活性允许适应不同的输入大小和数据类型,使其非常适合对WSI中固有的多分辨率特性进行建模。因此,本文结合ViT的上下文建模能力和多尺度特征在不同粒度级别表示NSCLC病理学特征的能力,通过整合细胞级别和组织级别的语义信息以及全局上下文线索,提高对NSCLC的细粒度和整体层面的理解,以应对其亚型内差异大和亚型间差异小的问题。
为了进一步利用实例级别的局部特征以及提高模型的可解释性,本文增加了一项基于注意力的实例级损失,为模型提供额外的实例级监督信号,以实现准确的亚型预测。基于以上思路,搭建的MCBAT-MIL总体框架如图3所示,主要包括数据预处理和亚型分类两个部分。
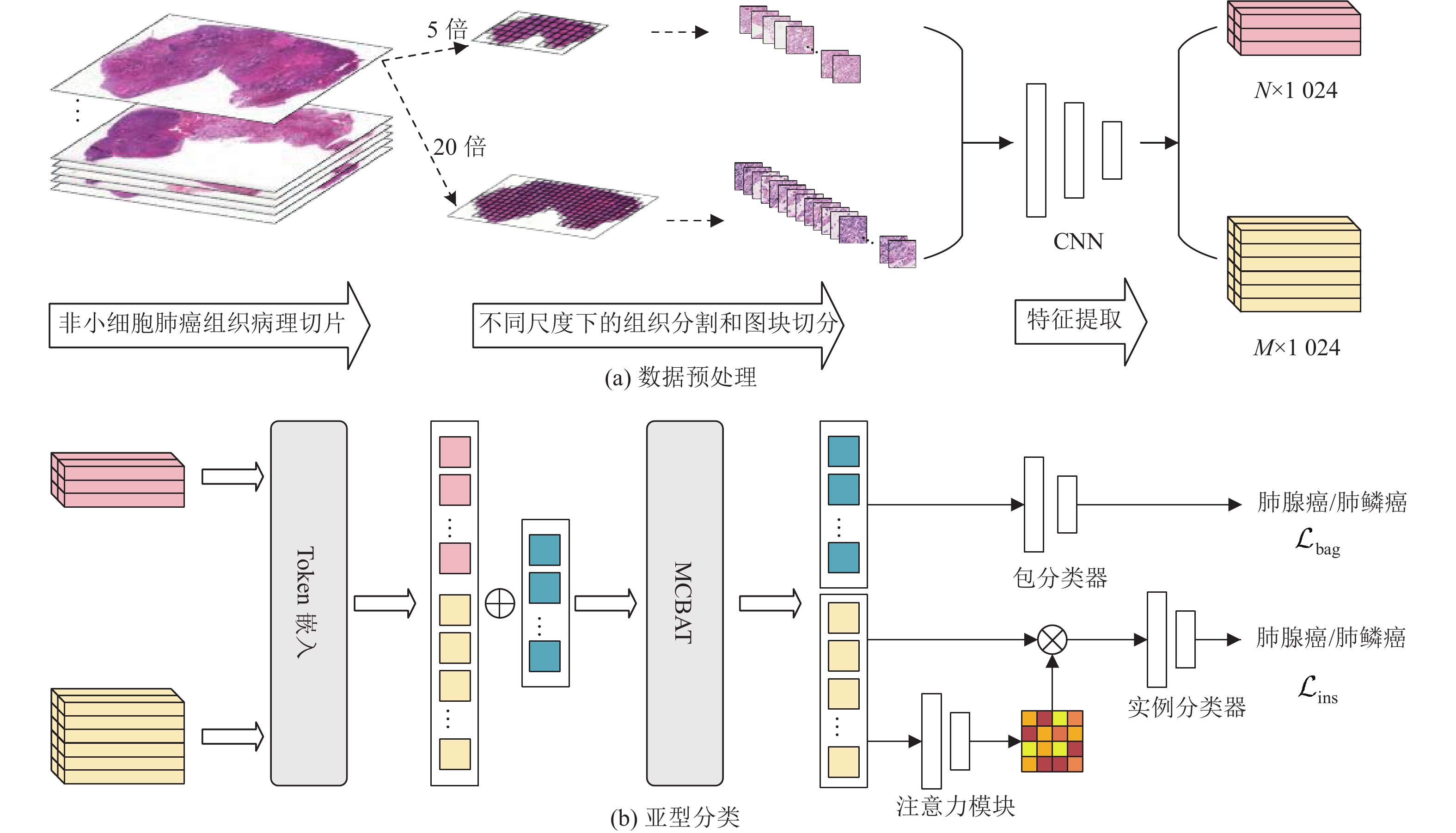
|
图 3 MCBAT-MIL网络架构的示意图 Figure 3 MCBAT-MIL network architecture |
由于肺癌WSIs图像分辨率极高,一张切片扫描文件体积往往达到GB甚至TB量级,直接作为网络输入将导致模型计算量巨大且训练难以收敛。因此,实际应用中常采用分割与采样策略,提取切片中的局部区域作为网络的输入,如图3(a)所示。
具体地,给定训练数据集
NSCLC亚型分类的总体框架如图3(b)所示,在预处理过程中,网络从多分辨率切片中分别采样了表达不同病理粒度(细胞级、组织级) 的图像块,并经过CNN提取对应的视觉特征。受启发于多模态学习中使用的瓶颈注意力Transfromer[22],本文添加额外的瓶颈tokens通过自注意力计算整合不同尺度的序列特征,并通过全连接层输出分类结果。这种token设计允许Transformers端到端的构建多尺度特征,是实现高效联合建模与融合的关键。
2.2.1 Token嵌入首先,模型使用线性投影层E将各维度图像块的CNN特征映射为适应输入维度的向量序列
| $ {\boldsymbol{T}}_{i,N}^{\mathrm{l}},{\boldsymbol{T}}_{i,M}^{\mathrm{h}} = E({\boldsymbol{X}}_{i,N}^{\mathrm{l}},{\boldsymbol{X}}_{i,M}^{\mathrm{l}}) $ | (1) |
如图4所示,本文使用基于瓶颈注意力的ViT,利用瓶颈tokens分别从5倍和20倍尺度下提取并融合组织级和细胞级特征,有效地建模全局上下文,获得对亚型分类任务更加敏感的鉴别性特征。
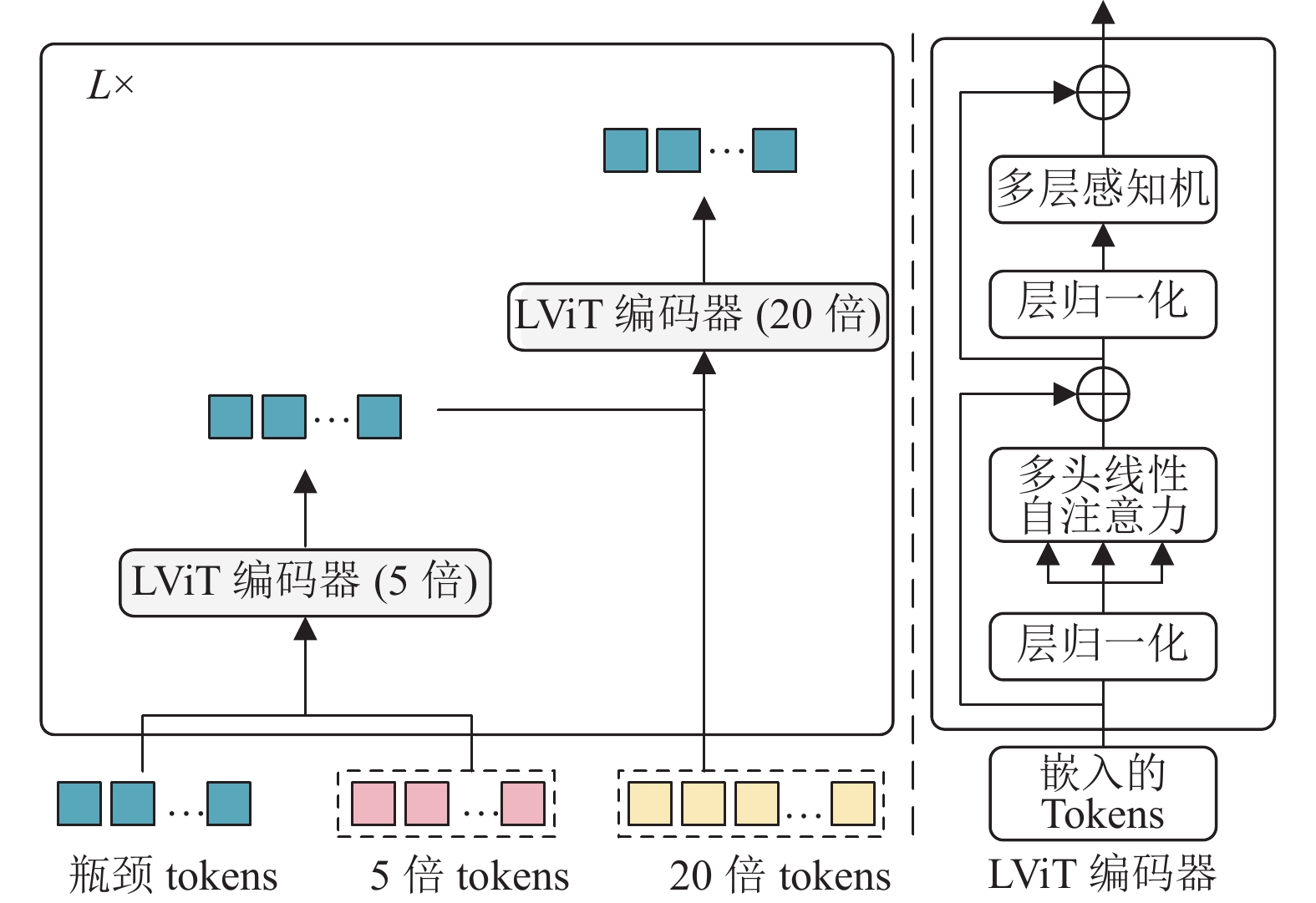
|
图 4 MCBAT结构示意图 Figure 4 Schematic structure of MCBAT |
首先,本文初始化B个瓶颈tokens,
| $ {\boldsymbol{T}}_B^{(L) } = {\text{MCBA}}{{\text{T}}^{(L) }}\left( {{{\boldsymbol{T}}_B},{\boldsymbol{T}}_{i,N}^{\mathrm{l}},{\boldsymbol{T}}_{i,M}^{\mathrm{h}}} \right) $ | (2) |
式中:L表示MCBAT的层数,其中每一层包括2个不同参数的轻量级的ViT编码器(Linear ViT,LViT)。为了降低计算和内存成本,LViT中使用文献[23]中的线性自注意力通过低秩矩阵分解来近似原始的注意力运算,大幅减少了计算量和参数量。除此以外,LViT编码器的其他模块,如前馈网络、层归一化等与原始ViT结构保持一致。如式(3)~(4)所示。
| $ {\boldsymbol{T}}_B^{{\text{mid}}} = {\text{LVi}}{{\text{T}}^{\mathrm{l}}}\left( {[{{\boldsymbol{T}}_B},{\boldsymbol{T}}_{i,N}^{\mathrm{l}}],{\theta ^{\mathrm{l}}}} \right) $ | (3) |
| $ {\boldsymbol{T}}_B^{} = {\text{LVi}}{{\text{T}}^{\mathrm{h}}}\left( {[{\boldsymbol{T}}_B^{{\text{mid}}},{\boldsymbol{T}}_{i,M}^{\mathrm{h}}],{\theta ^{\mathrm{h}}}} \right) $ | (4) |
式中:
在这一节,本文将得到的融合特征对应的融合token
| $ {\mathcal{L}_{ {\mathrm{bag}}}} = - \sum\limits_{i = 1}^W {{{{y}}_i}\ln ({p_i}) } $ | (5) |
式中:
由于20倍率下实例数量多且包含更加精细的特征,本文实例级损失仅在20倍特征
| $ {\mathcal{L}_{ {\mathrm{ins}}}} = - \sum\limits_{j = 1}^{2k} {{y_j}\ln ({p_j}) } $ | (6) |
式中:
最后的总损失函数
| $ {\mathcal{L}_{ {\mathrm{total}}}} = {\mathcal{L}_{ {\mathrm{bag}}}}\lambda + {\mathcal{L}_{ {\mathrm{ins}}}}(1 - \lambda ) $ | (7) |
式中:
为了验证MCBAT-MIL模型在NSCLC亚型分类任务上的性能,本文收集了2个大型公开可得的肺癌病理图像数据集TCGA-LUNG和CPTAC-LUNG,2个数据集都只有亚型类别的0/1标签。TCGA-LUNG来自癌症基因组图谱(The Cancer Genome Atlas, TCGA) 的2个项目TCGA-LUAD和TCGA-LUSC,分别包含了413张LUAD和405张LUSC,共818张NSCLC病理切片图像,相关的数据和标签可以通过美国国立卫生研究院的基因组数据共享门户网站(https://portal.gdc.cancer.gov)获取。CPTAC-LUNG数据集源自美国国立癌症研究所临床肿瘤蛋白质组学分析联盟(Clinical Proteomic Tumor Analysis Consortium,CPTAC) 的项目CPTAC-LUAD和CPTAC-LSCC,包括437张LUAD和419张LUSC共856张NSCLC病理切片图像,数据和标签可以从美国国立卫生研究院的癌症成像档案(https://cancerimagingarchive.net/datascope/cptac)获取。本文按照8:1:1的比例将2个数据集分为训练集、验证集和测试集。此外,本文采用了五折交叉验证的方法来确保模型评估的严谨性和结果的可靠性。
3.2 实验设置本文的实验在配备了一张显存为24 GB的RTX3090Ti GPU上进行。模型使用Pytorch 1.9框架和python3.9搭建。在训练过程中,损失函数使用交叉熵损失,学习率初始化为
本文采用分类任务中常用的度量指标衡量模型亚型分类的性能,包括:接受操作特征曲线下面积(AUC)、准确度(Accuracy)、召回率(Recall) 以及精确率(Precision),指标值越接近于1,模型的分类性能越好。
3.3 分类结果与分析为了验证MCBAT-MIL亚型分类的性能,本文在TCGA-LUNG和CPTAC-LUNG数据集上对比了包括传统方法和最先进算法的多种MIL方法,结果以平均值(方差)的百分比形式报告,如表1所示。本文比较了以下方法:基于最大池化的Maxpooling-MIL,基于RNN聚合的RNNMIL[16],基于注意力机制的AB-MIL[14]和CLAM系列(CLAM-SB和CLAM-MB)[15],基于Transformer的TransMIL[17]以及基于多尺度特征的DSMIL[7]和MG-Trans[18]。
| 表 1 TCGA-LUNG和CPTAC-LUNG数据集上各方法的亚型分类性能 Table 1 Subtype classification performance obtained by different methods on the TCGA-LUNG and CPTAC-LUNG datasets |
表1中的数据显示,使用简单的最大池化操作的Maxpooling-MIL在两个数据集中的分类性能最差。RNNMIL的表现有所提升,但仍远落后于基于注意力的方法。注意力机制和Transformer结构的引入使得AB-MIL和TransMIL的性能进一步提高。例如,AB-MIL在TCGA-LUNG上的AUC达到了89.76%,而TransMIL的AUC为91.05%。基于多尺度特征的DSMIL方法在AUC上达到了92.71%,在CPTAC-LUNG数据集上的准确率为91.09%,显示了多尺度策略的有效性。MG-Trans方法同样采用了多尺度策略,并在TCGA-LUNG数据集上取得了94.12%的AUC和88.42%的准确率,进一步验证了多尺度特征的重要性。最终,本文的MCBAT-MIL显示出了最佳的综合分类性能,在TCGA-LUNG数据集上的AUC为95.09%,准确率为90.69%,精确率为90.00%。在CPTAC-LUNG数据集上,MCBAT-MIL在准确率、精确率和AUC指标上同样表现最好,显示出了对LUAD和LUSC强大的鉴别能力。这一结果体现了MCBAT-MIL跨尺度特征融合和自注意力机制的有效性,尤其是在处理不同尺度下肿瘤异质性特征时。
尽管MCBAT-MIL取得了优异的分类性能,本文仍对其错误分类的情况进行了分析。其中,大部分错误分类发生在组织学特征模糊不清的WSI上。这些WSI通常缺乏典型的LUAD或LUSC形态学特征,如细胞排列方式、细胞核的大小和形状等,导致模型难以做出准确判断。此外,还有部分错误分类的WSI存在病理切片质量问题,如组织褶皱、破损等,这可能干扰了模型对关键区域的特征提取和理解。这提示未来模型需要进一步扩大训练数据的规模和多样性,纳入更多有代表性的疑难病例,并探索更有效的数据增强和预处理技术。
3.4 消融实验为了验证MCBAT-MIL使用的多尺度特征、多尺度融合模块MCBAT以及实例损失
| 表 2 TCGA-LUNG上的消融实验结果 Table 2 Ablation results on the TCGA-LUNG dataset |
消融实验结果显示,使用单一尺度特征的5×和20×分类性能均较差,这说明单一尺度的特征表达是不足的,无法有效建模样本的细粒度或全局特征。当直接拼接不同尺度特征时(5×+20×),AUC提高到92.31%,准确率提升到83.60%,召回率为85.75%,精确率为83.17%。这种多尺度方法虽略有改进,但由于缺乏跨尺度的信息交互和融合,分类效果仍然有限。与之相比,本文设计的MCBAT融合模块可以捕捉不同尺度特征之间的依赖关系,实现更好的特征互补和融合,使得AUC进一步提高到93.55%,准确率为86.21%,召回率为86.67%,精确率为85.00%。此外,本文还探索了仅使用实例损失而不加入MCBAT特征融合的效果。结果表明,虽然实例损失可以提供细粒度监督,但如果没有MCBAT的跨尺度特征融合,模型的性能提升仍然有限。
最后,综合利用MCBAT的跨尺度特征融合以及实例损失的监督,模型在所有指标上均得到了最佳表现,AUC达到了95.09%,准确率、召回率和精确率分别提高到90.69%、88.88%和90.00%。这验证了本文设计的模块和损失函数的有效性,多尺度建模与特征融合是提高NSCLC亚型分类性能的关键,加入实例损失可以提供额外的细粒度监督信号,进一步提升模型的区分能力。
3.5 分类结果可视化本节通过实例损失计算过程中的注意力为预测样本生成注意力图,突显模型最为关注的区域,提高模型的可解释性。如图5所示,5(a)为一个肺腺癌亚型分类正确的样本。5(b)为病理学家使用数字病理学查看软件Aperio ImageScope对该切片癌症区域的注释结果,深色部分代表癌症区域。5(c)表示模型在20倍视野下生成的注意力图,颜色越红表示注意力值越高。5(d)表示从样本中提取的8个最高注意力得分的图块。

|
图 5 肺腺癌可视化示例 Figure 5 An example of LUAD visualization |
可视化结果显示,MCBAT-MIL生成的高注意力区域与病理学家标注的癌症区域有较高的重叠度,Top 8注意力得分的图块正确关注了腺癌的典型腺体和乳头状结构等显著特征。这表明模型在仅有切片级别注释的情况下仍能关注到肿瘤细胞的形态特征和组织结构信息。但是注意力区域与癌症区域之间仍然存在一定的差异,这可能源于模型学习过程中的局限性,以及病理图像的复杂性和多样性。总体而言,MCBAT-MIL通过生成解释性的注意力图,不仅可在一定程度上直观判断单个样本的分类质量,还可以总结不同亚型肿瘤的整体注意力分布模式,这为进一步理解模型行为与生物学机制的关系提供了有价值的定量工具。
4 结论本文提出了一种基于多实例学习的非小细胞肺癌亚型病理图像分类方法。方法上,考虑到病理学家是以多层次方式观察病理切片,设计了一种多尺度特征提取和融合框架。该框架在细胞级和组织级不同尺度采样实例,提取异构特征。然后利用Transformer网络建模实例之间的依赖关系,实现端到端的多源信息融合与目标分类。在公开数据集上的实验结果表明,本方法可以有效利用病理切片的多尺度信息,实现对主要肺癌亚型的准确划分。这为提高这一领域的计算机辅助诊断水平提供了有效解决方案。未来工作将探索如何有效区分更多亚型间的过渡类型或混合类型样本,并将进一步扩大数据集规模,探究不同种族人群样本的差异性,提高方法的鲁棒性。
| [1] |
ZHENG R, ZHANG S, ZENG H, et al. Cancer incidence and mortality in China, 2016[J].
Journal of the National Cancer Center, 2022, 2(1): 1-9.
DOI: 10.1016/j.jncc.2022.02.002. |
| [2] |
SUNG H, FERLAY J, SIEGEL R L, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].
CA: A Cancer Journal for Clinicians, 2021, 71(3): 209-249.
DOI: 10.3322/caac.21660. |
| [3] |
COUDRAY N, OCAMPO P S, SAKELLAROPOULOS T, et al. Classification and mutation prediction from non-small cell lung cancer histopathology images using deep learning[J].
Nature Medicine, 2018, 24(10): 1559-1567.
DOI: 10.1038/s41591-018-0177-5. |
| [4] |
ZHAO L, XU X, HOU R, et al. Lung cancer subtype classification using histopathological images based on weakly supervised multi-instance learning[J].
Physics in Medicine & Biology, 2021, 66(23): 235013.
|
| [5] |
WANG X, CHEN H, GAN C, et al. Weakly supervised deep learning for whole slide lung cancer image analysis[J].
IEEE Transactions on Cybernetics, 2019, 50(9): 3950-3962.
|
| [6] |
VASWANI A, SHAZEER N, PARMAR N, et al. Attention is all you need[C]//31st Conference on Neural Information Processing Systems. Long Beach: MIT Press, 2017: 5998-6008.
|
| [7] |
LI B, LI Y, ELICEIRI K W. Dual-stream multiple instance learning network for whole slide image classification with self-supervised contrastive learning[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. Nashville: IEEE, 2021: 14318-14328.
|
| [8] |
DING S, WANG J, LI J, et al. Multi-scale prototypical transformer for whole slide image classification[C]//GREENSPAN H, MADABHUSHI A, MOUSAVI P, et al. Medical Image Computing and Computer Assisted Intervention-MICCAI 2023. Cham: Springer Nature Switzerland, 2023: 602-611.
|
| [9] |
DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16×16 words: transformers for image recognition at scale[EB/OL]. arXiv: 2010.11929(2021-06-03) [2024-04-10]. https://doi.org/10.48550/arXiv.2010.11929.
|
| [10] |
CAO L, WANG J, ZHANG Y, et al. E2EFP-MIL: end-to-end and high-generalizability weakly supervised deep convolutional network for lung cancer classification from whole slide image[J].
Medical Image Analysis, 2023, 88: 102837.
DOI: 10.1016/j.media.2023.102837. |
| [11] |
叶紫璇, 肖满生, 肖哲. 基于EfficientNet模型的多特征融合肺癌病理图像分型[J].
湖南工业大学学报, 2021, 35(2): 51-57.
YE Z X, XIAO M S, XIAO Z. Lung cancer pathological image classification based on an efficientnet model with multi-feature fusion[J]. Journal of Hunan University of Technology, 2021, 35(2): 51-57. DOI: 10.3969/j.issn.1673-9833.2021.02.008. |
| [12] |
YU K H, ZHANG C, BERRY G J, et al. Predicting non-small cell lung cancer prognosis by fully automated microscopic pathology image features[J].
Nature Communications, 2016, 7(1): 12474.
DOI: 10.1038/ncomms12474. |
| [13] |
朱滋陵. 基于细胞病理图像的肺癌亚型分类方法研究[D]. 沈阳: 沈阳工业大学, 2023.
|
| [14] |
ILSE M, TOMCZAK J, WELLING M. Attention-based deep multiple instance learning[C]// Proceedings of the 35th International Conference on Machine Learning. Stockholm: PMLR, 2018: 2127-2136.
|
| [15] |
LU M Y, WILLIAMSON D F, CHEN T Y, et al. Data-efficient and weakly supervised computational pathology on whole-slide images[J].
Nature Biomedical Engineering, 2021, 5(6): 555-570.
DOI: 10.1038/s41551-020-00682-w. |
| [16] |
CAMPANELLA G, HANNA M G, GENESLAW L, et al. Clinical-grade computational pathology using weakly supervised deep learning on whole slide images[J].
Nature Medicine, 2019, 25(8): 1301-1309.
DOI: 10.1038/s41591-019-0508-1. |
| [17] |
SHAO Z, BIAN H, CHEN Y, et al. Transmil: transformer based correlated multiple instance learning for whole slide image classification[J].
Advances in Neural Information Processing Systems, 2021, 34: 2136-2147.
|
| [18] |
SHI J, TANG L, GAO Z, et al. MG-Trans: multi-scale graph transformer with information bottleneck for whole slide image classification[J].
IEEE Transactions on Medical Imaging, 2023, 42(12): 3871-3883.
DOI: 10.1109/TMI.2023.3313252. |
| [19] |
OTSU N. A threshold selection method from gray-level histograms[J].
IEEE Transactions on Systems, Man, and Cybernetics, 1979, 9(1): 62-66.
DOI: 10.1109/TSMC.1979.4310076. |
| [20] |
DENG J, DONG W, SOCHER R, et al. Imagenet: a large-scale hierarchical image database[C]//2009 IEEE Conference on Computer Vision and Pattern Recognition. Miami: IEEE, 2009: 248-255.
|
| [21] |
HE K, ZHANG X, REN S, et al. Deep residual learning for image recognition[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016: 770-778.
|
| [22] |
NAGRANI A, YANG S, ARNAB A, et al. Attention bottlenecks for multimodal fusion[J].
Advances in Neural Information Processing Systems, 2021, 34: 14200-14213.
|
| [23] |
CHOROMANSKI K, LIKHOSHERSTOV V, DOHAN D, et al. Rethinking attention with performers[EB/OL]. arXiv: 2009.14794(2012-11-19) [2024-04-10]. https://doi.org/10.48550/arXiv.2009.14794.
|
| [24] |
KINGMA D P, BA J. Adam: a method for Stochastic Optimization[EB/OL]. arXiv: 1412.6980(2017-01-30) [2024-04-10]. https://doi.org/10.48550/arXiv.1412.6980.
|
 2025, Vol. 42
2025, Vol. 42

