2. 广东工业大学 自动化学院, 广东 广州 510006;
3. 广东省人民医院, 广东 广州 510080;
4. 广东技术师范大学 电子与信息学院, 广东 广州 510665
2. School of Automation, Guangdong University of Technology, Guangzhou 510006, China;
3. Guangdong Provincial People's Hospital, Guangzhou 510080, China;
4. School of Electronics and Information, Guangdong Polytechnic Normal University, Guangzhou 510665, China
在恶性肿瘤中,肺癌是一种高度恶性且广泛存在的癌症[1],其原因是肺部存在较多的病种,在医学影像上的表现较为复杂,小病灶早期特征难以发现,导致在临床中具有较高的漏诊率。由于发病诱因的增多,癌症的患病率大大增加,已成当今世界范围内主要的公共卫生问题,肺癌的发病率和死亡率增长最快[2],对人类的生命健康构成巨大威胁。计算机断层(Computer Tomography, CT) 扫描以其无创的成像方式被广泛应用于肺部肿瘤的早期检测、诊疗评估等多个阶段。在临床上,高质量的肺部肿瘤分割对肺癌的诊断和患者的诊疗至关重要。
目前针对肺部肿瘤的分割方法主要分为基于传统图像的分割方法和基于深度学习的分割方法。在传统图像分割中有基于阈值[3-5]、聚类[6-7]、随机游走[8]和边界[9-10]的分割方法,但是步骤相对复杂,需要人工干预,而且对于噪声较大和形状复杂的图像分割精度较低。近年来,深度学习飞速发展,基于卷积神经网络[11](Convonlution Nerural Network, CNN) 的方法在医学图像分割领域取得了很好的结果,如Ronneberge等[12]在2015年提出的编码−解码网络(U-Net),采用左右对称的U形网络结构,通过连续的下采样和上采样操作,实现了端到端的输出。Dutande等[13]基于U-Net结构引入了深度残差可分离卷积,并使用空洞卷积来增加感受野。Yang等[14]提出了一种多层次深度监督U-Net,将现有的U-Net模型与双路径深度监督机制相结合,以实现更准确的分割性能。Zhang等[15]提出了一种改进的3D密集连接U-Net,采用嵌套密集跳跃连接为编码器和解码器提供相似的特征映射。Kamal等[16]提出了一种基于多个卷积长短期记忆层的3D DenseUNet,利用卷积长短期层组成的循环块来提取细粒度的时空信息,然后再针对分割结果使用形态学操作进行后处理。然而上述方法存在2个问题:(1) 主要侧重于局部信息的传递和特征融合,没有关注全局语义信息,使得网络对病灶和周围组织的对比度不敏感;(2) 在编码器阶段未考虑不同尺度对特征的表达能力,影响模型对肺部肿瘤的分割性能。
最近,Transformer模型被广泛应用于图像分割任务[17-18]中,与传统的卷积神经网络不同,Transformer 模型并不依赖于卷积操作,而是通过自注意力机制来学习序列数据中元素之间的关系。在医学图像分割领域,Transformer和CNN结合的混合架构展现了很好的分割性能,如Hatamizadeh等[19]提出了一种名为UNETR的模型,使用Transformer作为编码器,无需依赖CNN,能够捕捉图像的全局多尺度信息,在肿瘤和脾脏数据中取得了很好的性能。Wang等[20]提出一种CNN和Transformer的混合模型,并结合边缘检测算子用来分割肺结节。STC-Net[21] 将Swin Transformer处理的全局图像信息与CNN处理的低层次细节特征相结合,在脑肿瘤分割、息肉分割和皮肤病变分割中都取得了良好的效果。但是Transformer结构的自注意力机制注重全局信息的交互,较远像素之间的信息传递会受到限制,导致在局部信息建模中丢失细粒度的细节和空间上下文[22]。
针对上述U-Net和Transformer方法的不足,本文提出基于Transformer和通道优先卷积注意力机制的Transformer-UNet网络(Channel Prior Convolutional Attention Transformer-UNet, CPCATNet),引入了基于Global-Local 注意力机制的Transformer,对输入全局建模以及提取更有效的局部特征,有助于了解全局语义和上下文信息;在跳跃连接中使用通道优先卷积注意力机制,利用多尺度的深度可分离卷积模块构成空间注意力,可以在通道和空间维度上动态分配注意权重,以此来有效地提升分割的准确率。
1 方法 1.1 CPCATNet模型CPCATNet的整体架构如图1所示,其框架是基于TransUNet[23]设计的,由编码器、解码器和跳跃连接3个核心部分组成。区别于TransUNet的是:(1) 在编码器阶段,本文设计了局部−全局注意力机制,并且将其嵌入在Transformer结构中;(2) 在跳跃连接阶段,本文引入了通道优先卷积注意力机制(Channel Prior Convolutional Attention, CPCA),使得网络可以对输入的特征在空间和通道维度上进行动态分配权重;(3) 在每个卷积层后加入了批归一化(Batch Normalization, BN),以便提高模型泛化性能[24]。
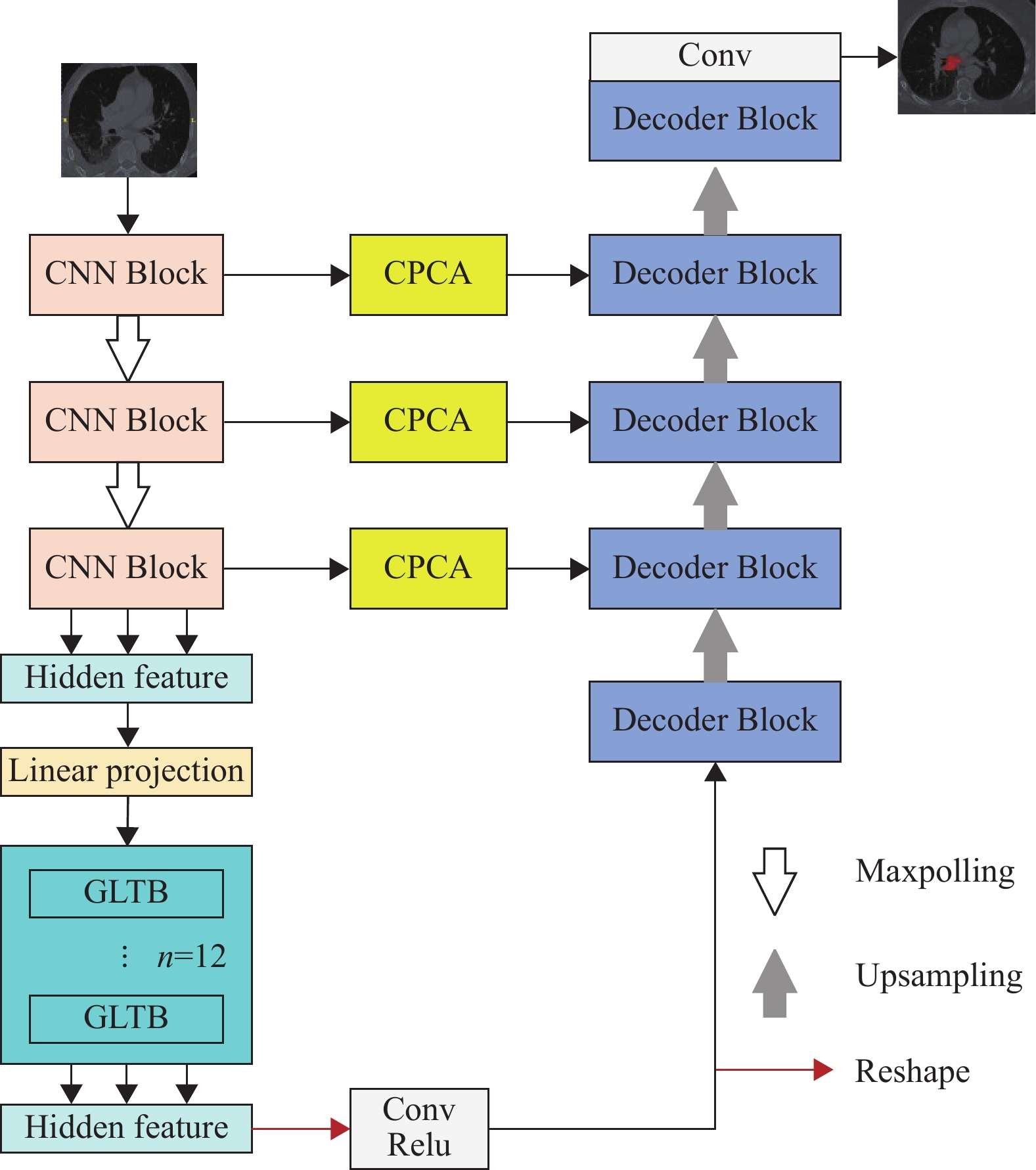
|
图 1 CPCATNet 模型结构 Figure 1 The structure of the CPCATNet model |
CPCATNet的编码器将传统的卷积神经网络和Transformer层融合在一起,包括3个CNN Block和12个Global-Local Transformer模块。编码器输出的特征图的维度为16×196×768,然后解码器对编码器生成的特征进行解码。解码器包括4个解码块和输出层,上采样是由双线性插值算法实现,并且在逐步解码过程中Decoder Block会拼接CNN Block经过CPCA模块处理的不同尺度特征信息,可以逐渐还原图像的分辨率,最后通过卷积核大小为1×1的卷积和Sigmoid,以此来生成精细的分割结果。
CNN Block的内部结构如图2(a)表示,由3个卷积层、3个BN层和2个Relu组成,最后经过跳跃连接输出。图2(b)表示Decoder Block的内部结构,由2个卷积层、2个BN层和2个Relu层组成。
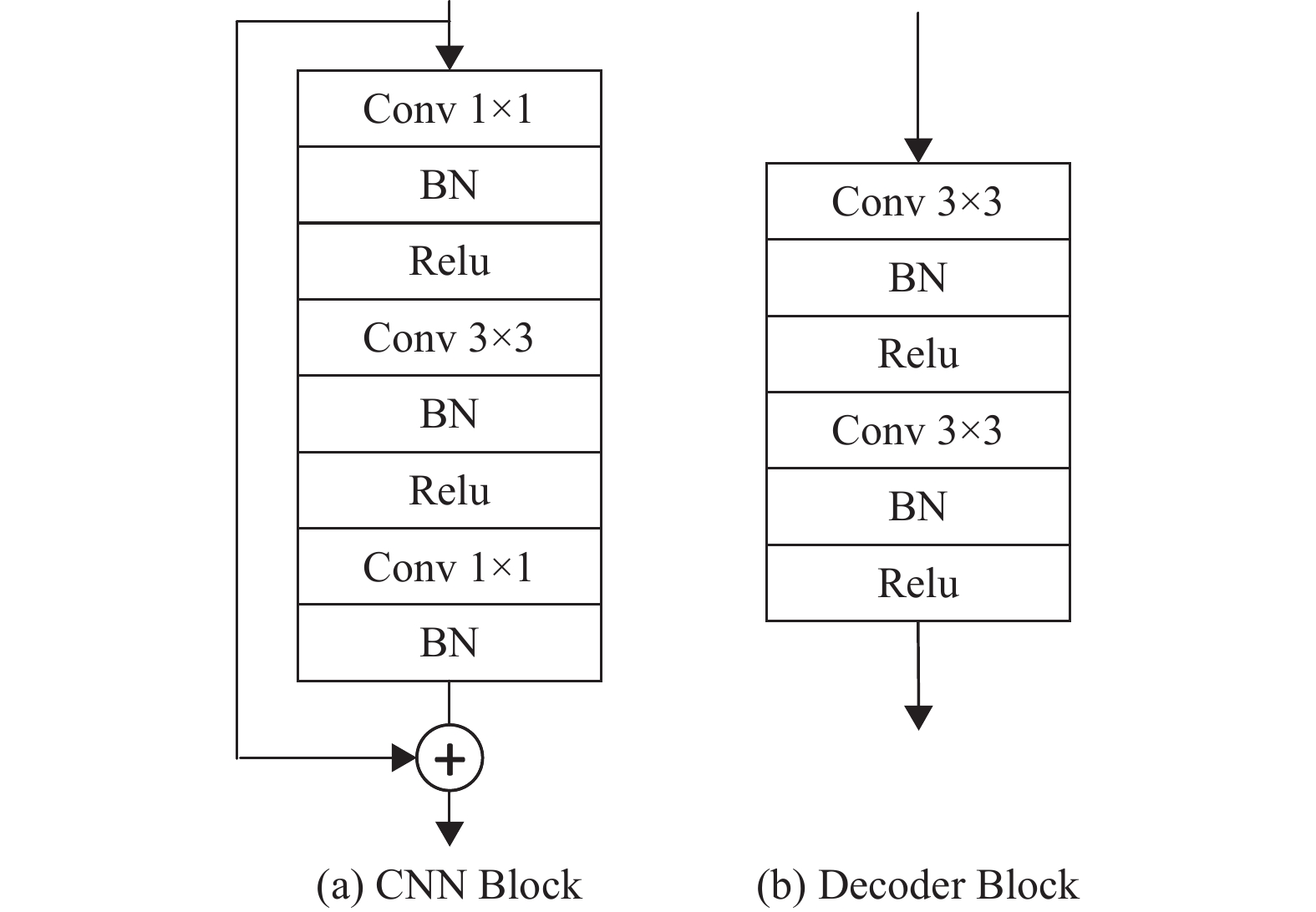
|
图 2 CNN Block和Decoder Block模块内部结构 Figure 2 Internal structure of CNN Block and Decoder Block modules |
GLTB的网络结构如图3所示,主要由4部分组成:2个组归一化、全局−局部注意力机制(Global-local Attention, GLA)和多层感知机。
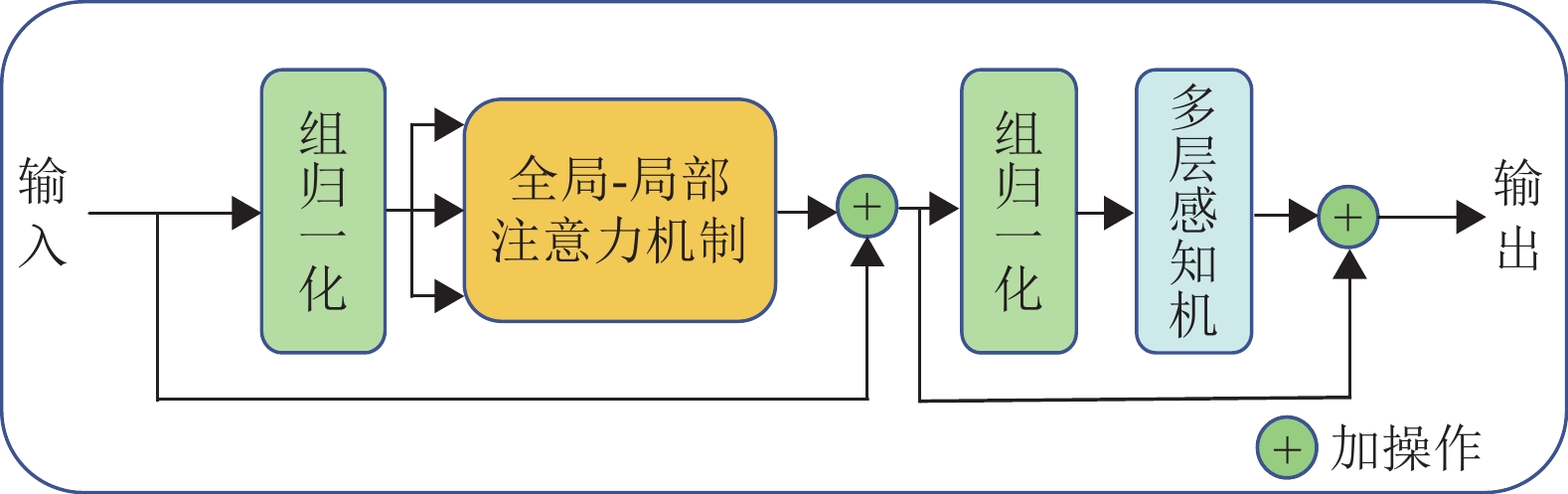
|
图 3 GLTB 网络结构 Figure 3 GLTB network structure |
在CNN层输出的特征输入GLTB之前,需要先把输入X进行矢量化,即将输入X重新构造为一系列的扁平的2D 块,如式(1)所示,其中,N为块的总数,P×P为每个块的大小,C为图像的通道数。然后需要将矢量化的块映射到潜在的D维嵌入空间中,同时为了对块空间信息进行编码,还加入了位置信息,以保留每个块之间的位置。过程表示如式(1)所示。
| $ {Z_0} = [X_p^1E;X_p^2E;{\mathbf{\cdot\cdot\cdot}};X_p^NE] + {E_{{\text{pos}}}} $ | (1) |
式中:E为块的嵌入映射,
GLTB编码器包括L层GLA和多层感知机(Multi-Layer Perceptron, MLP),在标准Transformer的多头自注意力机制中,多头注意力机制虽然可以捕获全局的上下文信息,但是却无法关注局部的尺度特征,导致分割精度降低。针对这个问题,本文在Transformer中引入了一个局部−全局注意力机制,具体架构如图4所示,与标准Transformer中的多头自注意力机制不同,所引入的GLA模块开发了一种有效的全局−局部注意机制,该机制包括全局分支和卷积局部分支,以捕获视觉感知的全局和局部上下文。

|
图 4 全局−局部注意力机制 Figure 4 Global-local Attention |
在全局分支部分,依然采用多头自注意力机制,自注意力机制通过对图像形状的重塑,能够缩短远距离依赖特征之间的距离。在局部分支部分,利用2个并行的卷积层和组归一化来提取局部上下文,每个卷积层负责捕捉不同类型的局部信息,组归一化将通道划分为多个组,每个组进行归一化,使得每个组内保持局部的统计特性。局部分支有助于捕捉局部依赖关系,即当前位置与其周围位置之间的关联,而相对较远位置之间的关联则被忽略。卷积层的卷积核的大小分别为3×3和1×1,最后对2个分支提取的特征进行求和。
GLA的具体计算过程如式(2)所示。
| $ {\text{GLA = MSA(}}{Z_i}{\text{) + }} {\text{ (GN(Conv1(}}{Z_i}{\text{) ) + GN(Conv3(}}{Z_i}{\text{) ) ) }} $ | (2) |
式中:MSA为多头注意力机制的处理过程,
因此GLTB的第l层的输出可由式(3)和(4)表示:
| $ Z'_l = {\text{GLA}}({\text{GN}}({Z_{l - 1}}) ) + {Z_{l - 1}} $ | (3) |
| $ {Z_l} = {\text{MLP(GN}}(Z'_l) ) + Z'_l $ | (4) |
GLTB编码器一共有L层,故
在跳跃连接中,编码器与解码器对应的特征映射存在较大的语义差距,如果编码器的特征映射不经过处理直接与解码器的特征映射相拼接,会增加网络的学习难度,降低分割性能[25],而在跳跃连接中引入注意力机制是一个很好的解决方案。为了提高模型的特征提取能力,Hu等[26]设计了一种挤压和激励网络(Squeeze-and-Excitation Networks, SE), 通过在通道维度上增加注意力机制来提高模型的表现能力。SE只结合了通道注意力机制,限制了网络关注其他重要区域。Woo等[27]提出了卷积模块注意力(Convolutional Block Attention Module, CBAM),依次结合了通道注意力机制和空间注意力机制[28],使网络能够专注于空间信息和通道重要区域。然而,空间注意力特征图是通过压缩通道来计算的,这导致在与输入特征逐元素相乘期间每个通道的空间注意力权重的一致分布。这限制了注意力的自适应能力,因为空间注意力权重不能基于每个通道的特定特性进行动态调整,为此Huang等[29]提出了一种称为通道优先卷积注意力的网络结构,能够在通道和空间维度上动态分布注意力权重。不同层级的跳跃连接具有不同尺度的特征信息,而由于通道优先卷积注意力具有自适应调整注意力权重的特性,在跳跃连接中引入通道优先卷积注意力可以使解码器更好地融合不同尺度的信息。通道优先卷积注意力具体的网络结构如图5所示。

|
图 5 通道优先卷积注意力机制 Figure 5 Channel prior convolutional attention |
CPCA顺序结合了通道注意力模块和空间注意力模块,但与CBAM不同的是,CPCA采用深度卷积模块来构造空间注意力,深度卷积模块采用不同尺度的条形卷积核提取像素间的空间映射关系。多尺度深度条形卷积核的使用保证了有效的信息提取,同时降低了计算复杂度。输入特征首先经过通道注意力模块,获得通道注意力特征图,然后与输入特征进行相乘,随后,深度卷积模块依次提取每个通道的关键空间区域,从而在每个通道上产生动态分布的空间注意力特征图,这些动态分布在每个通道上的空间注意力图与实际特征分布非常接近,有效地提高了网络的分割性能。最后空间注意力特征图经过1×1的卷积后与通道优先特征逐元素相乘生成最终的细化特征。
2 实验设置 2.1 数据集及数据预处理本文使用2个不同的数据集对模型分割性能以及泛化性能进行验证,包括1个公开数据集和1个医院的私有数据集,公开数据集是使用文献[30]公布在癌症影像档案(The Cancer Imaging Archive, TCIA) 的肺部肿瘤数据。TCIA由美国国家癌症研究所创建和管理,收集了来自世界各地的多个研究机构和医疗中心的医学影像数据。本文使用的公开数据集的名称为LUNG1,包含来自422名非小细胞肺癌患者的图像。私有数据集是来自广东省人民医院的内部数据GDPH,并且已获得授权使用。GDPH包含824例3D图像,该数据集的肺部肿瘤区域由2名放射科医师手动勾画,他们在CT肺癌解释方面有5年的经验。注释由放射科医生交叉检查,并通过讨论达成最终协议。图像的大小、分辨率、层厚等信息如表1所示。
| 表 1 数据的具体信息 Table 1 Data specific information |
数据预处理主要由裁剪、重采样和归一化3部分组成。首先对数据进行裁剪,在肿瘤的肺部区域以外有很多无关的信息,会影响模型的分割结果,把肺部以外的区域裁剪掉,排除那些无关信息的影响,然后再对数据进行重采样。由于不同客户端的数据采集的设备不同,会导致不同客户端的数据之间会有异质性,采用最近邻插值法获得轴向平面的各向同性空间分辨率,使每个体素的间距为1×1×1。最后对数据进行归一化,把肺部肿瘤窗宽设置为150豪斯菲尔德单位(Hounsfiled Unit, HU),窗位设置为−
为了评估CPCATNet性能,采用Dice相似系数和豪斯多夫距离(Hausdorff Distance,HD)来对肿瘤的分割结果进行评估,具体公式分别为
| $ {\text{Dice}} = \frac{{2|P \cap G|}}{{|P| + |G|}} $ | (5) |
| $ \begin{split} &{\text{HD}} = \max \{ {d_{PG}} + {d_{GP}}\} = \max \{ \mathop {\max }\limits_{p \in P} \mathop {\min }\limits_{g \in G} d(p,g) ,\\ &\mathop {\max }\limits_{g \in G} \mathop {\min }\limits_{p \in P} d(g,p) \} \end{split} $ | (6) |
式中:∩为交集算子,P为预测结果,G为标签图像。Dice系数范围是0到1之间,Dice系数值越高,说明模型的分割性能越好。在计算Dice系数时考虑了以下2种情况:(1) 对于真阴性,即没有肿瘤,并且算法也正确地检测到没有肿瘤,Dice系数将为1;(2) 对于假阳性,即没有肿瘤,但是算法错误地预测出了肿瘤,Dice系数将为0。HD的计算如式(5)所示,其中
数据按照7∶3的比例划分为训练集和测试集,实验均在RTX3090的GPU平台上进行,Python版本为3.7,CUDA版本为12.0,PyTorch的版本为1.7.1。使用Adam优化器来最小化损失函数,迭代次数为150,学习率设置为0.000 1,并且每经过10次学习率就衰减1×
| $ {L_{{\text{dice}}}} = 1 - \frac{{2|P \cap G| + s}}{{|P| + |G| + s}} $ | (7) |
| $ {L_{{\text{cs}}}} = - P\log G - (1 - P) \log (1 - G) $ | (8) |
式中:s为平滑系数。本文使用的混合损失函数如式(8)所示。
| $ L = \alpha {L_{{\text{dice}}}} + (1 - \alpha ) {L_{{\text{cs}}}} $ | (9) |
式中:权重系数
本文在GDPH和LUNG1数据集上进行实验,将CPCATNet(本文方法)与其他8种医学图像分割方法进行比较,包括:(1) U-Net;(2) DenseUNet[32];(3) KiU-Net[33];(4)TransUNet;(5) Swin-UNet[31];(6) FocalUNETR[34];(7)HSN[35];(8)RRc-UNet[36]。其中U-Net是一个对称的网络结构,解码器和编码器都包含了4个卷积模块,最后采用输出层来输出分割结果。DenseUNet是在U-Net的基础上将每个子模块都改进成具有跳跃连接和密集连接的形式。KiU-Net在U-Net的基础上设计了一个全卷积网络Kite-Net,用来捕获输入的细节。TransUNet的编码器是ResNet与Transformer级联组成,其中ResNet的输出作为Transformer的输入,解码器与U-Net的编码器一致。Swin-UNet的编码器和解码器都是由Swin Transformer块组成,每个块都包含了窗口式的自注意力机制,同时采用跳跃连接来加入多尺度的特征,在医学图像分割领域取得了很好的结果。FocalUNETR是CNN与 Transformer组成的混合架构,利用焦点空间注意力来分层学习长短期视觉依赖的特征映射。HSN提出了一种混合特征融合模块,用来融合三维和二维特征,在肺部CT数据集上取得了很好的性能。RRc-UNet基于U-Net设计了一种残差循环模块,用来提取肺部肿瘤的特征。
不同方法在2个数据集上的分割性能如表2所示。在GDPH数据集中,CPCATNet方法的分割性能最好,Dice指标为90.96%,相比于对比方法,Dice性能分别提升了7.31%、7.18%、4.81%、5.10%、4.08%、3.49%、3.76%和3.14%;HD为59.52,相对于8个对比实验方法,分别降低了13.28%、11.71%、9.74%、18.40%、13.90%、6.93%、8.68%和3.02%。在LUNG1数据集上,本文方法也取得了最好的分割结果,Dice为88.18%,相比于对比方法,Dice系数分别提升了6.54%、7.73%、5.55%、6.94%、2.82%、3.43%、2.19%和1.96%;HD分别降低了11.77%、10.20%、12.45%、11.34%、7.61%、9.48%、13.49%和5.14%。通过对比其他8个模型,本文的方法对肺部的病变区域分割取得了最优的结果,说明CPCATNet对肺部肿瘤的分割能力更好。
| 表 2 不同方法在2个数据集上的结果对比 Table 2 Comparative results of different methods on two datasets |
图6展示了选取的4个数据在不同方法下分割结果的可视化,图6(a)为原始图像,图6(b)为真实标签。图6(c)~图6(j)分别是U-Net、DenseUNet、KiU-Net、TransUNet、Swin-UNet、FocalUNETR、HSN、RRc-UNet和CPCATNet的分割结果可视化。

|
图 6 分割结果可视化 Figure 6 Visualization of segmentation results |
对于第1行和第3行的数据,肿瘤与周围组织粘连,对比实验方法普遍表现出多分割的现象,在分割过程中错误地将一些边界组织也划分为肿瘤区域,这是由于肺部肿瘤与周围组织之间的边界模糊且对比度较低所致;对于第2行数据,HSN出现了多分割的情况,其他对比方法则出现了不同程度的少分割的情况;对于第4行数据,在背景噪声大的情况下,对比实验方法分割结果较为粗糙。而本文提出的CPCATNet,一方面在跳跃连接中更加细化了多级尺度的特征,另一方面在编码器中嵌入的全局−局部注意力机制使得网络对于肿瘤的形状和位置更加关注,分割的结果更接近标签,并且分割的精确度有一定的提升。
3.2 消融实验消融实验一共有6次实验,第1次实验是仅在TransUNet的基础上添加CPCA模块,即CPCANet;第2次实验是仅在TransUNet的基础上添加GLTB模块,即GLTBTNet;剩余4次实验是在使用GLTB模块的基础上评估通道优先卷积注意力的性能。实验3中,模型在不同的跳跃连接中只使用通道注意力机制,即CATNet;实验4中,模型在不同的跳跃连接中只使用空间注意力机制,即SATNet;实验5中,模型在不同的跳跃连接中全部使用卷积块注意力模块,即MHATNet;实验6中,模型在不同的跳跃连接中使用通道优先卷积注意力机制,即CPCATNet,也就是本文的方法。表3为不同注意力机制在2个数据集上的Dice指标结果。
| 表 3 不同注意力机制的对比 Table 3 Comparison of different attention mechanisms |
比较CPCANet与CPCATNet,在GDPH和LUNG1数据集上,GLTB模块使Dice分别提升了2.98%和2.66%,说明了GLTB模块的引入能有效地提高网络的分割性能。对比GLTBTNet与CPCATNet,在两个数据集上,通道优先卷积注意力机制模块使Dice分别提高了3.03%和3.05%,这表明将通道优先卷积注意力机制嵌入到跳跃连接有利于肿瘤分割任务。此外,CPCATNet在GDPH和LUNG1数据集上的Dice指标优于在跳跃连接中使用空间注意力机制、通道注意力机制和卷积模块注意力机制的模型,说明了引用通道优先卷积注意力机制的正确性。
3.3 损失函数权重系数的对比实验为了探究损失函数的权重系数
| 表 4 不同权重系数的对比 Table 4 Comparison of different weight coefficients |
由实验结果可知,当
本文提出一种基于Transformer和通道优先卷积注意力机制的肺部肿瘤分割网络,在TransUNet模型的基础上,通过在编码器引入GLTB模块,将局部注意力机制与自注意力机制结合,使网络同时关注局部信息与全局上下文信息,通过在网络跳跃连接引入通道优先卷积注意力机制,对不同尺度特征在空间和通道维度上进行动态分配权重,使网络更加关注肺部肿瘤相关区域和重要的特征,进一步提升模型的分割性能。实验表明,本文方法在GDPH和LUNG1这2个数据集上Dice指标可分别达到 90.96%和88.18%,相比于其他8种分割方法,本文方法对病灶与周围组织的对比度低、肿瘤与正常组织易粘连的情况均能很好地分割。本文方法对肺部肿瘤的诊断具有很好的应用价值,但仍存在一些待改进的地方,如相比于U-Net等方法,CPCATNet具有更复杂的结构且参数量更多,导致训练时间相对较长。在后续的研究中,主要工作是在保持高分割精度的同时,构建一个更加轻量化的模型,以缩短训练时间并提高模型的泛化能力,从而进一步优化肿瘤的诊断效果。
| [1] |
SIEGEL R L, MILLER K D, WAGLE N S, et al. Cancer statistics, 2023[J].
CA: A Cancer Journal for Clinicians, 2023, 73(1): 17-48.
DOI: 10.3322/caac.21763. |
| [2] |
KASINATHAN G, JAYAKUMAR S, GANDOMI A H. Automated 3-D lung tumor detection and classification by an active contour model and CNN classifier[J].
Expert Systems with Applications, 2019, 134: 112-119.
DOI: 10.1016/j.eswa.2019.05.041. |
| [3] |
刘桂红, 赵亮, 孙劲光, 等. 一种改进粒子群优化算法的Otsu图像阈值分割方法[J].
计算机科学, 2016, 43(3): 309-312.
LIU G H, ZHAO L, SUN J G, et al. Otsu image threshold segmentation method based on improved particle swarm optimization[J]. Computer Science, 2016, 43(3): 309-312. DOI: 10.11896/j.issn.1002-137X.2016.03.058. |
| [4] |
贾鹤鸣, 李瑶, 姜子超, 等. 基于改进共生生物搜索算法的林火图像多阈值分割[J].
计算机应用, 2021, 41(5): 1465-1470.
JIA H M, LI Y, JIANG Z C, et al. Multi-threshold segmentation of forest fire images based on modified symbiotic organisms search algorithm[J]. Journal of Computer Applications, 2021, 41(5): 1465-1470. |
| [5] |
MALATHI M, SINTHIA P, JALALDEEN K. Active contour based segmentation and classification for pleura diseases based on Otsu’s thresholding and support vector machine (SVM)[J].
Asian Pacific Journal of Cancer Prevention: APJCP, 2019, 20(1): 167.
DOI: 10.31557/APJCP.2019.20.1.167. |
| [6] |
GOPI K, SELVAKUMAR J. Lung tumor area recognition and classification using EK-mean clustering and SVM[C]//2017 International Conference on Nextgen Electronic Technologies: Silicon to Software (ICNETS2) . Tamil Nadu: IEEE, 2017: 97-100.
|
| [7] |
SANGAMITHRAA P B, GOVINDARAJU S. Lung tumour detection and classification using EK-Mean clustering[C]//2016 International Conference on Wireless Communications, Signal Processing and Networking (WiSPNET) . Chennai : IEEE, 2016: 2201-2206.
|
| [8] |
MI H, PETITJEAN C, DUBRAY B, et al. Automatic lung tumor segmentation on PET images based on random walks and tumor growth model[C]//2014 IEEE 11th International Symposium on Biomedical Imaging (ISBI) . Beijing: IEEE, 2014: 1385-1388.
|
| [9] |
JONES J L, XIE X, ESSA E. Combining region-based and imprecise boundary-based cues for interactive medical image segmentation[J].
International Journal for Numerical Methods in Biomedical Engineering, 2014, 30(12): 1649-1666.
DOI: 10.1002/cnm.2693. |
| [10] |
YANG B, XIANG D, YU F, et al. Lung tumor segmentation based on the multi-scale template matching and region growing[C]//Medical Imaging 2018: Biomedical Applications in Molecular, Structural, and Functional Imaging. Houston: SPIE, 2018, 10578: 645-651.
|
| [11] |
LONG J, SHELHAMER E, DARRELL T. Fully convolutional networks for semantic segmentation[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. Santiago: IEEE, 2015: 3431-3440.
|
| [12] |
RONNEBERGER O, FISCHER P, BROX T. U-net: convolutional networks for biomedical image segmentation[C]//Medical Image Computing and Computer-Assisted Intervention-MICCAI 2015. Munich: Springer International Publishing, 2015: 234-241.
|
| [13] |
DUTANDE P, BAID U, TALBAR S. Deep residual separable convolutional neural network for lung tumor segmentation[J].
Computers in Biology and Medicine, 2022, 141: 105161.
DOI: 10.1016/j.compbiomed.2021.105161. |
| [14] |
YANG J, WU B, LI L, et al. MSDS-UNet: a multi-scale deeply supervised 3D U-Net for automatic segmentation of lung tumor in CT[J].
Computerized Medical Imaging and Graphics, 2021, 92: 101957.
DOI: 10.1016/j.compmedimag.2021.101957. |
| [15] |
ZHANG G, YANG Z, JIANG S. Automatic lung tumor segmentation from CT images using improved 3D densely connected UNet[J].
Medical & Biological Engineering & Computing, 2022, 60(11): 3311-3323.
|
| [16] |
KAMAL U, RAFI A M, HOQUE R, et al. Lung cancer tumor region segmentation using recurrent 3d-denseunet[C]//Thoracic Image Analysis: Second International Workshop, TIA 2020. Lima: Springer International Publishing, 2020: 36-47.
|
| [17] |
DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16×16 words: Transformers for image recognition at scale[EB/OL]. arXiv:2010. 11929 (2021-06-03) [2023-11-11]. https://doi.org/10.48550.
|
| [18] |
CHENG B, MISRA I, SCHWING A G, et al. Masked-attention mask transformer for universal image segmentation[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. New Orleans: IEEE, 2022: 1290-1299.
|
| [19] |
HATAMIZADEH A, TANG Y, NATH V, et al. Unetr: transformers for 3D medical image segmentation[C]//Proceedings of the IEEE/CVF Winter Conference on Applications of Computer Vision. New Orleans : IEEE, 2022: 574-584.
|
| [20] |
WANG S, JIANG A, LI X, et al. DPBET: a dual-path lung nodules segmentation model based on boundary enhancement and hybrid transformer[J].
Computers in Biology and Medicine, 2022, 151: 106330.
DOI: 10.1016/j.compbiomed.2022.106330. |
| [21] |
WANG X, LI Y. STC-Net: fusing swin transformer and convolution neural network for 2D medical Image segmentation[C]//2022 2nd International Conference on Electronic Information Engineering and Computer Technology (EIECT) . Xian: IEEE, 2022: 317-320.
|
| [22] |
WANG L, LI R, ZHANG C, et al. UNetFormer: a unet-like transformer for efficient semantic segmentation of remote sensing urban scene imagery[J].
ISPRS Journal of Photogrammetry and Remote Sensing, 2022, 190: 196-214.
DOI: 10.1016/j.isprsjprs.2022.06.008. |
| [23] |
CHEN J, LU Y, YU Q, et al. TransUNet: transformers make strong encoders for medical image segmentation[EB/OL]. arXiv: 2102.04306(2021-02-08) [2023-11-11]. https://arxiv.org/abs/2102.04306.
|
| [24] |
BJORCK J, GOMES C, SELMAN B, et al. Understanding batch normalization[C]// NIPS’18: Proceedings of the 32nd International Conference on Neural Information Processing Systems. Red Hook, New York: Curran Associates Incorporation, 2018: 7705–7716.
|
| [25] |
CAI Y, WANG Y. Ma-unet: an improved version of unet based on multi-scale and attention mechanism for medical image segmentation[C]//Third International Conference on Electronics and Communication; Network and Computer Technology (ECNCT 2021) . Xiamen: SPIE, 2022, 12167: 205-211.
|
| [26] |
HU J, SHEN L, SUN G. Squeeze-and-excitation networks[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. Vancouver : IEEE, 2018: 7132-7141.
|
| [27] |
WOO S, PARK J, LEE J Y, et al. Cbam: convolutional block attention module[C]//Proceedings of the European Conference on Computer Vision (ECCV) . Munich: Springer, 2018: 3-19.
|
| [28] |
ZHU X, CHENG D, ZHANG Z, et al. An empirical study of spatial attention mechanisms in deep networks[C]//Proceedings of the IEEE/CVF International Conference on Computer Vision. California: IEEE, 2019: 6688-6697.
|
| [29] |
HUANG H, CHEN Z, ZOU Y, et al. Channel prior convolutional attention for medical image segmentation[EB/OL]. arXiv: 2306.05196 (2023-06-08) [2023-11-11]. https://arxiv.org/abs/2306.05196.
|
| [30] |
AERTS H J W L, VELAZQUEZ E R, LEIJENAAR R T H, et al. Decoding tumour phenotype by noninvasive imaging using a quantitative radiomics approach[J].
Nature Communications, 2014, 5(1): 4006.
DOI: 10.1038/ncomms5006. |
| [31] |
CAO H, WANG Y, CHEN J, et al. Swin-unet: unet-like pure transformer for medical image segmentation[C]//European Conference on Computer Vision. Cham: Springer Nature Switzerland, 2022: 205-218.
|
| [32] |
LI X, CHEN H, QI X, et al. H-DenseUNet: hybrid densely connected UNet for liver and tumor segmentation from CT volumes[J].
IEEE Transactions on Medical Imaging, 2018, 37(12): 2663-2674.
DOI: 10.1109/TMI.2018.2845918. |
| [33] |
VALANARASU J M J, SINDAGI V A, HACIHALILOGLU I, et al. Kiu-net: overcomplete convolutional architectures for biomedical image and volumetric segmentation[J].
IEEE Transactions on Medical Imaging, 2021, 41(4): 965-976.
|
| [34] |
LI C, QIANG Y, SULTAN R I, et al. FocalUNETR: a focal transformer for boundary-aware prostate segmentation using CT images[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer Nature Switzerland, 2023: 592-602.
|
| [35] |
CHEN W, WEI H, PENG S, et al. HSN: hybrid segmentation network for small cell lung cancer segmentation[J].
IEEE Access, 2019, 7: 75591-75603.
DOI: 10.1109/ACCESS.2019.2921434. |
| [36] |
LE V L, SAUT O. Rrc-unet 3D for lung tumor segmentation from CT scans of non-small cell lung cancer patients[C]//Proceedings of the IEEE/CVF International Conference on Computer Vision (ICCV) Workshops. Paris: IEEE, 2023: 2316-2325.
|
 2025, Vol. 42
2025, Vol. 42