单光子发射计算机断层扫描(Single-photon Emission Computerized Tomography,SPECT)是一种诊断成像技术,通过对投影数据进行断层重建来估计放射性示踪剂在体内的分布情况。放射性示踪剂会对人体造成特异性辐射损伤,目前在积极研究低剂量SPECT重建,希望能在减少辐射和图像质量之间找到最佳平衡点。为提高低剂量SPECT重建图像的质量,现有大量研究工作探索基于全监督的深度学习重建方法,该方法依赖大量的标记数据。然而,在医学图像处理领域,标记数据非常缺乏,而大量的未标记数据却很容易获得。为克服标记数据获取困难,本文提出一种新的半监督学习重建方法,使用预训练模型和GAN损失函数进行低剂量SPECT重建。
基于深度学习的重建方法已成为医学成像领域的主流。与基于模型的迭代重建[1-3]方法相比,基于深度学习方法的优点是可以捕获图像的复杂特征和相关依赖关系,从而提高图像质量并降低计算成本。正如文献[4]中介绍的,基于深度学习的方法可以分为4大类:图像域学习、弦图域学习、混合域学习和自动映射。
弦图域学习可以被视为一种预处理方法,采用神经网络(Neural Network,NN)来学习弦图域插值并抑制噪声[5-7]。文献[8-9]主要将残差学习与U-Net架构相结合,去除低光子数正弦图中的随机噪声或合成稀疏视角正弦图中的缺失数据。混合域学习是一种由物理和数据耦合驱动的方法。Shao等[10]提出“SPECTnet”,采用两步训练策略,学习从投影空间到图像空间的转换,作为传统OSEM算法的替代方案。Li等[11]使用一种用于SPECT图像重建的训练端到端展开迭代神经网络,这需要一个内存高效的前向−后向投影来实现高效的反向传播。Shao等[12]提出用网络来重建图像,输入投影数据最初被输送到两个全连接(Fully Connected,FC)层以执行基本重建。然后,FC层的输出和衰减图被传递到5个卷积层,进行信号衰减补偿和图像优化。自动映射使用神经网络学习从弦图域到图像域的直接映射。以前的工作[13-14]讨论利用通用深度神经网络的端到端图像重建。Häggström等[13]提出一种基于深度卷积编码器−解码器网络架构的端到端正电子发射计算机断层扫描(Positron Emission Computed Tomography,PET)图像重建技术,网络以PET正弦图数据为输入,有效地输出高质量的重建图像。Pribanić等[15]评估使用深度卷积神经网络(Deep Convolutional Neural Network,DCNN)来减少采集时间的可行性。欠采样图像数据集作为输入提供给神经网络,而缺失的投影作为目标提供。
图像域学习是一种后处理方法,应用神经网络从一些传统方法产生的损坏图像中学习噪声或伪影[16]。Zhang等[17]利用编码器−解码器框架逐步将输入图像压缩为特征表示,然后逐步将特征表示重建为完整的数据。对于低剂量医学成像,该方法采用深度学习来学习低剂量和正常剂量图像之间的映射,从而解决由于辐射剂量减少导致的图像质量差问题。Xie等[18]提出一种逐层 Transformer 网络,用于从3D少角度数据重建心脏 SPECT 图像。Shao等[19]提出一种神经网络,在给定SPECT投影数据和衰减图的情况下,可以为SPECT生成高分辨率和低噪声图像。
大多数基于深度学习的重建方法属于全监督学习,需要使用大量的标记数据训练。对于低剂量医学成像,标记数据包括配对的低剂量和正常剂量数据。然而,获取大量标记数据非常耗时且昂贵,并且临床上容易获得大量未标记数据。为解决标记数据缺乏,研究人员对医学成像的无监督和半监督学习方法进行研究。在无监督学习中,模型只能从可用数据中学习,生成高质量的去噪图像。Gu等[20]使用自适应归一化层将低剂量CT(Low-dose CT,LDCT) 图像转换为正常剂量CT图像。事实上,半监督学习方法被证明在医学图像分割与分类任务领域是有效的[21- 22],可以利用少量标记数据和大量未标记数据来进一步提高学习性能。半监督学习方法通过训练少量标记数据和大量未标记数据降低低剂量医学成像中的噪声[23]。此外,目前还没有针对低剂量SPECT的系统研究半监督学习方法。
本文在图像域学习提出一种带有GAN(Generative Adversarial Network)损失的预训练平均教师方法(Pre-trained Mean Teacher Method with GAN Loss,PMTG),以实现低剂量SPECT重建。所提方法在平均教师(Mean Teacher,MT) 模型引入基于Swin-Conv-Unet(SCUNet)[24]的预训练模型,以提高未标记训练数据的可靠性。教师模型指导学生模型进行一致性预测,预训练模型指导学生模型使用未标记数据进行相似性预测。GAN损失函数用于测量学生模型和预训练模型之间的相似度。数值实验验证所提方法在抑制噪声和保持特征方面的性能,数据集由SIMIND仿真软件使用XCAT数字模体生成。
1 方法与细节本节提出一种用于低剂量SPECT重建的半监督学习方法,包括预训练模型和MT模型。并进一步描述仿真数据集和使用SIMIND进行SPECT成像的仿真设置。
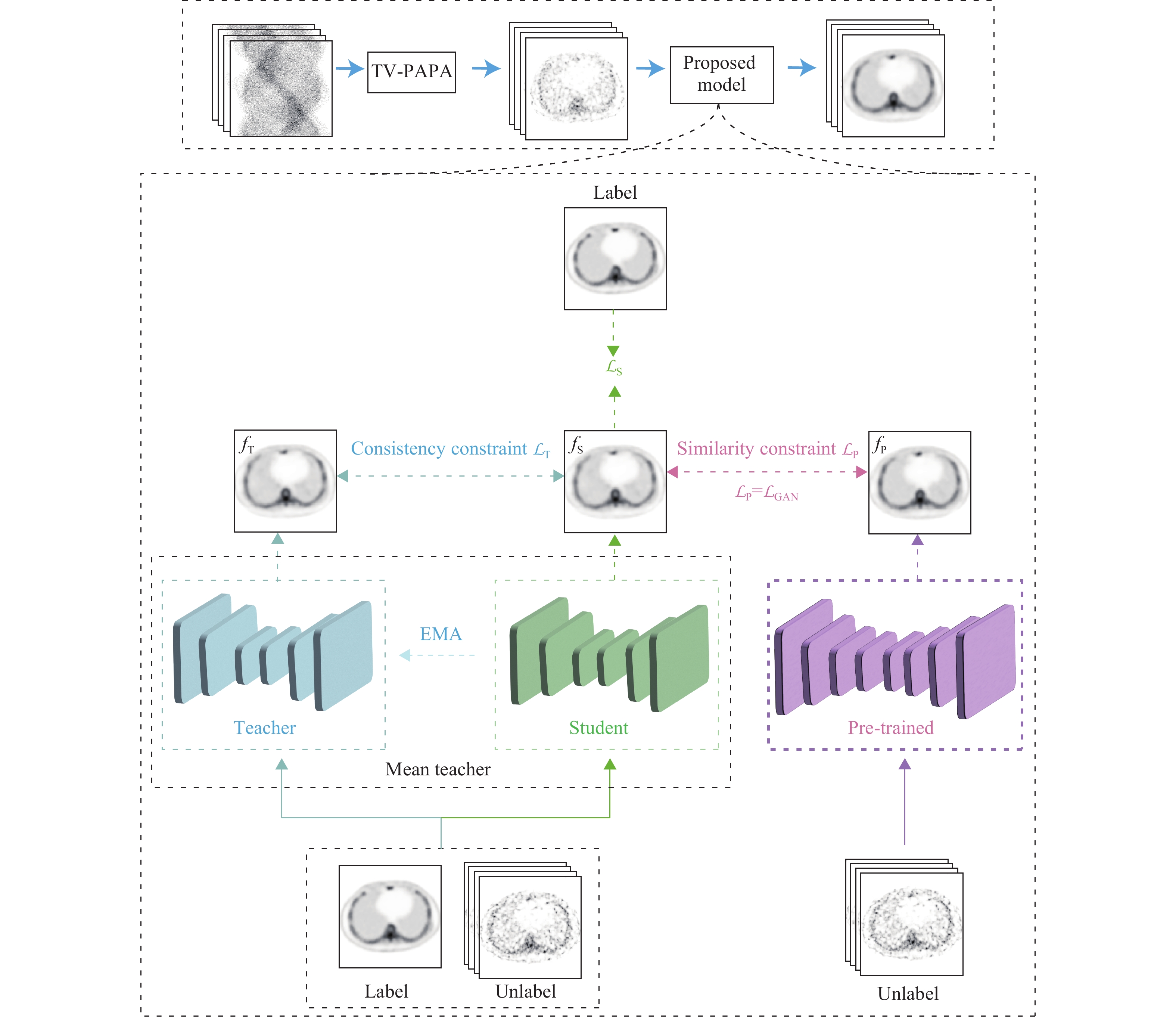
1.1 GAN 损失的预训练平均教师学习方法首先介绍所提PMTG方法的结构,包括3种学习模型如图1所示,学生模型(Student Model,SM)
|
图 1 带有GAN损失(PMTG)的预训练平均教师方法的结构 Figure 1 The architecture of the proposed pre-trained mean teacher method with GAN-loss (PMTG) |
首先,采用标记数据对PM进行训练,然后在所提PMTG方法中,将PM学习到的参数固定。另外,采用标记数据对SM进行训练,并将未标记数据输入到3个计算模型中。具体来说,PM的输出用于提高监督可靠性,TM的参数通过SM的指数移动平均(Exponential Moving Average,EMA)进行更新,其输出用于监督SM。
PMTG的复合损失函数设计算式如下:
| $ {\mathcal{L}}_{\mathrm{t}\mathrm{o}\mathrm{t}\mathrm{a}\mathrm{l}}={\mathcal{L}}_{\mathrm{S}}+{\lambda }_{\mathrm{T}}{\mathcal{L}}_{\mathrm{T}}+{\lambda }_{\mathrm{P}}{\mathcal{L}}_{\mathrm{P}} $ | (1) |
式中,
| $ {\lambda }_{{\rm{T}}}\left(t\right) =\mathrm{e}\mathrm{x}\mathrm{p}\left\{-5 \cdot {\left(1-\frac{t}{{t}_{\mathrm{m}\mathrm{a}\mathrm{x}}}\right) }^{2}\right\} $ | (2) |
式中,
在MT模型的训练阶段,由于SM学习能力不足,MT模型训练初期不稳定,导致TM学习的参数不可靠。为解决标记数据有限问题,本文添加一个PM,在训练的早期阶段通过未标记数据生成的伪标签指导SM。卷积神经网络(Convolutional Neural Network,CNN)仅具有局部建模能力,缺乏非局部建模能力[25]。为获得局部和非局部建模能力,使用SCUNet作为附加分支,学习数据分布的结构信息,并在每个解码层加入一个残差连接。使用标记数据预训练,然后在PMTG中利用未标记数据固定PM参数。
在PMTG的训练阶段,PM参数固定,为未标记数据生成伪标签。在没有标签的情况下,伪标签并非真正的标签,使用逐像素度量可能导致更大的误差。因此,在本文中,设计一种相似性度量的GAN损失函数,由下式给出
| $ {\mathcal{L}}_{\mathrm{P}}={\mathrm{E}}_{x}\left[\mathrm{l}\mathrm{g}D\left(x,{f}_{\mathrm{P}}\left(x\right) \right) \right]+{\mathrm{E}}_{x}\left[\mathrm{l}\mathrm{g}\left(1-D\left(x,{f}_{\mathrm{S}}\left({x}_{i},\theta \right) \right) \right) \right] $ | (3) |
式中,
在PMTG中,采用MT模型来实现低剂量SPECT重建的半监督学习方法。SM和TM的骨干都是UNet[26]。TM的权重(
| $ {{\theta }'_{t}}=\alpha {{\theta }'_{t-1}}+(1-\alpha ) {\theta }_{t} $ | (4) |
式中,
| $ {\mathcal{L}}_{\mathrm{T}}=\displaystyle\sum _{i=1}^{N+M}{\|{f}_{\mathrm{T}}({x}_{i},{\theta }') -{f}_{\mathrm{S}}({x}_{i},\theta ) \|}^{2} $ | (5) |
式中,
SM需要从少量标记数据
| $ {\mathcal{L}}_{\mathrm{S}}=\displaystyle\sum _{i=1}^{N}\left|{f}_{\mathrm{S}}({x}_{i},\theta ) -{y}_{i}\right| $ | (6) |
式中,
本文从XCAT(Extended Cardiac-torso)数字模体[27]获得仿真数据集,为医学成像研究提供真实的成像数据。模体的正弦图数据是使用SIMIND软件模拟SPECT成像产生的。
首先选择10个不同的XCAT数字模体来生成仿真数据集,这是真实的头部和躯干三维骨骼数据,共有
| 表 1 模体信息 Table 1 Phantoms information |
正常剂量水平的正弦图重建图像用作标签,低剂量水平的正弦图重建图像用作网络的输入。重建图像以4:1的比例划分为训练集和测试集。为扩大训练集并避免过度拟合,使用2种数据增强方法,包括水平翻转和垂直翻转。此外,采用无偏TV-PAPA[2]重建算法将所有正弦图重建到图像域。
2 实验结果及分析在本节中进行数值实验,证明所提方法的有效性,同时与对比方法相比有所提升。并给出实现其方法的参数集和评估指标。进一步在消融方面进行实验。
2.1 实验细节所提PMTG中,PM采用SCUNet,代码位于https://github.com/cszn/SCUNet。SM和PM采用UNet,由4个下采样模块、4个上采样模块和瓶颈层组成。每个下采样模块由2个连续卷积层组成,卷积核大小为3×3,步长为1,采用批量归一化层,激活函数为LeakyReLU函数。同时,在每个下采样模块中进行最大池化减小图像大小。瓶颈层结构与下采样模块相同,每个上采样模块通过跳连接将图像大小放大两倍,然后由2个连续的卷积层组成,卷积层结构与下采样模块相同。在本文中,所有基于深度学习模型的权重均使用Adam算法进行优化,Adam的动量参数设置为
所提模型在PyTorch平台上实现,并使用 NVIDIA
对于评估指标,采用3个全局图像质量指标和一个局部图像质量指标。全局图像质量指标为峰值信噪比(Peak Signal-to-Noise Ratio,PSNR)和结构相似性指数(Structural Similarity,SSIM)以及均方根误差(Root Mean Square Error,RMSE)。局部图像质量指标为平均对比度(Mean Contrast,MC)。
PSNR是评价图像重建的传统质量指标,PSNR值越高,重建图像的质量越高。SSIM是根据亮度、对比度和结构来评估两幅图像相似度的指标。SSIM值越高意味着结构信息匹配越好。RMSE用于测量图像之间的误差。
MC是计算高摄取区域ROI(Region of Interest)和低摄取区域背景之间的平均对比度差异。
| $ {C}_{a}=\left\langle{\left|{A}-{B}\right|}\right\rangle $ | (7) |
式中,A为重建图像的高摄取区域ROI的平均放射性,B为距离边缘或边界周围5个低摄取区域背景的平均放射性,
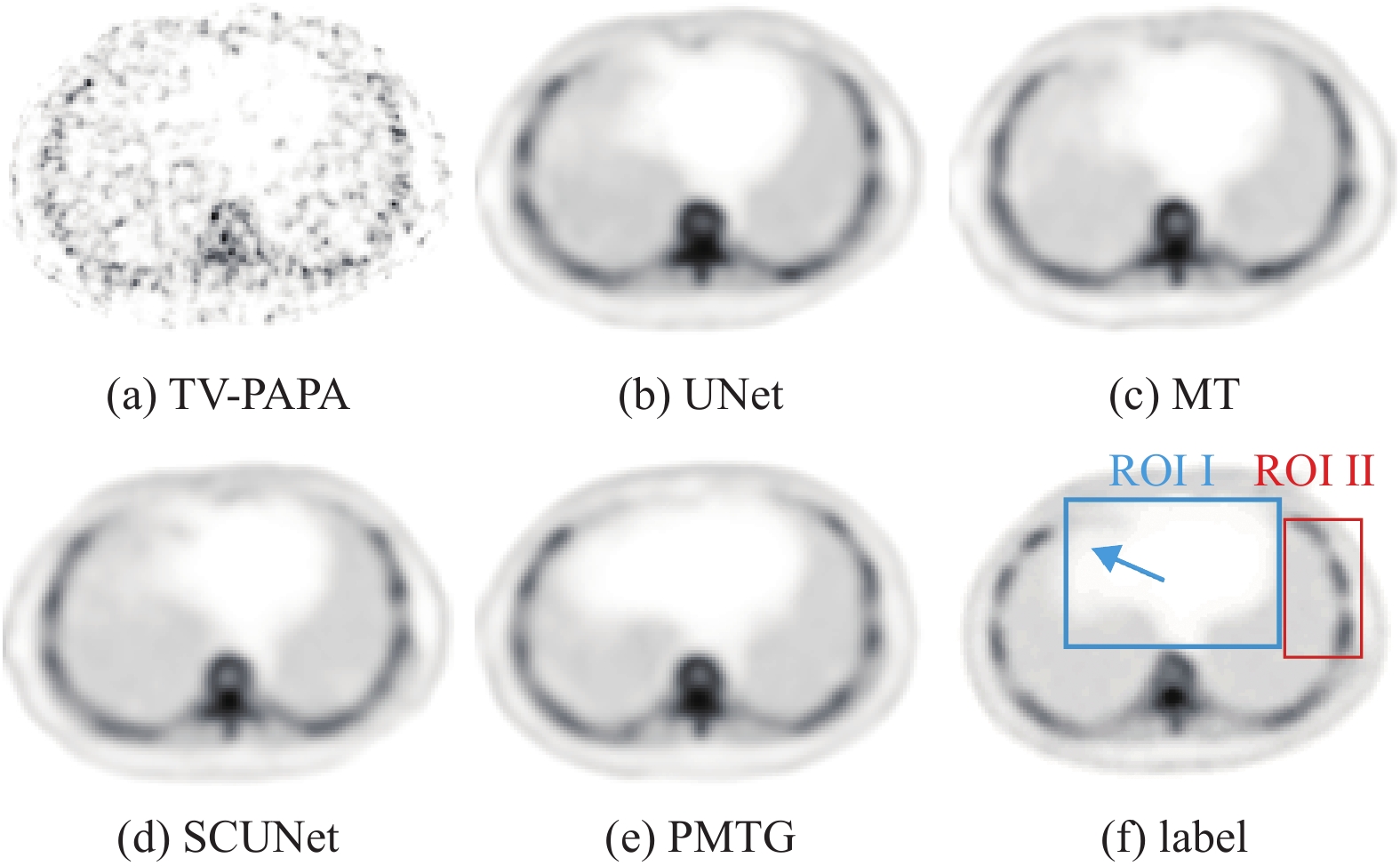
本文进行性能对比实验,验证提出的半监督学习方法优于其他对比方法。图2是使用不同方法重建的胸部图像。可以观察到TV-PAPA粗略地重建骨骼的整体轮廓,但丢失许多图像细节。相比之下,基于深度学习的方法既重建图像的整体结构,又恢复图像细节。为进一步评估不同方法的性能,选择两个ROI进行比较。对于图2(f)中标记的ROI II,可以观察到所提方法在保持骨骼结构和重建图像对比度方面优于其他方法。如图2(f)中标记的ROI I蓝色箭头所示,所提方法在获取图像特征方面相较于其他方法表现更为准确。
|
图 2 不同方法重建的胸部图像 Figure 2 The chest images reconstructed by different methods |
表2为不同方法的定量结果。所提方法在对比方法中取得最好的性能,相较于SCUNet,所提方法的PSNR提高了0.43 dB。与对比方法相比,定量结果表明所提方法取得更好的重建性能。MT方法使用不同骨干网络,性能提升并不明显。更换骨干网络并没有很大程度提升性能,是因为MT方法利用未标记数据的能力有限。相较于SCUNet作为MT方法的骨干网络,所提方法的PSNR提高了0.14 dB。
| 表 2 不同方法的定量结果 Table 2 Quantitative results of different methods |
本文进行消融研究使用有限的标记数据和大量的未标记数据,验证提出的半监督学习方法有效性。
表3为SM和PM之间使用不同度量方法的定量结果。如表3所示,使用逐像素MAE度量方法与MSE度量方法相当。相较于使用逐像素度量方法,使用GAN相似性度量方法,PSNR提高了0.21dB。由于未标记数据生成的伪标签在某种程度上是不可靠的,特别是在重建图像的器官边缘,这导致不准确的计算,从而影响模型性能。
| 表 3 不同度量方法的定量结果 Table 3 Quantitative results of different measure methods |
表4为不同方法的训练时间和内存使用情况。PM采用SCUNet网络,该网络具备学习局部和非局部能力的特性。从表4可以看出,当MT方法替换骨干网络为SCUNet,相较于MT方法,训练时间增加2.5 h,内存使用量增加14674 MB。所提方法相较于MT方法,训练时间增加1.9 h,内存使用量增加661 MB。所提方法使用未标记数据以提高性能,同时训练时间和资源消耗大大减少。
| 表 4 不同方法的训练时间和内存使用 Table 4 Training time and memory usage among different methods |
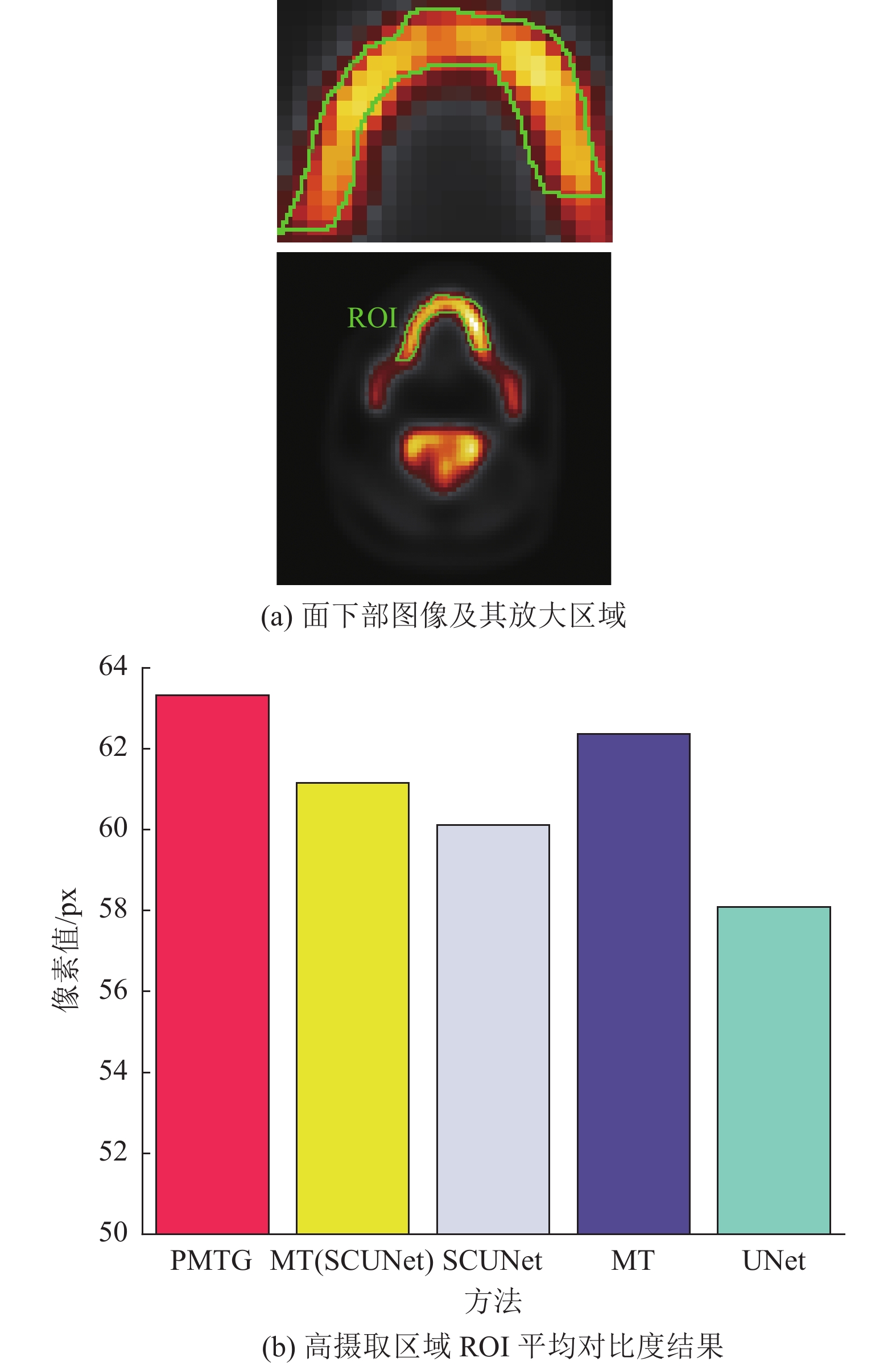
图3(a)中的放大区域表示颚骨的一部分,本文使用MC指标定量评估局部区域对比度恢复质量。在10个XCAT模体的面下部切片中选择了颚骨上的高摄取区域ROI(如图3(a)中的绿色ROI所示),高摄取区域ROI可能涵盖病灶区域,这为临床疾病应用提供潜在的帮助。本文计算10个XCAT模体的高摄取区域ROI和低摄取圆形背景之间的平均对比度差,并在图3(b)中绘制不同方法的MC结果,评估其局部对比度恢复能力。可以观察到,与其他方法相比,所提方法在高摄取区域ROI提供最佳的局部对比度。评估结果表明,与本文其他方法相比,所提方法可以有效地提高重建图像的局部对比度。
|
图 3 不同方法重建的面下部图像和高摄取区域ROI平均对比度结果 Figure 3 A basifacial image reconstructed by different methods and the comparative mean contrast results for the high-uptake ROI |
消融实验结果表明,所提GAN损失函数有助于提高重建性能。此外,本文验证GAN损失与其他逐像素度量损失之间的有效性。本文采用SCUNet作为预训练模型,通过有效地融合局部特征和非局部特征,实现最佳的图像重建性能。此外,通过实验证实得知,若采用过于强大的PM,由于其对图像细节的表现过多,可能导致SM的学习能力不足。
3 总结本文提出一种带有GAN损失的预训练平均教师方法,以提高低剂量SPECT重建图像的质量。在传统的平均教师框架中加入基于SCUNet的预训练模型,同时利用GAN损失函数度量学生模型和预训练模型之间的相似度。实验结果表明,所提方法在不影响重建图像质量的前提下,能够降低SPECT成像所需的示踪剂剂量。所提方法有助于减轻大规模医学图像标注的需求。
| [1] |
ZHANG J H, LI S, KROL A, et al. Infimal convolution-based regularization for SPECT reconstruction[J].
Medical Physics, 2018, 45: 5397-5410.
DOI: 10.1002/mp.13226. |
| [2] |
KROL A, LI S, SHEN L X, et al. Preconditioned alternating projection algorithms for maximum a posteriori ECT reconstruction[J].
Inverse Problems, 2012, 28: 115005.
DOI: 10.1088/0266-5611/28/11/115005. |
| [3] |
JIANG Y, LI S, XU Y S. A higher-order polynomial method for SPECT reconstruction[J].
IEEE Transactions on Medical Imaging, 2018, 38: 1271-1283.
|
| [4] |
RAVISHANKAR S, YE J C, FESSLER J A. Image reconstruction: from sparsity to data-adaptive methods and machine learning[J].
Proceedings of the IEEE, 2019, 108: 86-109.
|
| [5] |
CHEN X, ZHOU B, XIE H, et al. DuDoSS: deep-learning-based dual-domain sinogram synthesis from sparsely sampled projections of cardiac SPECT[J].
Medical Physics, 2023, 50: 89-103.
DOI: 10.1002/mp.15958. |
| [6] |
LI S, PENG L, LI F, et al. Low-dose sinogram restoration enabled by conditional GAN with cross-domain regularization in SPECT imaging[J].
Mathematical Biosciences and Engineering, 2023, 20: 9728-9758.
DOI: 10.3934/mbe.2023427. |
| [7] |
叶文权, 李斯, 凌捷. 基于多级残差U-Net的稀疏SPECT图像重建[J].
广东工业大学学报, 2023, 40(1): 61-67.
YE W Q, LI S, LING J. Sparse-view SPECT image reconstruction based on multilevel-residual U-Net[J]. Journal of Guangdong University of Technology, 2023, 40(1): 61-67. |
| [8] |
DONG X, VEKHANDE S, CAO G. Sinogram interpolation for sparse-view micro-CT with deep learning neural network[C]//CHEN G H. Medical Imaging 2019: Physics of Medical Imaging. San Diego: SPIE, 2019, 10948: 692-698.
|
| [9] |
YUAN H, JIA J, ZHU Z. SIPID: a deep learning framework for sinogram interpolation and image denoising in low-dose CT reconstruction[C]//AMINI A. 2018 IEEE 15th International Symposium on Biomedical Imaging (ISBI 2018) . Washington: IEEE, 2018: 1521-1524.
|
| [10] |
SHAO W, ROWE S P, DU Y. SPECTnet: a deep learning neural network for SPECT image reconstruction[J].
Annals of Translational Medicine, 2021, 9(9): 819.
DOI: 10.21037/atm-20-3345. |
| [11] |
LI Z, DEWARAJA Y K, FESSLER J A. Training end-to-end unrolled iterative neural networks for SPECT image reconstruction[J].
IEEE Transactions on Radiation and Plasma Medical Sciences, 2023, 7(4): 410-420.
DOI: 10.1109/TRPMS.2023.3240934. |
| [12] |
SHAO W, POMPER M G, DU Y. A learned reconstruction network for SPECT imaging[J].
IEEE Transactions on Radiation and Plasma Medical Sciences, 2020, 5(1): 26-34.
|
| [13] |
HÄGGSTRÖM I, SCHMIDTLEIN C R, CAMPANELLA G, et al. DeepPET: a deep encoder–decoder network for directly solving the PET image reconstruction inverse problem[J].
Medical Image Analysis, 2019, 54: 253-262.
DOI: 10.1016/j.media.2019.03.013. |
| [14] |
ZHU B, LIU J Z, CAULEY S F, et al. Image reconstruction by domain-transform manifold learning[J].
Nature, 2018, 555: 487-492.
DOI: 10.1038/nature25988. |
| [15] |
PRIBANIĆ I, SIMIĆ S D, TANKOVIĆ N, et al. Reduction of SPECT acquisition time using deep learning: a phantom study[J].
Physica Medica, 2023, 111: 102615.
DOI: 10.1016/j.ejmp.2023.102615. |
| [16] |
夏皓, 蔡念, 王平, 等. 基于多分辨率学习卷积神经网络的磁共振图像超分辨率重建[J].
广东工业大学学报, 2020, 37(6): 26-31.
XIA H, CAI N, WANG P, et al. Magnetic resonance image super-resolution via multi-resolution learning[J]. Journal of Guangdong University of Technology, 2020, 37(6): 26-31. |
| [17] |
ZHANG Z, LIANG X, DONG X, et al. A sparse-view CT reconstruction method based on combination of DenseNet and deconvolution[J].
IEEE Transactions on Medical Imaging, 2018, 37(6): 1407-1417.
DOI: 10.1109/TMI.2018.2823338. |
| [18] |
XIE H, THORN S, LIU Y H, et al. Deep-learning-based few-angle cardiac SPECT reconstruction using transformer[J].
IEEE Transactions on Radiation and Plasma Medical Sciences, 2022, 7(1): 33-40.
|
| [19] |
SHAO W, LEUNG K, POMPER M, et al. SPECT image reconstruction by a learnt neural network[J].
Journal of Nuclear Medicine, 2020, 61: 1478.
|
| [20] |
GU J, YE J C. AdaIN-based tunable CycleGAN for efficient unsupervised low-dose CT denoising[J].
IEEE Transactions on Computational Imaging, 2021, 7: 73-85.
DOI: 10.1109/TCI.2021.3050266. |
| [21] |
TARVAINEN A, VALPOLA H. Mean teachers are better role models: weight-averaged consistency targets improve semi-supervised deep learning results[C]//GUYON I. Advances in Neural Information Processing Systems 30. Long Beach: NeurIPS, 2017, 30.
|
| [22] |
YU L, WANG S, LI X, et al. Uncertainty-aware self-ensembling model for semi-supervised 3D left atrium segmentation[C]//SHEN D G. Medical Image Computing and Computer Assisted Intervention–MICCAI 2019. Shenzhen: Springer, 2019(11765) : 605-613.
|
| [23] |
CHEPLYGINA V, DE BRUIJNE M, PLUIM J P W. Not-so-supervised: a survey of semi-supervised, multi-instance, and transfer learning in medical image analysis[J].
Medical Image Analysis, 2019, 54: 280-296.
DOI: 10.1016/j.media.2019.03.009. |
| [24] |
ZHANG K, LI Y, LIANG J, et al. Practical blind image denoising via Swin-Conv-UNet and data synthesis[J]. Machine Intelligence Research, 2023: 1-14.
|
| [25] |
甘孟坤, 曾安, 张小波. 基于Swin-Unet的主动脉再缩窄预测研究[J].
广东工业大学学报, 2023, 40(5): 34-40.
GAN M K, ZENG A, ZHANG X B. Aortic re-coarctation prediction research based on Swin-Unet[J]. Journal of Guangdong University of Technology, 2023, 40(5): 34-40. |
| [26] |
RONNEBERGER O, FISCHER P, BROX T. U-net: convolutional networks for biomedical image segmentation[C]// NAVAB N. Medical Image Computing and Computer-Assisted Intervention–MICCAI 2015. Munich: Springer, 2015: 234-241.
|
| [27] |
SEGARS W P, BOND J, FRUSH J, et al. Population of anatomically variable 4D XCAT adult phantoms for imaging research and optimization[J].
Medical Physics, 2013, 40(4): 043701.
DOI: 10.1118/1.4794178. |
 2024, Vol. 41
2024, Vol. 41

