药物不良反应(Adverse Drug Reaction, ADR)是全球重要的公共卫生问题,是导致死亡的重大原因之一[1]。全球范围内因ADR导致的伤残或死亡患者每年近80万例,占所有入院患者的3.6%[2]。在美国,每年约200余万名住院患者发生严重ADR,造成5284亿美元经济损失,约占当年医疗总支出的16%[3]。我国每年也有超过250万人因ADR入院, 其中死亡人数高达19.2万人[4];2018年中国药品不良反应监测网络收到149.9万份药品不良反应/事件报告[5],且数量呈逐年增长趋势。尽管药物在被批准上市之前,经过严格试验,但由于样本数量及试验时间限制,许多严重ADR直到药物上市后才出现[6]。此外,高达50%与ADR相关的住院,可以通过避免不适当的处方来预防[7]。因此,如何有效识别和预测药物潜在的不良反应,预防ADR发生以及降低经济损失,提高临床用药的合理性和安全性,是当前智慧健康医疗领域的一个研究重点[8-9]。基于此,本文开发一种基于知识图谱嵌入和深度学习的ADR预测模型,并与多种常用基准模型及已有研究结果进行对比分析,同时检验本文预测模型的有效性和稳定性。本文的贡献可以概括如下。
(1) 本文结合知识图谱嵌入和深度学习开发了一种稳定且高效的ADR预测模型,将所有类型ADR进行统一预测,减少过往研究需要为每种ADR单独开发预测模型的冗余工作量,提高预测效率和精度。
(2) 本文通过对比评估不同嵌入策略对ADR分类模型的影响,选择最佳嵌入策略,所开发的ADR预测模型能够有效预测药物潜在的不良反应,为医生在用药时提供建议,提高患者的用药安全。
1 相关研究根据世界卫生组织的定义,药物不良反应是指在使用正常剂量的药物用于预防、诊断、治疗疾病或调节生理机能过程中,出现有害和非预期的且与用药目的无关的反应[10];且ADR可能是药物化学物质与蛋白质反应的结果[11]。早期对于ADR的研究,主要基于自发报告系统(Spontaneous Reporting Systems, SRSs) 的临床案例数据[7, 12],使用比例失衡分析[13]等方法评估药物与ADR之间的关联性和因果性,以挖掘相关药物不良反应信号。但SRSs的数据往往是不完整或不准确的,可能会导致研究结果有所偏差;此外加之数据量有限,缺乏对数据的深度挖掘,使得早期基于简单统计方法的研究结论缺乏说服力[14]。随着人工智能技术日趋成熟和生物医学数据量不断增长,一方面,研究人员基于文献、ADR报告等文本数据,结合自然语言处理技术挖掘药物潜在的不良反应[15-17];另一方面,基于药物的化学、生物学以及表型特征,使用机器学习或深度学习方法进行ADR预测研究 [18-21]。基于文本挖掘的研究常用于识别和监测相关ADR,其假定相关ADR已出现,但无法预测药物潜在的ADR;而基于药物特征和机器学习的ADR预测研究,有助于探索药物未知的ADR,这也是本文的研究主题。
机器学习相关方法能够提升ADR预测效果,但这些研究仍存在可改进的关键点:(1) 未考虑药物之间关联关系,可能导致有用信息丢失;(2) 使用大量独热编码的特征数据,而高维稀疏特征矩阵降维难度大,模型计算效率低;(3) 绝大多数需要为每种ADR单独构建分类器。而知识图谱(Knowledge Graph, KG)这种由节点和关系构成的特殊网络结构及其嵌入技术,通过将实体嵌入连续低维的特征空间,捕获特征实体之间非结构化语义关系,在不同类型信息之间实现融合和计算,能有效缓解高维稀疏特征数据带来的计算低效问题,提高分类器预测性能[22-24]。
近年来,知识图谱及其嵌入技术逐渐被应用于药物研究领域的知识发现和知识库构建,这些研究通过获取药物特征数据,构建含有不同类型节点的知识图谱,通过知识图谱嵌入技术结合分类模型进行相关研究主题的目标预测。基于 KG的ADR预测,相关典型研究如表1所示。通过文献综述,当前研究仍存在以下有待改进的要点:(1) 使用KG中未出现的“drug-ADR”组合作为ADR预测模型的负样本,但KG中不存在的“drug-ADR”组合可能只是目前尚未被发现[21];(2) 使用简单的机器学习模型;(3) 所覆盖的药物数量较少,特征局限于药物靶点和适应症,诸如酶和载体蛋白之类的重要信息尚未在先前的研究中使用。
| 表 1 相关典型研究 Table 1 Relevant typical studies |
基于此,本文采用知识图谱嵌入与深度学习相结合的方法实现ADR预测,除靶点和适应症之外,还整合了酶和载体蛋白信息构建知识图谱;并开发一个强大的深度神经网络,提高ADR的预测性能。
2 数据与方法在本文提出的方法中,参考文献[25]和[26],将药物的副作用(Side Effect) 视为ADR。鉴于结合药物的生物学特征和表型特征能够提升ADR预测模型性能[18,25],从DrugBank(v5.18) [30]和SIDER(v4.1) [31]数据库分别选择靶点(Target) 、载体(Transporter) 、酶(Enzyme) 等生物学特征和适应症(Indication) 和不良反应(ADR) 等表型特征,以及药物(drug) 作为知识图谱实体节点。然后,为规避为每种ADR构建单独分类器所增加的沉重工作量,将ADR预测视作一个统一的二分类问题,并使用“drug-ADR”组合和“drug-Indication”组合分别作为分类模型的正样本和负样本,样本标签分别记作“1”和“0”。由此开发一个基于知识图谱嵌入和深度学习的ADR预测模型,通过5次重复实验,检验卷积神经网络(Convolutional Neural Networks, CNN)模型稳定性。最后,以药物性肾功能损伤为例进行预测,并通过真实世界数据验证模型预测的有效性。具体研究思路如图1所示。
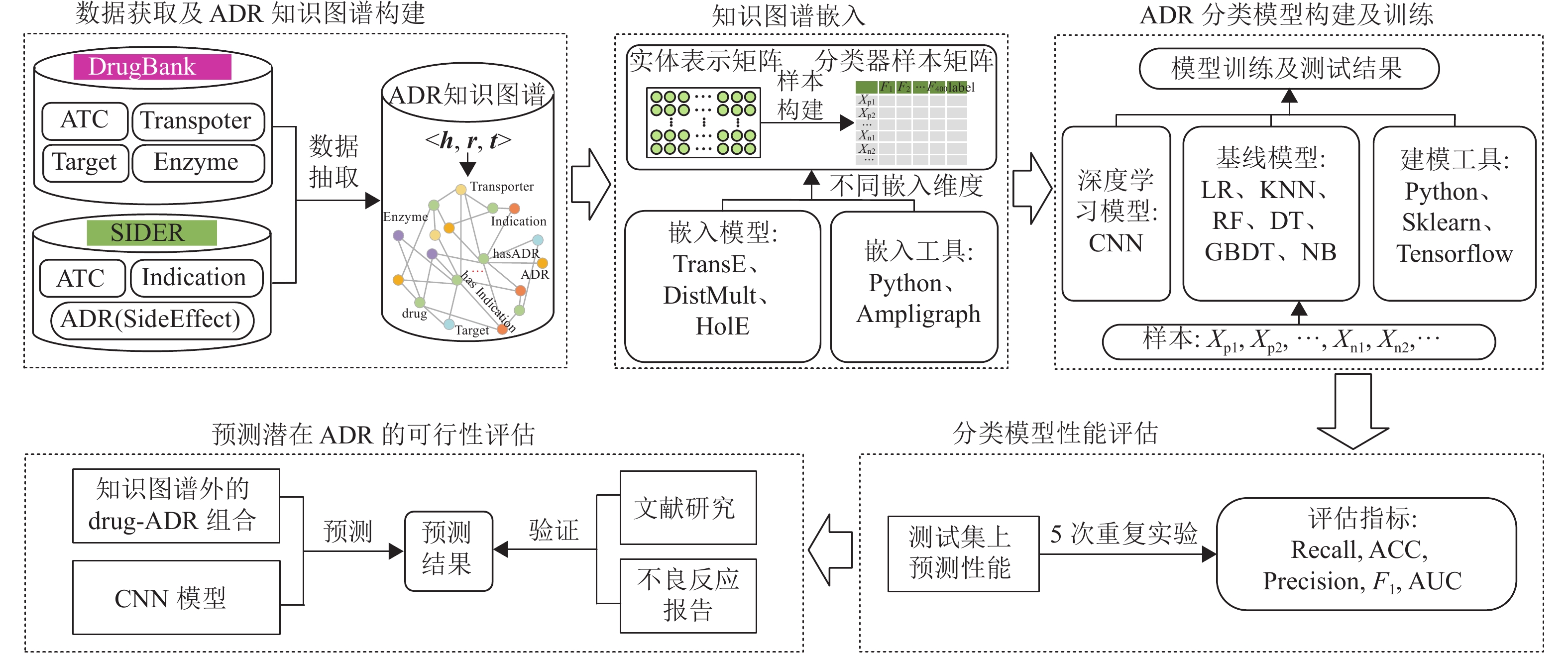
|
图 1 ADR预测研究框架 Figure 1 Research framework of ADR prediction |
DrugBank数据库涵盖丰富的生物和化学信息学资源,SIDER数据库收录了1430种药物,6000余种副作用。通过下载DrugBank中xml数据文件和SIDER中tsv文件,使用Python程序解析并获得药物的相关特征数据。根据药物解剖治疗化学代码(Anatomical Therapeutic Chemical, ATC) 整合2个数据库的相关数据,并筛选至少具有1种药物特征的药物记录。最终构建5类三元组:<drug, hasTransporter, Transporter>、<drug, hasADR, ADR>、<drug, hasEmzyme, Emzyme>、<drug, hasTarget, Target>、<drug, hasIndication, Indication>;将三元组储存至Neo4j图数据库,获得可视化知识图谱,如图2所示。该图谱共包含了7916种drug、5454种ADR以及158121个三元组,具体如表2所示。
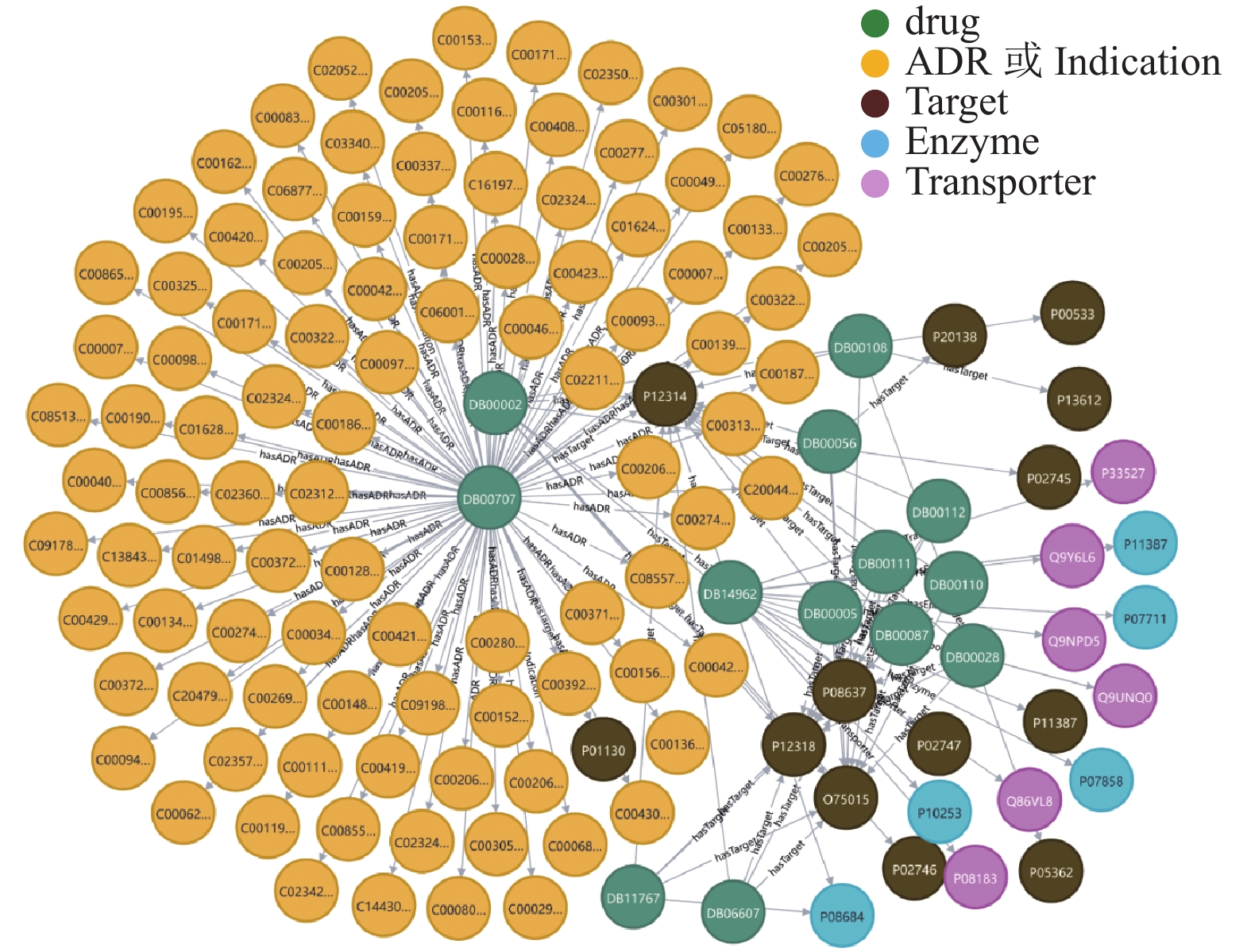
|
图 2 ADR知识图谱中的部分实体和关系 Figure 2 Local entities and relationships in the knowledge graph |
| 表 2 ADR知识图谱包含的实体、关系及其数量 Table 2 Entities, relationships and quantities included in the ADR knowledge graph |
知识图谱嵌入技术逐渐被应用于预测研究[22],其中基于张量分解的DistMult[32]模型和HolE[33]模型应用最为广泛。DistMult模型通过实体之间的双线性变换来描述实体之间的语义相关性,其中头实体和尾实体分别由向量
研究设计了一个具有2个卷积层,4个全连接层的CNN模型,如图3所示。由于ReLU激活函数计算效率和收敛速度等特性远高于sigmoid、Tanh等函数;因此,卷积层和全连接层均使用ReLU激活函数。同时,为使得每一层神经网络的输入保持相同分布和提高网络优化效率,卷积层均使用批归一化处理(Batch Normalization) ,模型具体参数如表3所示。本文使用式(1) 所示的二元交叉熵作为模型训练的损失函数,式中:
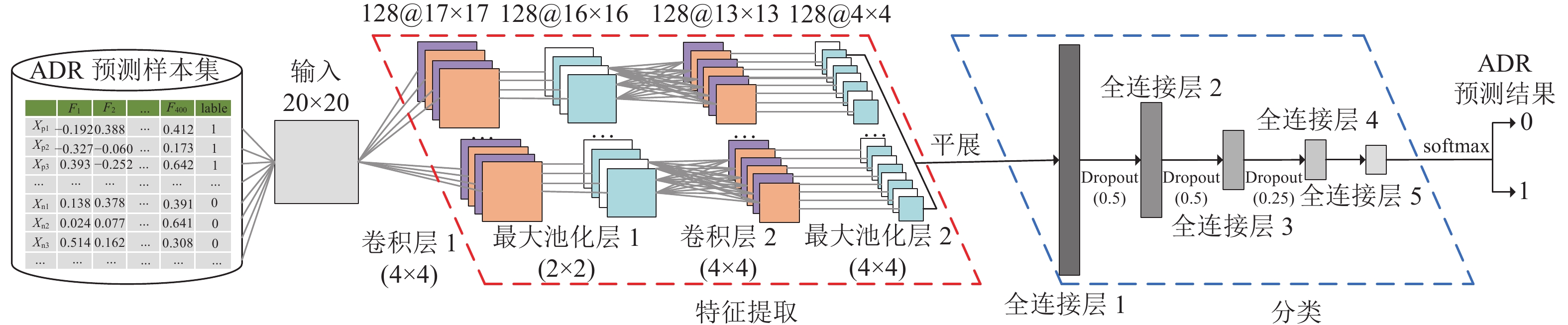
|
图 3 用于ADR预测的CNN模型结构图 Figure 3 CNN model structure diagram for ADR prediction |
| 表 3 CNN模型参数 Table 3 Parameters of CNN model |
| $ J\left(W,b\right) =-\frac{1}{n}\sum _{i}^{n}\left[{y}_{i}\mathrm{l}\mathrm{o}\mathrm{g}{\widehat{y}}_{i}+\left(1-{y}_{i}\right) \mathrm{l}\mathrm{o}\mathrm{g}\left(1-{\widehat{y}}_{i}\right) \right] $ | (1) |
采用逻辑回归(Logistic Regression, LR) 、K近邻(k-Nearest Neighbor, KNN) 、决策树(Decision Tree, DT) 、随机森林(Random Forest, RF) 、朴素贝叶斯(Naive Bayes, NB) 、梯度提升决策树(Gradient Boosting Decision Tree, GBDT) 等6种基准模型进行对比分析,上述模型被广泛应用于ADR预测 [8]。
3 实验与结果分析 3.1 模型评价指标本文采用混淆矩阵计算召回率(Recall) 、准确率(Accuracy, ACC) 、精确率(Precision, P) 、F1值(F1-Score, F1) 和曲线下面积(Areas Under the Curve, AUC) 作为模型的评价指标。
3.2 知识图谱嵌入及样本向量表示嵌入操作基于Python语言,调用AmpliGraph工具库实现。在嵌入操作前,需要确定ADR预测模型的训练集和测试集;训练集被用于知识图谱嵌入操作和ADR预测模型训练,测试集被用于评估ADR预测模型的预测性能。
知识图谱中正样本为119233个,负样本为12498个(见表4) 。由于正负样本数量相差1个数量级,故以负样本的总数为基础,按照9:1的比例,将负样本随机划分为11249个训练样本和1249个测试样本,并随机从正样本中取1249个作为测试样本;则测试集包含正负样本各1249个;训练集包括117984个正样本和11249个负样本。为解决训练集样本不平衡问题,采用过采样(Oversampling) 将负样本复制10倍。样本划分结果如表4所示。
| 表 4 用于知识图谱嵌入以及ADR分类器训练和测试的数据 Table 4 Data used for KG embedding and ADR classifier training and testing |
本文在知识图谱嵌入过程中,采用不同的嵌入策略获得嵌入向量。并分别使用
| 表 5 ADR分类器部分样本的表示向量(DistMult, dim=20) Table 5 Representation vector of partial samples of ADR classifier(DistMult, dim=20) |
本文通过组合不同嵌入模型和不同嵌入维度(10至800) ,探索不同嵌入策略对基准ADR分类模型在测试集上预测性能的影响。如图4所示,在不同嵌入模型下,随着嵌入维度增大,各基准模型在测试集上的AUC值也逐渐增大;并且ACC、F1指标值也存在不同程度的波动增大;Recall值没有明显增大,相对稳定。然而,当嵌入维度大于400时,各基准模型的AUC、ACC、F1指标值趋于稳定。通过综合分析,适当增大嵌入维度,能够在一定程度上提升ADR分类模型的预测性能。同时,为避免分类器出现过拟合和实验硬件设备资源浪费,本文选择400维为最佳嵌入维度,并结合CNN模型进行ADR预测。
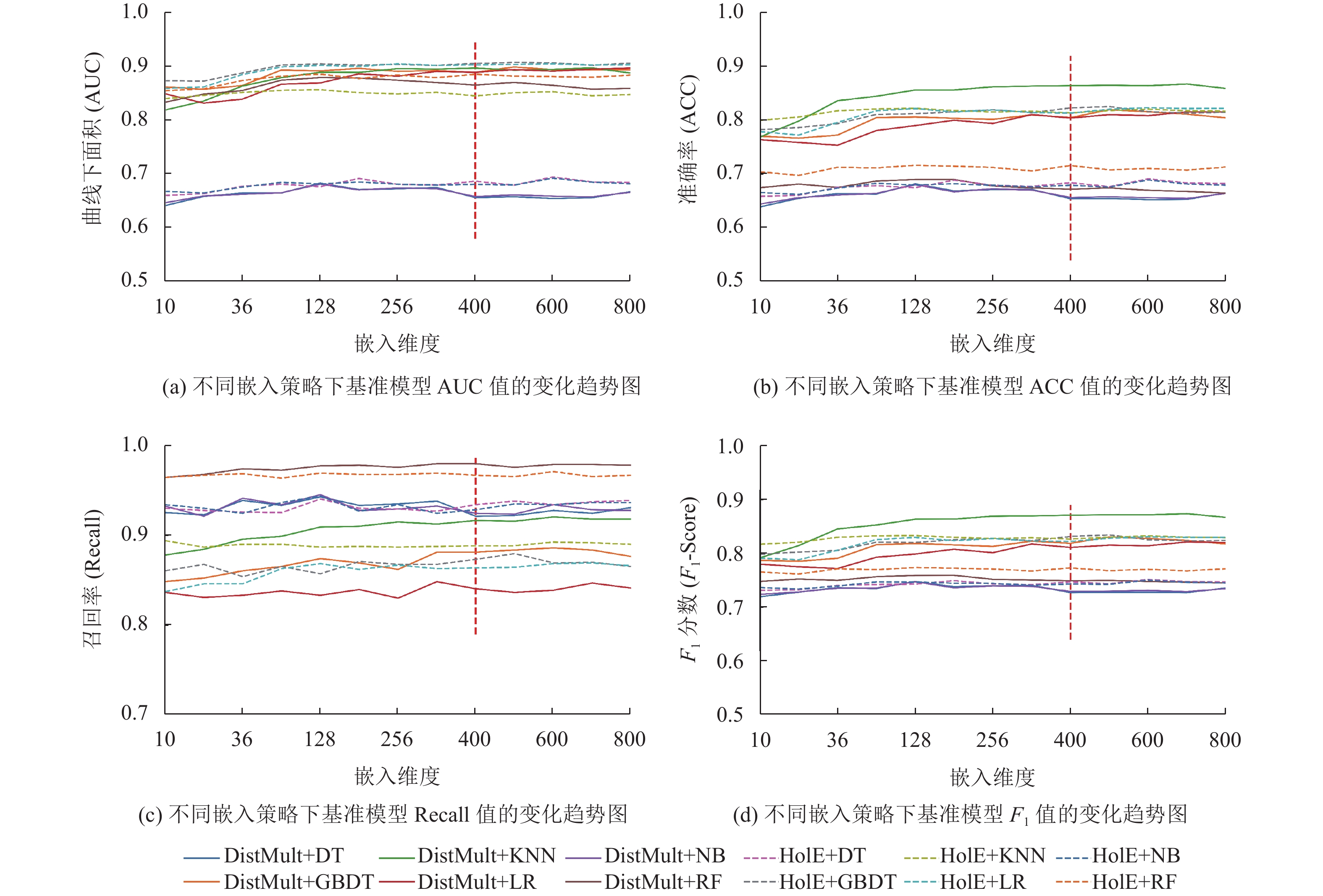
|
图 4 不同嵌入维度下各基准ADR分类模型在测试集上的性能表现 Figure 4 The performance of each baseline ADR classification model on the test set with different embedding dimensions |
基于Python语言,使用scikit-learn和深度学习框架Tensorflow2.0开发ADR分类模型,6种基准模型将使用默认参数。固定嵌入维度为400维,通过嵌入模型获得样本的表示向量,并将其输入到ADR分类模型进行训练和预测,各分类模型在测试集上的预测结果如表6所示。综合分析发现,在DistMult嵌入模型下,CNN分类模型在测试集上的AUC值为0.942,优于所有基准模型。
| 表 6 嵌入维度为400时各ADR预测模型比较 Table 6 Comparison of ADR prediction models when the embedding dimension is 400 |
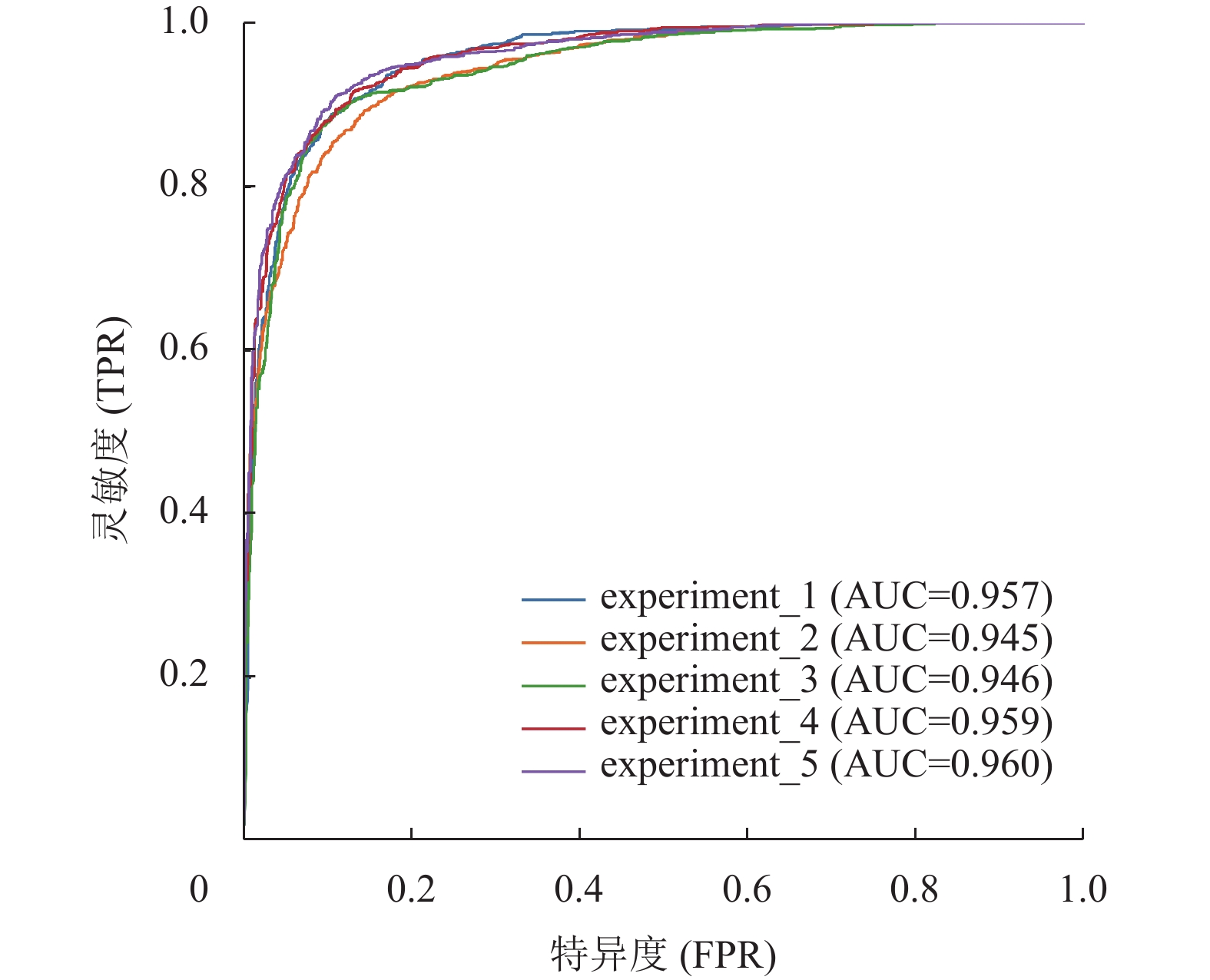
研究采用5次重复实验,评估CNN模型的稳定性。具体步骤:(1) 设定随机种子,构建训练集和测试集;(2) 采用“DistMult模型+400维”组合策略进行嵌入操作;(3) 将所得样本表示向量用于CNN分类模型训练和预测。结果如表7所示,本文CNN模型的AUC平均值为0.957,比Zhang等[26]的研究(平均AUC=0.863)高出0.094,提升了10.89%;F1均值为0.890,Recall均值为0.913,各指标值波动较小。同时,ROC曲线(见图5)表现也非常稳定,表明本文所开发的CNN模型具有较高稳定性。
| 表 7 5次重复实验CNN模型在测试集上的表现 Table 7 The performance of the CNN model on the test set for five repeated experiments |
|
图 5 CNN模型5次重复实验在测试集上的ROC曲线 Figure 5 ROC curve of five repeated experiments of CNN model |
本文通过现实世界数据,对 CNN模型的有效性进行检验。以“肾损伤”或“kidney injury”为关键词,在中国知网、PubMed等文献数据库中随机检索相关的ADR研究,获得5个未被SIDER数据库收录的“
| 表 8 使用CNN模型对文献中的drug-ADR组合的预测结果 Table 8 Prediction results of drug-ADR pairs in literature through CNN model |
由于目前缺乏用于检验ADR预测模型性能的标准数据集,本文将从所覆盖的药物、ADR种类数量,以及预测模型的AUC值等方面,与相关典型研究进行对比(见表9)。通过对比分析,本文开发的CNN模型的AUC高于相关研究所提供的结果,预测性能更好。同时,本文的实验数据集包含7916种药物和5454种ADR,所覆盖的药物信息多于绝大多数同类研究。此外,以往的研究大多需要针对每个ADR单独构建预测模型,增加了ADR预测任务的工作量;相比之下,本文通过构建药物知识图谱,使用知识图谱嵌入技术将药物、ADR等实体编码成特征向量;最终使用一个统一的CNN模型对各“
| 表 9 与现有典型研究对比 Table 9 Comparison with advanced ADR prediction models |
针对既往ADR预测模型研究的预测精度低、需要为每种ADR单独构建分类器导致工作量繁重等问题,本文将不同类型ADR预测简化为一个二分类问题,并开发一个基于知识图谱嵌入和深度学习的CNN预测模型。本文的预测模型比已有研究的预测精度更高,此外通过真实世界数据验证模型预测结果的有效性和可行性,有望在临床安全用药中发挥重要的辅助作用。下一步研究将考虑使用类似的方法,对中成药潜在的不良反应进行研究;或以患者为中心,评估导致临床患者发生ADR的潜在风险因素,并预测患者在具体用药情况下出现特定ADR的风险程度;或探究不同场景下的ADR预测模型。
| [1] |
WESTER K, JONSSON A K, SPIGSET O, et al. Incidence of fatal adverse drug reactions: a population based study[J].
British Journal of Clinical Pharmacology, 2008, 65(4): 573-579.
DOI: 10.1111/j.1365-2125.2007.03064.x. |
| [2] |
COCOS A, FIKS A G, MASINO A J. Deep learning for pharmacovigilance: recurrent neural network architectures for labeling adverse drug reactions in Twitter posts[J].
Journal of the American Medical Informatics Association, 2017, 24(4): 813-821.
DOI: 10.1093/jamia/ocw180. |
| [3] |
JONATHAN H W, TERRY M, JAN D H. Cost of prescription drug-related morbidity and mortality[J].
Annals of Pharmacotherapy, 2018, 52(9): 829-837.
DOI: 10.1177/1060028018765159. |
| [4] |
朱笑笑, 杨尊琦, 刘婧. 基于Bi-LSTM和CRF的药品不良反应抽取模型构建[J].
数据分析与知识发现, 2019, 3(2): 90-97.
ZHU X X, YANG Z Q, LIU J. Construction of an adverse drug reaction extraction model based on Bi-LSTM and CRF[J]. Data Analysis and Knowledge Discovery, 2019, 3(2): 90-97. |
| [5] |
国家药品不良反应监测年度报告(2018年) [J]. 中国药物评价, 2019, 36(6) : 476-480
|
| [6] |
郭凯. 基于深度学习和语义分析的药物不良反应发现[D]. 大连: 大连理工大学, 2017.
|
| [7] |
朱嘉静. 基于机器学习的药物不良反应关键问题研究[D]. 成都: 电子科技大学, 2020.
|
| [8] |
LEE C Y, CHEN Y P. Machine learning on adverse drug reactions for pharmacovigilance[J].
Drug Discovery Today, 2019, 24(7): 1332-1343.
DOI: 10.1016/j.drudis.2019.03.003. |
| [9] |
LEE C Y, CHEN Y P. Prediction of drug adverse events using deep learning in pharmaceutical discovery[J].
Briefings in Bioinformatics, 2020, 22(2): 1884-1901.
|
| [10] |
World Health Organization. International drug monitoring, the role of national centres: report of WHO Technical Group[R]. Geneva: WHO, 1972.
|
| [11] |
RING J, BROCKOW K. Adverse drug reactions: mechanisms and assessment[J].
European Surgical Research, 2002, 34(1-2): 170-175.
DOI: 10.1159/000048905. |
| [12] |
谈志远, 赵荣生. 人工智能技术在药物不良反应监测与上报中应用的研究进展[J].
临床药物治疗杂志, 2019, 17(2): 23-27.
TAN Z Y, ZHAO R S. Progress of studies of artificial intelligence in surveillance and report of adverse drug reactions[J]. Clinical Medication Journal, 2019, 17(2): 23-27. DOI: 10.3969/j.issn.1672-3384.2019.02.006. |
| [13] |
赵霞, 陈瑶, 廖俊, 等. 基于医药大数据的药品不良反应信号挖掘探讨[J].
中华医院管理杂志, 2017, 33(5): 4.
ZHAO X, CHEN Y, LIAO J, et al. Signal mining for adverse drug reactions based on healthcare big data: methodology and applications[J]. Chinese Journal of Hospital Administration, 2017, 33(5): 4. DOI: 10.3760/cma.j.issn.1000-6672.2017.05.016. |
| [14] |
VOSS E A, BOYCE RD, RYAN P B, et al. Accuracy of an automated knowledge base for identifying drug adverse reactions[J].
Journal of Biomedical Informatics, 2017, 66: 72-81.
DOI: 10.1016/j.jbi.2016.12.005. |
| [15] |
陈瑶, 吴红, 葛卫红, 等. 基于深度学习模型的我国药品不良反应报告实体关系抽取研究[J].
中国药科大学学报, 2019, 50(6): 753-759.
CHEN Y, WU H, GE W H, et al. Research on entity relation extraction of Chinese adverse drug reaction reports based on deep learning method[J]. Journal of China Pharmaceutical University, 2019, 50(6): 753-759. DOI: 10.11665/j.issn.1000-5048.20190617. |
| [16] |
申晨, 林鸿飞. 基于图嵌入的社交媒体药物不良反应事件检测方法[J].
大连理工大学学报, 2020, 60(5): 547-554.
SHEN C, LIN H F. Detection method of adverse drug events from social media based on graph embedding[J]. Journal of Dalian University of Technology, 2020, 60(5): 547-554. DOI: 10.7511/dllgxb202005011. |
| [17] |
仲雨乐, 马诗雯, 陆豪杰, 等. 基于机器学习的药品不良反应实体识别研究综述[J].
软件工程, 2022, 25(8): 1-6.
ZHONG Y L, MA S W, LU H J, et al. Survey of research on entity recognition of adverse drug reaction based on machine learning[J]. Software Engineering, 2022, 25(8): 1-6. DOI: 10.19644/j.cnki.issn2096-1472.2022.008.001. |
| [18] |
LIU M, WU Y H, CHEN Y K, et al. Large-scale prediction of adverse drug reactions using chemical, biological, and phenotypic properties of drugs[J].
Journal of the American Medical Informatics Association, 2012, 19(e1): e28-35.
DOI: 10.1136/amiajnl-2011-000699. |
| [19] |
VILAR S, HRIPCSAK G. The role of drug profiles as similarity metrics: applications to repurposing, adverse effects detection and drug-drug interactions[J].
Briefings in Bioinformatics, 2017, 18(4): 670-681.
|
| [20] |
MUNOZ E, NOVACEK V, VANDENBUSSCHE P Y. Using drug similarities for discovery of possible adverse reactions[J]. Amia Annual Symposium Proceedings, 2016, 2016: 924-933
|
| [21] |
WANG C S, LIN P J, CHENG C L, et al. Detecting potential adverse drug reactions using a deep neural network model[J].
Journal of Medical Internet Research, 2019, 21(2): e11016.
DOI: 10.2196/11016. |
| [22] |
DAI Y F, WANG S P, NEAL N, et al. A survey on knowledge graph embedding: approaches, applications and benchmarks[J].
Electronics, 2020, 9(5): 750-778.
DOI: 10.3390/electronics9050750. |
| [23] |
ZENG X X, TU X Q, LIU Y S, et al. Toward better drug discovery with knowledge graph[J]. Current Opinion in Structural Biology, 2022, 72: 114-126
|
| [24] |
SHTAR G, ROKACH L, SHAPIRA B. Detecting drug-drug interactions using artificial neural networks and classic graph similarity measures[J].
PloS One, 2019, 14(8): e0219796.
DOI: 10.1371/journal.pone.0219796. |
| [25] |
JOSHI P, MASILAMANI V, MUKHERJEE A. A knowledge graph embedding based approach to predict the adverse drug reactions using a deep neural network[J].
Journal of Biomedical Informatics, 2022, 132: 104122-104131.
DOI: 10.1016/j.jbi.2022.104122. |
| [26] |
ZHANG F, SUN B, DIAO X L, et al. Prediction of adverse drug reactions based on knowledge graph embedding[J].
BMC Medical Informatics and Decision Making, 2021, 21(1): 38-48.
DOI: 10.1186/s12911-021-01402-3. |
| [27] |
WANG M, MA X Y, SI J W, et al. Adverse drug reaction discovery using a tumor-biomarker knowledge graph[J]. Frontiers in Genetics, 2021, 11: 625659
|
| [28] |
DEY S, LUO H, FOKOUE A, et al. Predicting adverse drug reactions through interpretable deep learning framework[J].
BMC Bioinformatics, 2018, 19(Suppl 21): 476-488.
|
| [29] |
BEAN D M, WU H H, IQBAL E, et al. Knowledge graph prediction of unknown adverse drug reactions and validation in electronic health records[J].
Scientific Reports, 2017, 7(1): 16416.
DOI: 10.1038/s41598-017-16674-x. |
| [30] |
WISHART D S, FEUNANG Y D, GUO A C, et al. DrugBank 5.0: a major update to the DrugBank database for 2018[J].
Nucleic Acids Research, 2018, 46(D1): D1074-D1082.
DOI: 10.1093/nar/gkx1037. |
| [31] |
KUHN M, LETUNIC I, JENSEN L J, et al. The SIDER database of drugs and side effects[J].
Nucleic Acids Research, 2016, 44(D1): D1075-D1084.
DOI: 10.1093/nar/gkv1075. |
| [32] |
YANG B S, YIH W T, HE X D, et al. Embedding entities and relations for learning and inference in knowledge bases[EB/OL]. arXiv: 1412.6575 (2015-08-29) [2023-02-22]. https://doi.org/10.48550/arXiv.1412.6575.
|
| [33] |
NICKEL M, LORENZO R, TOMASO P. Holographic embeddings of knowledge graphs[C] //Thirtieth AAAI Conference on Artificial Intelligence. California: AAAI. 2016: 1955-1961.
|
| [34] |
高飞萌, 宋智慧. 艾司奥美拉唑镁肠溶片致肾损伤[J].
药物不良反应杂志, 2020, 22(6): 381-382.
GAO F M, SONG Z H. Kidney injury caused by esomeprazole magnesium enteric-coated tablets[J]. Adverse Drug Reactions Journal, 2020, 22(6): 381-382. DOI: 10.3760/cma.j.cn114015-20200305-00219. |
| [35] |
DHAKRIT R, IVES A L, HARRIOTT N G, et al. Comparative incidence of acute kidney injury in patients on vancomycin therapy in combination with cefepime, piperacillin-tazobactam or meropenem[J].
Journal of Chemotherapy (Florence, Italy), 2021, 34(2): 1-7.
|
| [36] |
NIITSU T, HAYASHI T, UCHIDA J, et al. Drug-induced kidney injury caused by osimertinib: report of a rare case[J].
Nephron, 2021, 146(1): 58-63.
|
| [37] |
WAKABAYASHI K, YAMAMOTO S, HARA S, et al. Nivolumab-induced membranous nephropathy in a patient with stage IV lung adenocarcinoma[J].
CEN Case Rep, 2022, 11(2): 171-176.
DOI: 10.1007/s13730-021-00645-3. |
| [38] |
苏伟, 苏培. 碘海醇注射液致患者获得性急性肾功能衰竭的不良反应[J].
中国生化药物杂志, 2016, 36(10): 92-94.
SU W, SU P. Effect of adverse reaction of acquired acute renal failure caused by iohexol injection[J]. Chinese Journal of Biochemical and Pharmaceuticals, 2016, 36(10): 92-94. |
 2024, Vol. 41
2024, Vol. 41

