命名实体识别(Named Entity Recognition,NER)是自然语言处理中的核心任务之一,目的是从一段序列文本中提取出实体,并且对实体进行正确的分类。例如在“细胞减少与肺内病变程度及肺内炎性病变吸收程度密切相关”这段文字中,出现了两个表示疾病的命名实体,分别是“肺内病变”和“肺内炎性病变”,还出现了表示身体部位的命名实体“细胞”,此外还有一个临床表现类型的命名实体“细胞减少”。
医学命名实体识别任务是对电子病历中的医学实体进行自动识别和分类,这些实体在自然语言处理的许多下游任务中发挥着重要的作用,比如信息检索、推荐系统、知识图谱[1]等。
在基于深度学习[2]的医学命名实体识别方法中,Wu等[3]提出了一个两阶段模型,首先预测句子中的实体,然后用Transformer网络结合上下文信息表示,并引入多尺度的句子表示再次预测实体类型。Wang等[4]将医学命名实体识别任务分解为两个子任务,并引入超图模型,首先提取所有可能的实体组合,然后预测实体组合之间的关系并分类。但超图模型在模型训练过程中,容易出现误差传播问题。因此,人们提出了基于跨度的方法,避免误差传播,这一类方法在预测实体时,只需要预测实体在字符中的开始位置、结束位置和实体的类别,实体相互是独立的。Li等[5]提出了能较好地识别命名实体的模型,首先枚举句子中所有的字符跨度,初步确定跨度的实体类型,再识别出实体跨度之间的关系,继而预测出文本中所有的医学命名实体。Gu等[6]使用两个模块来抽取实体,分别是规则感知模块和规则无感模块,用规则感知模块捕获跨度内部的规律性,确定实体的类型,通过规则无感模块定位实体的边界,缓解对于跨度内部规律性的过度关注,并结合正交空间,进一步加强模块提取不同方面特征的能力。Luan等[7]使用多任务学习,用动态构造的跨度图来识别命名实体,通过枚举所有可能的跨度,识别出句子中所有的实体。Yu等[8]基于图的依存分析方法,引入双仿射模型做分类器,在字符的开始和结束位置做标记,使用这些标记枚举所有的实体。
Wang 等[9]结合数据驱动的深度学习方法和知识驱动的字典方法,引入医学领域的字典知识,提出了扩展的LSTM模型,在医学数据集上取得了不错的结果。Li等[10]利用BERT对上下文嵌入向量的提取能力,结合汉字的字根特征和字典知识,构建了一个用于医学命名实体识别的模型。Li等[11]将命名实体识别任务转换为阅读理解任务,用回答问题的方式识别出文本中的命名实体。Pan等[12]使用语义引导的注意力方法,对标签和文本分别进行编码,并且将标签知识融入到文本中,从而改善了Li等[11]提出的模型的效率。Hang等[13]提出了一种针对命名实体识别任务的“序列−序列”模型框架,该框架将命名实体识别问题转化为实体跨度生成问题。Shen等[14]提出了一个并行查询网络模型,能够并行地预测输入文本中所有的命名实体,模型在训练过程中自动学习与实体类型和实体位置相关的医学知识,不需要借助外部知识构造特征。Li等[15]提出了融入词汇信息的Transformer,把医学领域的专业词汇作为模型的输入,并且在一个网格结构中构建了新的位置编码。陈明等[16]设计的混合神经网络模型,利用自注意力、线性注意力和改进的WLSTM网络提取多层次的语义特征,将特征融合后,利用条件随机场识别出实体。梁倬骞等[17]从词语属性描述、词语关系组织和相关知识链接3个维度构建财务报告领域本体,将非结构化的信息提取为结构化的信息并表示。Liu等[18]提出基于MRC模型扩展后的模型,该方法通过图注意力网络建模标签之间的关系,将标签信息和文本信息相融合,进一步改善了MRC模型的效果。
以上的这些模型在提取全局特征和局部特征方面效果显著,但是忽略了医学命名实体之间隐含的关联性,以及医学命名实体特征内在的联系。针对以上问题,本文提出了一个模型,将命名实体识别转换为集合查询任务,本文模型一共包括3个部分:文本编码器、实体集合解码器和基于二分图匹配的损失函数。首先,文本编码器从输入的句子中获取上下文信息,并引入实体查询矩阵。再利用实体集合解码器对上下文信息和实体查询矩阵进行预测,解码预测实体的开始位置、结束位置和实体类别。最后,利用二分图的匹配算法,计算出模型训练过程中的预测损失。本文提出的模型在3个数据集上进行实验,并通过和其他模型的对比,验证了本文模型的有效性。
1 基于门控注意力单元的模型 1.1 模型整体结构本文提出的模型由3个部分组成,分别是文本编码器、实体集合解码器和基于二分图匹配的损失函数。整体结构如图1所示。
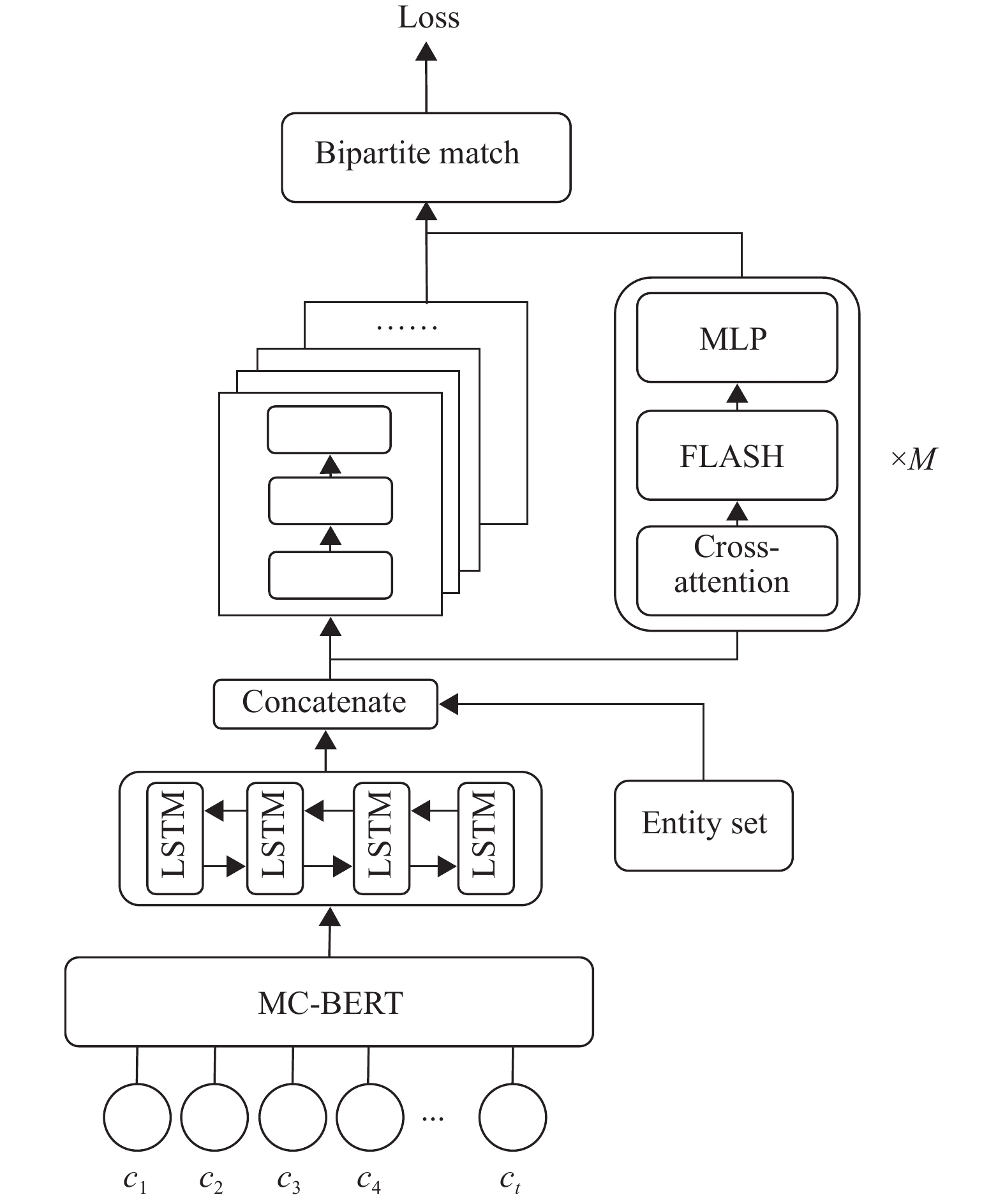
|
图 1 模型框架 Figure 1 The architecture of the proposed model |
与英文文本相比,中文命名实体识别中的汉字字符的边界不清晰,并且需要结合中文分词和语法分析才能准确识别命名实体。由于一般领域数据集的实体分布与生物医学文献的实体分布情况不同,所以很难估计一般领域的预训练模型在生物医学文本数据集上的性能。在一般领域和医学领域中有很多相同的词语、短语,但是它们属于不同的实体类型。例如,“腹部”在一般领域中可以表示为器官、位置,在医学文本中则可以作为解剖部位的医学实体。因此,本文采用生物医学领域专用的MC-BERT模型对命名实体进行识别。
MC-BERT能够处理医学领域的长尾概念和术语,并且能更好地适应生物医学中文语料库。MC-BERT首先使用不同的掩码生成器来掩盖词元的范围,而不仅仅是随机的;其次引入了全实体掩码和全跨度掩码,全实体掩码是指在医学文本数据集中生成的候选医学实体,使用医学知识图谱对生物医学领域的实体进行筛选,包括检查、治疗等。最后进行全实体掩码,并将医学实体知识注入到模型学习过程中。全跨度掩码使用完整的跨度掩码为模型注入细粒度的生物医学知识,例如,短语“肚子有点疼”和实体“腹痛”具有相同的意思。如果只使用全实体掩码则不能将上述的短语知识注入到模型中,因此在训练过程中,引入全跨度掩码学习,将输入文本中所有的短语字符都进行遮盖操作,因此模型在训练过程中能够学习到短语和实体的先验知识,MC-BERT通过引导的方式让模型明确地学习医学领域的知识信息,这使得MC-BERT模型在生物医学领域具有较好的适应性。
本文使用MC-BERT和BiLSTM作为模型的编码器。模型输入文本最大长度为510个字符,对于超过最大长度的文本,在标点符号处对文本进行截断,将超过最大长度的文本部分,作为一段新的输入文本,最大程度保留数据的完整性。对于中文文本,以字为最小单元对文本内容进行拆分,并且在文本的开头和结尾部分添加上“CLS”和“SEP”两个特殊字符作为标记。对于命名实体识别任务,针对输入的序列文本
| $ {\overrightarrow {\boldsymbol{H}} _{i}} = {{\rm{LST}}}{{{\rm{M}}}_{{\rm{f}}}}({{\boldsymbol{x}}_{i}},{\overrightarrow {\boldsymbol{H}} _{{i + 1}}};{\theta _{{\rm{f}}}}) $ | (1) |
| $ {\overleftarrow {\boldsymbol{H}} _{i}} = {{\rm{LST}}}{{{\rm{M}}}_{{\rm{b}}}}({{\boldsymbol{x}}_{i}},{\overleftarrow {\boldsymbol{H}} _{{i + 1}}};{\theta _{{\rm{b}}}}) $ | (2) |
| $ {{\boldsymbol{H}}_{i}} = {\overleftarrow {\boldsymbol{H}} _{i}} \oplus {\overrightarrow {\boldsymbol{H}} _{i}} $ | (3) |
式中:
本文提出的实体集合解码器包括交叉注意力机制和FLASH结构,其中FLASH结构由两层门控注意力单元组成。
1.3.1 交叉注意力为了加强模型对医学词汇的敏感性,本文采用交叉注意力机制,计算H和查询矩阵之间的交互性,将编码器中提取的医学命名实体信息通过交叉注意力的方式融入到查询矩阵中,提升模型对于医学命名实体的识别效果。
将实体查询矩阵
| $ {{\rm{Attention}}}({\boldsymbol{Q}},{\boldsymbol{K}},{\boldsymbol{V}}) = {\rm{{softmax}}}\left(\frac{{{\boldsymbol{Q}}{{\boldsymbol{K}}^{{\rm{T}}}}}}{{\sqrt {{d_{{\boldsymbol{K}}}}} }}\right) {\boldsymbol{V}} $ | (4) |
式中:
| $ {{\rm{hea}}}{{{\rm{d}}}_i} = {{\rm{Attention}}}({\boldsymbol{QW}}_i^{\boldsymbol{Q}},{\boldsymbol{KW}}_i^{\boldsymbol{K}},{\boldsymbol{VW}}_i^{\boldsymbol{V}}) $ | (5) |
| $ {\boldsymbol{R}} = {{\rm{Concat}}}({{\rm{hea}}}{{{\rm{d}}}_{1}}, \cdots ,{{\rm{hea}}}{{{\rm{d}}}_h}) $ | (6) |
式中:
门控注意力单元GAU结构将Transformer中的注意力机制和前反馈层进行融合,并引入了门控线性单元GLU、GAU,结构如图2所示,图中B为神经网络中的偏置项,表示偏置矩阵。
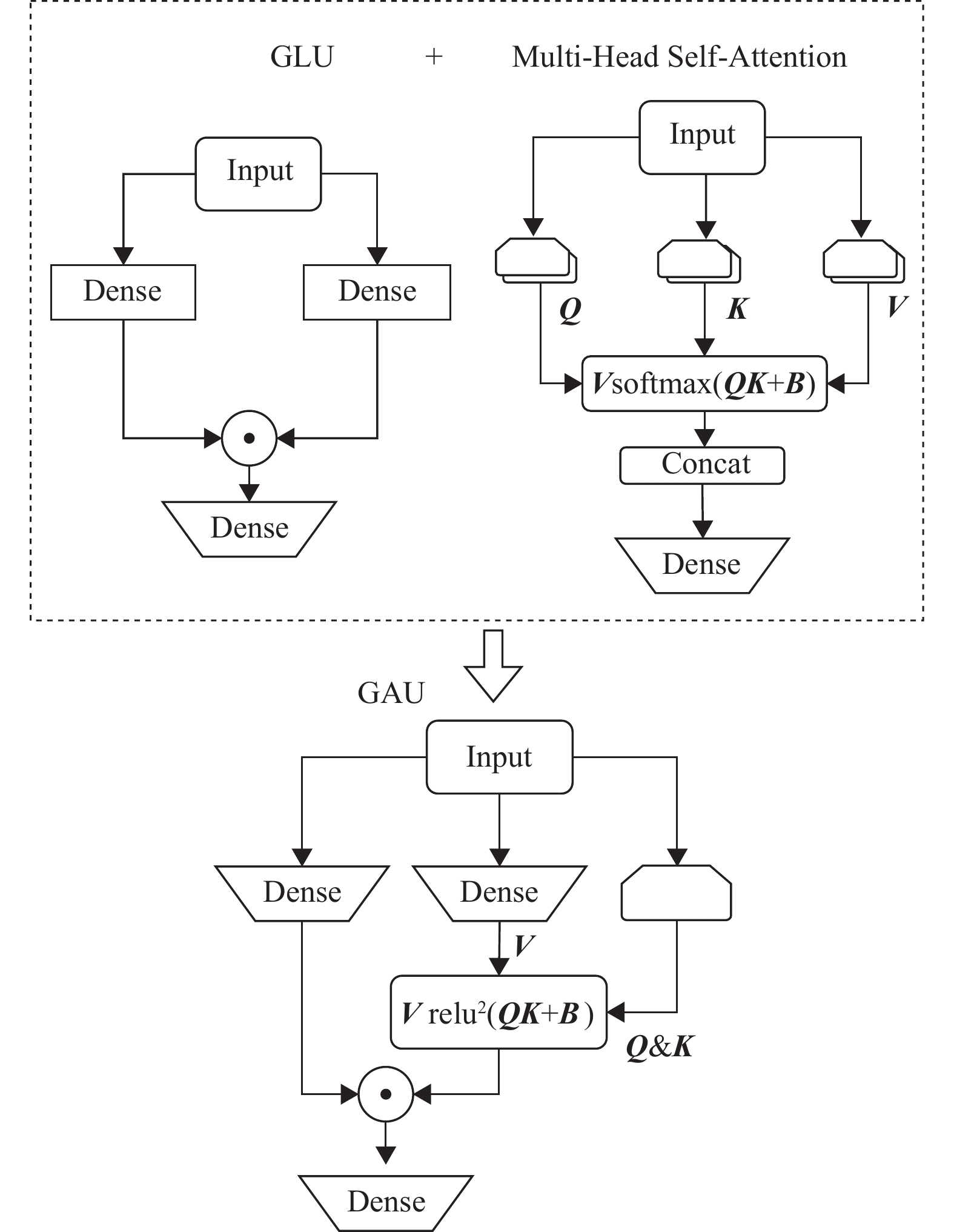
|
图 2 门控注意力单元结构 Figure 2 Structure of the gated attention unit |
GAU结构将多头自注意力机制和GLU结构进行融合作为统一的层,计算过程如式(7)~(8)所示。
| $ {\boldsymbol{U}}={\psi }_{{\boldsymbol{U}}}({\boldsymbol{R}}{{\boldsymbol{W}}}_{{\boldsymbol{U}}}) \text{,}{\boldsymbol{V}}={\psi }_{{\boldsymbol{V}}}({\boldsymbol{R}}{{\boldsymbol{W}}}_{{\boldsymbol{V}}}) $ | (7) |
| $ {\boldsymbol{O}} = ({\boldsymbol{U}} \odot {\boldsymbol{AV}}) {{\boldsymbol{W}}_{\boldsymbol{O}}} $ | (8) |
式中:
| $ {\boldsymbol{Z}} = {\psi _{\boldsymbol{Z}}}({\boldsymbol{R}}{{\boldsymbol{W}}_{\boldsymbol{Z}}}) $ | (9) |
| $ {\boldsymbol{A}} = \frac{1}{n}{{\rm{rel}}}{{{\rm{u}}}^2}(f({\boldsymbol{Z}}) g({{\boldsymbol{Z}}^{{\rm{T}}}}) ) $ | (10) |
式中:
通过叠加两层门控注意力单元,组成了本文提出的实体集合解码器中的FLASH结构。结合查询矩阵中融入的医学词汇信息,FLASH结构能够进一步挖掘医学命名实体间隐含的关联性,通过GAU中的门控机制,可以提高模型对输入文本中与医学信息相关的汉字字符的关注度,对多头自注意力机制进行稀疏化操作,减弱了FLASH结构对查询矩阵中与医学命名实体无关信息的关注,通过以上这些操作,可以加强模型对医学命名实体的解码效果。
在命名实体之间,通常存在着依赖关系和关联信息,例如在医学电子病历中,身体部位类型的命名实体和临床表现类型的命名实体经常同时出现在一段文本中,因此,可以通过学习两种命名实体类型之间的依赖关系,提高对命名实体识别的准确度。例如“咽痛”“鼻塞”这两个临床表现类型的实体中,都包含了身体部位类型的命名实体。
本文利用FLASH结构去提取实体间的依赖关系和关联信息。因为GAU结构对Transformer中的多头自注意力和前反馈层进行了融合,因此GAU结构也能通过和注意力机制类似的方式学习命名实体间的依赖关系。此外,GAU中的门控机制GLU能够从长序列文本中提取局部信息和相对位置信息,加强模型对上下文语义特征的提取能力。
在命名实体识别任务中,命名实体之间的依赖关系主要是局部和短距离内的,因此,模型对局部信息的提取能力更加重要。在FLASH结构中,采用了一种“局部−全局”分块混合的注意力机制,这种注意力机制能更大程度地提高模型对局部关联信息的提取能力,使得模型更多地关注命名实体间的依赖关系,但是又不会完全舍弃文本特征中的全局信息。因此在FLASH结构中,采用分块混合的注意力机制提取命名实体间的依赖关系。
分块混合注意力的计算过程如下:将输入长度为k的序列,划分成长度为e的块,设
| $ {{\boldsymbol{V}'}_g^{{{\rm{quad}}}}} = \frac{1}{e}{{\rm{rel}}}{{{\rm{u}}}^2}({\boldsymbol{Q}}_g^{{{\rm{quad}}}}{\boldsymbol{K}}_g^{{{\rm{quad}}}}) {{\boldsymbol{V}}_g} $ | (11) |
接下来利用
| $ {\boldsymbol{V}'}_g^{{{\rm{lin}}}} = \frac{1}{k}{\boldsymbol{Q}}_g^{{{\rm{lin}}}}\sum\limits_{h = 1}^{{n}/{c}} {{\boldsymbol{K}}_g^{{{\rm{lin}}}}{{\boldsymbol{V}}_h}} $ | (12) |
最后,将上面两种注意力计算过程相结合,整合到GAU中,按照式(8)进行计算就得到了具有线性复杂度的GAU,如式(13) 所示。
| $ {\boldsymbol{O}} = [{{\boldsymbol{U}}_g} \odot ({\boldsymbol{V}'}_g^{{{\rm{quad}}}} + {\boldsymbol{V}'}_g^{{{\rm{lin}}}}) ]{{\boldsymbol{W}}_{\boldsymbol{O}}} $ | (13) |
式中:
通过交叉注意力和FLASH结构,实体集合解码器的输出嵌入表示为
| $ {p^{\rm{c}}} = {{\rm{ML}}}{{{\rm{P}}}_{\rm{c}}}({\boldsymbol{O}}) $ | (14) |
| $ {p^{\rm{l}}} = {{\rm{ML}}}{{{\rm{P}}}_{\rm{l}}}({\boldsymbol{O}}) $ | (15) |
| $ {p^{\rm{r}}} = {{\rm{ML}}}{{{\rm{P}}}_{\rm{r}}}({\boldsymbol{O}}) $ | (16) |
式中:
在计算训练损失时,本文方法将实体集合解码器预测出的医学命名实体和数据集中标签命名实体分为两个部分,将每一部分看作是二分图的一部分,在计算模型训练损失时,使用匈牙利算法来计算真实实体和预测实体之间的最大匹配。
| $ \alpha = {{\rm{argmin}}}\sum\limits_{i = 1}^N {{L_{{{\rm{match}}}}}({y_i},{{y'}_i}) } $ | (17) |
式中:用y表示真实标签集合,用
计算出实体间的最大匹配
| $ {{\rm{loss}}} = \sum\limits_{i = 1}^N { - [{{\rm{log}}}p_{\alpha i}^{\rm{c}} + {{\rm{log}}}p_{\alpha i}^{\rm{l}} + {{\rm{log}}}p_{\alpha i}^{\rm{r}}]} $ | (18) |
为了验证本文模型对实体识别的有效性,实验部分采用了CMeEE、CMQNN和MSRA数据集。
CMeEE数据集是生物医学领域的常用数据集,包括身体部位(bod)、疾病(dis)等9个实体类别,CMeEE数据集的实体统计如表1所示。
| 表 1 CMeEE数据集实体分布统计 Table 1 CMeEE dataset statistics |
CMQNN数据集是医学领域的大型数据集,主要来源是医院的电子病历,CMQNN数据集的实体分布情况如表2所示。
| 表 2 CMQNN数据集实体分布统计 Table 2 CMQNN dataset statistics |
为了验证本文模型的适用性,在MSRA数据集上进行了实验。MSRA数据集的文本来源包括博客、论坛等,包含了金融、娱乐、体育等多个领域。MSRA数据集实体类型的统计情况如表3所示。
| 表 3 MSRA数据集实体分布 Table 3 MSRA dataset statistics |
本文采用精确匹配,只有当预测实体的实体类型、起始下标和结束下标都正确时,才认为实体预测正确。
2.2 参数设置与评估标准本文模型的实验环境如下:CPU型号是Intel(R) Xeon(R) Silver 4210R CPU @ 2.40 GHz,GPU型号是NVIDIA GeForce RTX3090 (24 G),Python版本是3.7.13,Pytorch版本是1.13,模型的超参数设置如表4所示。
| 表 4 实验参数设置 Table 4 Experimental parameter setting |
本文在验证模型有效性时,采用了3个评价指标,分别是精确率P,召回率R和F1值,计算公式如式(19)~(21)。
| $ P = \frac{{{\text{TP}}}}{{{\text{TP + FP}}}} $ | (19) |
| $ R = \frac{{{\text{TP}}}}{{{\text{TP + FN}}}} $ | (20) |
| $ F_1 = \frac{{2 \times P \times R}}{{P + R}} $ | (21) |
式中:TP、FP分别为识别正确的实体个数和识别错误的实体个数,FN为正例识别为反例的实体个数。
2.3 对比实验本文在3个数据集上进行了实验,为保证实验结果的可信度,采用了交叉验证的方式,将多次实验结果取平均值。由于对比模型原论文中使用了不同的数据集去验证模型效果,因此本文在数据集的对比实验中选择的模型不一致。首先在CMeEE数据集上进行了实验,实验的结果对比如表5所示。对比实验的模型介绍如下:
| 表 5 CMeEE数据集实验对比 Table 5 Comparative experimental results on the CMeEE dataset |
RICON[6]:通过规律感知模块和规律无感模块同时提取实体内部和边界的规律性,并在正交空间内对特征进行融合。
BERT-Biaffine[8]:用双仿射模型对标记的开始和结束位置评分,通过词元的标记评分来提取句子中的实体。
LEAR[12]:分别对文本和标签进行独立编码,将标签知识显式地集成到文本中。
FLAT[15]:将网格结构转换为跨度提取,并且在网格结构中构建了新的位置编码。
MANM[16]:利用稀疏化的注意力和改进的WLSTM网络提取多层次的语义特征。
FLR-MRC[18]:通过图注意力网络提取实体间关系,并且将实体信息和上下文语义信息相融合。
由表5可知,本文提出的SGAM模型在CMeEE数据集上取得了最佳的效果,F1值达到了70.74%,相比FLR-MRC模型的F1值提高了4.22%,与LEAR模型相比,F1值提升了4.95%。主要是因为SGAM模型使用交叉注意力将医学领域专业知识融入到查询矩阵中,提高了模型对医学命名实体的解码效果,此外,SGAM模型使用了生物医学领域的预训练模型,对于医学文本中的长尾术语有更强的上下文信息提取能力。
为了验证本文模型在医学命名实体识别上的优势,也在CMQNN数据集上进行了实验,对比实验结果如表6所示。对比实验的模型介绍如下:
| 表 6 CMQNN数据集实验对比 Table 6 Comparative experimental results on the CMQNN dataset |
BiLSTM+MultiCRF:结合双向长短期记忆网络和多层次的CRF网络实现命名实体预测。
RoBERT+CRF:使用预训练语言模型RoBERT编码命名实体,用CRF解码出文本中的命名实体。
表6实验结果表明本文提出的SGAM模型与FLAT模型相比,F1值提高了0.09%。本文模型在提取命名实体方面的优势相比其他模型有较大的提升,这是由于SGAM模型不再使用传统的序列标注方案,序列标注的方法对每个字符只能分配唯一的预测,而本文模型采用基于跨度的方法预测文本中的实体,每个实体都标注为(实体类型,起始下标,结束下标)这种三元组的形式,能更加准确地预测出文本中的命名实体,因此SGAM模型在CMQNN和CMeEE数据集上均获得了更佳的实验结果。
为了验证本文SGAM模型的在其他领域的应用性,本文在MSRA数据集上进行了实验,在这部分的实验中,文本编码器中预训练模型替换为BERT。实验结果如表7所示,对比模型介绍如下:
| 表 7 MSRA数据集实验对比 Table 7 Comparative experimental results on the MSRA dataset |
AAMwWIE[19]:用注意力机制动态地调整模型对实体的关注分布。
MECT[20]:模型中引入汉字结构信息,不仅能学习实体的边界和语义信息,也能学习汉字字根的结构信息。
PLTE[21]:结合多孔网格Transformer和多孔注意力机制,增强模型对局部上下文信息的建模能力。
Softlexicon[22]:对字符表征层进行小部分调整就能引入词库信息。
由表7可知,本文提出的SGAM模型在MSRA数据集上的F1值达到了95.53%,优于目前主流的模型。本文模型在CMeEE、CMQNN和MSRA数据集上的实验结果都达到了最佳的效果,主要源于以下几个方面:
(1) SGAM模型将实体提取转换为集合分配问题,对于命名实体有着更强的识别能力,从而在3个数据集上达到了更佳的效果。
(2) 本文提出的交叉注意力和FLASH结构相结合的实体集合解码器模型,将医学领域的词汇信息更好地融合到模型中,并且能提取医学命名实体之间隐含的关联性。
(3) 在模型训练过程中,二分图匹配算法在预测实体和真实实体之间寻找最大匹配,相比交叉熵损失,不会受到预测实体顺序变化的影响。
2.4 消融实验首先研究实体集合解码器层数对模型效果的影响,实验结果如图3所示。通过分析可知,当层数达到5层时,模型效果达到了最优,在CMeEE数据集上F1值达到了70.74%,当层数继续增加时,模型效果开始下降。表明实体集合解码器层数在5层时,模型能够充分地提取命名实体间的依赖关系。
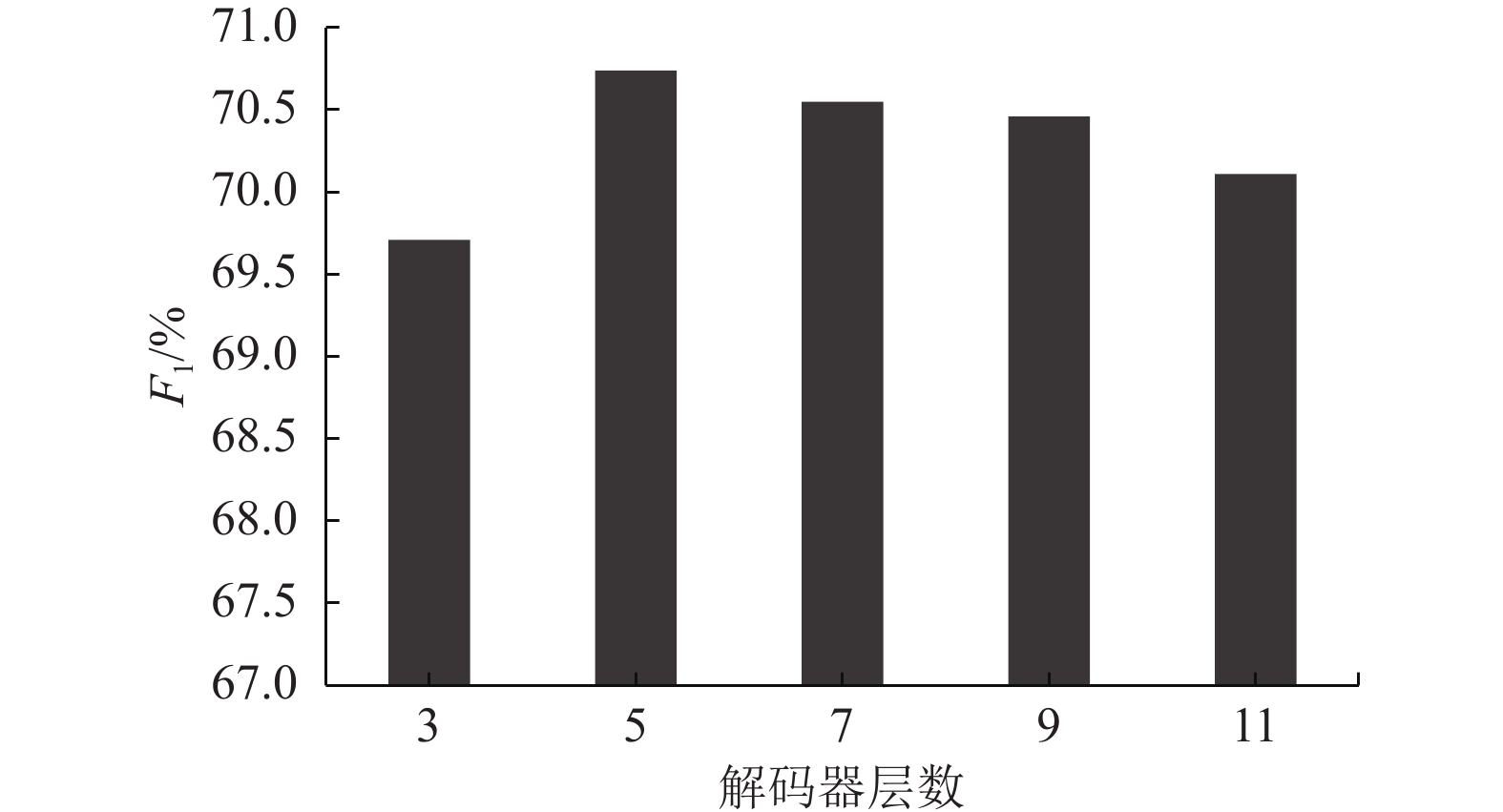
|
图 3 解码器层数对比实验 Figure 3 Comparative experiment of decoder layer |
在模型训练过程中,为了选择合适的实体查询矩阵数量,本文进行了消融实验,实验结果如图4所示。从实验结果表明,当实体查询的数量明显大于文本中的真实实体数量时,模型的效果开始下降。
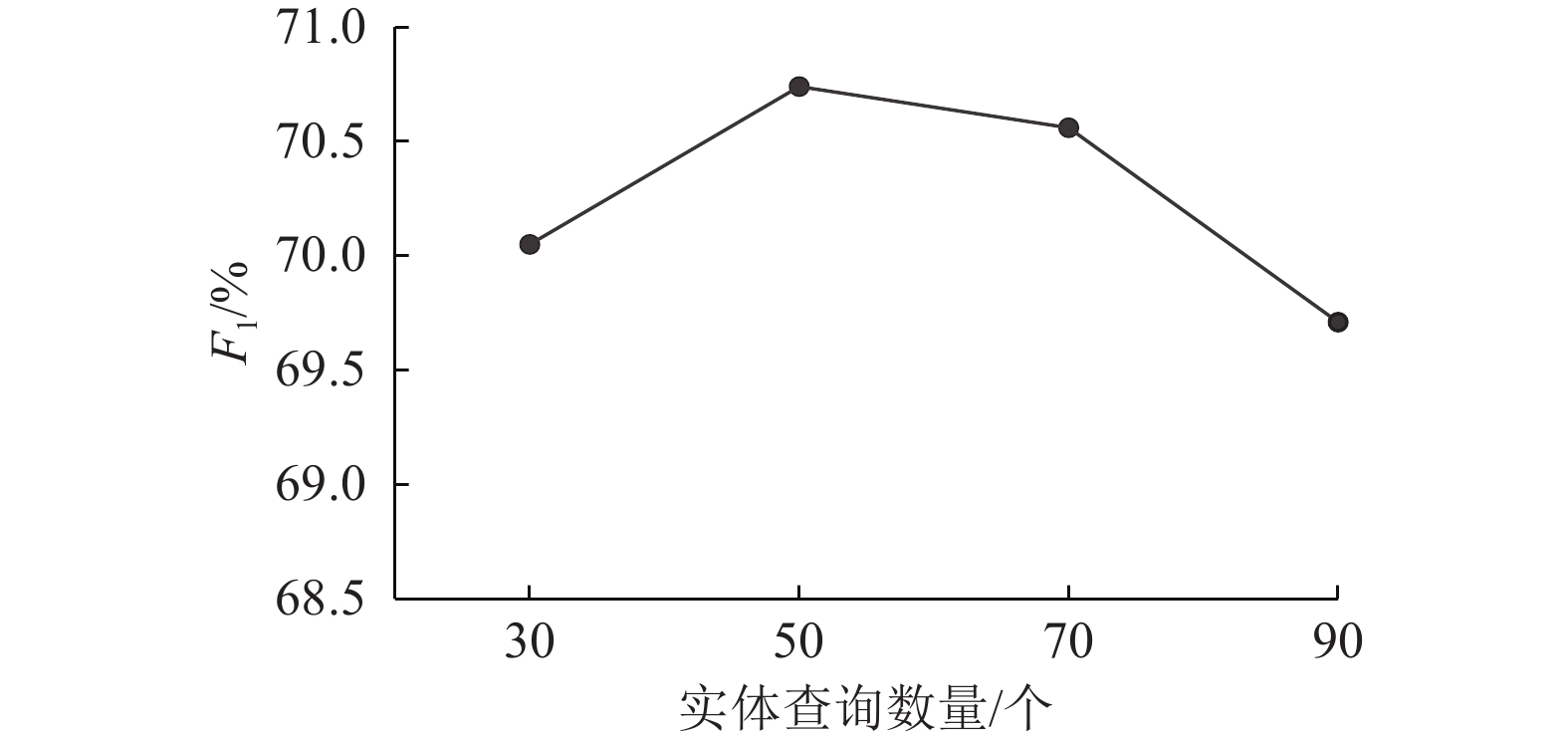
|
图 4 实体查询数量对比实验 Figure 4 Comparative experiment of entity query |
为了验证本文模型的有效性,对所提出的SGAM模型进行了模块的消融实验,编码器层数设置为5,实体查询数量设置为50,在CMeEE数据集上进行模块的消融实验,实验结果如表8所示,消融后的模型具体如下。
| 表 8 CMeEE数据集消融实验 Table 8 Ablation experiment on the CMeEE dataset |
CE loss:用交叉熵损失替代模型中的二分图匹配损失。
-attention:去掉本文模型中的交叉注意力机制。
-FLASH:去掉FLASH模块,并用Transformer中的多头自注意力机制和前反馈层代替。
表8中的各模块消融实验的结果表明:
(1) 损失函数用交叉熵损失替代后,模型的F1值下降了1.21%,证明了二分图的匹配算法对实体顺序不敏感,预测实体的排列不会影响模型训练过程中损失的计算,能够利用基于二分图的匹配损失函数较好地完成命名实体识别。
(2) 本文模型中attention去掉之后,模型F1值下降了0.28%,表明加强实体查询和医学领域词汇信息之间的交互计算有利于命名实体识别,可以提升本文模型对命名实体识别的准确率。
(3) 本文模型中的FLASH模块用多头自注意力和前反馈层替换后,模型F1值下降了0.78%,验证了FLASH模块在提取上下文语义信息和命名实体间的依赖关系上的有效性。
3 结束语本文针对中文医学命名实体识别任务设计了一个基于门控注意力单元的模型。首先,利用生物医学领域的预训练模型MC-BERT和BiLSTM网络对输入文本进行编码,提取输入文本中医学领域的专业词汇信息。其次,利用交叉注意力机制将医学命名实体信息融入到查询矩阵中,用FLASH结构提取医学命名实体之间的依赖关系并捕捉医学命名实体上下文相关的语境信息,提高模型对医学命名实体识别的准确性。最后,用匈牙利算法计算模型训练过程中的损失。实验结果表明,与目前主流模型相比,本文模型在效果上达到了最佳,证明了本文模型在中文医学命名实体识别任务上的有效性。在未来的工作中,将会尝试在少样本时,利用提示学习和对比学习进一步提升模型的性能。
| [1] |
苏攀予, 黄健文. 基于知识图谱的BIM技术在工程造价领域的应用研究[J].
广东工业大学学报, 2023, 40(2): 103-110.
SU P Y, HUANG J W. The application of BIM technology in the field of engineering cost based on knowledge graph[J]. Journal of Guangdong University of Technology, 2023, 40(2): 103-110. |
| [2] |
曹智雄, 吴晓鸰, 骆晓伟, 等. 融合迁移学习与YOLOv5的安全帽佩戴检测算法[J].
广东工业大学学报, 2023, 40(4): 67-76.
CAO Z X, WU X L, LUO X W, et al. Helmet wearing detection algorithm integrating transfer learning and YOLOv5[J]. Journal of Guangdong University of Technology, 2023, 40(4): 67-76. |
| [3] |
WU S H, SHEN Y L, TAN Z Q, et al. Propose-and-refine: a two-stage set prediction network for nested named entity recognition[C]//Proceedings of the 31th International Joint Conference on Artificial Intelligence. Montreal: IJCAI, 2022: 4418-4424.
|
| [4] |
WANG B L, LU W. Combining spans into entities: a neural two-stage approach for recognizing discontiguous entities[C]//Proceedings of the 2019 Conference on Empirical Methods in Natural Language Processing and the Ninth International Joint Conference on Natural Language Processing. Hong Kong: ACL, 2019: 6217-6225.
|
| [5] |
LI F, LIN Z C, ZHANG M S, et al. A span-based model for joint overlapped and discontinuous named entity recognition[C]//Proceedings of the 59th Annual Meeting of the Association for Computational Linguistics and the 11th International Joint Conference on Natural Language Processing (Volume 1: Long Papers) . Bangkok: ACL, 2021: 4814-4828.
|
| [6] |
GU Y J, QU X Y, WANG Z F, et al. Delving deep into regularity: a simple but effective method for Chinese named entity recognition[C]//Findings of the Association for Computational Linguistics: NAACL 2022. Seattle: ACL, 2022: 1863-1873.
|
| [7] |
LUAN Y, WADDEN D, HE L H, et al. A general framework for information extraction using dynamic span graphs[C]//Proceedings of the 2019 Conference of the North American Chapter of the ACL. Florence: ACL, 2019: 3036-3046.
|
| [8] |
YU J T, BOHNET B, POESIO M. Named entity recognition as dependency parsing[C]//Proceedings of the 58th Annual Meeting of the Association for Computational Linguistics. Seattle: ACL, 2020: 6470-6476.
|
| [9] |
WANG Q, ZHOU Y M, RUAN T, et al. Incorporating dictionaries into deep neural networks for the Chinese clinical named entity recognition[J].
Journal of Biomedical Informatics, 2019, 92: 103133.
DOI: 10.1016/j.jbi.2019.103133. |
| [10] |
LI X Y, ZHANG H, ZHOU X H. Chinese clinical named entity recognition with variant neural structures based on BERT methods[J].
Journal of Biomedical Informatics, 2020, 107: 103422.
DOI: 10.1016/j.jbi.2020.103422. |
| [11] |
LI X Y, FENG J R, MENG Y X, et al. A unified MRC framework for named entity recognition[C]//Proceedings of the 58th Annual Meeting of the Association for Computational Linguistics. Seattle: ACL, 2020: 5849-5859.
|
| [12] |
PAN Y, XIN C, SUN Z Y, et al. Enhanced language representation with label knowledge for span extraction[C]//Proceedings of the 2021 Conference on Empirical Methods in Natural Language Processing. Punta Cana: ACL, 2021: 4623-4635.
|
| [13] |
HANG Y, GUI T, DAI J Q, et al. A unified generative framework for various NER subtasks[C]//Proceedings of the 59th Annual Meeting of the Association for Computational Linguistics and the 11th International Joint Conference on Natural Language Processing (Volume 1: Long Papers) . Bangkok: ACL, 2021: 5808-5822.
|
| [14] |
SHEN Y L, WANG X B, TAN Z Q, et al. Parallel instance query network for named entity recognition[C]//Proceedings of the 60th Annual Meeting of the Association for Computational Linguistics (Volume 1: Long Papers) . Dublin: ACL, 2022: 947-961.
|
| [15] |
LI X N, YAN H, QIU X P, et al. FLAT: Chinese NER using flat-lattice transformer[C]// Proceedings of the 58th Annual Meeting of the Association for Computational Linguistics. Seattle: ACL, 2020: 6836-6842.
|
| [16] |
陈明, 刘蓉, 张晔. 基于多重注意力机制的中文医疗实体识别[J].
计算机工程, 2023, 49(6): 314-320.
CHEN M, LIU R, ZHANG Y. Chinese medical entity recognition based on multiple attention mechanism[J]. Computer Engineering, 2023, 49(6): 314-320. |
| [17] |
梁倬骞, 王东, 朱慧, 等. 基于领域本体的网络财务报告文本信息抽取研究[J].
广东工业大学学报, 2017, 34(3): 89-95.
LIANG Z Q, WANG D, ZHU H, et al. A research on text information extraction from annual report based on domain ontology[J]. Journal of Guangdong University of Technology, 2017, 34(3): 89-95. DOI: 10.12052/gdutxb.170029. |
| [18] |
LIU S Y, DUAN J W, GONG F, et al. Fusing label relations for Chinese EMR named entity recognition with machine reading comprehension[C]//18th International Symposium on Bioinformatics Research and Applications. Berlin: Springer, 2023: 41-51.
|
| [19] |
赵萍, 窦全胜, 唐焕玲, 等. 融合词信息嵌入的注意力自适应命名实体识别[J].
计算机工程与应用, 2023(8): 167-174.
ZHAO P, DOU Q S, TANG H L, et al. Attention adaptive model with word information embedding for named entity recognition[J]. Computer Engineering and Applications, 2023(8): 167-174. |
| [20] |
WU S, SONG X N, FENG Z H. MECT: multi-metadata embedding based cross-transformer for Chinese named entity recognition[C]//Proceedings of the 59th Annual Meeting of the Association for Computational Linguistics and the 11th International Joint Conference on Natural Language Processing (Volume 1: Long Papers) . Bangkok: ACL, 2021: 1529-1539.
|
| [21] |
XUE M G, YU B W, LIU T W, et al. Porous lattice transformer encoder for Chinese NER[C]//Proceedings of the 28th International Conference on Computational Linguistics. Barcelona: ICCL, 2020: 3831-3841.
|
| [22] |
MA R T, PENG M L, ZHANG Q, et al. Simplify the usage of lexicon in Chinese NER[C]//Proceedings of the 58th Annual Meeting of the Association for Computational Linguistics. Seattle: ACL, 2020: 5951-5960.
|
 2023, Vol. 40
2023, Vol. 40