2. 广东技术师范大学 电子与信息学院, 广东 广州 510665;
3. 广东省人民医院(广东省医学科学院) 心外科, 广东 广州 510080
2. School of Electronics and Information Technology, Guangdong Technical Normal University, Guangzhou 510665, China;
3. Department of Cardiovascular Surgery, Guangdong Provincial People’s Hospital (Guangdong Academy of Medical Sciences), Guangzhou 510080, China
在医学影像分析领域,心脏分割具有至关重要的意义,因为它能够为心脏疾病的准确诊断和治疗计划提供精确的解剖细节信息。先天性心脏缺陷是最常见的出生缺陷[1],研究人员将影像学和图形学应用于心脏病的临床实践和医学研究[2]。深度学习自2012年AlexNet[3]问世以来,在计算机视觉领域取得了巨大的发展。医学图像分割使医学影像分析自动化,辅助医生诊断,提高放射科医生效率,支持临床决策。
U-Net[4]是医学影像分割领域代表性深度学习架构,以对称的“U”字型的编码器−解码器结构为基础,引入跳跃连接增强特征融合。然而,编码器池化层中下采样导致深层网络中关键特征损失,而解码器难以有效恢复这些特征,降低了医学图像分割的精度。TransUNet[5]和Swin-UNet[6]通过引入自注意力机制来保留网络深层的细微纹理、不规则形状的分割目标和目标轮廓,但对于处理三维图像,二维切片模型容易丢失三维空间上下文信息,从而难以准确提取特征。UNETR[7]是基于patch块输入的三维模型,但网络在深层结构没有针对性改进,导致分割过程因为下采样丢失深层的细微血管和器官的轮廓等特征。
为了解决上述问题,本文提出一种结合三维卷积和自注意力机制的深度学习模型(3DCSNet),用于先天性心脏病多类分割。具体地,本文提出三维特征融合模块(Hybrid Block),从权重分配的角度出发,自注意力机制可以更好地在特征图的通道里的位置信息内分配权重,而卷积更加强调的是在通道间分配权重,而不是在通道内的位置信息。本文使用并行相加的形式,可以更好地对下采样后的信息进行特征提取,能够有效地调整特征图通道内部和通道之间的权重。此外,本文还提出了三维空间感知模块(3D Spatial-Aware Transformer Block),将神经网络深层维度的信息进行尺度统一,再分割成不同的小块,合并到一维空间上,然后使用自注意力机制捕捉不同维度之间的相关性,减少下采样带来的特征损失,进一步提升模型的分割性能。
1 相关工作心脏结构分割方式主要有3类:人工分割、传统图像处理分割法以及基于深度学习的语义分割。医生手动标注心血管CT影像需要专业知识,耗时、疲劳易导致准确性不足,限制了效率和可靠性。
1.1 传统方法医学计算机辅助技术发展,结合传统区域算法与人工操作,能显著提升心脏分割的准确性。传统分割法可分为弱先验信息法与强先验信息法。弱先验信息法涉及到较少的空间、强度以及解剖知识,包括基于图像的方法[8]、像素分类法[9]、可变形模型法[10]以及图割法[11]。这些方法可能受到先验信息准确性和数据多样性等方面的限制,心脏分割结果可能出现边界模糊等问题。强先验信息法[12]运用统计模型学习约束分割结果,从而弥补弱先验信息法的不足。此外,基于形状先验的可变形模型[13]、基于心脏运动外观的模型[14]和基于地图集的方法[15],其效果受原始数据质量、心脏运动和形状规律影响,容易过拟合。
1.2 深度学习方法深度学习方法依据输入的尺寸可分为基于二维切片的模型、基于全尺寸输入的模型、基于patch块的模型。基于二维切片的模型有TransUNet、Swin-UNet、APFormer[16]等。其中,TransUNet将Transformer[17]与U-Net结合,运用Transformer的全局自注意力机制,弥补U-Net难以建模长距离依赖的不足。Swin-UNet以Swin Transformer[18]替代U-Net中的卷积块,通过分层块内的窗口式注意力机制,强调了局部区域内的特征交互。APFormer采用自监督策略改进了Transformer中的自注意力矩阵,以加速模型收敛;运用高斯先验知识引入位置信息,以降低训练复杂度。上述模型有着计算效率高和内存占用小的优势,但在应用于三维心脏数据时,因为切片而丢失三维上下文信息。
基于完整尺寸图像输入(Full-image)神经网络有3D U-Net[19]跟Attention U-Net[20]。其中,3D U-Net是基于U-Net框架的变种,其中所有的二维操作被替换为三维操作,以适应三维医学图像分割任务。Attention U-Net提出了一种新的关注门(Attention Gate,AG)模型,可以抑制医学影像中的不相关区域,同时突出对目标器官有益的显著特征。以上模型分割结果有较多误分类,是因为更大神经网络尺寸和处理更大心脏图像尺寸所需的复杂性增加。
基于三维体积块(patch)的模型有UNETR、D-Former[21]、nnFormer[22]。其中,UNETR使用Transformer作为编码器,直接运用嵌入的三维体积来有效地捕获长期依赖关系。D-Former则引入了一种扩张式Transformer,通过交替地应用自注意力机制于局部和全局补丁(patches)的成对关系,实现了扩张式的全局自注意。nnFormer运用局部和全局自注意力构建特征金字塔提供大的感受野,提出了用跳跃注意取代传统的拼接和求和运算。以上网络都普遍存在一个问题,即在分割过程中因为下采样可能会丢失深层信息,这种信息丢失的情况可能会随着网络的加深而逐渐恶化。
2 3DCSNet 2.1 总体架构3DCSNet的整体架构如图1所示,其框架基于nnFormer设计,由编码器、瓶颈层和解码器组成。与nnFormer不同的是,在编码器部分,nnFormer主要是由2个局部的Transformer块组成,而本文在第1层使用一个大尺寸卷积核的三维卷积块(Large Kernel Convolutional Module,LKCM),如图2所示,第2层用2个LKCM串行。在瓶颈层部分,nnFormer主要由3个全局的Transformer块组成,而本文主要由三维特征融合模块(Hybrid Block)、三维空间感知模块(3D Spatial-Aware Transformer Block,3D-SATB)和信息融合模块(Cat)组成。在解码器部分,nnFormer主要由2个局部的Transformer块组成,而本文每层是由1个LKCM构成。下采样采用步长为2、卷积核为2的卷积,上采样则采用步长为2、卷积核为2的转置卷积。
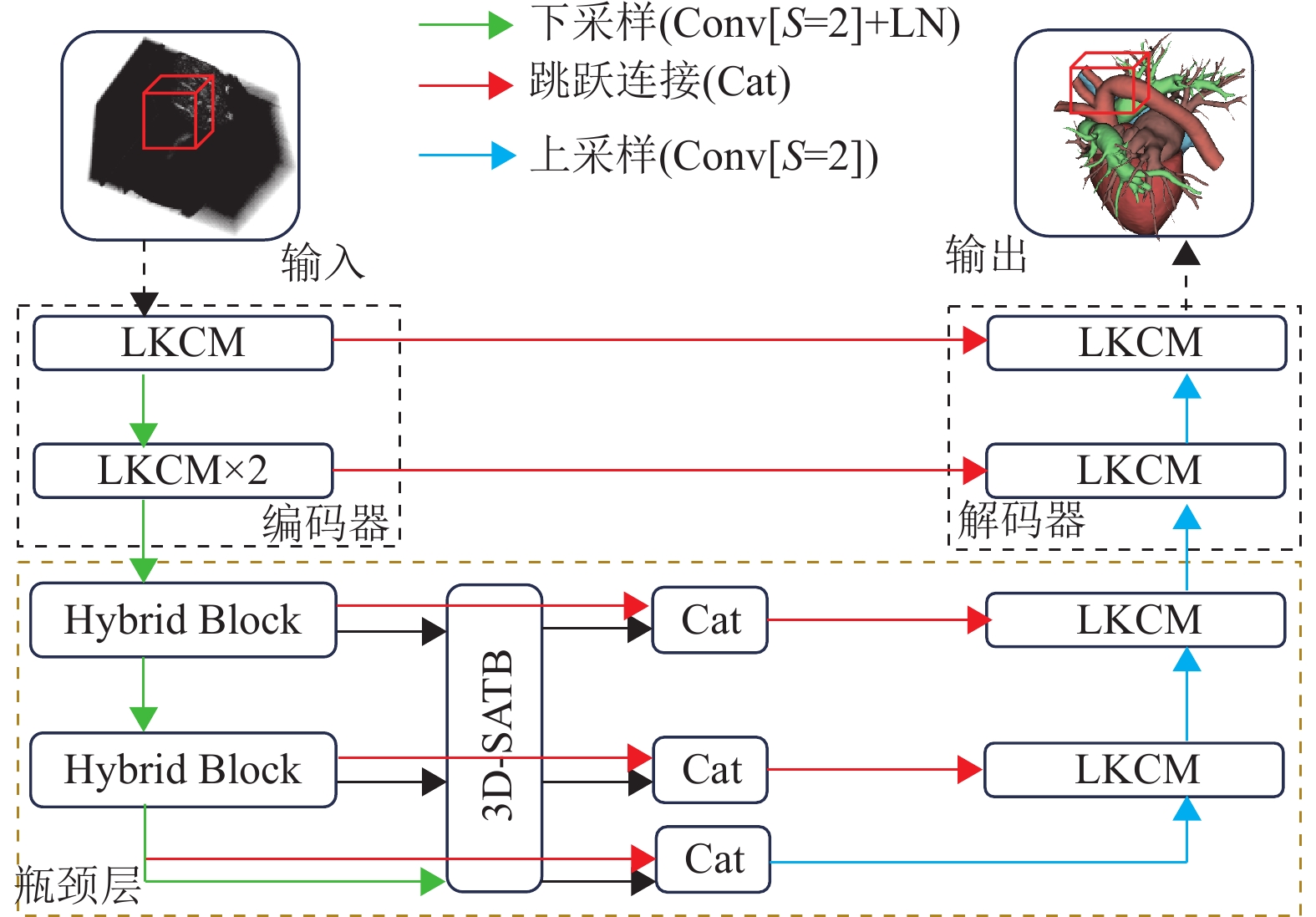
|
图 1 3DCSNet网络架构的示意图 Figure 1 Overview of the 3DCSNet architecture |
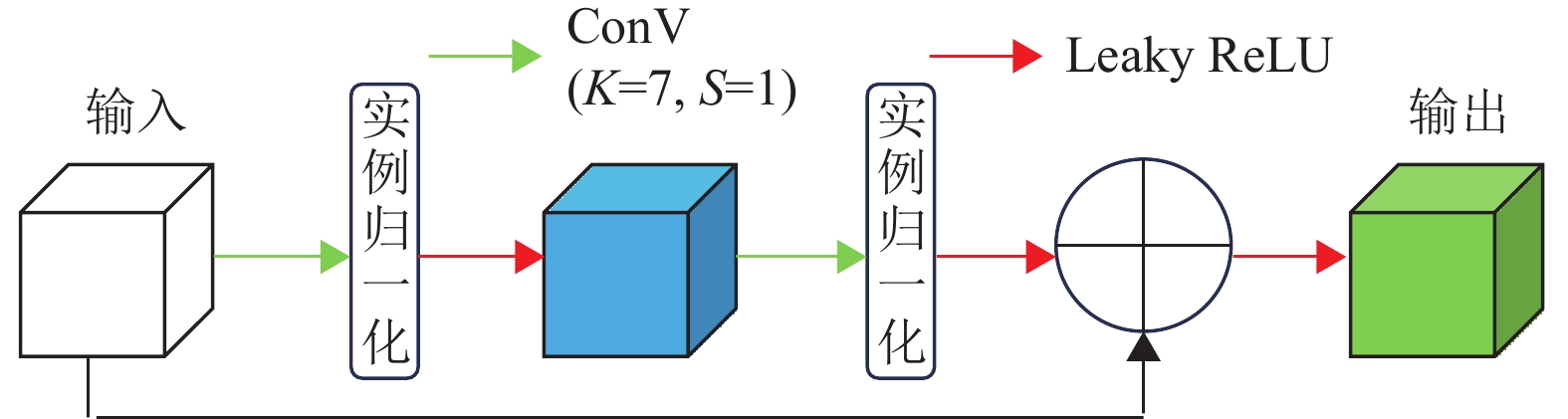
|
图 2 LKCM网络结构 Figure 2 Structure of the LKCM network |
受到Swin Transformer的启发,本文注意到其有效之处在于将图像分割成不同的窗口,然后在每个窗口内进行自注意力机制计算。因此,在LKCM中使用大卷积核(7×7×7)的三维卷积提取特征,捕获更广泛的上下文信息。残差连接使医学图像信息更容易在网络的不同层之间流动,可以更专注于去捕捉到图像中的心脏器官,如图2所示。其计算过程如式(1)所示。
| $ {{\boldsymbol{Z}}}_{{{{l}} + 1}} = {f_{{{\rm{LR}}}}}({{\boldsymbol{Z}}}_{{{l}}} + {f_{{{\rm{IN}}}}}({f_{{3{\rm{DC}}}}}({f_{{{\rm{LR}}}}}({f_{{{\rm{IN}}}}}({f_{{3{\rm{DC}}}}}({{\boldsymbol{Z}}}_{{{l}}}) ) ) ) ) ) $ | (1) |
式中:
受到UNet-2022[23]的启发,本文提出三维特征融合模块,由2个LKCM和注意力模块(Swin Transformer Block)组成,通过并行计算将它们的结果相加,形成一个非同构块,如图3所示。与UNet-2022不同的是,本文采用了基于patch块的三维模型,而UNet-2022则采用了二维切片模型。三维模型更能有效地利用三维空间的上下文信息进行特征提取。此外,3DCSNet没有使用深度可分离卷积进行特征提取,因为深度可分离卷积会有部分信息损失。
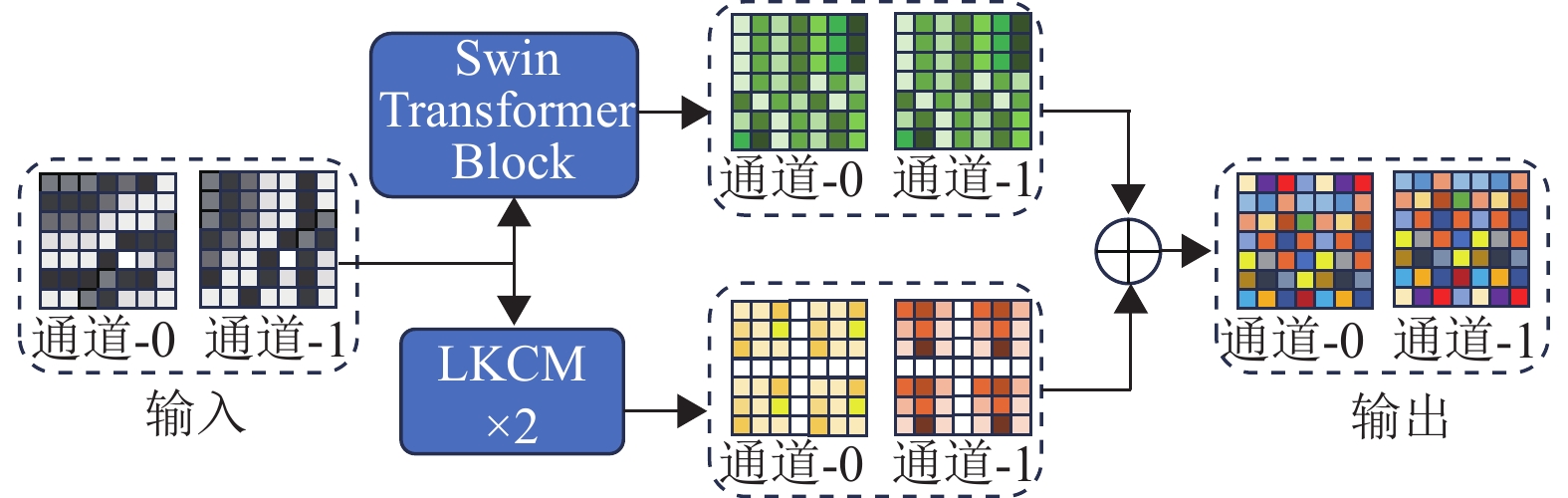
|
图 3 三维特征融合模块 Figure 3 3D feature fusion module |
从权重分配的角度来看,注意力模块的优势在于自注意力机制可以计算通道内不同位置之间的相关性,将权重动态分配到不同的空间位置,可以使模型更有针对性地关注图像中重要的区域。然而,自注意力机制也有弊端,就是在同一位置的不同通道内权重是一样的,会导致模型无法充分捕捉多通道特征之间的差异性。因为每个通道代表了图像中的不同信息(例如纹理、形状等),如果在同一位置上使用相同的权重,模型可能无法对这些不同通道的特征差异进行精细地建模,可能会影响模型的分割效果。与此相反的是,三维卷积模块可以在同一位置的不同通道内分配权重,可以更好地探索在不同通道内的特征信息,但是在同一通道内的不同位置是共享相同的卷积核权值的,所以会缺乏描述深层复杂空间的能力。因此,本文提出三维特征融合模块,可以在同一维度的通道内以及通道间更有效去分配动态权重,从而更好地去捕捉三维医学图像中的复杂特征。其计算过程如式(2)所示。
| $ {{\boldsymbol{Z}}}_{{{{l}} + }{{1}}}= {f_{{{\rm{Swin}}}}}({{\boldsymbol{Z}}}_{{{l}}}) + {f_{{{\rm{LKCM}}}}}({f_{{{\rm{LKCM}}}}}({{\boldsymbol{Z}}}_{{{l}}}) ) $ | (2) |
式中:
在解决医学图像分割中的尺度变化问题时,多维度之间的特征交互非常重要。受到ScaleFormer[24]的启发,本文提出了三维空间感知模块,使用自注意力机制,结合不同维度的特征信息,进行多维度的位置相似性计算,对不同维度的特征进行权重分配,这样可以避免下采样导致的关键特征丢失,如图4所示。与ScaleFormer不同,本文采用了基于patch块的三维模型,而ScaleFormer是基于二维切片模型,这种差异导致了在三维医学图像上进行语义分割时ScaleFormer容易丢失深度信息,无法表现出良好的性能。
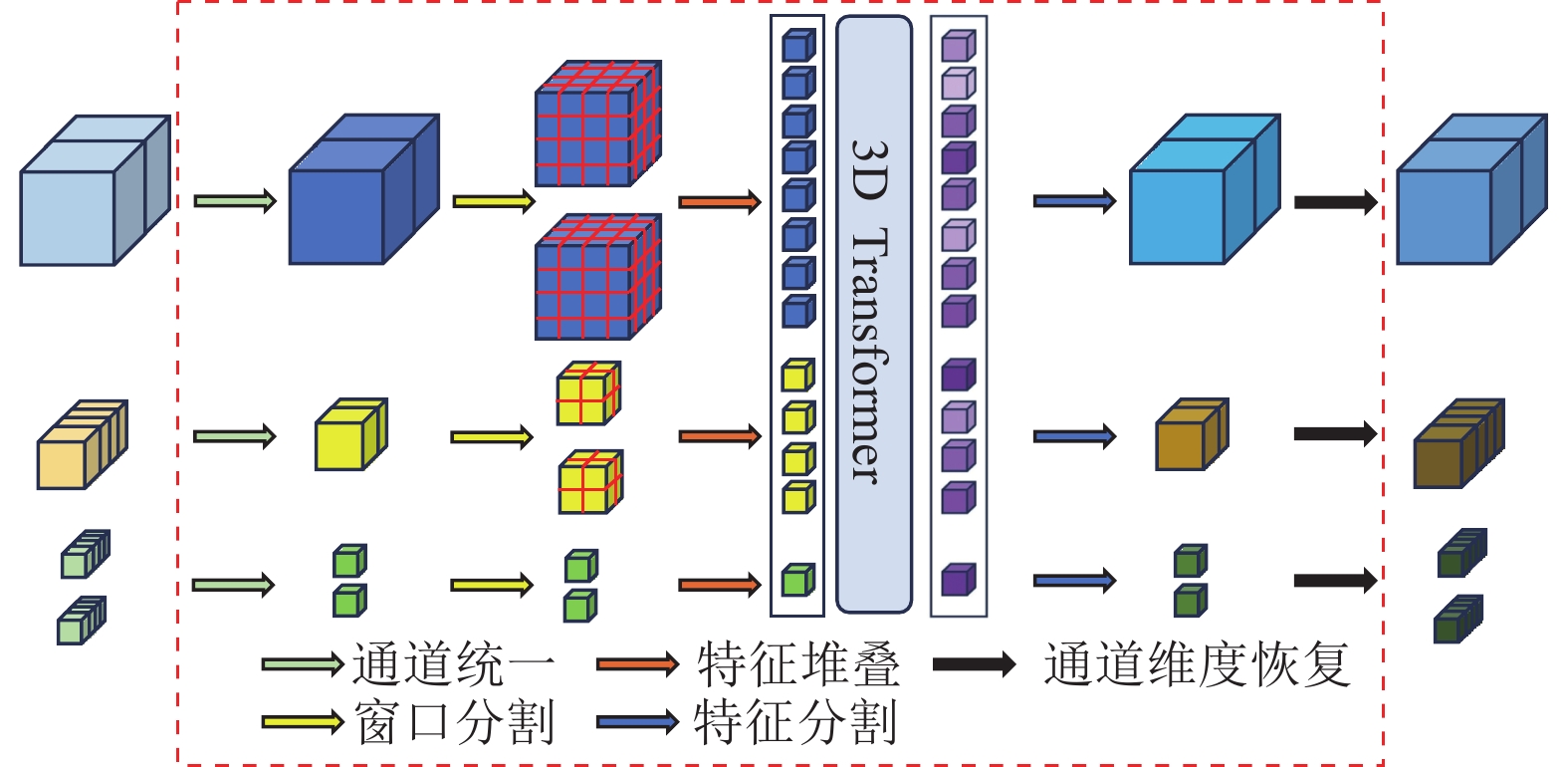
|
图 4 三维空间感知模块 Figure 4 3D spatial-aware transformer block |
首先,本文采用瓶颈层经过三维特征融合模块处理后的3个特征(j=0,1,2),假设输入大小是
| $ {{\boldsymbol{X}}}_{j}^{\tfrac{{H}}{{{{2}}^{j}}}{ \times }\tfrac{{W}}{{{{2}}^{j}}}{ \times }\tfrac{{D}}{{{{2}}^{j}}}{ \times 96}} = {f_{{{\rm{FC}}}}}({{\boldsymbol{X}}}_{j}^{\tfrac{{H}}{{{{2}}^{j}}} \times \tfrac{{W}}{{{{2}}^{j}}} \times \tfrac{{D}}{{{{2}}^{j}}} \times {(}{{2}}^{j}{C) }}) $ | (3) |
式中:
使用窗口分割法,对3个特征向量分别进行分割,使用窗口大小为6的立方体进行分割。3个特征向量在数量维度上不一样,其计算过程如式(4)所示。
| $ {{\boldsymbol{X}}}_{j}^{{o}^{{3}} \times {b} \times {{96}}}{{ = }}{f_{{{\rm{reshape}}}}}({{\boldsymbol{X}}}_{j}^{\tfrac{{H}}{{{{2}}^{j}}} \times \tfrac{{W}}{{{{2}}^{j}}} \times \tfrac{{D}}{{{{2}}^{j}}} \times {{96}}}{{) }} $ | (4) |
式中:
在特征堆叠部分,把不同维度的张量在数量维度上进行拼接,堆叠成一个更大的特征向量。其计算过程如式(5)所示。
| $ {{\boldsymbol{X}}}^{{o}^{{3}} \times {S} \times {{96}}}{{ = }}{C_{{{\rm{concat}}}}}{{(}}{{\boldsymbol{X}}}_{j}^{{o}^{{3}} \times {b} \times {{96}}}, - 2) $ | (5) |
式中:
使用注意力机制提取不同维度堆叠后的特征,获取当前各个位置的相关系数,经过3D Transformer处理后,可以得到相似性矩阵。其计算过程如式(6)、(7)所示。
| $ {\boldsymbol{Z}}^{{/}}_{{{{l}}}}={f}_{\rm{Atten}}({f}_{\rm{LN}}({\boldsymbol{Z}}_{{l}}) ) +{\boldsymbol{Z}}_{{l}} $ | (6) |
| $ {\boldsymbol{Z}}_{{{l}}+{1}}={f}_{\rm{MLP}}({f}_{\rm{LN}}({\boldsymbol{Z}}^{{/}}_{{l}}) )+{\boldsymbol{Z}}^{{/}}_{{l}} $ | (7) |
式中:为了简化公式表示,这里用向量
对
在瓶颈层部分的信息融合(Cat)部分,是将三维特征融合模块和三维空间感知模块在通道维度进行拼接,然后再经过一个三维点卷积降维回原来的通道维度。其计算过程如式(8)所示。
| $ {{{\boldsymbol{Y}}}_{{{i}}}} = {f_{{{\rm{Conv}}}{{1} \times {1} \times {1}}}}({C_{{{\rm{concat}}}}}({{{\boldsymbol{H}}}_{{{i}}}},{{{\boldsymbol{T}}}_{{{i}}}}) , - 1) $ | (8) |
式中:
公开的先天性心脏病分类的数据集[25] (ImageCHD) 是由Siemens Biograph 64位机器从110名患者中采集的三维计算机断层图像组成。该数据集主要有8个标签,其中标签0表示背景,标签1~7分别是左心室(Left Ventricle,LV)、右心室(Right Ventricle,RV)、左心房(Left Atrium,LA)、右心房(Right Atrium,RA)、心肌(Myocardium,Myo)、主动脉(Aorta,AO)和肺动脉(Pulmonary Artery,PA),如图5所示。根据数据筛选要求,保留了标签为0~7的数据,并剔除了一些异常样本。经过筛选后,最终保留了94个符合条件的样本。先天性心脏病数据集灰度值(Hounsfield Units,HU)一般在0~4 095之间,由于范围太大,需要将数据范围限制在一个较小的区间,模型训练可以集中注意力在感兴趣区域上。因此,需要对其进行截断,截断后范围为500~2 500,然后需要对数据进行归一化处理,使图像数值范围为0~1。考虑到图像尺寸过大且不均匀,本文需要对数据集进行下采样,采用双三次插值将所有数据和标签的大小调整至256×256×128,以确保一致性,并将标签超过7的所有标签值置为0。
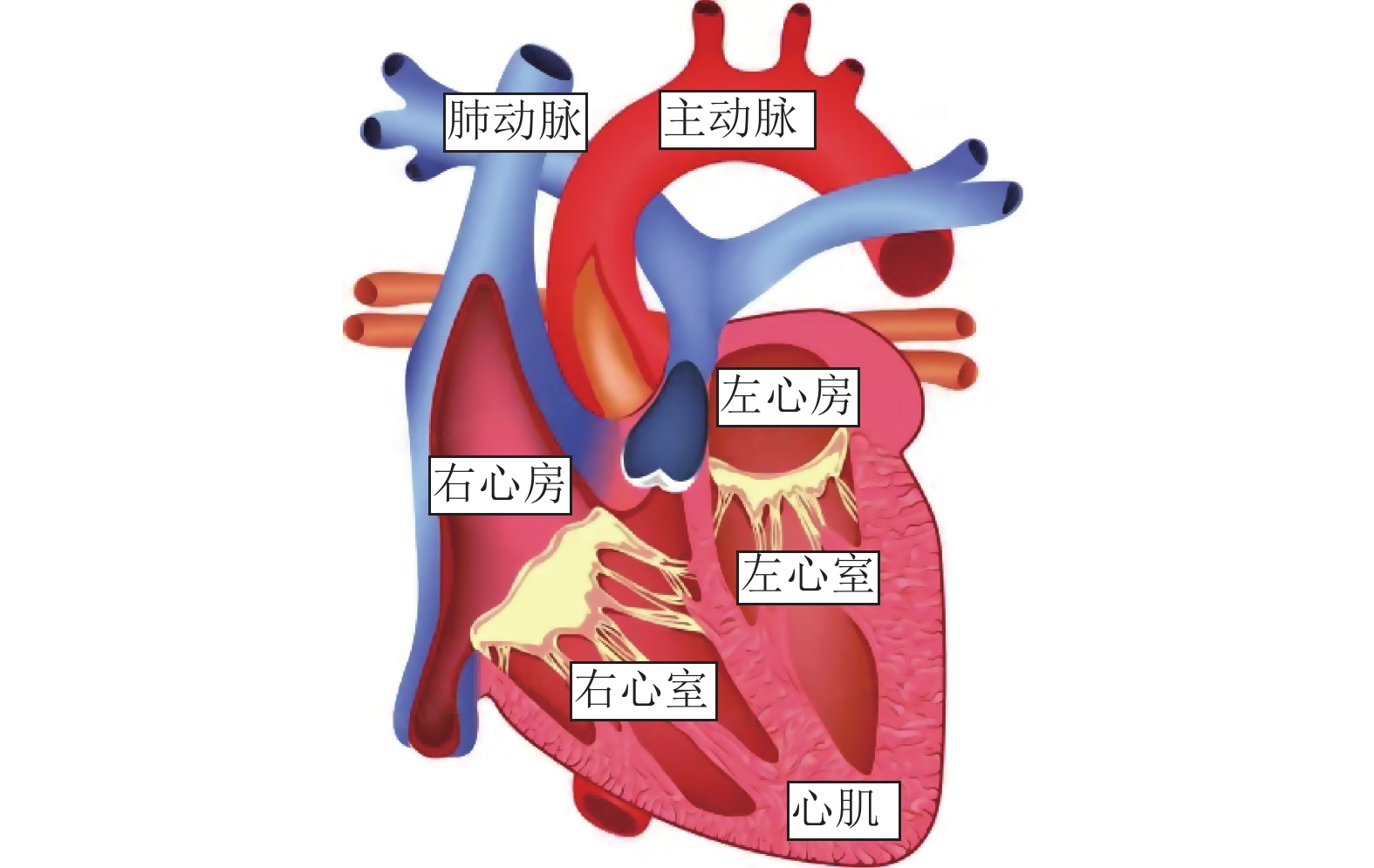
|
图 5 ImageCHD标签展示图 Figure 5 ImageCHD label visualization image |
本文使用的是Pytorch 1.9框架,代码使用python3.6编写,GPU使用的是RTX3090。在训练过程中,将数据集分为5份,其中4份作为训练集,1份作为测试集,采用5折交叉验证。考虑到2D分割丢失空间信息,本文决定采用3D卷积训练。使用原始体数据进行随机裁剪,每次裁剪一个96×96×96的三维patch块进行训练。损失函数使用DiceCELoss,优化器采用AdamW,学习率使用余弦退火策略,训练迭代次数为400代,批量大小为2,目标是充分运用3D卷积提取空间信息,并通过余弦退火优化模型训练。本文的评价指标是Dice,计算过程如式(9)所示。
| $ {{\rm{Dice}}} = \frac{{2 \times (\left| {{{\rm{Pre}}} \cap {{\rm{GT}}}} \right| + a) }}{{\left| {{{\rm{Pre}}}} \right| + \left| {{{\rm{GT}}}} \right| + a}} $ | (9) |
式中:
模型训练完成后,本文使用滑动窗口的方式进行推断。对于patch投票,本文采用高斯重要性加权策略,可以在softmax聚合过程中给予中心像素。在所有网络模型进行Dice计算之前,使用scikit-image[26]分析体素形态连通性,提取前三大连通域,忽略其他连通域,从而优化分割结果。
3.4 实验结果及分析为了证明3DCSNet的有效性,实验结果如表1所示。在ImageCHD数据集上分别使用基于2D模型:2D U-Net[4]、TransUNet[5]、Swin-Unet[6]、APFormer[16]、UNet-2022[23]、ScaleFormer[24];基于full-image(完整图像尺寸输入)模型:3D U-Net-full[19]、Attention U-Net[20];基于patch块的分割方法:UNETR[7]、D-Former[21]、nnFormer[22]、3D U-Net-patch(使用963patch的3D U-Net[19]模型),进行对比实验。以上模型以及3DCSNet都进行了五折交叉验证,指标使用Dice系数,这是一个比例指标,表格中的数值以(平均值±标准差)%的形式呈现。
| 表 1 ImageCHD数据集上不同方法的分割性能 Table 1 Segmentation performance of different methods on the ImageCHD dataset |
通过表1可以发现,3DCSNet在ImageCHD数据集上7类心脏器官分割的平均Dice系数是84.44,达到了最好的分割水平,分别在左右心室、左心房、心肌以及肺动脉上都获得最佳,右心房和主动脉均为第2。通过提高心脏多类分割结果的准确性,为先天性心脏病患者的早期预测和医疗辅助提供了有力支持。通过比较基于卷积的2D U-Net、3D U-Net-patch、3D U-Net-full,可以发现基于随机裁剪成96³的patch的方法能够取得不错的结果。基于二维神经网络的方法存在一个较大的问题,即丢失了切片间的信息,无法充分运用3D空间信息,从而导致分割效果不佳。
3.5 分割结果可视化本文展示了随机选取3个不同的数据,UNet-2022、ScaleFormer、3D U-Net-full、nnFormer、3DCSNet的分割结果与真实标签的可视化对比。每行表示单个数据实例在各个模型中的语义分割图,每列则展示了同一模型下3个不同数据的语义分割图,具体示例如图6所示。从可视化图中可以得到,UNet-2022和ScaleFormer在其分割结果中表现出明显的切片堆积问题,这归因于这些模型使用二维切片作为输入进行训练,导致三维上下文信息的丢失。3D U-Net-full表现出明显错误的大连通域,这是由于更大的神经网络尺寸和处理更大的心脏图像尺寸所需的复杂性增加。此外,基于补丁的方法nnFormer减少了分割错误,但很难捕捉心脏深层特征中的细微血管或心房心室等器官的不规则性。本文提出的3DCSNet不仅可以准确地分割器官,还可以有效地分离器官之间的边缘,没有明显的像素错误分类。

|
图 6 分割结果可视化 Figure 6 Visualization of segmentation results |
为了验证三维特征融合模块和三维空间感知模块的有效性,本文使用ImageCHD数据集进行了五折交叉验证的消融实验。表2中的3DCSNet-SA指的是瓶颈层三维特征融合模块中移除自注意力机制层,仅保留卷积部分。另一方面,3DCSNet-SW表示移除三维空间感知模块和随后特征融合模块。在比较3DCSNet-SA和3DCSNet时,很明显,自注意力机制模块的结合将平均Dice提高了0.58%。这种现象表明,将自注意力模块整合到网络的更深层有利于心脏分割任务。在比较3DCSNet-SW和3DCSNet时,观察到三维空间感知模块的引入将平均Dice提高了0.98%。这验证了心脏分割在探索跨维度的信息特征相互作用的必要性。
| 表 2 消融实验ImageCHD结果 Table 2 Ablation results on the ImageCHD dataset |
本文提出3DCSNet用于心脏分割任务,通过引入三维特征融合模块,将三维卷积和自注意力机制有机结合,运用自注意力和三维卷积并行进行特征提取,有效地分配特征图通道内部和通道之间的权重,以更好地保留深层关键特征信息。另外,3D-CSNet还提出三维空间感知模块,通过结合三维自注意力机制,能够提取不同维度之间的相关性信息,有效解决了图像下采样操作可能导致的信息丢失问题,并提高心脏分割的准确性。尽管在分割过程中取得了一些进展,但仍然面临着数据稳定性和个体差异性等挑战。未来,将继续研究更先进的方法,特别是将卷积和自注意力机制相结合,以提升分割效果,为临床医学图像诊疗提供可靠的支持。
| [1] |
BHAT V, BELAVAL V, GADABANAHALLI K, et al. Illustrated imaging essay on congenital heart diseases: multim-odality approach part III: cyanotic heart diseases and complex congenital anomalies[J].
Journal of Clinical and Diagnostic Research, 2016, 10(7): TE01-TE06.
|
| [2] |
NESSER H J, SUGENG L, CORSI C, et al. Volumetric analysis of regional left ventricular function with real-time three-dimensional echocardiography: validation by magnetic resonance an- d clinical utility testing[J].
Heart, 2007, 93(5): 572-578.
DOI: 10.1136/hrt.2006.096040. |
| [3] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E. Imagenet classification with deep convolutional neural networks[J].
Advances in Neural Information Processing Systems, 2012, 25(2): 84-90.
|
| [4] |
RONNEBERGER O, FISCHER P , BROX T. U-net: convolutional networks for biomedical image segmentation[C]//Proceedings of International Conference on Medical Image Computing and Computer-Assisted Intervention. New York: Springer; 2015: 234–241.
|
| [5] |
CHEN J, LU Y, YU Q, et al. Transunet: Transformers make strong encoders for medical image segmentation[EB/OL]. arXiv: 2102.04306(2021-02-08) [2023-09-04]. https://arxiv.org/abs/2102.04306.
|
| [6] |
CAO H, WANG Y, CHEN J, et al. Swin-unet: Unet-like pure transformer for medical image segmentation[C]//Proceedings of European Conference on Computer Vision. Cham: Springer Nature Switzerland, 2022: 205-218.
|
| [7] |
HATAMIZADEH A, TANG Y, NATH V, et al. Unetr: Transfor-mers for 3d medical image segmentation[C]//Proceedings of the IEEE/CVF Winter conference on Applications of Computer Vision. New Orleans: IEEE Computer Society 2022: 574-584.
|
| [8] |
LIU H, HU H, XU X, et al. Automatic left ventricle segmen-tation in cardiac MRI using topological stable-state thresholding and region restricted dynamic programming[J].
Academic Radiology, 2012, 19(6): 723-731.
DOI: 10.1016/j.acra.2012.02.011. |
| [9] |
ULEN J, STRANDMARK P, Kahl F. An efficient optimization framework for multi-region segmentation based on lagrangian duality[J].
IEEE Transactions on Medical Imaging, 2012, 32(2): 178-188.
|
| [10] |
CHEN T, BABB J, KELLMAN P, et al. Semiautomated segmentation of myocardial contours for fast strain analysis in cine displacement-encoded MRI[J].
IEEE Transactions on Medical Imaging, 2008, 27(8): 1084-1094.
DOI: 10.1109/TMI.2008.918327. |
| [11] |
AYED I B, CHEN H, PUNITHAKUMAR K, et al. Max-flow segmentation of the left ventricle by recovering subject-specificdistributions via a bound of the Bhatta-charyya measure[J].
Medical Image Analysis, 2012, 16(1): 87-100.
DOI: 10.1016/j.media.2011.05.009. |
| [12] |
PETITJEAN C, DACHER J N. A review of segmentation methods in short axis cardiac MR images[J].
Medical Image Analysis, 2011, 15(2): 169-184.
DOI: 10.1016/j.media.2010.12.004. |
| [13] |
QUEIROS S, BARBOSA D, HEYDE B, et al. Fast automatic myocardial segmentation in 4D cine CMR datasets[J].
Medical Image Analysis, 2014, 18(7): 1115-1131.
DOI: 10.1016/j.media.2014.06.001. |
| [14] |
MITCHELL S C, BOSCH J G, LELIEVELDT B P F, et al. 3D active appearance models: segmentation of cardiac MR and ultrasound images[J].
IEEE Transactions on Medical Imaging, 2002, 21(9): 1167-1178.
DOI: 10.1109/TMI.2002.804425. |
| [15] |
BAI W, SHI W, LEDIG C, et al. Multiatlas segmentation withaugmented features for cardiac MR images[J].
Medical Image Analysis, 2015, 19(1): 98-109.
DOI: 10.1016/j.media.2014.09.005. |
| [16] |
LIN X, YU L, CHENG K T, et al. The lighter the better: Rethinking Transformers in medical image segmentation through adaptive pruning[J].
IEEE Transactions on Medical Imaging, 2023, 42(8): 2325-2337.
DOI: 10.1109/TMI.2023.3247814. |
| [17] |
VASWANI A, SHAZEER N, PARMAR N, et al. Attention is all you need[C]//Proceedings of Neural Information Processing Systems, Long Beach: MIT Press; 2017: 5998-6008.
|
| [18] |
LIU Z, LIN Y, CAO Y, et al. Swin transformer: Hierarchical vision transformer using shifted windows[C]//Proceedings of IEEE/CVF International Conference on Computer Vision. Montreal: IEEE Computer Society, 2021: 10012-10022.
|
| [19] |
ÇICEK Ö, ABDULKADIR A, LIENKAMP S S, et al. 3D U-Net: learning dense volumetric segmentation from sparse annotation[C]//Proceedings of International Conference on Medical Image Computing and Computer-Assisted Intervention. New York: Springer; 2016: 424-432.
|
| [20] |
OKTAY O, SCHLEMPER J, FOLGOC L L, et al. Attention U-Net: Learning where to look for the pancreas[EB/OL]. arXiv: 1804.03999(2018-04-11) [2023-09-04]. https://arxiv.org/abs/1804.03999.
|
| [21] |
WU Y, LIAO K, CHEN J, et al. D-former: a u-shaped dilated transformer for 3d medical image segmentation[J].
Neural Computing and Applications, 2023, 35(2): 1931-1944.
DOI: 10.1007/s00521-022-07859-1. |
| [22] |
ZHOU H Y, GUO J, ZHANG Y, et al. nnformer: Interleaved transformer for volumetric segmentation[EB/OL]. arXiv: 2109.03201(2021-09-07) [2023-09-04]. https://arxiv.org/abs/2109.03201
|
| [23] |
GUO J, ZHOU H Y, WANG L, et al. UNet-2022: exploring dynamics in non-isomorphic architecture[EB/OL]. arXiv: 2210.15566(2022-10-27) [2023-09-04]. https://arxiv.org/abs/2210.15566
|
| [24] |
HUANG H, XIE S, LIN L, et al. ScaleFormer: revisiting the transformer-based backbones from a scale-wise perspective for medical image segmentation[EB/OL]. arXiv: 2207.14552(2022-01-29) [2023-09-04]. https://arxiv.org/abs/2207.14552
|
| [25] |
XU X, WANG T, ZHUANG J, et al. Imagechd: A 3D computedtomography image dataset for classification of congenital heart disease[C]//Proceedings of Medical Image Computing and Computer-Assisted Intervention. New York: Springer; 2020: 77-87.
|
| [26] |
VAN D W S, SCHONBERGER J L, NUNEZ J, et al. scikit-image: image processing in Python[J].
PeerJ, 2014, 2: e453.
DOI: 10.7717/peerj.453. |
 2023, Vol. 40
2023, Vol. 40