单光子发射型断层扫描 (Single-Photon Emission Computed Tomography,SPECT) 作为一种非侵入型分子成像技术已经广泛应用于心血管疾病、骨扫描、肺灌注显像等临床诊断中[1]。它的原理是将一定浓度的放射性示踪剂注射到病人体内,放射性示踪剂在衰减过程中会发射出γ光子。当γ光子沿着准直器确定的方向运动时可以被围绕在人体周围的探测器所记录,记录下来的数据可以被处理成一组沿投影角方向的投影数据。对投影数据利用重建算法进行迭代重建便可得到用于辅助临床诊断的重建图像。
然而,放射性示踪剂会对人体造成特异性辐射损伤甚至有可能增加诱发癌症的风险,因此在SPECT成像中如果能降低示踪剂的剂量则能减少对病人的损害,但是低剂量的示踪剂得到的投影数据重建后的图像往往会存在质量劣化的情况,如:图像可能存在泊松噪声、射线伪影以及空间分辨率低等问题,从而会影响临床诊断的准确性[2]。因此,如何在保持重建图像质量能满足临床诊断需要的前提下降低SPECT成像中的放射性示踪剂的剂量已成为一个热门的研究问题。基于模型迭代重建方法是低剂量投影数据重建的传统方法之一[3-5]。这种方法具有良好的数学解释性,但是传统的迭代重建方法中用到的模型参数如:变换滤波器、非线性收缩算子以及正则化参数等都需要通过先验知识经验地选取,不能满足不同剂量下的成像需要。
近年来,深度学习技术不仅在视觉领域有大量的应用[6],同时也极大地推动了医学图像重建领域的进展。在分析和迭代重建方法的基础上[7],传统重建方法和基于深度学习的结合已成为一个更加热门的趋势。在基于深度学习方法中,无论是传统重建模型还是神经网络框架中用到的模型参数都能在给定的数据集上进行优化。在存在高质量标签的大型数据集的条件下,优化的参数能让图像重建模型发挥更加强大的作用,提高图像重建任务的技术水平。目前,深度学习技术在医学图像重建领域有以下几种应用:
(1) 利用神经网络对图像进行后处理。神经网络可以学习低质量图像和高质量图像之间的映射关系。在网络训练完后,将低质量图像作为神经网络的输入,利用神经网络来提升图像的质量。在低剂量SPECT心肌灌注成像研究中,Ramon等[8]用监督学习方法训练卷积神经网络来抑制成像后的图像噪声,以此提高图像质量。Chen等[9]将自动编码器、反卷积网络和捷径连接等技术结合到残差编码−解码卷积神经网络中,用来提升低剂量计算机断层扫描(Computed Tomography,CT) 图像的质量。在编码−解码结构的基础上,Zhang等[10]进一步应用了DenseNet中的cell结构来构建编码器模块,模块中特征的重用可以增加网络的深度同时也增强了网络的表达能力。编码−解码网络结构能逐步地将输入图像压缩成特征表示,然后将特征表示重建为完整的数据集,该网络在低剂量CT研究中可以有效地抑制噪声,去除条纹伪影。在磁共振(Magnetic Resonance,MR)图像处理研究中,Basty等[11]提出了一种基于U-Net和长短期记忆网络的超分辨率网络结构,利用动态MRI数据的时序特征将低分辨率的MR图像恢复成高分辨率的MR图像。同时多分辨率网络也被提出用于磁共振图像的超分辨率重建任务[12]。
(2) 基于神经网络的端对端图像重建。利用神经网络学习投影数据与重建图像之间的映射。网络训练好后,可以将投影数据作为神经网络的输入,直接输出重建后的图像。Yang等[13-14]提出了一种定义在数据流图上的深度网络结构。该网络是基于MRI重建领域的交替方向乘子算法(Alternating Direction Method of Multipliers,ADMM)的迭代过程来设计的,作者将其称为ADMM-Net,稀疏采样的k空间数据作为ADMM-Net的输入,网络的输出为重建后的MR图像。此外,Zhang等[15]将原始−对偶混合梯度算法(Primal-Dual Hybrid Gradient Algorithm,PDHG)的迭代过程以神经网络的结构展开用于低剂量CT的重建任务中,并将该网络称为PD-Net。在PET研究中,Häggström等[16]提出了一种基于深度编码−解码网络的端对端正电子发射型计算机断层显像(Positron Emission Computed Tomography,PET) 图像重建网络结构,称为DeepPET。网络以投影数据中的正弦图作为输入,输出一个重建后的PET图像。端对端的重建方法需要大量且有良好标注的数据集,这种类型的无模型方法严重依赖数据,且容易导致网络结构泛化错误。
(3) 利用神经网络对投影数据进行预处理。神经网络将低剂量的投影数据处理成正常剂量的投影数据后再使用重建算法进行图像重建。目前已有研究[17-19]在低剂量CT中使用神经网络来合成稀疏采样中投影数据缺失的部分数据,以此来抑制成像后的噪声和伪影。Shiri等[20]使用了卷积编码器网络结构对稀疏采样的PET投影数据进行数据插值,但由于原始投影数据获取难度大,所以现有的基于深度学习的低剂量研究大多集中于图像后处理方面。
目前深度学习在医学图像重建领域中广泛应用于CT、MR以及PET中,但对于SPECT应用较少,因此本文设计了一种基于多级残差的U-Net网络结构(Multilevel-Residual U-Net,MRU-Net)应用于低剂量SPECT中。在本文中使用MRU-Net来学习低剂量SPECT中稀疏角度采样的投影数据到全角度投影数据的映射关系来对稀疏投影数据进行处理,最后再使用预处理交替投影算法[4](Preconditioned Alternating Projection Algorithm,PAPA)进行图像重建,同时将重建结果与经过残差U-Net处理后重建的图像以及用传统迭代重建算法PAPA重建的图像进行比较。
1 改进的多级残差U-Net 1.1 问题描述临床中,可以通过降低放射性示踪剂的活性来降低SPECT成像的辐射剂量。临床检查时,在固定的SPECT扫描时间内,较低活性的示踪剂会导致探测器上的检测计数率降低。低计数率通常会导致存在泊松噪声的测量投影数据。如果直接用传统的图像重建方法重建,成像后的图像质量会存在高噪声、低分辨率等情况,这种低质量图像不符合临床诊断标准。另一方面,低计数率的问题可以通过减少SPECT扫描期间的投影角度数量来解决。一个投影角度的成像时间越长,该视角的计数率越高,因此投影数据的泊松噪声越低。然而在临床实践中,总的SPECT成像时间不应进一步延长,因为总成像时间越长,会带来额外的缺点,包括由于更多患者运动、更多患者不适和更少患者吞吐量而导致图像质量变差。因此,在固定的SPECT扫描时间周期内,可以采用稀疏角度采样,每隔固定个角度采取投影数据,才能让探测器在每个投影角度获得更多的成像时间。
然而,SPECT扫描中稀疏角度采样会丢失某些角度的投影数据,从而容易导致重建后的图像产生高噪声、射线伪影等现象。为了在投影数据域合成缺失的投影数据,本文设计了多级残差U-Net网络结构,经过该网络结构处理后得到的投影数据再通过预处理交替投影算法(PAPA)进行重建。将基于神经网络的预处理方法与图像重建算法PAPA相结合,既可以利用正弦图合成,又可以利用图像的先验信息,从而得到噪声低、伪影少、精度较高的重建图像。
1.2 一种新的网络结构本文设计了基于多级残差的U-Net网络,来学习稀疏角度采样投影数据与全角度采样投影数据之间的映射关系。U-Net是一种具有编码和解码结构的经典网络,下采样过程包含多个连续的卷积层和池化层,上采样过程包含多个连续卷积层和反卷积层。对应的层之间具有拼接连接,是一种对医学图像处理非常有效的网络结构,主要用于医学图像分割[21],并且在图像去噪应用中表现出了良好的性能[22-23]。本文设计的网络结构如图1所示,网络由7个卷积模块组成。每个卷积模块由3个连续的卷积层组成,为了在训练过程中保持输入图像与输出图像大小一致,在卷积过程中使用了0填充。同时,每个卷积层的滤波器大小为3×3,采用了批归一化层,激活函数为ReLU函数,最大池化的作用是在下采样时减小图像的大小,反卷积用于上采样时扩大图像的大小。本文设计的网络结构在U-Net的基础上加入了多级残差连接,残差学习技术可以使网络更快且更有效地收敛,同时在医学图像上能减少伪影[24-25]。每一级的残差连接可以保证在下采样时图像中的重要信息不会丢失,确保能把需要的信息完整地保存至对应的上采样过程中。
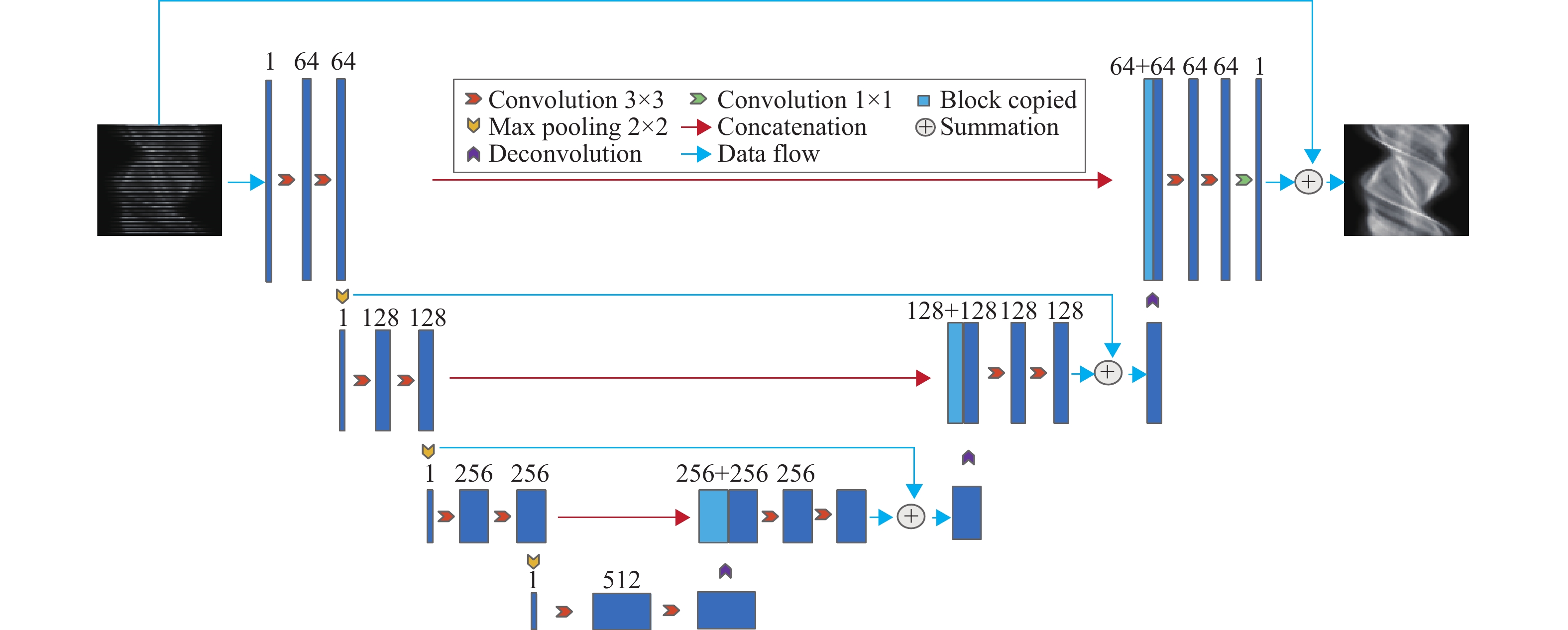
|
图 1 本文的新网络结构 Figure 1 The new network architecture |
本文中使用的实验数据是通过蒙特卡罗模拟软件Simind获得的。该软件可以模拟一台临床SPECT机对各种模体进行基于蒙特卡罗随机模拟方法的成像。Simind软件中使用了3个模体,分别是人体整身(Mdp WB) 、人体躯干(Mdp ECT) 和由多个几何形状组成的Jaszczak模体。3个模体的三维投影数据如图2所示。
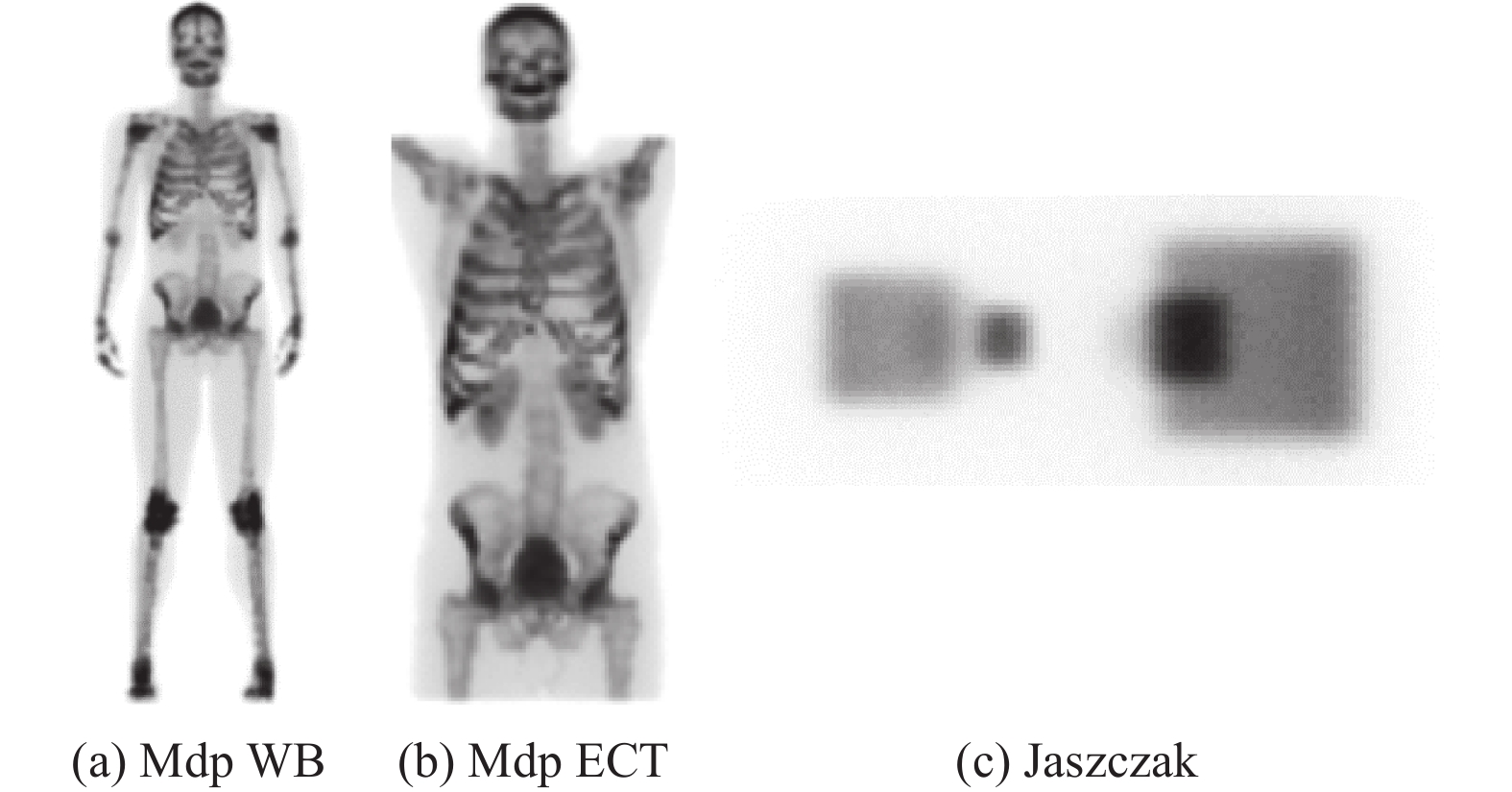
|
图 2 Mdp WB、Mdp ECT和Jaszczak模体三维投影数据 Figure 2 The projection data of Mdp WB, Mdp ECT and Jaszczak phantoms |
从三维投影数据中共提取790张二维的投影数据(正弦图),其中选取了625张数据分布清晰的图像用于实验。Simind模拟的SPECT的检测板上128个检测单元,120个投影角度,因此获得的正弦图的大小为120 × 128。
本文在3个不同的投影角度数等级下测试了所提出的网络结构的性能,分别是60个投影角度、40个投影角度以及30个投影角度,因此正弦图的大小分别是60×128,40×128,30×128。以全角度的正弦图 (120个投影角度) 作为标签图像,让MRU-Net来分别学习60×128、40×128、30×128大小的正弦图与标签图像之间的映射关系。将625张正弦图分为训练集和测试集,其中训练集包含528张正弦图数据,占总数据的84.5%;测试集包含97张正弦图数据,占总数据的15.5%,数据集的详细情况汇总于表1。
| 表 1 实验数据集 Table 1 Experimental data set |
本文通过每隔一行或多行的方式对正弦图数据进行多个投影角度数等级的稀疏角度采样。用该方法获得了全角度数二分之一、三分之一以及四分之一级别的正弦图数据,如图3所示。为了保证这些数据的大小符合神经网络的输入需要,对空行填充0值以保证正弦图数据的尺寸大小一致。

|
图 3 各个投影角度等级的正弦图 Figure 3 Sinogram of different projection views |
本文提出的网络结构是基于Pytorch深度学习框架实现的,损失函数为均方误差函数(MSE)。
| $ {\mathcal{L}}_{\mathrm{M}\mathrm{S}\mathrm{E}}=\frac{1}{n}\sum\nolimits_{i = 1}^n {{{\left( {{x_i} - {y_i}} \right)}^2}} $ | (1) |
式中:
将测试集中每个稀疏角度采样数量等级的正弦图分别输入到训练好的MRU-Net网络中,网络的输出就是处理后的正弦图。图4展示了Mdp ECT模体的胸腔部位和Jaszczak模体经过网络处理后的正弦图。

|
图 4 Mdp ECT模体胸腔区域(左列)以及Jaszczak模体(右列)的正弦图 Figure 4 Sinogram of the chest region of Mdp ECT (left) and Jaszczak (right) phantom |
将120个投影角度的正弦图重建后的图像作为标签图像,对30个投影角度的正弦图分别做如下处理:(1) 直接用传统迭代重建算法PAPA进行重建;(2) 用U-Net处理后再进行重建;(3) 用本文提出的MRU-Net处理后再进行图像重建,以上重建算法均为在同一正则化参数下的PAPA。图5中展示了MRU-Net与传统迭代重建算法对Mdp ECT模体胸腔部位的重建结果,图6则是Jaszczak模体的重建图像。从图5及图6中可以看出,与传统迭代重建算法相比,MRU-Net网络在保持平滑区域方面表现出优越的性能,比传统的迭代重建算法表现出更好的阶梯伪影抑制性能。
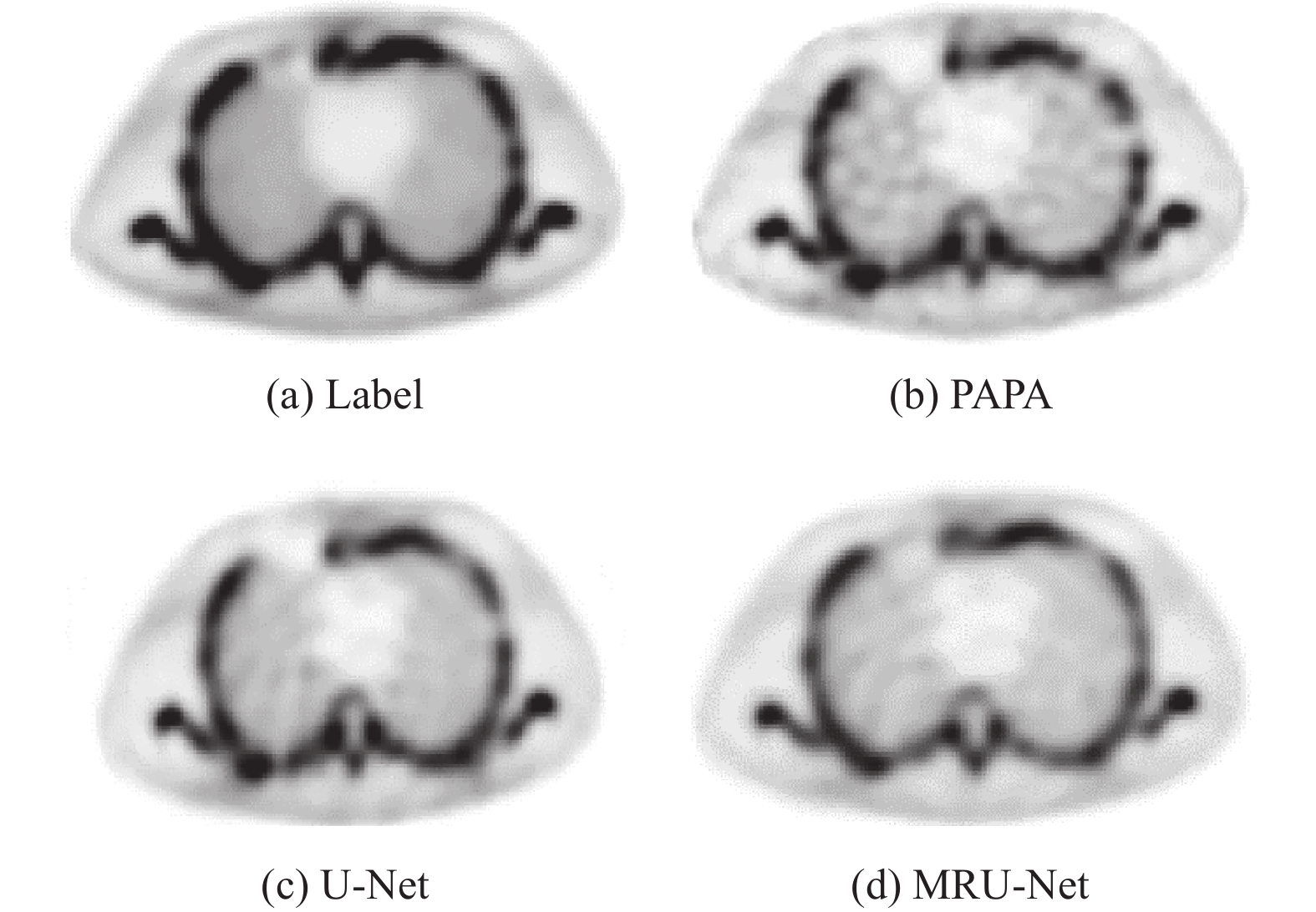
|
图 5 胸腔部位正弦图重建图像结果 Figure 5 Reconstructed images of the chest sinogram |

|
图 6 Jaszczak模体的正弦图的重建图像结果 Figure 6 Reconstructed images of the Jaszczak |
为了更直观地表现MRU-Net在低剂量SPECT图像重建中的作用,本文计算了多个投影角度数量等级的重建图像的标准均方误差(Normalized Mean-Square-Error,NMSE)、峰值信噪比(Peak Signal-to-Noise Ratio,PSNR)以及结构相似性(Structural Similarity,SSIM)。NMSE是一个用来评估重建图像质量的全局指标,表达式为
| $ {\rm{NMSE}}=\frac{{\displaystyle\sum }_{i=1}^{N}{\left({f}_{i}-{g}_{i}\right) }^{2}}{{\displaystyle\sum }_{i}^{N}{g}_{i}^{2}} $ | (2) |
式中:
PSNR是一个用来表示图像噪声等级的客观指标,表达式为
| $ {\rm{PSNR}}=10 \mathrm{lg}\left(\frac{{I}_{{\rm{max}}}^{2}}{{V}_{{\rm{MSE}}}}\right) $ | (3) |
式中:
SSIM是用来测量两个图像直接相似性的指标,表达式为
| $ {\rm{SSIM}}\left(x\text{,}y\right) =\frac{\left(2{\mu }_{x}{\mu }_{y}+{c}_{1}\right) \left(2{\sigma }_{xy}+{c}_{2}\right) }{({\mu }_{x}^{2}+{\mu }_{y}^{2}+{c}_{1}) ({\sigma }_{x}^{2}+{\sigma }_{y}^{2}+{c}_{2}) } $ | (4) |
式中:
计算结果汇总于表2至表4。以表2为例,本文分别对PAPA以及MRU-Net处理后的正弦图重建的图像计算了表中数值,计算的数值结果是测试集上进行平均后的结果。从表2中可以看出,在SSIM和PSNR方面,PAPA的结果最低,MRU-Net的表现优于PAPA。对于NMSE方面,MRU-Net的结果最低,PAPA的结果显著高于MRU-Net。以上结果表明,本文提出的网络在低剂量SPECT重建任务中能够保持较高的重建图像质量。表3和表4也有类似的结论。
| 表 2 30个投影角度正弦图的重建图像的数值指标 Table 2 The metrics of images reconstructed by two competing methods from 30 views projection data |
| 表 3 40个投影角度正弦图的重建图像的数值指标 Table 3 The metrics of images reconstructed by two competing methods from 40 views projection data |
| 表 4 60个投影角度正弦图的重建图像的数值指标 Table 4 The metrics of images reconstructed by two competing methods from 60 views projection data |
本文提出的多级残差U-Net是在U-Net的基础上加入了三级残差连接技术的网络结构,为了探究多级残差U-Net的最佳性能,分别测试了一级、二级以及三级残差U-Net(MRU-Net)在稀疏角度SPECT成像中的表现,结果如表5所示。
| 表 5 60个投影角度正弦图不同等级残差U-Net的性能 Table 5 The performance of U-Net with different levels of residuals for 60 views projection data |
本文提出了一种基于多级残差的U-Net网络(MRU-Net),旨在研究MRU-Net对稀疏角度采样的正弦图的处理能力。数据重建的实验结果表明,在不同稀疏采样角度数量等级(1/2、1/3和1/4)的正弦图处理中,本文提出的网络结构优于残差U-Net和传统的迭代重建方法,证明了所提出的网络架构在抑制噪声和伪影方面具有更好的性能。同时,从逐步加入不同等级的残差连接的实验结果可得本文提出的拥有三级残差连接的MRU-Net表现最优。因此,该网络结构能在不牺牲过多重建图像质量的前提下,显著降低SPECT成像所需示踪剂的剂量,有效保障患者健康。
| [1] |
O’MALLEY J, ZIESSMAN H, THRALL J. Nuclear medicine: the requisites [M]. 3rd ed. Maryland: Mosby, 2006.
|
| [2] |
WELLS R G. Dose reduction is good but it is image quality that matters[J].
Nucl Cardiol, 2018, 27: 238-240.
|
| [3] |
ZHANG J H, LI S, KROL A, et al. Infimal convolution-based regularization for SPECT reconstruction[J].
Medical Physics, 2018, 45: 5397-5410.
DOI: 10.1002/mp.13226. |
| [4] |
KROL A, LI S, SHEN L X, et al. Preconditioned alternating projection algorithms for maximum a posteriori ECT reconstruction[J].
Inverse Problems, 2012, 28: 115005.
DOI: 10.1088/0266-5611/28/11/115005. |
| [5] |
JIANG Y, LI S, XU Y S. A higher-order polynomial method for SPECT reconstruction[J].
IEEE Transactions on Medical Imaging, 2019, 38(5): 1271-1283.
DOI: 10.1109/TMI.2018.2881919. |
| [6] |
高俊艳, 刘文印, 杨振国. 结合注意力与特征融合的目标跟踪[J].
广东工业大学学报, 2019, 36(4): 18-23.
GAO J Y, LIU W Y, YANG Z G. Object tracking combined with attention and feature fusion[J]. Journal of Guangdong University of Technology, 2019, 36(4): 18-23. DOI: 10.12052/gdutxb.190039. |
| [7] |
ZHANG H M, DONG B. A review on deep learning in medical image reconstruction[J].
Journal of the Operations Research Society of China, 2020, 8: 311-340.
DOI: 10.1007/s40305-019-00287-4. |
| [8] |
RAMON A J, YANG Y Y, PRETORIUS P H, et al. Improving diagnostic accuracy in low-dose SPECT myocardial perfusion imaging with convolutional denoising networks[J].
IEEE Transactions on Medical Imaging, 2020, 39: 2893-2903.
DOI: 10.1109/TMI.2020.2979940. |
| [9] |
CHEN H, ZHANG Y, KALRA M K, et al. Low-dose CT with a residual encoder-decoder convolutional neural network[J].
IEEE Transactions on Medical Imaging, 2017, 36: 2524-2535.
DOI: 10.1109/TMI.2017.2715284. |
| [10] |
ZHANG Z C, LIANG X K, DONG X, et al. A sparse-view CT reconstruction method based on combination of densenet and deconvolution[J].
IEEE Transactions on Medical Imaging, 2018, 37: 1407-1477.
DOI: 10.1109/TMI.2018.2823338. |
| [11] |
BASTY N, GRAU V. Super resolution of cardiac cine MRI sequences using deep learning[C]// Image Analysis for Moving Organ, Breast, and Thoracic Images. Barcelona: Springer, 2018: 23-31.
|
| [12] |
夏皓, 蔡念, 王平, 等. 基于多分辨率学习卷积神经网络的磁共振图像超分辨率重建[J].
广东工业大学学报, 2020, 37(6): 26-31.
XIA H, CAI N, WANG P, et al. Magnetic resonance image super-resolution via multi-resolution learning[J]. Journal of Guangdong University of Technology, 2020, 37(6): 26-31. DOI: 10.12052/gdutxb.200011. |
| [13] |
YANG Y, SUN J, LI H B, et al. Deep ADMM-Net for compressive sensing MRI[C]//Proceedings of the 30th International Conference on Neural Information Processing Systems. Barcelona: ACM, 2016: 10-18.
|
| [14] |
YANG Y, SUN J, LI H B, et al. ADMM-CSNet: a deep learning approach for image compressive sensing[J].
IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 42: 521-538.
|
| [15] |
ADLER J, OKTEM O. Learned primal-dual reconstruction[J].
IEEE Transactions on Medical Imaging, 2018, 37: 1322-1332.
DOI: 10.1109/TMI.2018.2799231. |
| [16] |
HAEGGSTROEM I, SCHMIDTLEN C R, CAMPANELLA G, et al. DeepPET: a deep encoder-decoder network for directly solving the PET reconstruction inverse problem[J].
Medical Image Analysis, 2018, 54: 253-262.
|
| [17] |
DONG X, VEKHANDE S, CAO G. Sinogram interpolation for sparse-view micro-CT with deep learning neural network[C]//Physics of Medical Imaging. San Diego: SPIE, 2019: 1094820.
|
| [18] |
YUAN H Z, JIA J Z, ZHU Z X. SIPID: a deep learning framework for sinogram interpolation and image denoising in low-dose CT reconstruction[C]//2018 IEEE 15th International Symposium on Biomedical Imaging. Washington: IEEE, 2018: 1521-1524.
|
| [19] |
LEE H, LEE J, KIN H, et al. Deep-neural-network based sinogram synthesis for sparse-view CT image reconstruction[J].
IEEE Transactions on Radiation & Plasma Medical Sciences, 2019, 3: 109-119.
|
| [20] |
SHIRI I, SHEIKHZADEH P, AY M R. Deep-Fill: deep learning based sinogram domain gap filling in positron emission tomography[EB/OL]. (2019-06-16) [2021-12-20]. https://arxiv.org/abs/1906.07168v1.
|
| [21] |
RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation[C]//Medical Image Computing and Computer-Assisted Intervention. Munich: MICCAI, 2015: 234-241.
|
| [22] |
黄兴, 杨瑞梅. 结合残差U-Net神经网络和DIP的PET图像降噪[J].
光学技术, 2021, 47(2): 209-216.
HUANG X, YANG R M. PET image denoising based on residual U-Net neural network and DIP[J]. Optical Technique, 2021, 47(2): 209-216. DOI: 10.13741/j.cnki.11-1879/o4.2021.02.014. |
| [23] |
陈钧荣, 林涵阳, 陈羽中. 基于U-Net融合的保留纹理的图像去噪方法[J].
小型微型计算机系统, 2021, 42(4): 791-797.
CHEN J R, LIN H Y, CHEN Y Z. Texture-preserving image denoising method based on U-Net fusion[J]. Journal of Chinese Computer Systems, 2021, 42(4): 791-797. DOI: 10.3969/j.issn.1000-1220.2021.04.020. |
| [24] |
MCCANN M T, JIN H K, UNSER M. Deep convolutional neural network for inverse problems in imaging: A Review[J].
IEEE Transactions on Image Processing, 2017, 34: 85-95.
|
| [25] |
HAN S Y, YOO J, YE C J. Deep residual learning for compressed sensing ct reconstruction via persistent homology analysis[EB/OL]. (2016-11-25) [2021-12-20]. https://arxiv.org/abs/1611.06391.
|
 2023, Vol. 40
2023, Vol. 40

