基因在各项生理机能的调控作用中起决定性作用。DNA甲基化是一种常见的基因表观遗传修饰,在基因的表达和关闭的调节、蛋白质功能的调控以及RNA加工等生命过程中有重要影响,且与各类重大疾病的发展过程密切相关[1-2],例如在癌细胞发育过程中,甲基化水平过高是致使抑癌基因沉默进而导致癌变的主要因素。因此基因的表观遗传修饰研究,尤其是基因序列上甲基化位点的识别,一直是生物信息学中一项重要的研究内容。研究基因甲基化位点有助于了解细胞事件的发展规律,对肿瘤致病机理的研究具有重要意义[3-7]。
DNA甲基化最常见的修饰方法包括4-甲基胞嘧啶(4-mC)、5-甲基胞嘧啶(5-mC)和6-甲基腺嘌呤(6-mA)[8]。6-mA是指在DNA序列上,腺嘌呤被DNA腺嘌呤甲基转移酶催化,导致腺嘌呤的第6碳原子上添加了一个甲基。6-mA修饰广泛参与了多细胞真核生物的生物学调控,例如DNA修复和复制、基因表达和限制性修饰系统,并与癌症和其他疾病息息相关[9-13]。由于6-mA修饰在真核生物的基因序列上分布较为稀疏[14],受限于6-mA的检测技术,目前对6-mA修饰的研究工作较少。
为寻找和发现基因上的6-mA位点,研究者设计了一系列试验测定方法,如激光诱导荧光的毛细管电泳、偶联免疫沉淀的下一代测序、质谱单分子实时测序和超高效液相色谱测序等技术[15-19],这些方法很好地测定了甲基化位点及程度,但通常非常耗时并且成本高昂,同时存在实验通量低、假阳性率高等问题。因此,生物信息学工作者利用机器学习(Machine Learning, ML)方法,通过实验获得的6-mA甲基化位点数据,预测潜在的6-mA位点,为实验提供计算参考。
机器学习作为一种高效、成本低的预测方法,在6-mA甲基化数据分析及位点预测上取得了一定成果,例如Bhasin等[20]利用支持向量机(Support Vector Machine,SVM)对甲基化位点附近的基因序列进行研究,并且开发了Methylator甲基化位点预测工具;Zhang等[21]利用随机森林(Random Forest,RF)作为分类器,根据甲基化位点和周围的基因调控因子信息设计了甲基化位点预测模型;Feng等[22]利用朴素贝叶斯模型,根据伪三核苷酸组合信息,对CpG岛甲基化水平进行预测。机器学习方法可以通过较低成本实现6-mA位点的预测,而且在处理轻量级基因序列库时有良好的性能表现,成功解决了许多生物学问题[23-24]。但随着基因样本数量剧增,机器学习方法处理这些基因大数据时,计算速度有所下降,准确率也无法进一步提升。更重要的是,机器学习通过人工设计的特征提取方法难以实现基因大数据的高维特征处理。因此,为提高基因大数据背景下6-mA的预测能力,需要增加模型对基因数据的特征提取能力,实现高性能、自动化的数据挖掘功能。
深度学习(Deep Learning,DL)作为改进的人工神经网络方法,可以自动挖掘样本中隐含的特征信息,在处理大数据时有良好的性能表现[25]。在6-mA位点预测相关领域,深度学习方法在预测精度和可分析数据规模上较传统方法均取得了一定的进步。例如Tian等[26]设计了可实现自定义特征的6-mA甲基化位点预测的CNN模型;Zeng等[27]利用卷积神经网络(Convolutional Neural Network,CNN)对 6-mA甲基化位点进行预测,并开发了CpGenie甲基化位点预测工具;Angermueller等[28]在CpGenie基础上,通过在CNN模型结构中设置双向长短时记忆网络(Bidirectional Long Short Term Memory,Bi-LSTM)层,形成了双向门控制循环网络,开发了DeepCpG预测工具,可用于预测细胞6-mA甲基化变异的相关信息。现有文献工作主要使用CNN处理基因序列(One Dimensional Convolutional Neural Network Model,1D-CNN),这种一维序列提供的特征种类和数量有限,为进一步提高深度学习模型的数据挖掘效果,需要为模型提供更多基于序列模式的特征。
综上所述,为充分利用6-mA基因大数据,增加深度学习模型从基因序列上提取到的特征种类和数量,本文提出了一种基于LSTM神经网络的基因甲基化预测的深度学习方法。首先,使用2-mer编码方法将碱基序列从包含4种字符、长度为41 bp的1D字符序列处理为包括16种字符、长度为40的1D格式;然后通过应用One-hot编码方法,将40×1的1D基因序列扩展成40×16的2D数据矩阵,使用向量作为序列特征的数值表达。在模型设计方面,根据甲基化位点预测任务序列长度短、序列数量多、序列模式复杂的特点,在LSTM功能层之前,设置向量编码处理层优化对向量矩阵的处理性能,并对初始化方法及多种优化器进行最佳模型参数的择优,以提高LSTM模型的处理性能。本文首次结合使用K-mer复合编码及One hot编码方法对甲基化位点序列进行编码优化,通过人工编码设计实现短基因序列任务的特征增强,有助于提高深度学习方法对甲基化任务的分析性能。
1 材料和方法 1.1 基因数据集收集在文献[29]中,研究者使用6-mA位点数据集对一系列的基因长度窗口进行对比测试,最终选取了表现性能最佳的长度为41 bp的基因序列作为实验样本。本实验使用58670条蔷薇基因序列作为训练集,使用另外14600条蔷薇基因作为测试集,每次实验前使用随机排序将数据集中的数据顺序打乱,并保证正负样本以1:1的比例随机分配至训练集和测试集,正样本的序列上该位点经实验验证存在甲基化,负样本表示该位点不会发生甲基化。本方法使用水稻和拟南芥基因样本作为测试模型,并在其他物种数据集上进行实验。
1.2 K-mer编码基因为了进一步挖掘基因序列潜在的物种特异性信息,使用K-mer编码对基因序列预处理。由于A、T、C、G四种碱基在表示特征种类时不利于模型提取特征,本文使用K-mer编码基因序列,将基因序列分成包含k个碱基的片段和对碱基片段,如式(1)所示。
| $ n = m - k + 1 $ | (1) |
式中:n为序列经过编码后k-mer片段数量,m为原序列长度,k为截取子序片段长度。在本文方法中,m为41,k为2,n为40,即在实验中使用的基因序列长度为41 bp,通过 k=2的2-mer编码方式,将长度为41 bp的基因序列处理为长度为40的2-mer编码序列,处理方法如图1(a)所示。2-mer对应字符如图1(b)所示。

|
图 1 2-mer编码示意图 Figure 1 Encoding process of 2-mer format diagram |
根据图1(b)中的对应字符,将由A、T、C、G四种碱基组成的41bp序列编码为长度为40的由A ~P字符组成的序列,可以在保持序列基本长度的同时,将序列中字符的种类从4种提高至16种,解决了A、T、C、G四种碱基表示的特征种类太少的问题。
1.3 One-hot编码经过K-mer处理后的基因样本数据格式为40×1,为了让模型从数据中获取更多特征,使用One-hot对样本维度进一步扩展。
One-hot编码,又称作一位有效编码。该编码方法将x种不同字符种类编码成长度为x的二进制序列,每个二进制序列中仅有一位是1,使用1在二进制中的不同位置来对应x种字符。One-hot编码用于从低维度数据中提取高维度特征,使模型可以从数据中提取更多的特征。
使用One-hot方法,进一步对长为40的字符序列进行特征向量编码,采用16位0/1值来表示,每一组数字中只有一个1,其余15个为0。用不同位上的1值代表16个不同的字母。最后,使用One-hot方法将字符序列编码为行数16、列数40的一维矩阵作为深度学习模型的输入。
1.4 LSTM预测方法设计生物序列问题通常使用循环神经网络(Recurrent Neural Network, RNN)处理,但RNN存在序列短期记忆问题,使用RNN处理较长的基因序列会出现梯度消失或梯度爆炸的现象,进而影响结果的准确度[30]。为解决RNN记忆短、处理长序列效果不佳等问题,本文方法在RNN的结构基础上增加了包括输入门、输出门,以及遗忘门在内的门控单元,建立具有长记忆力且具有遗忘功能的LSTM模型,通过学习基因长距离之间的隐藏关系特征,来完成对序列数据的预测工作。LSTM神经网络结构如图2(a)所示。
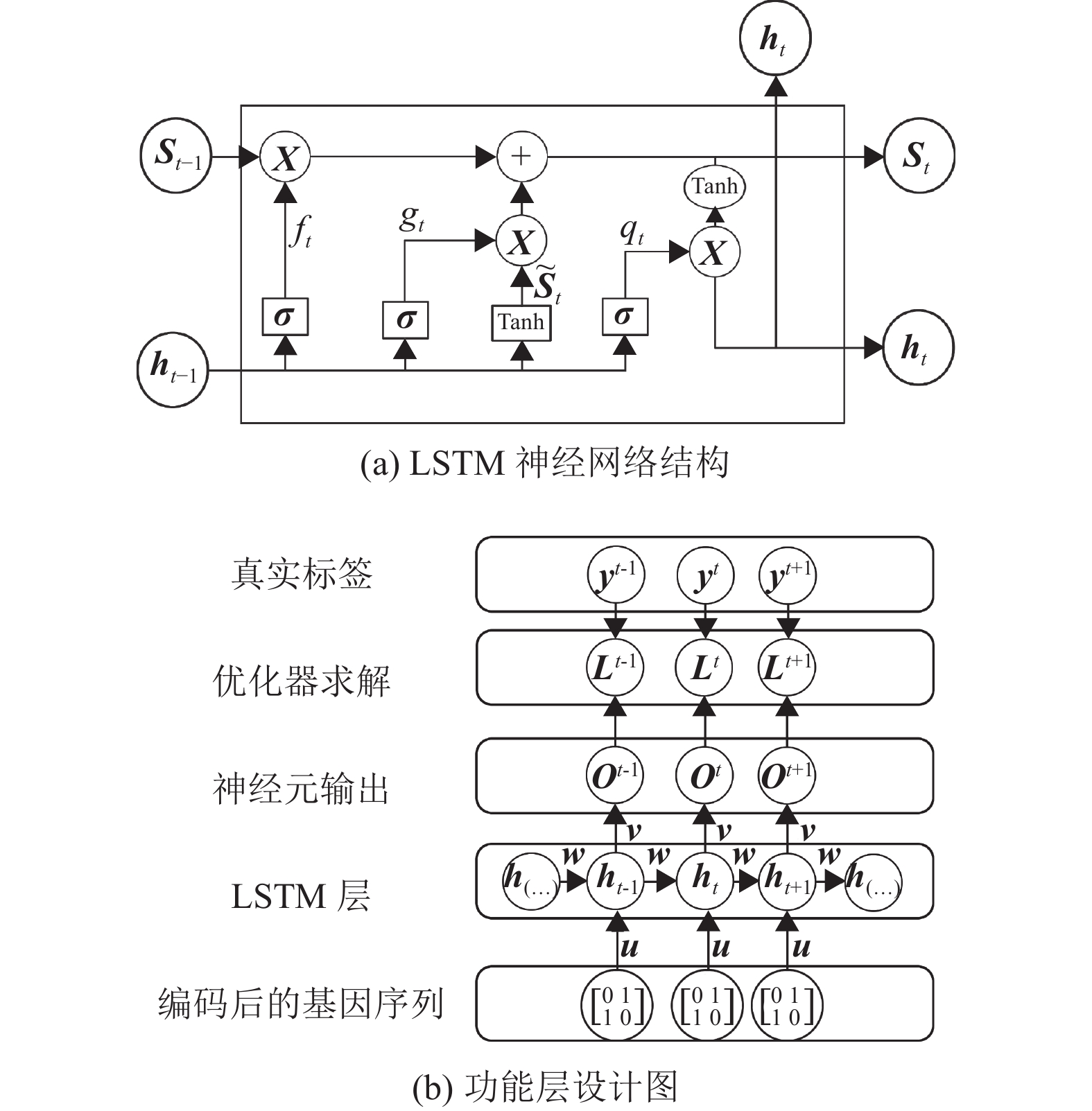
|
图 2 模型设计示意图 Figure 2 Model design diagram |
使用LSTM模型处理基因序列的流程为:先对基因样本序列进行复合编码预处理,将每一条41 bp长度的基因序列编码为40×16的One-hot矩阵。然后将预处理好的数据集输入LSTM层,建立序列中前后单元的关联信息,参与模型计算并提取样本高维特征。通过使用优化器,对训练生成的权重参数进行更新,不断降低损失函数值,提高模型准确率,实现对序列高维特征的有效学习。功能层设计如图2(b)所示。
1.5 模型评价指标正负样本的输出结果使用以下指标统计:TP(True Positive)为正样本被模型正确分类;FP(False Positive)为正样本被模型错误分类;TN(True Negative)为负样本被模型正确分类;FN(False Negative)为负样本被模型错误分类。通过统计正负样本的分类结果,使用以下指标进行结果的对比分析,包括准确性Ac(Accuracy)、敏感性Se(Sensitive)、特异性Sp(Specificity)、精确度Pr(Precision)、综合评价指标Fm和马氏相关系数Mc(Matthews Correlation Coefficient)、受试者工作特性曲线(Receiver Operating Char-acteristic curve,ROC)、曲线线下面积(Area Under Curve,AUC),以及准确率−召回率曲线(Precision-recall Curve,PR)。各指标的定义如式(2)~(7)所示。
| $ {{A_{\rm{c}}}} = \frac{{{{T_{\rm{P}}}} + {{T_{\rm{N}}}}}}{{{{T_{\rm{P}}}} + {{T_{\rm{N}}}} + {{F_{\rm{P}}}} + {{F_{\rm{N}}}}}} $ | (2) |
| ${S_{\rm{e}}} = \frac{{{T_{\rm{P}}}}}{{{T_{\rm{P}}} + {F_{\rm{N}}}}}$ | (3) |
| ${S_{\rm{p}}} = \frac{{{T_{\rm{N}}}}}{{{T_{\rm{N}}} + {F_{\rm{P}}}}}$ | (4) |
| ${P_{\rm{r}}} = \frac{{{T_{\rm{P}}}}}{{{T_{\rm{P}}} + {F_{\rm{P}}}}}$ | (5) |
| ${F_{\rm{m}}} = \frac{{{\rm{2}} \times {T_{\rm{P}}}}}{{{\rm{2}} \times {T_{\rm{P}}} + {F_{\rm{P}}} + {F_{\rm{N}}}}}$ | (6) |
| ${M_{\rm{c}}} = \frac{{{T_{\rm{P}}} \times {T_{\rm{N}}} - {F_{\rm{P}}} \times {F_{\rm{N}}}}}{{\sqrt {{\rm{(}}{T_{\rm{P}}} + {F_{\rm{P}}}{\rm{)(}}{T_{\rm{P}}} + {F_{\rm{N}}}{\rm{)(}}{T_{\rm{N}}} + {F_{\rm{P}}}{\rm{)(}}{T_{\rm{N}}} + {F_{\rm{N}}}{\rm{)}}} }} $ | (7) |
式中:Ac为该模型做出的判断属于正确判断的比例,即模型的预测准确率;Se与Sp分别为该模型对于正样本以及负样本做出正确判断的比例;Pr与Se相类似,反映模型对正样本分类的准确率;Fm指数用于评判模型分类好坏;Mc为一个综合考虑到TP、TN、FP、FN四个参数的指标,取值结果越接近1,表示模型预测越接近真实值。
受试者工作特性曲线ROC,又称感受性曲线,横坐标为FR,纵坐标为TR,公式为
| $ {F_{\rm{R}}} = \frac{{{F_{\rm{P}}}}}{{{F_{\rm{P}}} + {F_{\rm{N}}}}} $ | (8) |
| ${T_{\rm{R}}} = \frac{{{T_{\rm{P}}}}}{{{T_{\rm{P}}} + {F_{\rm{N}}}}} $ | (9) |
FR值为负样本判错的比例,TR为正样本判断正确的比例。曲线越弯曲,越趋向于坐标系左上角,模型所预测的结果越接近真实值。
1.6 最佳模型参数的选择模型权重参数的初始化方法对深度学习模型的训练过程有重要影响,本节将对模型初始化参数以及模型优化器参数进行对照实验,选择合适的参数初始化方法以及优化器,从而加快模型收敛至全局最优的速度,并提高模型性能。
在实验中使用蔷薇科数据集进行训练,并使用拟南芥数据集作为校验集,进行5次交叉验证,分别使用不同的初始化方法训练模型300轮,实验结果如图3(a)所示。

|
图 3 模型参数性能表现示意图 Figure 3 Model parameter performance diagram |
本实验共使用11种权重参数初始化方法,分别是:Glorot 正态分布初始化器(Glorot Normal)、Glorot 均匀分布初始化器(Glorot Uniform)、He 正态分布初始化器(He Normal)、He 均匀方差缩放初始化器(He Uniform)、Le Cun 正态分布初始化器(Le Cun Normal)、Le Cun 均匀初始化器(Le Cun Uniform)、截断正态分布初始化器(Truncated Normal)、标准正态分布初始化器(Random Normal)、均匀分布初始化器(Random Uniform)、单位矩阵初始化器(Identity)、随机正交矩阵初始化器(Orthogonal)。
从图3(a)中的数据可得Glorot均匀分布初始化方法在Ac、Pr、Sp、Fm和Mc这5个指标上均优于其他初始化方法。从综合角度考虑,本实验选用Glorot均匀分布初始化方法作为模型权重的初始化方法。
为确定最适合分析基因序列数据的优化器,对7种优化器进行性能分析,分别是:RMSprop算法、Adam算法、随机梯度下降算法(Stochastic Gradient Descent,SGD)、Adagrad算法、Adadelta算法、Adamax算法和Nadam算法。本实验对水稻以及拟南芥数据集分别进行5次随机实验,五折交叉验证的结果如图3(b)所示。
从图3(b)的对比结果可以看出,Adam优化器的性能在各项指标上优于其他优化器。Adam优化器综合考虑一阶矩估计、二阶矩估计,以及计算步长的更新,能加快梯度下降的速度。使用Adam优化器,梯度伸缩变换不会影响模型参数的更新,适用于基因数据集这种数据规模较大的任务,且可以在训练过程中解决存在的梯度稀疏或梯度噪声问题[31]。因此选择Adam优化器作为模型的优化器。Adam优化器的学习率设置为1×10−4,同时设置模糊因子参数为None,权重衰减为1×10−6[32]。
2 结果 2.1 模型训练性能本实验利用蔷薇科基因作为训练集,将水稻基因与拟南芥基因分别作为独立测试集,对水稻基因以及拟南芥基因进行预测,以评估模型在甲基化数据集上的分类性能。使用Glorot均匀分布初始化方法以及Adam优化器的LSTM模型经过300轮训练后,模型性能表现如图4所示。当使用一般规模(63748条拟南芥基因数据)的数据集时,测试集和训练集的Ac曲线平稳上升,且无明显波动,说明训练效果较好,从损失值Ls曲线可以看出模型在训练过程中并未因使用Adam优化器(该优化器的梯度优化方向是随机的)而产生较大扰动,说明模型在训练过程中并未出现欠拟合或过拟合的情况,训练效果良好。

|
图 4 模型性能表现 Figure 4 Model performance |
进一步分析模型在不同数据集上的ROC曲线以及计算AUC值来验证泛化能力,使用的数据集来自6个不同物种[14],分别为Caenorhabditis elegans(C.elegans,包含3108条数据),Drosophila melanogaster(D.melanogaster,包含3538条数据),Arabidopsis thaliana(A.thaliana,包含3956条数据),Escherichia coli(E.coli,包含776条数据),Geoalkalibacter subterraneus(G.subterruneus,包含1812条数据) 以及Geobacter pickeringii(G.pickeringii,包含1138条数据)。分别对每个物种基因进行预处理,并且以5:1:1的比例划分训练集、测试集和校验集。
本方法在不同数据集上的准确率如图5所示。根据图5可得,在不同种类且不同规模的基因数据集上,本方法的Ac可稳定地保持在0.82以上,在这6个数据集上的ROC曲线如图6所示。

|
图 5 不同数据集准确率结果图 Figure 5 Accuracy result of each dataset |

|
图 6 模型在6种不同物种基因数据集上的ROC曲线 Figure 6 ROC vaules of LSTM model on 6 different species |
图6表明对于不同物种的基因数据集,本方法的分类性能较为稳定。6个数据集的AUC值分别为0.924、0.9262、0.9109、0.9067、0.9055及0.9181,训练数据和测试数据的AUC值均大于0.90,结合ROC曲线可知基于复合特征的LSTM模型可以应用于较大的数据集中,而且在不同规模和不同物种数据集上的预测性能较高,在规模较小的数据集上仍然可以保持稳定的性能表现。
2.2 与其他方法的对比本方法与传统的机器学习方法和最近发表的深度学习方法[29]进行了对比。所有的方法均使用相同的蔷薇科benchmark数据集进行训练及校验,分别就Mc、Ac、Se以及Sp四个指标对模型性能进行评价,本方法与这些方法的5次交叉验证实验结果对比见图7,RF为随机森林方法(Random Forest),GB为梯度提升方法(Gradient Boosting),LR为逻辑回归方法(Logistic Regression),ERT为回归树算法(Ensemble of Regression Trees),SVM为支持向量机方法(Support Vector Machines),NB为朴素贝叶斯算法(Naive Bayes)。

|
图 7 各种算法分类器性能对比 Figure 7 Comparison between the proposed method with other algorithm classifier |
图7中复合编码的LSTM模型的性能均优于传统机器学习方法、深度学习方法和单独使用One-hot编码的LSTM模型。实验结果表明复合编码LSTM的Mc值约为0.87,比其他方法都更接近于1,预测更接近真实值;并且Ac达到了约0.938,高于其他模型方法,预测更加准确;Sp和Se也优于其他方法,说明复合编码LSTM模型能够更加准确地对正负样本进行预测。
此外,与7种在线6-mA预测工具进行了对比[33-35]。使用Mc、Ac、Se以及Sp 四个指标综合考究模型对样本的分析能力。实验结果对比如表1所示,其中MK指马尔科夫模型方法(Markov Model)。
| 表 1 本方法与在线工具对比结果 Table 1 Comparison with other 6-mA online prediction tools |
表1说明了处理较大规模的数据集时,本方法可以提供更精准的预测结果,在性能上优于其他预测工具。复合特征的LSTM模型也优于传统的机器学习方法(RF,MK和SVM),说明复合特征可以有效增强深度学习模型的性能。
2.3 对潜在甲基化位点的预测能力为进一步验证提出的LSTM模型对未知基因的6-mA位点预测能力,在美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)获取水稻粳稻基因组测序计划中编号为NC_029258.1的水稻基因,对应第三号染色体上28512604位至28 515 156位的基因序列,并对这条序列中的所有A碱基进行预处理,形成404条长度为41 bp的基因片段。将这404条序列输入训练好的LSTM模型中,共训练300轮,模型预测404个A碱基位点上有29个位点存在甲基化的可能。分别使用4种工具对这29个位点进行验证,验证结果如表2所示。在4种工具对29个位点的预测中,均超过25个结果为正(86%),证明该模型的预测结果得到当前最新预测工具的支持,结论可靠。
| 表 2 不同6-mA预测工具对潜在甲基化位点的验证结果 Table 2 Verification results of potential methylation sites with different 6-mA prediction tools |
本研究以甲基化位点预测为目标,设计了一种结合使用One-hot编码与K-mer编码的序列编码方法,使用LSTM模型学习序列数据特征,实现了快速、精准的甲基化位点预测功能。
甲基化位点预测的常用方法是使用机器学习方法和深度学习方法,从甲基化位点数据集中学习数据特征规律,进而对未知的碱基位点做出分类判断。然而,甲基化的形成过程受诸多因素影响,甲基化位点上下游序列的碱基组成具有多种模式。从甲基化酶作用甲基化位点角度来看,甲基化位点前后数位的碱基组成会影响碱基位点与甲基化酶的结合能力,进而影响甲基化位点的形成,因此越靠近甲基化位点的碱基,对分析模型的权重影响越大,甲基化位点上下游3~5位的侧翼位序列、以甲基化位点为中心总长度在11 bp以内的碱基序列对甲基化位点的判断影响较大[36]。但是从机器学习角度出发,训练数据的信息丰度对模型训练的效果有明显的影响,序列数据越长,则信息量越大,机器学习模型的数据特征越多,这与靠近甲基化位点的碱基影响权重高、远离甲基化位点的影响碱基权重低的事实存在冲突,因此本研究首先就训练数据的长度进行了选择。文献中有部分关于最佳序列长度的讨论[37-39],在以上研究基础上,进一步参考文献[40],最终将训练序列的长度设置为41 bp,即以甲基化位点为中心,上下游各20个碱基,既可以为LSTM提供足够的数据特征,也可以将序列特征体现出的甲基化规律限制在甲基化位点上下游较小的范围内,从而确保预测结论的合理性。
结果表明,采取复合编码后,LSTM模型可以实现精准的基因序列数据分析。经过模型最佳初始化方法和最佳优化器的选择,可以有效提升LSTM模型的处理性能。从模型训练过程中的准确率和损失函数值变化可以看出训练过程平滑无明显抖动,且收敛速度较快(300轮迭代训练过程中前100轮以内可实现收敛)。通过ROC曲线可以看出使用复合编码的LSTM模型具有较好的分类性能(AUC均大于0.9)。与文献中报道的较新分析工具相比,本方法在准确率、特异性等指标上均存在优势,同时本方法在水稻基因NC_029258.1上的预测结果也受到这些预测工具的支持,表明本方法具有一定的应用潜力。
同时,本方法仍存在两处不足:首先,缺乏基于碱基序列折叠动力学的分析,预测出的位点是否暴露在甲基化酶可以作用到的催化腔并没有得到理论证明或计算依据;其次,本方法的结论是通过甲基化数据集的数据内容分析得到,预测的甲基化位点在碱基序列组成模式上可能与现有的数据具有相似性,而忽略了其他潜在的具有不同模式规律的甲基化位点。因此,在后续工作中,考虑进一步结合现有研究数据,研究如何关联更多数据规律,挖掘碱基序列的更多模式。
| [1] |
KULIS M, ESTELLER M. DNA methylation and cancer[J].
Advances in Genetics, 2010, 70(10): 27-56.
|
| [2] |
ROBERTSON, KEITH D. DNA methylation and human disease[J].
Nature Reviews Genetics, 2005, 6(8): 597-610.
DOI: 10.1038/nrg1655. |
| [3] |
LOPEZ-SERRA P, ESTELLER M. DNA methylation-associated silencing of tumor-suppressor micro-RNAs in cancer[J].
Oncogene, 2012, 31(13): 1609-1622.
DOI: 10.1038/onc.2011.354. |
| [4] |
LYU H, DAO F Y, ZHANG D, et al. Advances in mapping the epigenetic modifications of 5-methylcytosine (5mC), N6-methyladenine (6mA), and N4‐methylcytosine (4mC)[J].
Biotechnology and Bioengineering, 2021, 118(11): 4204-4216.
DOI: 10.1002/bit.27911. |
| [5] |
DAY J J, CHILDS D, GUZMAN-KARLSSON M C, et al. DNA methylation regulates associative reward learning[J].
Nature Neuroscience, 2013, 16(10): 1445-1452.
DOI: 10.1038/nn.3504. |
| [6] |
YANG X J, LAY D F, HAN H, et al. Targeting DNA methylation for epigenetic therapy[J].
Trends Pharmacol Sci, 2010, 31(11): 536-546.
DOI: 10.1016/j.tips.2010.08.001. |
| [7] |
MEISSNER A, MIKKELSEN T S, GU H C. Genome-scale DNA methylation maps of pluripotent and differentiated cells[J].
Nature, 2008, 454: 766-770.
DOI: 10.1038/nature07107. |
| [8] |
LIANG Z, SHEN L S, CUI X A, et al. DNA N6-adenine methylation in arabidopsis thaliana[J].
Developmental Cell, 2018, 45(3): 406-416.
DOI: 10.1016/j.devcel.2018.03.012. |
| [9] |
LIU M C, OXNARD G R, KLEIN E A, et al. Sensitive and specific multi-cancer detection and localization using methylation signatures in cell-free DNA[J].
Ann Oncol, 2020, 31: 745-759.
DOI: 10.1016/j.annonc.2020.02.011. |
| [10] |
CATANIA S, PHILLIP A D, HAROLD P, et al. Evolutionary persistence of DNA methylation for millions of years after ancient loss of a de novo methyltransferase
[J].
Cell, 2020, 180(20): 263-277.
|
| [11] |
CHAI P W, YU J, GE S F, et al. Genetic alteration, RNA expression, and DNA methylation profiling of coronavirusdisease 2019 (COVID-19) receptor ACE2 in malignancies: a pan-cancer analysis[J].
Journal of Hematology Oncol, 2020, 13: 1-5.
DOI: 10.1186/s13045-019-0838-y. |
| [12] |
IZZO F, LEE S C, PORAN A, et al. DNA methylation disruption reshapes the hematopoietic differentiation landscape[J].
Nature Genetics, 2020, 52(4): 1-10.
|
| [13] |
JOSÉ A E, MENENDEZ J A. Potential drugs targetingearly innate immune evasion of SARS-coronavirus 2 via 2'-O-methylation of viral RNA[J].
Viruses, 2020, 12(5): 525.
DOI: 10.3390/v12050525. |
| [14] |
YANG J L, LANG K, ZHANG G L, et al. SOMM4mC: a second-order markov model for DNA N4-methylcytosine site prediction in six species[J].
Bioinformatics, 2020, 36(14): 4103-4105.
DOI: 10.1093/bioinformatics/btaa507. |
| [15] |
KRAIS A M, CORNELIUS M G, SCHMEISER H H. Genomic N6- methyladenine determination by MEKC with LIF[J].
Electrophoresis, 2010, 31(21): 3548-3551.
DOI: 10.1002/elps.201000357. |
| [16] |
SMITH Z D, MEISSNER A. DNA methylation: roles in mammalian development[J].
Nature Reviews Genetics, 2013, 14(3): 204-220.
DOI: 10.1038/nrg3354. |
| [17] |
LUO G Z, WANG F, WENG X C, et al. Characterization of eukaryotic DNA N6-methyladenine by a highly sensitive restriction enzyme-assisted sequencing[J].
Nature Communications, 2016, 7(1): 1-6.
|
| [18] |
ZHANG G Q, HUANG H, LIU D, et al. N6-methyladenine DNA modification in Drosophila[J].
Cell, 2015, 161: 893-906.
DOI: 10.1016/j.cell.2015.04.018. |
| [19] |
FANG G, MUNERA D, FRIEDMAN D I, et al. Genome-wide mapping of methylated adenine residues in pathogenic Escherichia coli using single-molecule real-time sequencing[J].
Nature Biotechnology, 2012, 30(12): 1232-1239.
DOI: 10.1038/nbt.2432. |
| [20] |
BHASIN M, ZHANG H, REINHERZ E L, et al. Prediction of methylated CpGs in DNA sequences using a support vector machine[J].
FEBS Letters, 2005, 579(20): 4302-4308.
DOI: 10.1016/j.febslet.2005.07.002. |
| [21] |
ZHANG Q Y, AIRES-DE-SOUSA J. Random forest prediction of mutagenicity from empirical physicochemical descriptors[J].
Journal of Chemical Information and Modeling, 2007, 47(1): 1-8.
DOI: 10.1021/ci050520j. |
| [22] |
FENG P M, CHEN W, LIN H. Prediction of CpG island methylation status by integrating DNA physicochemical propertyes[J].
Genomics, 2014, 104(4): 229-233.
DOI: 10.1016/j.ygeno.2014.08.011. |
| [23] |
YU H, WANG S, LEE X R, et al. Algorithm study of real-time detection of sleep apnea-hypopnea event based on long-short term memory-convolutional neural network[J].
Chinese Journal Biomedical Engineering, 2020, 39(3): 303-310.
|
| [24] |
AMIN R, RAHMAN C R, SHATABDA S, et al. i6mA-CNN: a convolution based computational approach towards identification of DNA N6-methyladenine sites in rice genome[J].
Sci Rep, 2020, 11(1): 10458.
|
| [25] |
WANG Y J, HUANG F L, HUANG S, et al. Breast cancer image classification based on fusion multi-network deep convolution features and sparse double relation regularization method[J].
Chinese Journal Biomedical Engineering, 2020, 39(5): 532-540.
|
| [26] |
TIAN Q, ZOU J X, TANG J X, et al. MRCNN: a deep learning model for regression of genome-wide DNA methylation[J].
BMC Genomics, 2019, 20(2): 1-10.
|
| [27] |
ZENG H Y, GIFFORD D K. Predicting the impact of non-coding variants on DNA methylation[J].
Nucleic Acids Research, 2017(11): 11.
|
| [28] |
ANGERMUELLER C, LEE H J, REIK W, et al. DeepCpG: accurate prediction of single-cell DNA methylation states using deep learning[J].
Genome Biol, 2017, 18(1): 1-13.
DOI: 10.1186/s13059-017-1189-z. |
| [29] |
HASAN M M, BASITH S, SHAMIMA K M, et al. Meta-i6mA: an interspecies predictor for identifying DNA N6-methyladenine sites of plant genomes by exploiting informative features in an integrative machine-learning framework[J].
Brief Bioinform, 2020, 22(3): bbaa202.
DOI: 10.1093/bib/bbaa202. |
| [30] |
HASAN M M, MANAVALAN B, SHOOMBUATONG W, et al. i6mA-Fuse: improved and robust prediction of DNA 6mA sites in the Rosaceae genome by fusing multiple feature representation[J].
Plant Molecular Biology, 2020, 103(1): 225-234.
|
| [31] |
CONG P, ZHANG G G, LI F, et al. MM-6mAPred: identifying DNA N6-methyladenine sites based on Markov model[J].
Bioinformatics, 2019, 36(2): 388-392.
|
| [32] |
BASITH S, MANAVALAN B, SHIN T H, et al. SDM6A: a web-based integrative machine-learning framework for predicting 6mA sites in the rice genome[J].
Molecular Therapy-Nucleic Acids, 2019, 18: 131-141.
DOI: 10.1016/j.omtn.2019.08.011. |
| [33] |
LYU H, DAO F Y, GUAN Z X, et al. iDNA6mA-Rice: a computational tool for detecting N6-methyladenine sites-in rice[J].
Frontiers in Genetics, 2019(10): 793.
DOI: 10.3389/fgene.2019.00793;. |
| [34] |
XUH D, HUR F, JIAP L, et al. 6mA-Finder: a novelonline tool for predicting DNA N6-methyladenine sites in genomes[J].
Bioinformatics, 2020, 36(10): 3257-3259.
DOI: 10.1093/bioinformatics/btaa113. |
| [35] |
CHEN W, LYU H, NIE F L, et al. i6mA-Pred: identifying DNA N6-methyladenine sites in the rice genome[J].
Bioinformatics, 2019, 35(11): 2796-2800.
|
| [36] |
CHENG M, SHU X, CAO J, et al. A mutation-based method for pinpointing a DNA N6-methyladenine methyltransferase's modification site at single base resolution[J].
Chem Bio Chem, 2021, 22(11): 1936-1939.
DOI: 10.1002/cbic.202100088. |
| [37] |
LEE H K, BARBAROSIE M, KAMEYAMA K, et al. Regulation of distinct AMPA receptor phosphorylation sites during bidirectional synaptic plasticity[J].
Nature, 2000, 405(6789): 955-978.
DOI: 10.1038/35016089. |
| [38] |
XUE Y, ZHOU F F, ZHU M J, et al. GPS: a comprehensive www server for phosphorylation sites prediction[J].
Nucleic Acids Research, 2005, 33: 184-187.
|
| [39] |
KIM J H, LEE J, OH B, et al. Prediction of phosphorylation sites using SVMs [J]. Bioinformatics, 2004, 20(17) : 3179-3184.
|
| [40] |
ZHU Q L, LI X L, CONESA A, et al. GRAM-CNN: a deep learning approach with local context for named entity recognition in biomedical text [J]. Bioinformatics, 2017, 34(9) : 1547-1554.
|
 2023, Vol. 40
2023, Vol. 40

