农业病虫害问题是我国主要的农业自然灾害之一,具有种类繁多、影响大且时常爆发成灾的特点。目前的农作物病虫害识别手段主要是人工识别或计算机辅助识别,但由于病虫害的种类繁多、识别起来速度慢、错误率高,而且对专业知识要求高,现有的病虫害识别手段越来越离不开计算机的辅助。为了解决该问题,赵国华等[1]利用进阶精简指令集机(Advanced RISC Machine,ARM)对农作物病虫害的识别系统构建进行研究,该方法通过图像扫描提取图像的数字化特征,然后遍历数据库,通过K-means聚类计算特征相似度来输出相似度最高的病虫害种类。这种方法存在识别精度低和识别速度慢的问题。柴帅等[2]利用深度卷积神经网络(Deep convolutional neural network,DCNN)实现番茄叶片的病虫害识别,通过构建VGG-19网络缓和了识别番茄叶片病虫害时出现的过拟合现象,提取特征后利用支持向量机进行分类,进一步提高了识别准确率。但该方法存在对一些特定类别的识别率低的问题。孙军[3]用目标识别的方式来对茶树病虫害进行识别,通过基于目标分割算法实现目标位置锁定,确定阈值调整目标位置的图像比例尺寸,再对目标属类进行划分,效果优于普通的卷积神经网络(Convolutional Neural Network,CNN)模型。但均匀的密集采样的一个重要缺点是训练比较困难,因为正样本与负样本(背景)极其不均衡,导致模型准确度稍低。因此,设计出训练速度快、识别准确率高和分类效果好的农作物病虫害识别算法是急需解决的问题,对于改善种植环境、提高粮食产量以及推动我国精准农业的发展都具有重要意义。
当前的计算机视觉任务主要采用深度学习的方法,在图像识别和分类任务上都取得了较好的效果。2012年ImageNet竞赛冠军获得者Hinton和他的学生Alex Krizhevsky[4]设计的AlexNet是深度学习用于图像分类的一个划时代算法。与传统的人工特征方法的图像分类相比,AlexNet使得分类的准确率大幅度提升。Szegedy等[5]另辟蹊径,用回归的方法解决目标识别问题,将深度卷积网络作为特征提取器,然后回归出目标的种类。Dumitru Erhan等[6]将感兴趣的区域信息包装成边界框,然后给出每个边界框的信息进行回归训练,利用深度卷积网络进行回归预测。为了解决输入图像尺寸限制问题,He等[7]提出一种空间金字塔池化(Spatial Pyramid Pooling,SPP)方法,可以把不同尺寸的输入图像映射到一个固定尺寸的特征向量上,提高了识别速率。以上方法均需要提取候选区域,这一步是与识别无关的耗时步骤,为了避免候选区域的提取耗时过多,一种端到端的识别方法陆续提出。Cirshick等[8]提出一种端到端的快速区域卷积神经网络,将整个图像归一化后直接送入CNN网络,卷积层不进行候选区的特征提取,而是在最后一个池化层加入候选区域坐标信息,进行特征提取的计算,加快了识别速率。Liu等[9]提出一种单点多盒探测器的方法,把不同卷积核用于不同的宽高比进行识别,然后将特征图用于网络高层的多个特征映射当中,采用多尺度特征映射用于识别。而面对农作物病虫害的识别任务中,当新增种类时便需要模型迁移训练,这就要求模型的训练速度要快。同时对于类别较多的问题,需要一种更有效的特征提取网络以及更有效的多分类器。
针对上述问题,本文提出一种基于深度学习的InceptionNet网络的Inception结构和ResNet网络的Residual结构组合的Inception-resnetl-v2网络以及softmax分类器的农作物病虫识别方法,改善了传统深度学习中的普通卷积结构训练慢、容易过拟合以及分类效果差的问题,实现更精准高效的农作物病虫害识别。最后通过实验对比传统深度学习算法,验证了本文方法的有效性。
1 病虫害图像识别原理随着精准农业的兴起,运用信息技术辅助农业生产为农作物病虫害的识别提供了新思路,图像处理技术就是其中之一[10]。一种高效的农作物病虫害识别算法不仅能有效提取病虫害的特征,还能均衡区分不同类别。传统的图像分类算法大多采用普通CNN或DCNN提取图像特征,然后使用支持向量机(Support Vector Machine,SVM)进行多个二分类。与人工特征方法相比,取得了一定的效果。但是存在过拟合导致特征提取效果差和模型训练成本高的问题,有较大的改进空间。本文以Inception结构和Residual结构组合的Inception-resnetl-v2网络和Softmax分类器构建了一种用于多分类的农作物病虫害识别算法,达到准确识别和分类的均衡化。所提出的农作物病虫害识别算法中,利用特征提取网络(Inception-resnet-v2)以及Softmax分类器是实现农作物病虫害识别算法的关键环节,识别流程如图1所示。
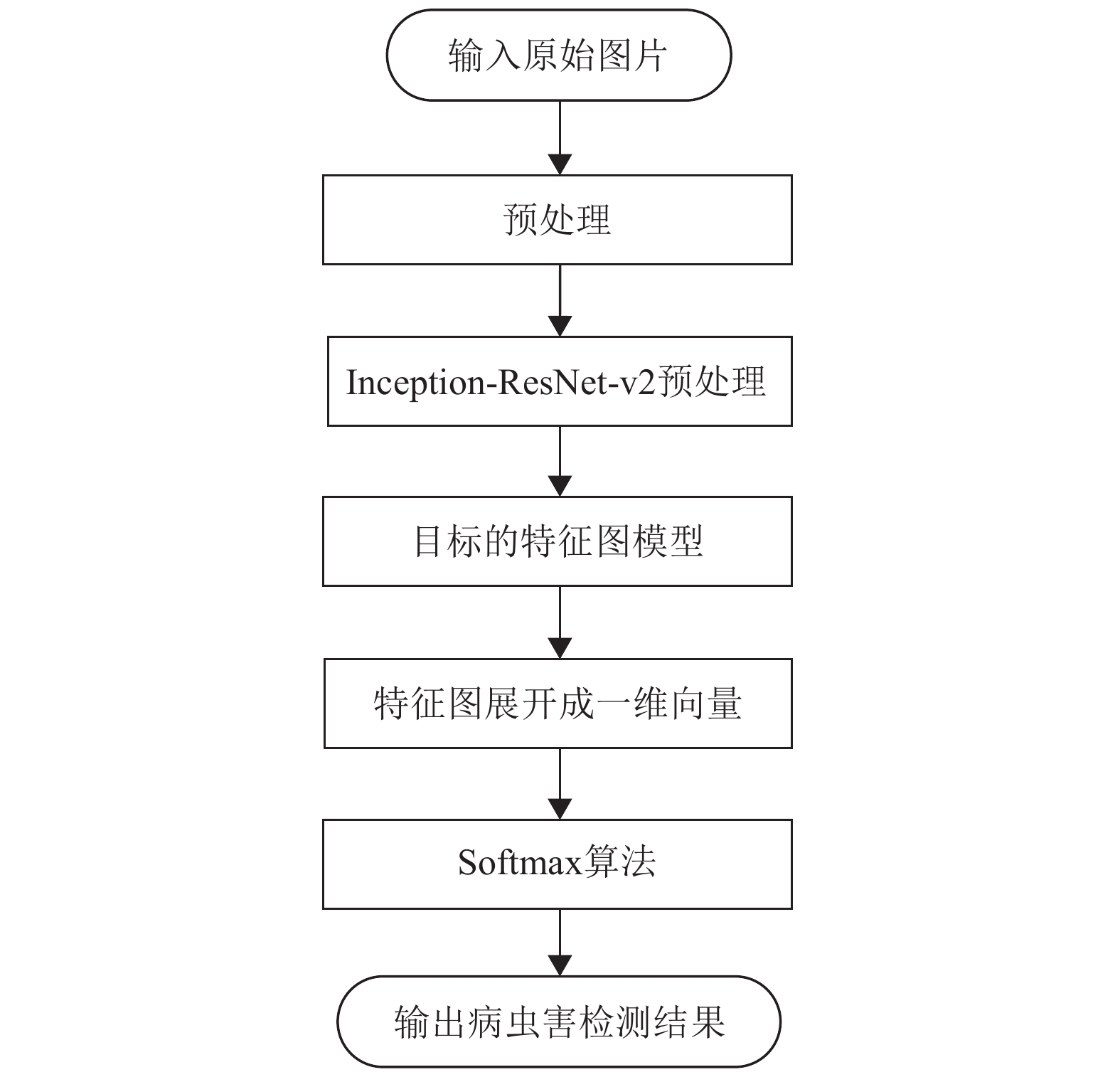
|
图 1 农作物病虫害识别流程 Figure 1 Crop pest and disease identification process |
提取农作物病虫害图像的特征以及学习相应的图像模式是农作物病虫害算法实现敏感识别和精准分类的关键。为了可以提取更高级的病虫害深度特征表达,产生更多不同的特征组合学习得到病虫害特征映射,本文构建了病虫害深度特征提取网络,网络结构采用了Szegedy等[11]提出的Inception结构,在性能不降低的前提下降低参数数量,加快训练速度。同时采用了残差网络的连接思想,引入了He等[12]提出的Residual结构,在保证网络结构很深的情况下仍能收敛。农作物病虫害特征提取网络如图2所示。
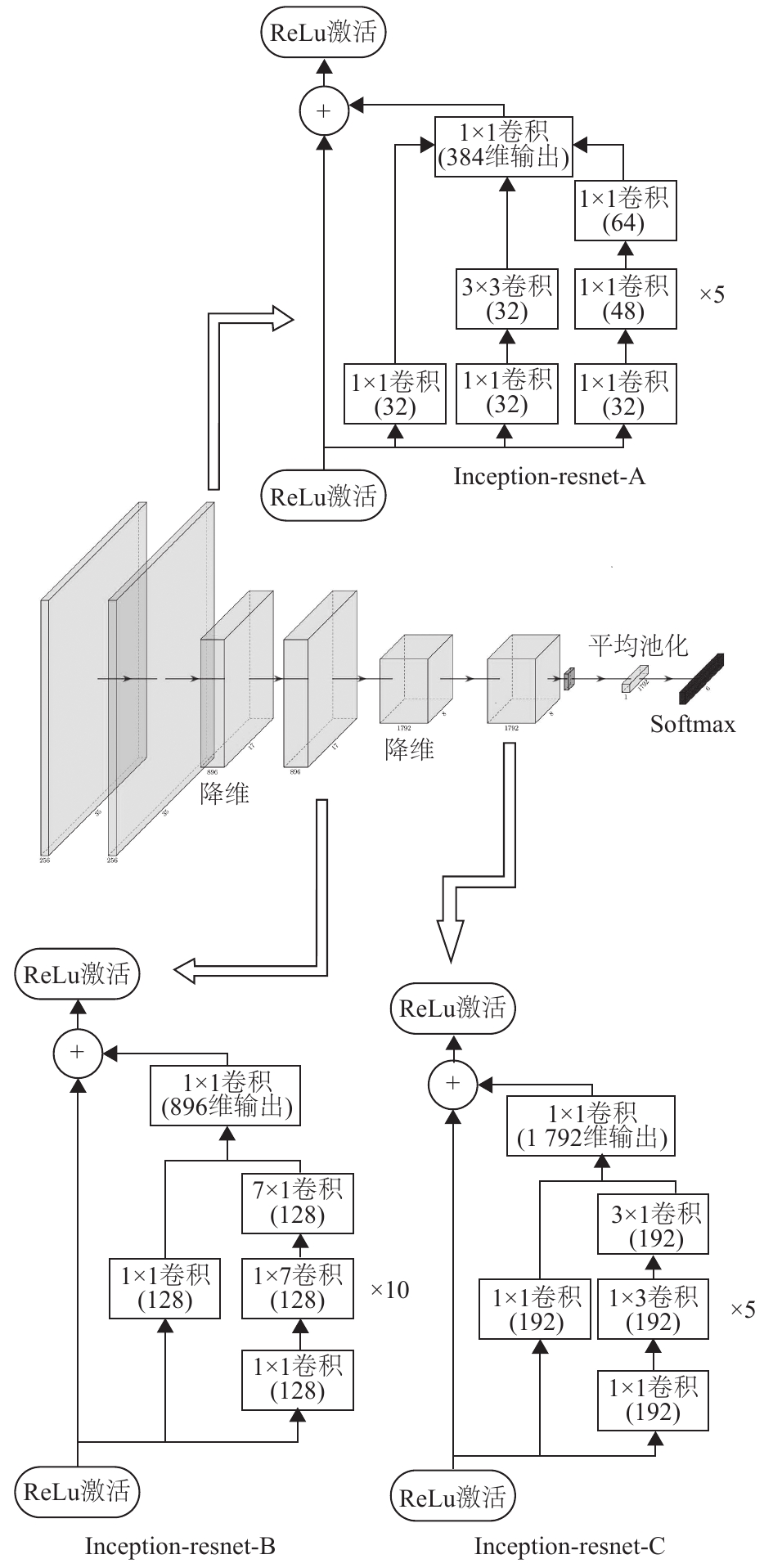
|
图 2 病虫害特征提取网络 Figure 2 Pest and disease feature extraction network |
网络包含3种Inception-resnet块,3种网络块区别在于Inception结构卷积核的大小和数量不同。对于Inception-resnet-A块,首先输入上一个深度网络层输出的病虫害特征映射,Inception结构有3路卷积通道,分别提取出尺寸相同通道数不同的特征映射进行堆叠,然后对堆叠后的特征映射进行卷积得到Inception结构的输出特征,通过残差连接与输入特征映射相加,最后通过激活函数得到这个块提取的病虫害更高级的特征映射。以水稻稻瘟病为例,通过Inception-resnet-A块把纹枯病的低级特征映射高度抽象得到高级特征映射如图3所示。网络的特征提取核心结构由Inception-resnet-A/B/C块组成,该结构由一系列卷积和残差连接实现,公式为

|
图 3 水稻稻瘟病低级特征到高级特征 Figure 3 Low-grade to advanced features of rice blast |
| ${{{x}}^b} = f\left({h^k}\left({{{x}}^{b - 1}}\right) + {{{x}}^{b - 1}}\right),k = A,B,C$ | (1) |
| ${{A}}_n^l = f\left(\sum\limits_{i = 1}^m {{{A}}_i^{l - 1}*{{W}}_n^l + {{b}}_n^l} \right)$ | (2) |
其中,
通过Inception-resnet结构提取的病虫害高级特征,存在邻域大小受限造成的估计值方差增大,为了缩小该误差,使用平均值池化卷积层输出的病虫害特征映射。池化特征映射表达式为
| ${{{f}}_{u,v}} = {\rm{Avg}}\left({{S}}_{n\{ i + (u - 1) \times d,j + (v - 1) \times d\} }^r\right)$ | (3) |
其中,
对原始图像进行亮度归一化[13]预处理后,输入到Inception-resnet-v2网络对农作物病虫害图像采用端到端的方式直接提取从低级到高级的特征,得到特征映射后再通过平均池化层进行池化,池化输出后将特征映射进行一维展开得到相应的特征向量,最后将特征向量通过分类器进行农作物病虫害分类。
由于农作物病虫害种类多,而且类别之间相似度较高,容易造成误分类。本文选用多类别分类的Softmax分类器,它是由逻辑回归分类器的基础发展而来,能够一次完成对多个类别的分类任务。
经过特征提取网络提取后输出的第
| $P({y^{(i)}} = j|{{{x}}^{(i)}};{{\theta}} ) = \frac{{{{\rm{e}}^{\theta _j^{}{x^{(i)}}}}}}{{\sum\limits_{j = 1}^c {{{\rm{e}}^{\theta _j^{}{x^{(i)}}}}} }},j = 1,2,\cdots,c$ | (4) |
其中,
Softmax分类器的损失函数是对函数中类别标记的
| $L({{\theta}} ) = - \frac{1}{m}\left\{ \sum\limits_{i = 1}^m {\sum\limits_{j = 1}^c {1({y^{(i)}} = j)\ln (P({y^{(i)}} = j|{{{x}}^{(i)}};{{\theta}} ))} } \right\} $ | (5) |
其中,
本文实验采用的数据集来源于农业病虫害研究图像数据库(Image Database for Agricultural Diseases and Pests Research,IDADP)[14],IDADP包含大量的水稻、小麦、玉米等作物病害图像资源,每种病害有几百乃至上千幅图片,其原始图片分辨率达到2 000万像素,建立了一个可为机器学习建模提供训练和测试样本的农作物病虫害识别研究图像数据集。本文选取水稻和小麦作为研究对象,每个研究对象各选取3种常见的病虫害类别,共6个类别,分别水稻白叶枯病、水稻胡麻斑病、水稻稻瘟病、小麦白粉病、小麦赤霉病、小麦叶锈病,其中水稻每个类别600张图片,小麦每个类别400张图片,共计3 000张图片用于训练加验证,另外有单独300张用于测试。每一张样本图片分辨率为3 000×2 000×3。部分样本示例如图4所示。

|
图 4 水稻与小麦的图像样例 Figure 4 Samples of images of rice and wheat |
本实验的实验平台为Ubuntu16.04操作系统,32 GB RAM,CPU处理器主频为3.7 GHz,GPU为GTX1060,深度学习框架为Pytorch1.1.0。
4.2 参数选择训练过程中,为了更明显地进行对比,本文选用CNN和DCNN两种传统网络与本文方法进行对比,均使用Adam优化器进行参数更新,损失函数为交叉熵(Cross Entropy, CE)函数。其中Adam学习率初始化为
为了验证本文方法的有效性,本文采用传统神经网络和深度卷积神经网络方法对农作物病虫害识别做实验对比,评价指标包括准确率、精度、召回率和
| 表 1 平均准确率对比 Table 1 Mean accuracy comparison |
| 表 2 特定类别的识别准确率对比 Table 2 Comparison of recognition accuracy for specific categories % |
| 表 3 精度、召回率和F1分数 Table 3 Precision, Recall and F1 score |
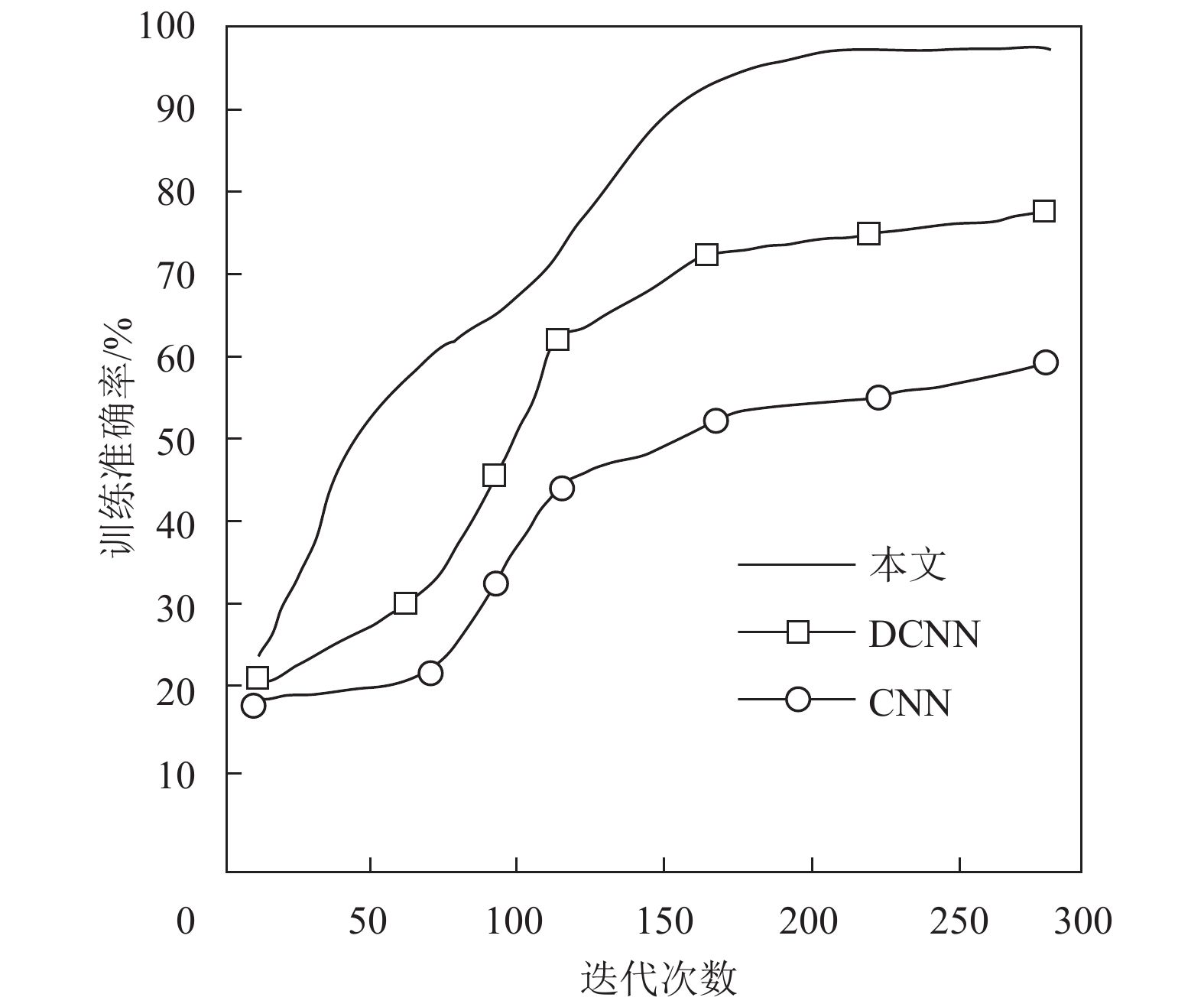
|
图 5 模型迭代曲线 Figure 5 Model iteration curve |
通过表1可知,在包含水稻和小麦的共6种类型农作物的病虫害识别任务中,传统的CNN算法表现最差,测试平均准确率仅为79.56%。DCNN通过更深的网络能达到94.56%的平均准确率。而本文方法引入了残差连接,在深度一定的情况下有效的缓和过拟合,准确率达到了96.67%,比没有残差结构的DCNN多2.11%的准确率。
由表2可知,不同模型对不同类别的识别效果有明显差异,CNN对稻瘟病的识别准确率与赤霉病识别准确率相差达到23.66%,对稻瘟病的识别表现最差,存在明显的识别不均衡现象。本文方法的各个类别的识别准确率均匀,而且每个类别的识别准确率比DCNN高。
由表3可知,本文方法的模型精度(precision,预测为正的样本当中真实为正的比例)达到90.77%,在预测的正样本中准确率最高。同时,本文方法的召回率(recall,真实为正的样本中被预测正确的比例)为89.72%,在样本中找到各个病虫害类别的能力最强。本文方法的
本文提出的基于Inception和Residual结构组合的Inception-resnet-v2网络的农作物病虫害识别分类方法在面向多类别的任务中,不仅有效提升了识别准确率,还平衡了不同类别带来的差异特征。与传统算法的对比表明,本文模型的训练能够更快收敛,能减少训练成本,同时又有很好的准确性和鲁棒性。本研究是基于组合结构网络在农作物病虫害上的识别研究,并未对其他更多的农作物种类和病虫害种类进行更大规模的识别研究。后续工作将采用规模更大的数据集和其他组合网络进行更深入的研究。
| [1] |
赵国华, 王婷. 基于ARM的作物病虫害自动识别系统设计[J].
南方农机, 2019, 50(5): 62.
DOI: 10.3969/j.issn.1672-3872.2019.05.046. |
| [2] |
柴帅, 李壮举. 基于迁移学习的番茄病虫害检测[J].
计算机工程与设计, 2019, 40(6): 1701-1705.
CHAI S, LI Z J. Detection of tomato pests and diseases based on transfer learning[J]. Computer Engineering and Design, 2019, 40(6): 1701-1705. |
| [3] |
孙军. 基于图像目标检测的茶树病虫害预警研究[J].
福建茶叶, 2018, 40(12): 11.
SUN J. Study on early warning of insect pests in tea yard based on image target detection[J]. Tea in Fujian, 2018, 40(12): 11. DOI: 10.3969/j.issn.1005-2291.2018.12.011. |
| [4] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E. Image net classification with deep convolutional neural networks[C]//NIPS 2012: Proceedings of the 25th International Conference on Neural Information Processing Systems. North Miami Beach, FL: Curran Associates Inc, 2012: 1097-1105.
|
| [5] |
SZEGEDY C, TOSHEV A, ERHAN D. Deep neural networks for object detection[C]// Proceedings of the 2013 International Conference on Neural Information Processing Systems. Cambridge, MA: MIT Press, 2013: 2553-2561.
|
| [6] |
ERHAN D, SZEGEDY C, TOSHEV A, et al. Scalable object detection using deep neural networks[C]//CVPR 2014: Proceedings of the 2014 IEEE Conference on Computer Vision and Pattern Recognition. Washington, DC: IEEE Computer Society, 2014: 2155-2162.
|
| [7] |
HE K M, ZHANG X Y, REN S Q, et al. Spatial pyramid pooling in deep convolutional networks for visual recognition[J].
IEEE Transactions on Pattern Analysis and Machine Intelligence, 2015, 37(9): 1904-1916.
DOI: 10.1109/TPAMI.2015.2389824. |
| [8] |
ROSS G. Fast R-CNN[C]// The IEEE International Conference on Computer Vision (ICCV). Washington, DC: IEEE Computer Society, 2015: 1440-1448.
|
| [9] |
LIU W, ANGUELOV D, ERHAN D, et al. SSD: single shot multibox detector[C]//ECCV 2016: Proceedings of the 2016 European Conference on Computer Vision, LNCS 9905. Cham: Springer, 2016: 21-37.
|
| [10] |
汪京京, 张武, 刘连忠, 等. 农作物病虫害图像识别技术的研究综述[J].
计算机工程与科学, 2014, 36(7): 1363-1370.
WANG J J, ZHANG W, LIU L Z, et al. Summary of crop diseases and pests image recognition technology[J]. Computer Engineering & Science, 2014, 36(7): 1363-1370. DOI: 10.3969/j.issn.1007-130X.2014.07.026. |
| [11] |
SZEGEDY C, LIU W, JIA Y Q, et al. Going deeper with convolutions[C]//The IEEE Conference on Computer Vision and Pattern Recognition. Washington (CVPR), DC: IEEE Computer Society, 2015: 1-9.
|
| [12] |
HE K, ZHANG X, REN S, et al. Deep Residual Learning for Image Recognition[C]//The IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Washington, DC: IEEE Computer Society, 2016: 770-778.
|
| [13] |
蔡建新, 汪仁煌, 黄颖怡. 亮度归一化在图像处理中的应用[J].
广东工业大学学报, 2008, 25(4): 65-68.
CAI J X, WANG R H, HUANG Y Y. Application of brightness normalization in image processing[J]. Journal of Guangdong University of Technology, 2008, 25(4): 65-68. DOI: 10.3969/j.issn.1007-7162.2008.04.017. |
| [14] |
陈雷, 袁媛. 大田作物病害识别研究图像数据集[DB/OL]. Science Data Bank, 2019. (2019-03-20)[2019-07-12]. DOI: 10.11922/sciencedb.745.
|
 2020, Vol. 37
2020, Vol. 37