2 复旦大学, 现代人类学教育部重点实验室, 上海 200438;
3 陕西省考古研究院, 陕西 西安 710054;
4 西北大学文化遗产学院, 陕西 西安 710069)
在古代, 马既是国家重要的战略军备物资, 标志着一个国家或一个时代军事实力的强弱, 又是人们日常生活中主要的交通工具和生产资料, 标志着一个家族或个人的身份地位和财富水平[1]。马与印欧语[2]和冶铜术[3]的传播有关, 马的驯化对人类社会的政治和经济发展轨迹产生了深远的影响。学术界比较公认的观点是家马在约5500年前于新石器时代哈萨克北部大草原被驯化, 博泰文化(Botai Culture)遗址出土的材料为此提供了最有力的考古学证据[4]。家马在4000~3600年前传入了中国西北的甘青地区, 在大何庄遗址和秦魏家墓地等齐家文化晚期遗址中均发现其遗骸[5];之后的3300~2000年前, 家马的分布范围已扩散至中国北方的大部分地区[6], 并被赋予了社会功能、文化意涵, 特别是以马祭祀的规制[7]。
在过去的10年, 古DNA研究发生了革命性的变化, 并开创了古基因组学这一新领域[8]。相比分子克隆和聚合酶链式反应(Polymerase Chain Reaction, 简称PCR)以及一代测序技术, 高通量测序避免了对古代样本中获取的DNA进行直接PCR, 而是将提取物用于构建可以测序或者杂交捕获的DNA文库[9]。古基因组学具有如下优势: 1)检出率显著提高。传统方法需要有足够的空间让引物与DNA片段相结合, 古代样本中大量的DNA片段降解严重[10], 扩增子的长度(大于100 bp)已经超过了古DNA降解后的长度(小于70 bp), 时常检测不出;2)检测通量高, 传统方法一次检测的位点数有限(常染色体STR、Y-STR & Y-SNP、X-STRs、线粒体DNA等), 迭代的测试容易造成有限DNA模板的大量损耗, 高通量测序技术能够对不同遗传标记组合和全基因组水平进行并行测序, 所需的模板数量可以低至1 ng或更少[11];3)便于污染评估。可以利用古DNA的特性来判断污染情况, 例如DNA片段长度、序列碱基一致性, 以及末端损伤情况;随着时间流逝这种损伤会逐步积累[12], 现代人DNA往往不存在损伤, 因此, 损伤可视为判断古DNA序列的标准。当发现提取液中存在污染时, 可以只分析那些携带损伤的古DNA片段[13]。污染评估非常重要, 保证了实验结果的真实性。
在动物古基因组学研究方面, 洞熊[14]、地懒[15]、剑齿虎[16]、猛犸象[17]、马[18]、欧洲野牛[19]、猪[20]和狼/狗[21]等的全基因组序列均以获取, 它们为厘清物种的进化关系、功能性适应、驯化的遗传机理、家畜/伴生动物的起源和扩散提供了可能[8]。1995年, 第一届马的基因图谱国际研讨会(International Equine Gene Mapping Workshop)促进了对马的基因组进行有组织、跨地区的合作研究[22]。25年的时间以来, 马的基因组学产生了有许多重要的成就[22], 里程碑是2009年参考基因组EquCab2.0装配成功[23]。纯种母马“黎明Twilight”提供了第一个家马和奇蹄动物的基因组, 并据此建立了家马参考序列草图[23]。马的古基因组学研究始于2011年, Orlando等[24]从加拿大育空蓟溪永久冻土层的一个更新世马骨化石(大于5万年)中成功获取了约100 Mb的DNA片段。值得注意的是, 马科的研究创造了古DNA研究史中的3个第一:第一次、最古老和最长时间跨度。1984年, Higuchi等[25]首次在斑驴组织中成功提取和分析了古DNA, 早于埃及木乃伊中获取的人类古DNA[26];其次, 最古老的古DNA基因组来自于70万年前的更新世马化石[18], 比43万年前的胡瑟古人的基因组还要古老[27];近期, Fages等[28]基于278个古马的大规模基因组数据, 追踪了5000年时间跨度中家马的驯化过程。该研究是目前非人类生物古基因组学的一个重要里程碑[29]。马的古基因组研究极大地扩展了我们关于马的认知, 包括马的自然史[18, 30]、马的驯化及其基因组的动态变化[28, 31~36]。但是, 前期研究主要聚焦于马的生物学属性, 缺乏或者忽视对历史文献和考古资料的综合考察, 导致马的古基因组学研究与考古学关心的问题存在“脱钩”现象。相比马的生物学问题, 考古学更关心是人类对于马的利用及其反映的社会现象, 例如祭祀用马中古代先民对于马匹的管理方式, 其中是否存在统一场所长期豢养马匹, 祭祀用马是否存在统一的挑选标准等, 进而探究中国古代祭祀文化和礼制文化。本文从马的遗传谱系和基因型-表型关联研究这两方面的最新进展出发, 尝试探讨马的古基因组学研究与考古学实践相结合的问题。
1 家马的遗传谱系研究马的基因组研究中最重要的一个方面是厘清马的遗传谱系, 进而回答家马的起源、迁徙和驯化等问题。马的基因组由细胞核中的核DNA和细胞质中的线粒体DNA共同组成。核基因组含有62条常染色体, 每一对常染色体都有一条来自父方, 一条来自母方, 两条染色体在传代过程中会发生部分交换, 从而造成混血效应, 即遗传学上的重组(图 1);还有一对性染色体, 由一条X染色体和一条Y染色体组成。在母马体内, X染色体也是成对出现, 所以不可避免的存在混血。Y染色体只在父子间传递, 呈现严格的父系遗传, 构成了父系遗传系统;细胞质中的线粒体DNA则由母马传递给子女, 构成母系遗传系统, 绝大部分情况下, 线粒体DNA也遵循严格的母系遗传(图 1)。单亲遗传系统(线粒体和Y染色体)在追踪研究群体的起源、迁徙和演化方面扮演着重要的角色[29]。尽管单亲遗传系统有不重组的优点, 但它们所包含的多态位点有限, 遗传信息的含量不能满足更精细的遗传结构研究, 而且正因为没有重组, 单亲遗传方式只能提供单个遗传座位的信息, 而从这种单个遗传座位的信息构建的遗传关系存在一些缺陷[37], 特别是祖先信息的丢失, 比如鉴定祖母和孙子、外祖父和孙女, 就很困难(图 1)。近年来, 随着全基因组数据分析水平的提高, 利用常染体遗传标记显示出极大的优势, 单核苷酸多态性(single nucleotide polymorphism, 简称SNP)遗传标记具有极高的密度, 蕴含极其丰富的遗传信息。一个个体会记录所有祖先的遗传历史[38]。国内学者对古马遗骸多用线粒体DNA进行母系亲缘鉴定与谱系树的构建[39~41], 而对全基因组或Y染色体的分析则多见于现代马的研究中[42~43]。
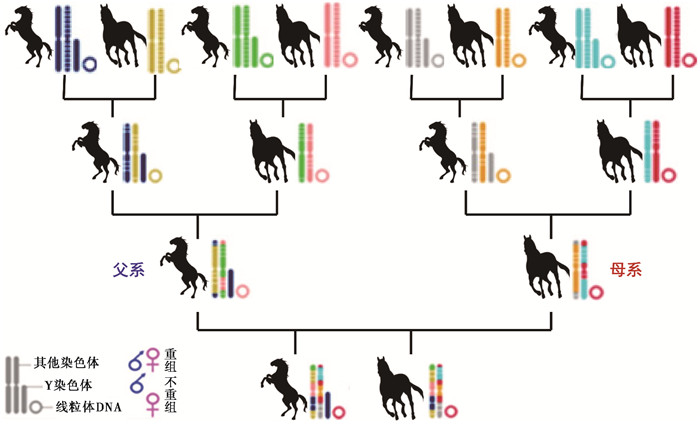
|
图 1 马的遗传物质的传递规律 Fig. 1 The transmission of genetic material in horses |
由于每个细胞内有数百到数千个拷贝, 扩增线粒体DNA模板往往比核基因组容易, 所以直到2000年代中期, 古DNA研究的重心还停留在线粒体调控区(D-loop:物理位置15469~16660)中的高变区(HVS-I:物理位置15469~15834;HVS-II:物理位置16351~16660)。2001年, Vila等[44]分析了10个品系191匹家马中线粒体调控区的355 bp序列, 首次发现家马存在6个不同的母系遗传谱系(A~F), 展示了较高的母系多样性;2002年, Jansen等[45]对分布在全球的25个马的品种的线粒体调控区进行了测序, 并结合Genbank上共计652个序列构建了家马的中介网络图, 发现了7个主要的谱系(A~G), 进一步可细化为17个支系;2006年, McGahern等[46]对欧亚中部、东北亚的7个家马群体和GenBank上共计962个线粒体调控区序列进行了分析, 发现谱系F在东方马中出现频率较多, 而欧洲马却聚在D谱系;2012年Achilli等[47]对欧亚大陆和美洲的83个线粒体全序进行了分析, 发现新石器时代以后, 出现了18个星状扩张的单倍群(A~R), 展示了极高的多样性, 其共祖时间为约13~16万年前;另外, 之前发现的一些支系和矮马种的对应关系不太可靠;Librado等[33]对4100~2300年前的14个早期家马进行了基因组测序, 发现这种极高的多样性在铁器时代的斯基泰马中就已经呈现出来;Gaunitz等[34]根据211个马的线粒体基因组重建的贝叶斯天际线图(Bayesian Skyline Plot, 简称BSP)表明, 马的群体扩张发生在大约4500年前。上述研究表明, 家马线粒体DNA多样性极高, 但是缺乏系统地理分布模式, 导致线粒体DNA单倍型、品种和地理之间关联性较低[48]。家马母系遗传谱系所呈现这一特点的原因有很多, 可能与人类的管理方式有关, 例如有性别差异的将野马补充到家马种群[48]。
1.2 Y染色体DNA研究揭示家马父系的极低多样性与线粒体DNA的多样性相比, 现代家马的Y染色体几乎变得同质, 只有少数单倍型在现代种群中被发现[36, 49]。早期研究表明, 现代欧洲马品种中仅存在6个Y染色体单倍型(HTs)[50], 微卫星的变异也非常有限[51~53], 暗示雄马的有效群体大小极低。基于高通量测序的方法, 除了细分为更多的Y染色体单倍型, 现代家马父系多样性极低的结论并没有改变。Wallner等[49]分析了21个品系52匹家马的Y染色体雄性特异区的1.46 Mb序列, 鉴定了36种单倍型, 发现各种现代马的Y染色体谱系的分化远远晚于该物种的驯化。除了一些北欧的单倍型, 所有的现代马都聚集在一个大约仅有700年历史的单倍群中。该单倍群来源于欧洲进口的东方种马;东方种马主要分有两个分支:阿拉伯血统和土库曼马血统[49]。Felkel等[54]在Wallner等[49]的数据基础上, 增加了52个样本, 包括5个欧洲品种, 2个美国品种和7个亚洲品种, 鉴定出了42个单倍型, 其中6个单倍型仅仅出现在亚洲品系中。亚洲谱系在距今约5500年前就分离出来, 亚洲家马显示出比欧洲品种更高的多样性, 因此, Felkel等[54]认为较早分离的亚洲单倍型(HG-O)是驯化早期具有高多样性的家马种群的残余。近期, Felkel等[55]重新组装了Y染色体雄性特异区的参考序列草图, 并扫描了精选的130个样本的5.8 Mb中Y染色体特异区的突变。在欧洲中部和北美的家马中鉴别出了一个年轻的冠状单倍群(crown haplogroup), 在这个单倍群中, Felkel等[55]根据211个突变定义了58个单倍型。除了前期研究发现的两个特征单倍群(阿拉伯马/冷血型马和土库曼/纯种马), Felkel等[55]还发现了以伊比利亚品系和北非倒钩马为主体的第三个单倍群。现代公马中Y染色体多样性极低, 但是铁器时代斯基泰马的Y序列表明, 在驯化阶段早期, 存在着大量的雄性家马奠基者[33]。Wutke等[36]对从驯化阶段早期(红铜和青铜时代)到中世纪的96个古代欧亚公马进行了Y染色体SNP分型, 观察Y染色体多样性随着时间是如何变化的。其结果表明现存家马中的多个种马谱系的业已缺失, 其原因并非奠基者效应或者随机人口效应, 而是始于铁器时代欧亚草原游牧民族的人工选择, 其后是罗马时期的大规模育种[36]。Fages等[28]基于105匹古代公马覆盖了1500个Y染色体位点的研究进一步证实了这一结论, 研究表明在过去大约2000年里, Y染色体的核苷酸多样性稳步下降, 但直到公元850~1350年, 才下降到现在的水平。目前所谓的“现代马种”是在过去的几百年内集中、有组织饲养的结果[28~29, 36]。
1.3 全基因组视野下家马的起源与驯化马的古基因组研究主要回答了3个关键问题: 1)除了现存的家马(Equus caballus)和普氏野马(E.przewalskii), 在驯化阶段早期还存在一些其他的马的谱系。其中一个谱系最初是在大约43000~5000年前的全北极区的遗骸中被鉴定出来[31], 其活动范围可能一直延伸到西伯利亚南部[30]。这一谱系在形态上与已经灭绝的勒那马(Equus lenensis)相似[28, 56]。此外, 公元前3000年到公元前2000年初期, 伊比利亚马(Iberian horse)与家马、普氏野马、勒那马在聚类上较为疏远, 并有着极高的线粒体和Y染色体多样性, 暗示在马的早期驯化阶段, 伊比利亚存在着另一个现在已经灭绝了的谱系[28]。2)现存马种(家马与普氏野马)之间的基因交流。基于现代和古代的基因组序列, 大体的共识是这两个马种的分化时间大约在45000年前[31, 57~58], 但是存在广泛的双向基因流动[48, 58]。家马不是普氏野马的直系后代[48]。2018年, Gaunitz等[34]和Damgaard等[59]基于欧亚大陆上40多匹古代马的古基因组研究非常重要, 其结果表明普氏野马并不是真正的野生马, 而是大约5500年前博泰文化驯养的马的后代;此外, 4000年前到现在的所有已研究的家马中只有2.7 %的博泰血统。这表明, 家马起源于一个不同于博泰文化驯养马种的谱系, 随后这个野生谱系灭绝了[28]。3)家马驯化的时间和地理框架。当代考古学和DNA证据表明, 马的驯化与多个遗址有关, 其中最早的(约5000~5500年前)位于欧亚大草原西部:哈萨克斯坦北部的博泰文化和东欧大草[28, 34, 48, 60~61], 其次的候选者位于伊比利亚半岛、安纳托利亚半岛东部、伊朗西部、黎凡特和东欧(匈牙利)[34]。然而, 即使驯化的主要候选地点被找到, 不等同于找到了现代家马的起源地。家马的起源问题仍然悬而未决。考虑到古代博泰马和伊比利亚马对于现代家马没有做出太大贡献, 对于公元前4千纪其他早期驯化候选地开展家马起源的研究也是非常有必要的[28]。
2 家马的基因型-表型关联研究人类根据马的性能特征(速度、耐力、力量、步态)、外观(大小、颜色、构造)和性情, 选择性地培育出400~500个不同的品种[62~63]。选择性繁殖产生的种群结构特点是种间高度多样化、种内较低多样化[63~64], 进而形成了一个马的形态和行为特征的研究队列。
马的基因型-表型关联研究是对其遗传因素和它们表现出的不同表型(生理、病理特征等)进行关联, 从而找到产生不同表型的遗传机理[65]。利用这种关联性, 我们可以通过基因型反推/预测古马的表型特征。完成这一目的的重要工具是马的参考基因组。家马参考基因组EquCab2.0为高质量、2.5 Gb的组装草图, 纳入了31条常染色体, X染色体和线粒体, 不包括Y染色体[23]。参考基因组的另一个任务是完成数百万个SNPs位点的鉴定。这些SNPs位点是由“黎明”的二倍体基因组和另外7匹不同品种的马的部分测序结果比较产生的[23]。基于EquCab2.0, 2011年上市了第一代和第二代DNA基因分型芯片, 分别包含54602个和74500个SNP标记。通过这些基因芯片识别了大量的表型和疾病性状, 例如毛色稀释症[66]、交替步态[65]、虹膜颜色差异[67]和眼部鳞状细胞癌等[68]。2017年, Affymetrix公司研制出了第三代SNP芯片, 含有670805个SNP标记(MNEc670k芯片)[69]。这款芯片是依据代表 24个不同品种的156匹马的全基因组序列设计而成。平均SNP间距估计为3756 bp, SNP选择旨在全基因组水平标记大约200万个SNP。到目前为止, 已发表的两项研究已经成功地应用MNEc670k芯片定位了性状, 然后通过全基因组测序进行了细化:其一是在阿克哈特克马中首次发现ST14中与毛色稀释症相关的无义突变[70];其次是发现KRT25和SP6基因中的遗传变异, 导致了马的卷毛[71]。
参考序列EquCab2.0存在一些缺陷, 该组装序列包含许多缺口, 主要位于结构复杂的基因组区域, 包括片段重复和CNV位点[72~73]。因此, 利用最新的基因组测序技术重新测序和组装“黎明”的基因组, 产生了参考序列EquCab3.0[74]。新组装的参考序列建立在EquCab2.0[23]、物理图谱[75]和BAC(Bacterial artificial chromosome)末端测序[76]的坚实基础上。比较已发表的13个古代家马的EquCab2.0和EquCab3.0映射统计[31~34], 有更多的读长被映射到EquCab3.0上, 这表明对于高度碎片化和受损的古DNA样本, EquCab 3.0的映射效果有了大幅度改进。目前, 共计1936个家马全基因组序列可在NCBI序列读长档案馆(NCBI Sequence Read Archive)中获得(https://www.ncbi.nlm.nih.gov/sra)。
古代马的基因型-表型关联应用方面也方兴未艾。Schubert等[31]分析了古马的全基因组数据, 找出了125个可能受驯化影响的基因位点, 这些位点涉及心脏和循环系统、骨骼、四肢/面部形态形成、大脑发育和行为以及毛色相关的基因。Liu等[77]也总结了家马品系的重要性状所对应的受到选择的基因组区域。比如MC1R、TYRP 1、ASIP、KIT等基因与马的毛色相关, MSTN、RALGAPA 2、INSM 1、PLDN、RIN 2等与运动能力相关, LCORL、ZFAT、HMGA 2、TBX 3等与体高相关, BARX2、PHIP等与极端环境适应相关, AMEL和SRY与性别相关。Wutke等[78]分析了107个古马的毛色相关的基因, 发现铜器-铁器时代的斑点色马显著增加, 但到了中世纪时期, 纯色(栗色)马变为主流;赵欣等[79]检测了MC1R、ASIP、MATP、KIT、SILV、EDNRB共6个基因的8个SNP位点, 发现陕西淳化西周遗址出土4匹马均为枣色, 而新疆石人子沟遗址[80]和木垒平顶山墓群[81]出土的马匹的毛色较为丰富。
3 考古学应用与意义中国地处亚洲大陆, 有着广阔的农牧地区, 马产资源十分丰富, 地方马品种23个, 培育马品种17个, 占世界马品种比重的13 %左右[82]。根据马种的历史来源、生态环境和尺寸类型等综合因素, 可以分为五大马种类型:蒙古马、西南马、河曲马、哈萨克马和藏马[83]。但是中国古代家马的源流问题仍是一个未解之谜。目前, 中国家马的起源主要有“舶来说”[84]与“独立起源说”[85]。前者主要基于考古学证据, 由于家马在商朝的突然大规模出现, 中国家马从境外传入是比较合理的解释;后者主要依据旧石器遗址出土了丰富的野生马骨化石, 古典文献记载追溯中国养马、驯马和用马的历史可至龙山文化时期。此外, 东方马与线粒体F单倍型组显著相关, 暗示可能存在一个独立驯化的中心[40]。
“国之大事, 在祀与戎”, 祭祀是中国农耕文明的重要组成部分。马在商周时期的遗址中多在殉葬坑或祭祀坑等宗教祭祀的考古背景中出现, 少见于灰坑等生活居址中, 说明商周时期的马非常珍贵, 代表主人的身份地位[41]。周礼记载中养马之官有“校人”、“廋人”及“圉人”等, 所养之马除供国家日常使役外, 还用于祭祀, 可见马在祭祀体系中占有重要的地位。传统考古学和动物考古学注重“眼见为实”。考古研究通过田野发掘探索马骨的出土地点, 利用动物考古学的方法判断家马最早出现的时间和地点、推测随葬马匹的年龄和性别、估算家马的身高等, 为探讨中国家马的起源、用途、祭祀用马的来源和挑选标准等方面的问题做出了重要的贡献。但是对于一些“看不见”的表型, 如马的性能特征、外观和性情等, 仍然知之甚少。结合前期基因组学成果, 我们认为古基因组学研究可以从7个方面与考古学实践相结合。遗传谱系可以追踪家马的源流和祭祀用马的来源;性别、毛色、运动能力、步态、肩高、高原适应这些表型可以推测所研究马匹的用途, 以及在祭祀遗址中祭祀用马是否存在统一的挑选标准, 进而达到“补史证经”的研究目的。
这里需要注意两个问题:一是如果展开全基因组水平研究, 所有的遗传数据都能获取, 但是成本比较昂贵, 特别是样本数目较多的墓葬, 经费很难支持;二是如果有些样本保存不佳, 马的内源性比例较低, 直接建库测序往往很难达到预期的数据质量。这时候就需要研究目的明确, 设计相应的基于探针/多重PCR的检测组合(panel)进行测试。
3.1 遗传谱系这一部分主要涉及两个尺度的问题, 一个较为宏观——研究马匹的品系来源, 一个较为微观——研究葬马坑内/间遗骸的亲缘关系。中国境内古马的研究主要集中线粒体DNA高变区与母系来源[39, 41, 79~81, 86]。如图 1所示, 对于单个个体而言, 只是追踪了母系遗传线索, 其他祖先信息均已丢失。下一阶段的工作重点是Y染色体DNA追踪父系遗传线索, 以及利用常染色体SNPs位点判断复杂亲缘关系。
从线粒体DNA和母系遗传的角度, 我们先看看中国境内家马谱系的分布情况。现代家马和古代家马线粒体世系在欧亚大陆中的分布如图 2所示, 现代家马中[86], 谱系A高频(31.4 % ~57.9 %)分布于欧亚大陆的不同群体, 北亚为最;谱系B不见于北亚马, 以中频(8.3 % ~12.7 %)分布于东亚、中亚、中东和欧洲, 俄罗斯马和韩国济州马中频率较高(27.8 %和17.6 %);谱系C也不见于北亚马, 以中频(6.7 % ~13.8 %)分布于东亚、中亚、中东和欧洲, 关山马和斯堪的纳维亚马中频率较高(36 %和30 %);谱系D以中到高频(15.7 % ~34.6 %), 从东亚到欧洲呈递增的分布频率, 其中非洲和伊比利亚半岛的马群中频率最高, 大约为40 %;谱系E不见于北亚马和中东马, 低频(3.7 % ~4.7 %)存在于东亚、中亚和欧洲, 在藏马中频率较高(18.7 %);谱系F以低到高频(5.2 % ~23.9 %), 从欧洲到东亚呈递增的分布频率, 其中以东亚的蒙古马(36.4 %)和济州马(32.4 %)中频率最高。古代家马中[39, 41, 79~81, 86], 除了于家庄遗址, 谱系A高频出现(大于25 %), 如毛园民宅二号车马坑(1/1)、大山前遗址(4/5)、秦公一号大墓车马坑(4/7)、小双古城遗址(2/4);谱系B高频出现于于家庄遗址(1/2), 中频出现于枣树沟脑遗址(1/4)、石人子沟遗址(1/5)和秦公一号大墓车马坑(1/7), 不见于其他已检测遗址;谱系C高频出现于板城墓地(2/7)和枣树沟脑遗址(1/4), 不见于其他已检测遗址;谱系D中频出现于秦公一号大墓车马坑(2/7)、板城墓地(2/7)和平顶山墓群(1/8), 不见于其他已检测遗址;谱系E高频出现于石人子沟遗址(3/5)、中频出现于和林格尔新店子墓地(3/12)、井沟子遗址西区墓葬(1/4)和平顶山墓群(2/8), 不见于其他已检测遗址;除了毛园民宅二号车马坑、石人子沟遗址和秦公一号大墓车马坑, 谱系F中高频出现(12.5 % ~50.0 %)。因此, 母系谱系在古代和现代家马中均有较高的多样性, 古代家马中东亚相关的谱系F的频率确实很高, 此外, 北亚相关的谱系A也经常被检出。
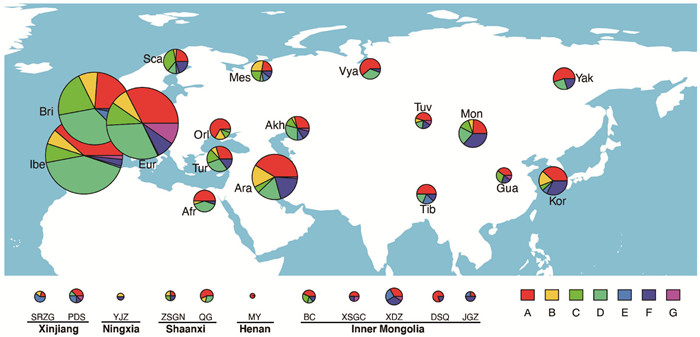
|
图 2 现代和古代家马线粒体世系在欧亚大陆的分布, 用不同的颜色代表不同的谱系(右下角) 图中现代马的英文缩写为, 蒙古马: Mon;关山马: Gua;藏马: Tib;济州马: Kor;图瓦马: Tuv;雅库特马: Yak;维亚茨卡亚马: Vya;梅森斯卡亚马: Mes;奥尔洛夫马: Orl;阿拉伯马: Ara;阿克哈特克马: Akh;突厥马: Tur;非洲马: Afr;伊比利亚马: Ibe;英伦马: Bri;斯堪的纳维亚马: Sca;欧洲大陆马: Eur。古代马的缩写为, 板城: BC;小双古城: XSGC;新店子: XDZ;毛园: MY;于家庄: YJZ;大山前: DSQ;井沟子: JGZ;枣树沟脑: ZSGN;平顶山: PDS;石人子沟: SRZG;秦公大墓: QG Fig. 2 Distributions of modern and ancient horse mtDNA lineages in Eurasia, with different colors representing different lineages(lower-right corner). The abbreviation for the breed/population of modern horses:Mongolian(Mon), Guan Mountain(Gua), Tibetan(Tib), Korea Cheju(Kor), Tuva(Tuv), Yakut(Yak), Vyatskaya(Vya), Mesenskaya(Mes), Orlov(Orl), Arabia(Ara), Akha-teke(Akh), Turkey(Tur), Africa(Afr), Iberia(Ibe), British Isles(Bri), Scandinavia(Sca), and Maniland Europe(Eur). The abbreviation for ancient horses in studied sites:Bancheng(BC), Xiaoshuanggucheng(XSGC), Xindianzi(XDZ), Maoyuan(MY), Yujiazhuang(YJZ), Dashanqian(DSQ), Jinggouzi(JGZ), Zaoshugounao(ZSGN), Pingdingshan(PDS), Shirenzigou(SRZG), and Qingong mausoleum(QG) |
祭祀用马的性别对于理解马在仪式中的含义、马与人的关系以及古代社会中马的地位和用途至关重要[87]。通常马骨遗存的性别鉴定基于形态特征的差异, 如骨盆的形状和犬齿的有无[88]。对幼年个体以及保存情况差的成年个体分析时容易产生偏差。
在已经发表的文献中[89~93], 祭祀用马一般会有意识地选择使用公马(有可能被阉割, 但是DNA无法证实), 这可能和公马勇猛、无畏的特性有关。例如, 冰岛维京时代墓葬中的葬马, 雄雌性别比为18 ︰ 1[89], 蒙古地区青铜时代晚期鹿石-石堆墓复合文化中的高规格墓葬中使用了相当高比例的成年公马[90], 贝雷尔墓葬[91]、丹麦Illerup Aadal遗址[92]、新疆喀拉苏墓地[93]的随葬马匹均为雄性。整体表现为高规格墓葬偏好使用公马, 非墓葬(如冰岛维京时代的性别比为0 :3)和一般墓葬(如蒙古地区青铜时代晚期鹿石-石堆墓复合文化、中世纪早期瑞典斯韦兰地区Vendel大型墓地遗址和Eketorp祭祀遗址[94])会出现高比例的雌马。对于中国不同时期、不同地域和不同用途的出土马骨的性别调查是有意义的, 其蕴含的考古学意义值得深思。
3.3 毛色毛色是人类正向选择产生的表型之一, 在遗传与进化过程中扮演着重要角色[95]。马的毛色分为基础毛色、淡化毛色以及白斑和褪色(包括白色)三大类型。马的毛色是由若干较深且单一的颜色被影响色素合成的修饰基因和突变位点作用而形成。基础毛色(base color)在马群中比较普遍, 可分为骝色(bay)、褐骝色(seal brown)、栗色(chestnut)和黑色(black)。有一些修饰基因不同程度地抑制皮肤或毛囊中真黑素、褐黑素合成。这些基因自由组合, 独立或共同发挥作用形成了马的淡化毛色(dilution), 包括奶酪色(cream)、香槟色(champagne)、银色(silver)和暗褐色(dun)。另一类修饰基因可在基础毛色及淡化毛色之上进行调控形成更为特殊的毛色表型, 如白斑(white spotting)、褪色(depigmentation)等, 包括珍珠色(pearl)、分背花色(overo pattern)、越背花色(tobiano pattern)、沙边毛色(sabino spotted)、豹点色(leopard complex)等[94]。
Wutke等[78]针对欧亚大陆上201个古代马的毛色进行了SNP分型检测, 发现在更新世/中石器时代(>4000 BC), 马的毛色以骝色和黑色为主, 欧洲马出现了骝色带豹点;新石器时代/铜器时代(4000~2700 BC), 在原有的基础上, 亚洲马出现了栗色, 毛色表型已经丰富至6种;到了青铜和铁器时代(2700~900 BC和900 BC~400 A.D.), 马的毛色表型已经达到9种, 但是到了中世纪时期以后(> 400 A.D.), 纯色(栗色)马变为主流。
中国境内马的毛色研究主要集中在以下4篇文献[79~81, 96]:青海省互助县丰台遗址(1000~800 BC)检出了4个样本, 1个为骝色带越背花, 3个是骝色;新疆石人子沟遗址(400~120 BC)检出了5个样本, 3个栗色, 1个骝色, 1个金黄色;陕西省淳化县枣树沟脑遗址(西周中晚期)检出了4个样本中均为骝色;新疆木垒县平顶山墓群(公元前一千纪至公元前六世纪)检出的8个样本3个为栗色, 2个栗色带越背花, 2个黑色, 1个金黄色。由此可见, 中国境内的马, 越靠近中原地区, 毛色越单一, 不同于西欧亚地区, 直到中世纪时期以后, 纯色马才占据主体。在欧洲, 这种毛色喜好的改变有多个原因[78]:1)特别是纯合子个体, 毛色减少的现象会被负选择所强化;2)斑点色作为区分野马与家马的区分标记, 随着野马的减少, 这个功能也就没有必要了;3)较为负面的宗教含义;4)与武器(如长弓)的发展有关, 淡色或有斑点色的马更容易成为敌人的目标。中国境内古代家马的毛色还没有系统的研究, 这种类似的毛色变化是否也存在, 如果存在反映了什么社会现象, 可能需要深入探讨。中国古代养马业发达, 有评价马匹外形外貌的相马术。仅在古籍中列名可考的养马、相马、疗马经典著作就有32种, 百余卷之多[97]。但是包括最著名的《伯乐相马经》均已失传, 仅存《司牧安骥集》所传《宝金篇》(明代改名宝金歌)[97]。篇中将毛色和凶吉挂钩, 如“白首黑身须可忌”、“耳白腰花实是凶”, 可见中国古代先民对花色马并不太喜好。
3.4 运动能力在过去的几百年里, 人类更偏爱具有较强的力量、耐力和比赛速度的马, 并精选了有着优质运动能力的品种。国际赛马协会定义了赛马的比赛距离的5个分类, 并可简化为三类:“短跑”、“中距离”和“长跑”[98]。Moon等[99]通过对240匹韩国纯血赛马的全基因组扫描发现, RALGAPA2基因(chr22:4632335)在提高赛马成绩方面起着至关重要的作用, 携带等位基因G的马有着更强的运动能力;Hill等[98]调查了148个注册赛马在不同赛距的表型, MSTN基因上的一个位点(chr18:66493737)与最佳赛距呈现强相关(P=1.91×10-6)。C/C基因型的马适合快速、短距离的比赛, C/T基因型的马在中距离比赛中表现良好, T/T基因型的马的耐力更强, 更适合长距离奔跑[98]。
《周礼·夏官·马质》:马量三物, 一曰戎马, 二曰田马, 三曰驽马。戎马也就是战马, 往往是身材高大、爆发力强的马种。田马是打猎所用的马, 运动能力次之。驽马是蹩脚马, 资质较差、不出众。古马运动能力相关基因的检测, 结合马匹用途的考古学推测, 有助于我们了解马在不同场景(如骑乘、拉车、祭祀等)所扮演的角色。
3.5 步态马的行走方式受到了多次人工选择, 特别是其交替步法(alternate gaits)[100]。交替步法是马的典型特征, 根据时间模式和落脚顺序, 可分为4类:溜蹄(pace)、有节奏漫步(regular rhythm ambling)、横向漫步(lateral ambling)、对角线漫步(diagonal ambling)。溜蹄是两拍步, 马以同步的横向运动移动身体同一侧的两条腿, 不同于小跑(对角线的前腿和后腿一起向前和向后移动)。漫步是一种四拍步, 在这种步法中, 特定的马种的脚步模式、脚的位置和时间往往是独一无二的。托尔特步(Tölt)是冰岛马的一种有规律的漫步。Andersson等[100]通过对30匹四步态(慢步、托尔特步、小跑和疾驰)和40匹五步态(慢步、托尔特步、小跑、疾驰和溜蹄)冰岛马的全基因组关联分析, 发现SNP位点(chr23:22967656)与马的步态差异有关;对其连锁区域的进一步重测序发现, DMRT 3基因上的一个突变(chr23:22999655)会导致DMRT3的转录在301号密码子处过早停止, 进而显著影响马在完成交替步态时的能力。随后, 对全世界的不同马种中对该突变进行了进一步的验证[101]。在芬恩马(Finn horses)中, 携带纯合突变型A/A的马有着更好的比赛成绩, 而携带C等位基因的马似乎更适合古典骑术比赛[102]。其他一些研究也发现该基因与马的慢跑[103]、盛装舞步[104]和跳跃[105]能力相关。
为了适应长途骑乘, 减少马背颠簸之苦, 古人对驿马进行了选择, 驯化可以走交替步法(溜蹄步)的马种, 让骑马人感觉到舒适[106], 东汉时期创作的“踏飞燕铜奔马”正反映了这种轻盈的步态。祁连山南北草原的大通马中, 有大量天生会走这种步法的马, 可能是人工选择的结果[106]。除了骑乘, 对于步态灵活度要求最高的是马的芭蕾舞表演——“盛装舞步”。在中国古代, 舞马(类似于盛装舞步, 属于马的特殊用途)是一种历史悠久的杂技艺术, 在史籍上有过零星记载[107]。到唐玄宗开元天宝年间, 舞马达到极盛。西安南郊何家村出土的唐代银壶上, 一匹金溜马体态丰盈, 后腿屈蹬, 口衔酒杯作跪拜状, 舞马的形象得到了证实[107]。唐朝统治者令教坊负责训练汗血宝马为舞马, 专门为祝贺唐玄宗的寿辰而表演舞蹈。张说的《舞马千秋万岁乐府词》云:“金天诞圣千秋节, 玉礼还分万寿觞。试听紫骝歌乐府, 何如騄骥舞华岗”。这些舞马“衣以文绣, 络以金银, 饰以鬃鬣, 间杂珠玉”, 伴随《倾杯乐》的节奏, 奋首鼓尾, 欢腾舞蹈, 步伐整齐, 令人瞠目。并有四百匹舞马上演“舞马登床”和“舞马衔杯”[107]。安史之乱后, 这些舞马死的死, 散的散, 不知所踪。通过出土马骨的步态相关基因的检测, 或许是我们寻找舞马踪迹的一个候选方案。
3.6 身体大小/马肩隆高度身高是马最容易观察到的一个表型之一。马肩隆高度的变异范围较广, 既有80 cm的设得兰矮种马(Shetland pony)又有超过2 m的夏尔马(Shire horse)和珀切龙马(Percheron horse)。对于矮马的定义是肩高低于148 cm[77]。Brooks等[108]收集了世界范围内1215匹代表 65种不同形态的马的测量数据, 发现马肩隆高度的中值为160 cm, 其中一半马匹的马肩隆高度在150~160 cm之间。因此, 我们定义120 cm以下的马为小矮马, 148 cm为小型马和大型马的临界值。
相比人类由数百个具有较小影响的基因座控制着身高变异, 在家养动物中, 通常少数几个具有较大影响的位点就可以解释遗传力的大部分。Makvandi-Nejad等[109]根据全基因组关联分析, 发现4个位点LCORL(chr3:105547002)、HMGA2(chr6:81481064)、ZFAT(chr9:75550059)和LASP1(chr11:23259732)联用可以解释高达85 %马的体型差异。其中, LCORL(chr3:105547002)位点可以解释18 %的变异[110]。基于全基因组芯片, Frischknecht等[111]发现设得兰矮种马共享793 kb的单倍型区域。已知与马和许多其他物种的身高有关的HMGA2基因也属于该单倍型, 其中突变(HMGA2:c.83G>A)在矮马中常见, 而在正常大小的马的品种中没有发现[111]。对于矮马来说, 在设得兰矮种马(Shetland pony)中, 矮小型在ACAN基因上为纯合突变型C/C, 正常亲本为G/C[112];在中国特有的德保矮马(Debao pony)中, 80.65 %在TBX3基因(chr8:18101000)上携带等位基因A, 其他马种中未见纯合子A/A, 70.97 %在TBX3(chr8:18120526)上携带等位基因G, 其他马种中罕见纯合子G/G[113];在佛利斯矮马(Friesian dwarf horses)中, B4GALT7(chr14:3761355)为纯合子TT的为矮种, 其余正常, 该基因也与人类的侏儒症有关[114]。最后, Dall'Olio等[115]在16个品系的396匹马中发现, MSTN基因上的两个位点(chr18:66495696和chr18:66495826)与马的体型有关, 前者的突变型等位基因与矮胖有关, 野生型等位基因与高瘦有关。
《酉阳杂俎·毛篇》:“旧种马:戎马八尺, 田马七尺, 驽马六尺。”可见戎马和身高直接相关。汉景帝时禁止高五尺九寸(约135.7 cm)以上的壮年马出关。汉代引入汗血马, 唐代引入二十几种不同类型的马种, 在陇西建马场育成唐马[116]。我们可以对祭祀用马、陪葬用马、拉车用马等进行分类, 通过综合观察其决定运动能力、步态和身材大小的基因的差异, 更加深入地展开考古学讨论。
3.7 高原适应生活在高海拔地区会承受生理和代谢方面的挑战, 例如缺氧、低温、高辐射和缺乏食物等, 这些挑战会给当地的物种带来强大的进化压力。在人[117]、猪[118]、狗[119]、牦牛[120]等的高原适应研究中, EPAS1基因均被发现与高海拔适应有关。Hendrickson[121]通过对比低地马和安第斯山马的基因组, 发现EPAS1内含子上的一个SNP位点(rs69041973;chr 15:52570648)与马的高海拔适应相关。Liu等[122]对138匹涵盖了中国的不同海拔的家马进行了全基因组测序, 发现EPAS1基因在藏马的基因组中显示出了极强的选择信号。其中两个错义突变与在缺氧条件下促进血液循环和低氧消耗的血液生理参数密切相关[122]。因此在研究高海拔地区及其邻近区域的遗址时, 马在基因上能否适应高原环境可能也是一个非常有趣的话题。
4 结语根据考古学所关心的谱系、性别、毛色、运动能力、步态、肩高、高原适应这7个方面, 我们优化了三款基于高通量测序的检测panel。Panel 1主要是完成母系线粒体鉴定(控制区和细胞色素b)和性别鉴定(SRY和AMEL)。如果所测样本为雄性, 我们将进一步检测Panel 2 (6个Y-STRs和40个Y-SNPs), 完成父系Y染色体分型鉴定。最后, 我们会对所有雄性样本进行Panel 3检测, 获取毛色(MC1R、ASIP、MATP、KIT、EDNRB、SILV、TRPM 1)、运动能力(MSTN、RALGAPA 2)、步态(DMRT3)、肩高(LCORL、HMGA 2、ZFAT、LASP 1、HMGA 2、ACAN、TBX 3、B4GALT7)、高原适应(EPAS1)相关基因的分型情况。基于这些Panel, 我们对陕西省雍山血池秦汉祭祀遗址北斗坊7号长条坑中26匹幼马马骨进行了古DNA检测和分析, 探讨该遗址祭祀用马的来源和挑选标准的问题[123]。结果表明其母系来源的多样性极高、马匹中雄性略多、栗色马占绝对主体、马匹的爆发力和步法灵活度一般。这些似乎暗示当时马匹的来源地不止一处, 对祭祀用马的性别要求并不严格, 对同一坑中马的毛色有明确规定。从运动能力上看, 这些马虽然耐力较好但受爆发力和灵活度的限制, 不太可能成长为出色的战马[123]。
在中国, 马的古基因组学研究必将更深入地与考古学实践相结合, 并为考古学带来新的活力和范式。但是有些问题值得我们思考: 1)起源地和驯化地的问题。家马的驯化地位于欧亚大陆西部, 但是家马的野生型祖先的起源地仍属未知。考虑到线粒体谱系F的分布, 驯化早期中国境内野马的谱系鉴定非常重要。2)中国现代马和古代马Y染色体数据极少, 全基因组数据也非常欠缺, 严重影响了马的古基因组学发展, 相关研究急需开展。3)上述基因型和表型的关联研究绝大部分是在西欧亚品系中开展, 中国境内现代马的验证工作并不充分, 也有待加强。4)马的古基因组学与考古学相结合的问题, 可能需要更多感兴趣的研究者加入进来, 集思广益才能做到真正的学科融合。马的古基因组研究能解决的不仅仅是上述几个方面的问题, 比如说马的古病理研究、伴生古病原菌的演化等, 也将是未来的研究热点。
| [1] |
Kelekna P. The Horse in Human History[M]. Cambridge: Cambridge University Press, 2009: 2-5.
|
| [2] |
Anthony D W, Ringe D. The Indo-European homeland from linguistic and archaeological perspectives[J]. Annual Review of Linguistics, 2015, 1(1): 199-219. |
| [3] |
Anthony D W. The Horse, the Wheel, and Language:How Bronze-Age Steppes Shaped the Modern World[M]. Princeton: Princeton University Press, 2007: 222-225.
|
| [4] |
Outram A K, Stear N A, Bendrey R, et al. The earliest horse harnessing and milking[J]. Science, 2009, 323(5919): 1332-1335. DOI:10.1126/science.1168594 |
| [5] |
袁靖. 中国古代家养动物的动物考古学研究[J]. 第四纪研究, 2012, 30(2): 298-306. Yuan Jing. Zooarchaeological study on the domestic animals in ancient China.[J]. Quaternary Sciences, 2012, 30(2): 298-306. |
| [6] |
任乐乐, 董广辉. "六畜"的起源和传播历史[J]. 自然杂志, 2016, 38(4): 257-262. Ren Lele, Dong Guanghui. The history for origin and diffusion of "six livestock"[J]. Chinese Journal of Nature, 2016, 38(4): 257-262. |
| [7] |
王维清. 先秦祭祀中用马现象探析[J]. 古籍研究, 2015(1): 271-275. Wang Weiqing. Analysis on the phenomenon of employing horses in sacrificial ceremony in pre-Qin Dynasty[J]. Study of Ancient Books, 2015(1): 271-275. |
| [8] |
Machugh D E, Larson G, Orlando L. Taming the past:Ancient DNA and the study of animal domestication[J]. Annual Review of Animal Biosciences, 2017, 5(1): 329-351. DOI:10.1146/annurev-animal-022516-022747 |
| [9] |
Burbano H A, Hodges E, Green R E, et al. Targeted investigation of the Neandertal genome by array-based sequence capture[J]. Science, 2010, 328(5979): 723-725. DOI:10.1126/science.1188046 |
| [10] |
Green R E, Briggs A W, Krause J, et al. The Neandertal genome and ancient DNA authenticity[J]. Embo Journal, 2009, 28(17): 2494-2502. DOI:10.1038/emboj.2009.222 |
| [11] |
Churchill J D, Schmedes S E, King J L, et al. Evaluation of the IlluminaⓇ Beta Version ForenSeqTM DNA Signature Prep Kit for use in genetic profiling[J]. Forensic Science International:Genetics, 2016, 20: 20-29. DOI:10.1016/j.fsigen.2015.09.009 |
| [12] |
Sawyer S, Krause J, Guschanski K, et al. Temporal patterns of nucleotide misincorporations and DNA fragmentation in ancient DNA[J]. PLoS One, 2012, 7(3): e34131. DOI:10.1371/journal.pone.0034131 |
| [13] |
Krause J, Briggs A W, Kircher M, et al. A complete mtDNA genome of an early modern human from Kostenki, Russia[J]. Current Biology, 2010, 20(3): 231-236. DOI:10.1016/j.cub.2009.11.068 |
| [14] |
Hanni C, Laudet V, Stehelin D, et al. Tracking the origins of the cave bear(Ursus spelaeus)by mitochondrial DNA sequencing[J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(25): 12336-12340. DOI:10.1073/pnas.91.25.12336 |
| [15] |
Höss M, Dilling A, Currant A, et al. Molecular phylogeny of the extinct ground sloth Mylodon darwinii[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(1): 181. DOI:10.1073/pnas.93.1.181 |
| [16] |
Janczewski D N, Yuhki N, Gilbert D A, et al. Molecular phylogenetic inference from saber-toothed cat fossils of Rancho La Brea[J]. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(20): 9769-9773. DOI:10.1073/pnas.89.20.9769 |
| [17] |
Miller W, Drautz D I, Ratan A, et al. Sequencing the nuclear genome of the extinct woolly mammoth[J]. Nature, 2008, 456(7220): 387-390. DOI:10.1038/nature07446 |
| [18] |
Orlando L, Ginolhac A, Zhang G, et al. Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse[J]. Nature, 2013, 499(7456): 74-78. DOI:10.1038/nature12323 |
| [19] |
Orlando L. The first aurochs genome reveals the breeding history of British and European cattle[J]. Genome Biology, 2015, 16(1): 225. |
| [20] |
Ramírez O, Burgos-Paz W, Casas E, et al. Genome data from a sixteenth century pig illuminate modern breed relationships[J]. Heredity, 2015, 114(2): 175-184. DOI:10.1038/hdy.2014.81 |
| [21] |
Frantz L A, Mullin V E, Pionnier-Capitan M, et al. Genomic and archaeological evidence suggest a dual origin of domestic dogs[J]. Science, 2016, 352(6290): 1228. DOI:10.1126/science.aaf3161 |
| [22] |
Finno C J, Bannasch D L. Applied equine genetics[J]. Equine Veterinary Journal, 2014, 46(5): 538-544. DOI:10.1111/evj.12294 |
| [23] |
Wade C M, Giulotto E, Sigurdsson S, et al. Genome sequence, comparative analysis, and population genetics of the domestic horse[J]. Science, 2009, 326(5954): 865-867. DOI:10.1126/science.1178158 |
| [24] |
Orlando L, Ginolhac A, Raghavan M, et al. True single molecule DNA sequencing of a Pleistocene horse bone[J]. Genome Research, 2011, 21(10): 1705-1719. DOI:10.1101/gr.122747.111 |
| [25] |
Higuchi R, Bowman B, Freiberger M, et al. DNA sequences from the quagga, an extinct member of the horse family[J]. Nature, 1984, 312(5991): 282-284. DOI:10.1038/312282a0 |
| [26] |
Pääbo. Molecular cloning of ancient Egyptian mummy DNA[J]. Nature, 1985, 314(6012): 644-645. DOI:10.1038/314644a0 |
| [27] |
Meyer M, Arsuaga J L, de Filippo C, et al. Nuclear DNA sequences from the Middle Pleistocene Sima de los Huesos hominins[J]. Nature, 2016, 531(7595): 504-507. DOI:10.1038/nature17405 |
| [28] |
Fages A, Hanghoj K, Khan N, et al. Tracking five millennia of horse management with extensive ancient genome time series[J]. Cell, 2019, 177(6): 1419-1435. DOI:10.1016/j.cell.2019.03.049 |
| [29] |
Raudsepp T, Finno C J, Bellone R R, et al. Ten years of the horse reference genome:Insights into equine biology, domestication and population dynamics in the post-genome era[J]. Animal Genetics, 2019, 50(6): 569-597. DOI:10.1111/age.12857 |
| [30] |
Jonsson H, Schubert M, Seguin-Orlando A, et al. Speciation with gene flow in equids despite extensive chromosomal plasticity[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(52): 18655-18660. DOI:10.1073/pnas.1412627111 |
| [31] |
Schubert M, Jonsson H, Chang D, et al. Prehistoric genomes reveal the genetic foundation and cost of horse domestication[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(52): E5661-5669. DOI:10.1073/pnas.1416991111 |
| [32] |
Librado P, Der Sarkissian C, Ermini L, et al. Tracking the origins of Yakutian horses and the genetic basis for their fast adaptation to subarctic environments[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(50): 6889-6897. DOI:10.1073/pnas.1513696112 |
| [33] |
Librado P, Gamba C, Gaunitz C, et al. Ancient genomic changes associated with domestication of the horse[J]. Science, 2017, 356(6336): 442-445. DOI:10.1126/science.aam5298 |
| [34] |
Gaunitz C, Fages A, Hanghoj K, et al. Ancient genomes revisit the ancestry of domestic and Przewalski's horses[J]. Science, 2018, 360(6384): 111-114. DOI:10.1126/science.aao3297 |
| [35] |
Janecka J E, Davis B W, Ghosh S, et al. Horse Y chromosome assembly displays unique evolutionary features and putative stallion fertility genes[J]. Nature Communications, 2018, 9(1): 2945. DOI:10.1038/s41467-018-05290-6 |
| [36] |
Wutke S, Sandoval-Castellanos E, Benecke N, et al. Decline of genetic diversity in ancient domestic stallions in Europe[J]. Science Advances, 2018, 4(4): eaap9691. DOI:10.1126/sciadv.aap9691 |
| [37] |
Pamilo P, M Nei. Relationships between gene trees and species trees[J]. Molecular Biology Evolution, 1988, 5(5): 568-583. |
| [38] |
Hellenthal G, Busby G B J, Band G, et al. A genetic atlas of human admixture history[J]. Science, 2014, 343(7172): 747-751. |
| [39] |
蔡大伟, 韩璐, 谢承志, 等. 内蒙古赤峰地区青铜时代古马线粒体DNA分析[J]. 自然科学进展, 2007, 17(3): 385-390. Cai Dawei, Han Lu, Xie Chengzhi, et al. Mitochondrial DNA analysis of Bronze Age horses in Chifeng area, Inner Mongolia[J]. Progress in Natural Science, 2007, 17: 17. |
| [40] |
蔡大伟.古DNA与家养动物的起源研究[D].长春: 吉林大学博士学位论文, 2007: 44-64. Cai Dawei. Ancient DNA and the Origins of Domestic Animals[D]. Changchun: The Doctoral's Dissertation of Jilin University, 2007: 44-64. http://cdmd.cnki.com.cn/article/cdmd-10183-2007095720.htm |
| [41] |
陈曦.陕西凤翔秦公一号大墓车马坑马骨遗骸古DNA研究[D].长春: 吉林大学硕士学位论文, 2014: 8-16. Chen Xi. Mitochondrial DNA Analysis of Horse Remains from Chariot Burial Pits of No.1 Qingong Mausoleum in Fengxiang, Shaanxi Province[D]. Changchun: The Master's Thesis of Jilin University, 2014: 8-16. |
| [42] |
黄金龙.马属基因组和染色体快速进化的研究[D].呼和浩特: 内蒙古农业大学博士学位论文, 2015: 9-10. Huang Jinlong. Equus Genomes and the Study of Fast Chromosomal Evolution[D]. Hohhot: The Doctoral's Dissertation of Inner Mongolia Agricultural University, 2015: 9-10. http://cdmd.cnki.com.cn/Article/CDMD-10129-1015427466.htm |
| [43] |
毛春春.马KIT基因与Y染色体遗传多样性研究[D].杨凌: 西北农林科技大学硕士学位论文, 2012: 10-12. Mao Chunchun. Genetic Diversity of KIT Gene and Y Chromosome in Horses[D]. Yangling: The Master's Thesis of Northwest A&F University, 2012: 10-12. http://cdmd.cnki.com.cn/Article/CDMD-10712-1012436856.htm |
| [44] |
Vila C L J, Gotherstrom A, Marklund S, et al. Widespread origins of domestic horse lineages[J]. Science, 2001, 291(5503): 474-477. DOI:10.1126/science.291.5503.474 |
| [45] |
Jansen T, Forster P, Levine M A, et al. Mitochondrial DNA and the origins of the domestic horse[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(16): 10905-10910. DOI:10.1073/pnas.152330099 |
| [46] |
McGahern A, Bower M A, Edwards C J, et al. Evidence for biogeographic patterning of mitochondrial DNA sequences in Eastern horse populations[J]. Animal Genetics, 2006, 37(5): 494-497. DOI:10.1111/j.1365-2052.2006.01495.x |
| [47] |
Achilli A, Olivieri A, Soares P, et al. Mitochondrial genomes from modern horses reveal the major haplogroups that underwent domestication[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(7): 2449-2454. DOI:10.1073/pnas.1111637109 |
| [48] |
Librado P, Fages A, Gaunitz C, et al. The evolutionary origin and genetic makeup of domestic horses[J]. Genetics, 2016, 204(2): 423-434. DOI:10.1534/genetics.116.194860 |
| [49] |
Wallner B, Palmieri N, Vogl C, et al. Y chromosome uncovers the recent oriental origin of modern stallions[J]. Current Biology, 2017, 27(13): 2029-2035. DOI:10.1016/j.cub.2017.05.086 |
| [50] |
Wallner B, Vogl C, Shukla P, et al. Identification of genetic variation on the horse Y chromosome and the tracing of male founder lineages in modern breeds[J]. PLoS One, 2013, 8(4): e60015. DOI:10.1371/journal.pone.0060015 |
| [51] |
Han H, Zhang Q, Gao K, et al. Y-single nucleotide polymorphisms diversity in Chinese indigenous horse[J]. Asian Australasian Journal of Animal Science, 2015, 28(8): 1066-1074. DOI:10.5713/ajas.14.0784 |
| [52] |
Kreutzmann N, Brem G, Wallner B. The domestic horse harbours Y-chromosomal microsatellite polymorphism only on two widely distributed male lineages[J]. Animal Genetics, 2014, 45(3): 460. DOI:10.1111/age.12149 |
| [53] |
Wallner B, Piumi F, Brem G, et al. Isolation of Y chromosome-specific microsatellites in the horse and cross-species amplification in the genus Equus[J]. Journal of Heredity, 2004, 95(2): 158-164. DOI:10.1093/jhered/esh020 |
| [54] |
Felkel S, Vogl C, Rigler D, et al. Asian horses deepen the MSY phylogeny[J]. Animal Genetics, 2018, 49(1): 90-93. |
| [55] |
Felkel S, Vogl C, Rigler D, et al. The horse Y chromosome as an informative marker for tracing sire lines[J]. Scientific Reports, 2019, 9(1): 6095. DOI:10.1038/s41598-019-42640-w |
| [56] |
Boeskorov G G, Potapova O R, Protopopov A V, et al. A study of a frozen mummy of a wild horse from the Holocene of Yakutia, east Siberia, Russia[J]. Mammal Research, 2018, 63(3): 1-8. |
| [57] |
Goto H, Ryder O A, Fisher A R, et al. A massively parallel sequencing approach uncovers ancient origins and high genetic variability of endangered Przewalski's horses[J]. Genome Biology and Evolution, 2011, 3: 1096-1106. DOI:10.1093/gbe/evr067 |
| [58] |
Der Sarkissian C, Ermini L, Schubert M, et al. Evolutionary genomics and conservation of the endangered Przewalski's horse[J]. Current Biology, 2015, 25(19): 2577-2583. DOI:10.1016/j.cub.2015.08.032 |
| [59] |
de Barros Damgaard P, Martiniano R, Kamm J, et al. The first horse herders and the impact of early Bronze Age steppe expansions into Asia[J]. Science, 2018, 360(6396): 7711. DOI:10.1126/science.aar7711 |
| [60] |
Outram A K, Stear N A, Bendrey R, et al. The earliest horse harnessing and milking[J]. Science, 2009, 323(5919): 1332-1335. DOI:10.1126/science.1168594 |
| [61] |
Warmuth V, Eriksson A, Bower M A, et al. Reconstructing the origin and spread of horse domestication in the Eurasian steppe[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(21): 8202-8206. DOI:10.1073/pnas.1111122109 |
| [62] |
Hendricks B L. International Encyclopedia of Horse Breeds[M]. Norman: University of Oklahoma Press, 2007: 124-125.
|
| [63] |
Petersen J L, Mickelson J R, Cothran E G, et al. Genetic diversity in the modern horse illustrated from genome-wide SNP data[J]. PLoS One, 2013, 8(1): e54997. DOI:10.1371/journal.pone.0054997 |
| [64] |
McCue M E, Bannasch D L, Petersen J L, et al. A high density SNP array for the domestic horse and extant Perissodactyla:Utility for association mapping, genetic diversity, and phylogeny studies[J]. PLoS Genetics, 2012, 8(1): e1002451. DOI:10.1371/journal.pgen.1002451 |
| [65] |
Andersson L, Archibald A L, Bottema C D, et al. Coordinated international action to accelerate genome-to-phenome with FAANG, the Functional Annotation of Animal Genomes project[J]. Genome Biology, 2015, 16: 57. DOI:10.1186/s13059-015-0622-4 |
| [66] |
Brooks S A, Gabreski N, Miller D, et al. Whole-genome SNP association in the horse:Identification of a deletion in myosin Va responsible for Lavender Foal Syndrome[J]. PLoS Genetics, 2010, 6(4): e1000909. DOI:10.1371/journal.pgen.1000909 |
| [67] |
Mack M, Kowalski E, Grahn R, et al. Two variants in SLC24A5 are associated with "tigereye" iris pigmentation in Puerto Rican Paso Fino horses[J]. G3—Genes, Genomes, Genetics(Bethesda, Md.), 2017, 7(8): 2799-2806. |
| [68] |
Bellone R R, Liu J, Petersen J L, et al. A missense mutation in damage-specific DNA binding protein 2 is a genetic risk factor for limbal squamous cell carcinoma in horses[J]. International Journal of Cancer, 2017, 141(2): 342-353. DOI:10.1002/ijc.30744 |
| [69] |
Schaefer R J, Schubert M, Bailey E, et al. Developing a 670 k genotyping array to tag~2 M SNPs across 24 horse breeds[J]. BMC Genomics, 2017, 18(1): 565. DOI:10.1186/s12864-017-3943-8 |
| [70] |
Bauer A, Hiemesch T, Jagannathan V, et al. A nonsense variant in the ST14 gene in Akhal-Teke horses with naked foal syndrome[J]. G3—Genes, Genomes, Genetics(Bethesda, Md.), 2017, 7(4): 1315-1321. |
| [71] |
Thomer A, Gottschalk M, Christmann A, et al. An epistatic effect of KRT 25 on SP 6 is involved in curly coat in horses[J]. Scientific Reports, 2018, 8(1): 6374. DOI:10.1038/s41598-018-24865-3 |
| [72] |
Doan R, Cohen N, Harrington J, et al. Identification of copy number variants in horses[J]. Genome Research, 2012, 22(5): 899-907. DOI:10.1101/gr.128991.111 |
| [73] |
Ghosh S, Qu Z, Das P J, et al. Copy number variation in the horse genome[J]. PLoS Genetics, 2014, 10(10): e1004712. DOI:10.1371/journal.pgen.1004712 |
| [74] |
Kalbfleisch T S, Rice E S, DePriest M S, et al. Improved reference genome for the domestic horse increases assembly contiguity and composition[J]. Communications Biology, 2018, 1: 197. DOI:10.1038/s42003-018-0199-z |
| [75] |
Raudsepp T, Gustafson-Seabury A, Durkin K, et al. A 4, 103 marker integrated physical and comparative map of the horse genome[J]. Cytogenetic and Genome Research, 2008, 122(1): 28-36. DOI:10.1159/000151313 |
| [76] |
Leeb T, Vogl C, Zhu B, et al. A human-horse comparative map based on equine BAC end sequences[J]. Genomics, 2006, 87(6): 772-776. DOI:10.1016/j.ygeno.2006.03.002 |
| [77] |
Liu X, Ma Yuehui, Jiang L. Genomic regions under selection for important traits in domestic horse breeds[J]. Frontiers of Agricultural Science and Engineering, 2017, 4(3): 289-294. |
| [78] |
Wutke S, Benecke N, Sandoval-Castellanos E, et al. Spotted phenotypes in horses lost attractiveness in the Middle Ages[J]. Scientific Reports, 2016, 6(1): 38548. DOI:10.1038/srep38548 |
| [79] |
赵欣, 李悦, 陈洪海, 等. 陕西省淳化县枣树沟脑遗址马坑出土马骨的DNA初步研究[J]. 南方文物, 2015(3): 70-76. Zhao Xin, Li Yue, Chen Honghai, et al. Preliminary aDNA study of unearthed horse remains from chariot pits of Zaoshugounao site in Chunhua County, Shaanxi Province[J]. Cultural Relics in Southern China, 2015(3): 70-76. DOI:10.3969/j.issn.1004-6275.2015.03.012 |
| [80] |
赵欣, Rodrigues A T, 尤悦, 等. 新疆石人子沟遗址出土家马的DNA研究[J]. 第四纪研究, 2014, 34(1): 187-195. Zhao Xin, Rodrigues A T, You Yue, et al. Ancient DNA analysis of ancient horse remains from Shirenzigou site, Xinjiang, China[J]. Quaternary Sciences, 2014, 34(1): 187-195. DOI:10.3969/j.issn.1001-7410.2014.01.22 |
| [81] |
赵欣, 东晓玲, 韩雨, 等. 新疆木垒县平顶山墓群出土马骨的DNA研究[J]. 南方文物, 2017(3): 187-191. Zhao Xin, Dong Xiaoling, Han Yu, et al. Ancient DNA analysis of unearthed horse remains from Pingdingshan site in Mulei County, Xinjiang[J]. Cultural Relics in Southern China, 2017(3): 187-191. DOI:10.3969/j.issn.1004-6275.2017.03.025 |
| [82] |
《中国畜禽遗传资源状况》编委会编. 中国畜禽遗传资源状况[M]. 北京: 中国农业出版社, 2004: 48-55. Editorial Board of Genetic Resources of Livestock and Poultry in China. Genetic Resources of Livestock and Poultry in China[M]. Beijing: China Agriculture Press, 2004: 48-55. |
| [83] |
谢成侠. 中国马驴品种志[M]. 上海: 上海科学技术出版社, 1986: 18-21. Xie Chengxia. Breeds of Horse and Donkey in China[M]. Shanghai: Shanghai Science and Technology Press, 1986: 18-21. |
| [84] |
袁靖, 安家瑗.中国动物考古学研究的两个问题[N].中国文物报, 1997-4-27, (3). Yuan Jing, An Jiayuan. Two Problems in Zooarchaeology in China[N]. Chinese Cultural Relics News, 1997-4-27, (3). |
| [85] |
王宜涛.也谈中国马类动物历史及相关问题[N].中国文物报, 1998-8-12, (3). Wang Yitao. Rethinking the History of Equid in China and Its Related Issues[N]. Chinese Cultural Relics News, 1998-8-12, (3). |
| [86] |
Cai D, Tang Z, Han L, et al. Ancient DNA provides new insights into the origin of the Chinese domestic horse[J]. Journal of Archaeological Science, 2009, 36(3): 835-842. DOI:10.1016/j.jas.2008.11.006 |
| [87] |
Ruscillo D. Zooarchaeology: Methods of collecting age and sex data[M]//Smith C. Encyclopedia of Global Archaeology. New York: Springer, 2014: 8000-8009.
|
| [88] |
Lyublyanovics K. Size variability in roman period horses from Hungary[M]//Ruscillo D. Recent Advances in Ageing and Sexing Animal Bones. Durham: Oxbow Books, 2006: 237-246.
|
| [89] |
Heidi N, Albína P, Bastiaan Star, et al. Sexing Viking Age horses from burial and non-burial sites in Iceland using ancient DNA[J]. Journal of Archaeological Science, 2019, 101: 115-122. DOI:10.1016/j.jas.2018.11.007 |
| [90] |
Taylor W. Horse demography and use in Bronze Age Mongolia[J]. Quaternary International, 2017, 436(A): 270-282. |
| [91] |
Sébastien L. Horse sacrifice in a Pazyryk Culture Kurgan:The princely tomb of Berel'(Kazakhstan). selection Criteria and slaughter procedures[J]. Anthropozoologica, 2013, 48(2): 309-321. DOI:10.5252/az2013n2a9 |
| [92] |
Andres S D, Doug P, Jacob K, et al. The four horses of an Iron Age apocalypse:War-horses from the third-century weapon sacrifice at Illerup Aadal(Denmark)[J]. Antiquity, 2014, 88(339): 191-204. DOI:10.1017/S0003598X00050304 |
| [93] |
尤悦, 于建军, 陈相龙, 等. 早期铁器时代游牧人群用马策略初探——以新疆喀拉苏墓地M15随葬马匹的动物考古学研究为例[J]. 西域研究, 2017(4): 108-120. You Yue, Yu Jianjun, Chen Xianglong, et al. A research on horse exploitation of nomadic people in the Early Iron Age—A zooarchaeological case study of Tomb 15 in Kalasu Cemetery, Xinjiang[J]. The Western Regions Studies, 2017(4): 108-120. |
| [94] |
Götherström A. The value of stallions and mares during the Early Medieval Time in upper class Svealand:Molecular sex identifications on horse remains from Vendel and Eketorp[J]. Journal of Nordic Archaeological Science, 2002, 13: 75-78. |
| [95] |
赵若阳, 赵一萍, 李蓓, 等. 马毛色遗传机理研究进展[J]. 遗传, 2018, 40(5): 21-32. Zhao Ruoyang, Zhao Yiping, Li Bei, et al. Overview of the genetic control of horse coat color patterns[J]. Hereditas, 2018, 40(5): 21-32. |
| [96] |
Ludwig A, Pruvost M, Reissmann M, et al. Coat color variation at the beginning of horse domestication[J]. Science, 2009, 324(5926): 485-485. DOI:10.1126/science.1172750 |
| [97] |
李卫平. 现代中国爱马人手册[M]. 北京: 中国铁道出版社, 2002: 20-27. Li Weiping. Handbook of Equestrian Fans in Modern China[M]. Beijing: China Railway Publishing House, 2002: 20-27. |
| [98] |
Hill E W, Gu Jingjing, Eivers S S, et al. A Sequence polymorphism in MSTN predicts sprinting ability and racing stamina in thoroughbred horses[J]. PLoS One, 2010, 5(1): e8645. DOI:10.1371/journal.pone.0008645 |
| [99] |
Moon S, Lee J W, Shin D H, et al. A genome-wide scan for selective sweeps in racing horses[J]. Asian Australasian Journal of Animal Sciences, 2015, 28(11): 1525-1531. DOI:10.5713/ajas.14.0696 |
| [100] |
Andersson L S, Larhammar M, Memic F, et al. Mutations in DMRT 3 affect locomotion in horses and spinal circuit function in mice[J]. Nature, 2012, 488(7413): 642-646. DOI:10.1038/nature11399 |
| [101] |
Promerová M, Andersson L S, Juras R, et al. Worldwide frequency distribution of the 'Gait keeper' mutation in the DMRT 3 gene[J]. Animal Genetics, 2014, 45(2): 274-282. |
| [102] |
Fegraeus K J, Johansson L, Mäenpää M, et al. Different DMRT 3 genotypes are best adapted for harness racing and riding in Finnhorses[J]. Journal of Heredity, 2015, 106(6): 734-740. |
| [103] |
Revold T, Mykkänen A K, Karlström K, et al. Effects of training on equine muscle fibres and monocarboxylate transporters in young Coldblooded Trotters[J]. Equine Veterinary Journal, 2010, 42(38): 289-295. |
| [104] |
Solé M, Cervantes I, Gutiérrez J P, et al. Estimation of genetic parameters for morphological and functional traits in a Menorca horse population[J]. Spanish Journal of Agricultural Research, 2014, 12(1): 125. DOI:10.5424/sjar/2014121-4686 |
| [105] |
Brard S, Ricard A. Genome-wide association study for jumping performances in French sport horses[J]. Animal Genetics, 2015, 46(1): 78-81. |
| [106] |
甘肃农业大学主编. 养马学[M]. 北京: 中国农业出版社, 1990: 70-75. Gansu Agricultural University. Horse Breeding[M]. Beijing: China Agriculture Press, 1990: 70-75. |
| [107] |
傅起凤, 傅腾龙著. 中国杂技[M]. 天津: 天津科学技术出版社, 1983: 76-78. Fu Qifeng, Fu Tenglong. Chinese Acrobatics[M]. Tianjin: Tianjin Science and Technology Press, 1983: 76-78. |
| [108] |
Brooks S A, Makvandi-Nejad S, Chu E, et al. Morphological variation in the horse:Defining complex traits of body size and shape[J]. Animal Genetics, 2010, 41(Suppl.2): 159-165. |
| [109] |
Makvandi-Nejad S, Hoffman G E, Allen J J, et al. Four loci explain 83 % of size variation in the horse[J]. PLoS One, 2012, 7(7): e39929. DOI:10.1371/journal.pone.0039929 |
| [110] |
Petersen J L, Mickelson J R, Rendahl A K, et al. Genome-wide analysis reveals selection for important traits in domestic horse breeds[J]. PLoS Genetics, 2013, 9(1): e1003211. DOI:10.1371/journal.pgen.1003211 |
| [111] |
Frischknecht M, Jagannathan V, Plattet P, et al. A non-synonymous HMGA 2 variant decreases height in shetland ponies and other small horses[J]. PLoS ONE, 2015, 10(10): 1-11. |
| [112] |
Metzger J, Gast A C, Schrimpf R, et al. Whole-genome sequencing reveals a potential causal mutation for dwarfsm in the miniature Shetland pony[J]. Mammalian Genome, 2017, 28(3-4): 143. DOI:10.1007/s00335-016-9673-4 |
| [113] |
Kader A, Li Y, Dong K, et al. Population variation reveals independent selection towards small body size in Chinese Debao pony[J]. Genome Biology and Evolution, 2015, 8(1): 42-50. |
| [114] |
Orr N, Back W, Gu J, et al. Genome-wide SNP association-based localization of a dwarfism gene in Friesian dwarf horses[J]. Animal Genetics, 2010, 41(Suppl.2): 2-7. |
| [115] |
Dall'Olio S, Fontanesi L, Costa L N, et al. Analysis of horse myostatin gene and identification of single nucleotide polymorphisms in breeds of different morphological types[J]. Journal of Biomedicine and Biotechnology, 2010(6628): 542945. DOI:10.1155/2010/542945 |
| [116] |
王俊. 中国传统民俗文化——中国古代养殖[M]. 北京: 中国商业出版社, 2015: 92-94. Wang Jun. Chinese Traditional Folk Culture—Cultivation in Ancient China[M]. Beijing: China Commercial Press, 2015: 92-94. |
| [117] |
Beall C M, Cavalleri G L, Deng L, et al. Natural selection on EPAS 1(HIF2alpha)associated with low hemoglobin concentration in Tibetan highlanders[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(25): 11459-11464. DOI:10.1073/pnas.1002443107 |
| [118] |
Dong K Z, Kang Y, Yao N, et al. Genetic variation of EPAS 1 gene in Tibetan pigs and three low-altitude pig breeds in China[J]. Journal of Integrative Agriculture, 2014, 13(9): 1990-1998. DOI:10.1016/S2095-3119(13)60577-8 |
| [119] |
Fan R, Fei L, Hong W, et al. A positive correlation between elevated altitude and frequency of mutant alleles at the EPAS 1 and HBB loci in Chinese indigenous dogs[J]. Journal of Genetics & Genomics, 2015, 42(4): 173-177. |
| [120] |
Wu X Y, Ding X Z, Min C, et al. Novel SNP of EPAS 1 gene associated with higher hemoglobin concentration revealed the hypoxia adaptation of yak(Bos grunniens)[J]. Journal of Integrative Agriculture, 2015, 14(4): 741-748. DOI:10.1016/S2095-3119(14)60854-6 |
| [121] |
Hendrickson S L. A genome wide study of genetic adaptation to high altitude in feral Andean Horses of the Páramo[J]. BMC Evolutionary Biology, 2013, 13(1): 273-273. |
| [122] |
Liu X, Zhang Y, Li Y, et al. EPAS1 gain-of-function mutation contributes to high-altitude adaptation in Tibetan horses[J]. Molecular Biology and Evolution, 2019, 36(11): 2591-2603. DOI:10.1093/molbev/msz158 |
| [123] |
文少卿, 俞雪儿, 孙畅, 等.雍山血池秦汉祭祀遗址北斗坊7号坑马骨的古DNA研究[J].考古与文物, 2020, 发表中. Wen Shaoqing, Yu Xue'er, Sun Chang, et al. Ancient DNA analysis of horse remains from the pit 7 of Beidoufang in Xuechi Sacrificial Site[J]. Archaeology and Cultural Relics, 2020, in press. |
2 Ministry of Education Key Laboratory of Contemporary Anthropology, School of Life Sciences, Fudan University, Shanghai 200433;
3 Shaanxi Academy of Archaeology, Xi'an 710054, Shaanxi;
4 School of Cultural Heritage, Northwest University, Xi'an 710069, Shaanxi)
Abstract
DNA analysis on degraded samples revealed itself as an important research tool in anthropology, molecular evolution, and population genetics. The past decade has witnessed a revolution in ancient DNA research. Although the field's focus was previously limited to mitochondrial DNA and a few nuclear markers, whole genome sequences from the deep past can now be retrieved. This breakthrough is tightly connected to the massive sequence throughput of next generation sequencing platforms and the ability to target short and degraded DNA molecules, giving rise to the new field of paleogenomics. Here, we reviewed the course of development of equine genomics, and described the state-of-the-art achievements in horses paleogenomic studies, with particular focus on revealing genetic lineages of ancient horses and predicting equine traits via the results of genotype-phenotype correlation analysis. Based on previous horse genome researches, we optimized three NGS-based panels to analyze the genes associated with horse lineages, gender, coat color, racing performance, gaits, body size/height at withers, and high-altitude adaptation and other aspect. Our results show that genetic lineages and phenotypic traits are crucial to the study of the origin and use of domestic horses in China, especially the source and selection criteria of sacrificial horses. This research contributes to our understanding of the evolution and spread of domestic horse in China, and its relationship to past social, political, economic, cultural and ideological developments. 2020, Vol.40
2020, Vol.40
