2 中国科学院大学, 北京 100049;
3 The Globe Institute, University of Copenhagen, Copenhagen, Denmark 1350;
4 中国科学院青藏高原地球科学卓越创新中心, 北京 100101;
5 兰州大学资源环境学院, 教育部西部环境重点实验室, 甘肃 兰州 730000)
古DNA(ancient DNA,简称aDNA)是指来源于古代生物遗存中的DNA分子[1]。最早涉及古DNA的研究出现在1980年,湖南医学院的研究人员对马王堆汉代女尸的古核酸进行了研究,但当时的技术条件限制了古DNA的检测进而与古DNA概念的提出失之交臂[2]。“ancient DNA”这一概念首先由现任德国马普学会莱比锡进化人类研究所所长的Pääbo[3]提出。1985年,Pääbo等[3]将现代DNA方法应用于古埃及木乃伊的研究中,虽然后续的研究证实其结果是现代DNA污染,但也展示了古DNA在考古学研究中的价值。之后,古DNA研究在考古领域涌现出大量的研究成果,如: Pääbo科研团队对尼安德特人和丹尼索瓦人基因组的解码[4~6],Raghavan等[7]对“幽灵人群”马耳他男孩基因组的发现,Miller等[8]对灭绝猛犸象基因组的恢复和Ginolhac等[9]对DNA损伤算法的开发等。在中国,世纪之交前后便有学者开始关注古DNA方法[10~11],之后,周慧等[12]、崔银秋等[13]相继报道了利用古DNA开展的中国古代人研究。随着测序技术和分析手段的进步,付巧妹及其团队[14~16]完成了中国4万年前北京田园洞人古基因组的提取和分析,利用45000年前人类的古基因组追溯冰河时代欧亚人群的遗传谱图,并参与实现了对晚期尼安德特人遗传历史的重建;同时,在动物古DNA研究中也取得了重要进展,如2018年,盛桂莲等[17]对古代大熊猫全基因组的测定等。
古环境DNA(ancient environmental DNA或palaeoenvironmental DNA,简称ancient eDNA)是散布于古环境样品中的生物古DNA[18]。不同于直接从古代动物骨骼、植物遗存内提取的古DNA,古环境DNA主要是从冰川、冻土、湖泊沉积物、泥炭沉积物、遗址文化层、牙结石、粪便化石等环境样品中提取得到的混合了多物种的古DNA。这些DNA随着生物的遗留物质(包括:遗体、毛发、粪便和尿液等)进入环境,在环境中迅速降解,缓慢变性,最终吸附在矿物等颗粒上或者被微生物整合到自身基因组上而长期保存,进而形成了古环境DNA[19~20]。最早的古环境DNA研究始于1998年Poinar等[21]对粪便化石中的古DNA研究。该研究反映出古环境DNA在恢复古生物食性中的重要作用;之后Rawlence等[18]在2014年对古环境DNA进行了重新命名而形成独立概念。古环境DNA具有明显的古DNA(aDNA)特征(年代久远、高度降解、片段化和化学损伤)和环境DNA特征(混合了多物种的DNA)[22]。古环境DNA在长时间的保存下出现强烈的降解和化学损伤,大部分古环境DNA因长时间的降解作用而消失,但少量的古环境DNA在寒冷干燥环境中得到了良好的保存——目前已知保存最古老的古环境DNA来自格陵兰冰芯大约80万年[23]。从环境样品中提取得到的古DNA包含了大量的古代生物的DNA信息,结合第二代高通量测序技术和生物信息学分析可以高效而准确地检测出其中包含的古代生物物种。在历经20年的发展后,古环境DNA技术已日臻成熟并逐渐成为环境考古学研究的主要研究手段之一。
目前古环境DNA研究的主要流程包括:样品采集,古环境DNA提取,古环境DNA定量及扩增,DNA测序,古环境DNA数据分析等内容。相对完善的古环境DNA研究体系,可以完成对古环境样品中大部分动物、植物和微生物物种的检测并广泛应用到古生态环境重建、古代农业发展、古代人类食性与健康、人类扩散历史和人类活动对环境影响等环境考古学研究中(图 1)。本文从古环境DNA研究方法、古环境DNA在环境考古学中的应用以及古环境DNA技术未来的发展方向等3个主要方面进行了综述,以期能为中国环境考古研究提供新的思路和启发。
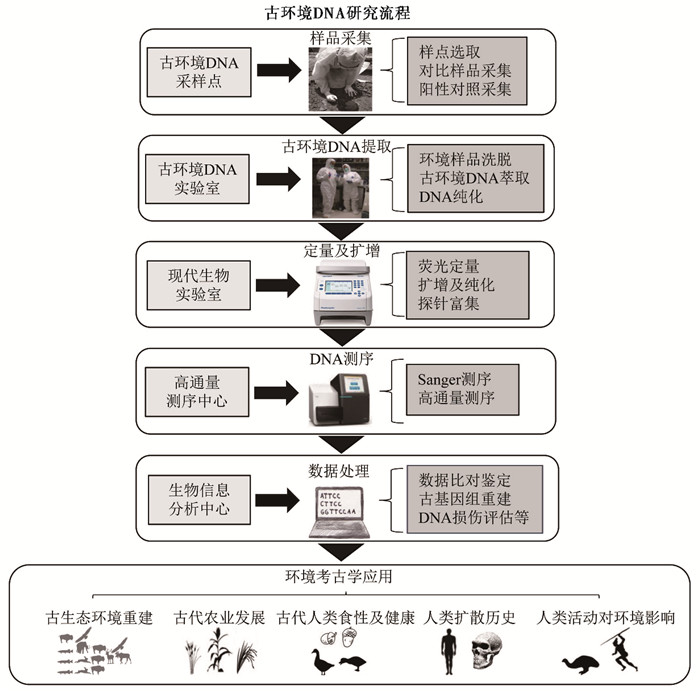
|
图 1 古环境DNA研究方法及环境考古学应用示意图 Fig. 1 Diagram for methods of paleoenvironmental DNA and its application in environmental archaeology |
古环境DNA样品采集前要对沉积剖面进行选择,首先排除存在迁移和淋溶的剖面,之后尽可能对沉积剖面的不同层位进行样品采集。为了排除表层现代DNA的污染,采样前将质粒DNA的聚合酶链式反应(Polymerase Chain Reaction,简称PCR)扩增产物溶液作为示踪物涂抹在剖面表面[24]。采样时,先采集表层样品作为阳性对照,之后将表层刮除并采集内部未受污染的样品。样品采集需要严格控制外源污染:采样时要佩戴发网、袖套、口罩和手套并穿着防护服;用来采集样品的器具要提前经过灭菌处理;在操作过程中要经常更换一次性手套,并使用消毒液(5 %的次氯酸钠溶液)来排除潜在的DNA污染;用无菌一次性手术刀或注射器对样品进行采集;采集的样品要直接放于无菌袋或无菌离心管中。样品获取后需及时进行DNA提取,如果长时间不处理需要放于-20℃冰箱冷冻保存[25]。
2.2 古环境DNA提取提取古环境DNA时希望较少受到现代生物DNA污染,同时获取样品中大部分生物残留的古DNA,因此提取过程需在未进行现代DNA扩增的超净古DNA实验室完成,同时提取方法需要尽可能多的移除腐殖质等DNA提取抑制因子。目前用于古环境DNA提取的方法主要有两种:一是改良的试剂盒提取法,一是改良的饱和苯酚-氯仿抽提法。
试剂盒提取法简便易行,改良后也因其便捷性而经常用于古环境DNA的提取。由于古环境DNA常常吸附于石英、蒙脱石、长石和方解石等矿物颗粒上[26],所以在提取前需要对样品进行洗脱再进行提取。Matisoo-Smith等[27]在提取湖泊沉积物古环境DNA时采用改良的土壤DNA(FastDNA Spin Kit for soil,Qbiogene)试剂盒提取法对沉积物古环境DNA进行了提取,在提取之前先使用陶瓷、二氧化硅颗粒裂解基质对环境样品进行破碎,之后加入含有十二烷基磺酸钠和磷酸盐缓冲液的裂解试剂和蛋白沉淀试剂进行洗脱和萃取。使用吸附剂(Binding Matrix)将萃取液中的DNA吸附出来,之后使用70 %的酒精和洗脱缓冲液进行抑制剂去除纯化从而获得古环境DNA。但由于商业试剂盒提取法采用试剂盒配备的试剂,在方法改进的空间上比较小,受裂解和洗脱试剂的限制,古环境DNA的获取效率并不是很高。
苯酚-氯仿抽提法,是一种传统的DNA提取方法,一般成本低、流程简单、提取效果理想[28~29]。基于苯酚-氯仿抽提法的改良方法也被应用到古环境DNA的研究中。Anderson-Carpenter等[30]在提取湖泊沉积物样品的古环境DNA时,采用了改良的酚氯仿法。该方法首先将样品放入缓冲液中孵育,随后加入一定比例的氯仿-异戊醇混合溶液进行萃取,通过超滤管离心过滤进行DNA截流浓缩,之后加入缓冲液洗脱以获取样品DNA。该方法操作简单,同时使用超滤管浓缩的方法提高了提取效率。Perderson等[24]则将Wales等[31]用于古植物遗存DNA提取的苯酚-氯仿法进行改良来提取古环境DNA,取得了更好的效果。其基本流程包括:样品震荡洗脱,裂解孵育,苯酚氯仿DNA萃取,离心柱浓缩和试剂盒纯化等。该方法采用常规试剂的同时缩减了操作步骤,可以保留大部分古环境样品中的DNA,并可以根据样品的理化性质选择纯化方式来提高DNA提取质量。
2.3 古环境DNA检测古环境DNA的研究技术主要经历了从DNA条形码技术(DNA barcoding)到宏条形码技术(metabarcoding)再到鸟枪法宏基因组(shotgun metagenomic)技术的发展过程[32~36]。目前主要应用宏条形码技术和鸟枪法宏基因组技术对古环境DNA进行研究。宏条形码和鸟枪宏基因组技术可以高效地识别古环境样品中的生物遗传物质,被广泛的应用到揭示古环境样品所包含的古代生物信息的研究中[36]。目前采用条形码技术和宏基因组技术进行的古环境DNA研究的对象包括冻土[37~38]、湖泊沉积物[39~44]、冰川下沉积物[45~47]、粪便化石[48~49]、牙结石[50~51]等环境样品。从环境样品中获得的DNA序列通过与标准数据库进行比对可以获得大量古代生物的分类信息[52]。
2.3.1 DNA条形码技术DNA条形码(DNA barcode)是指可以代表物种的、有足够变异的、易扩增且相对较短的生物DNA片段[53~54]。应用于古环境DNA研究的DNA条形码技术,主要通过特殊的引物对古环境DNA条形码序列进行PCR扩增,通过Sanger测序法来获得DNA条形码序列。得到的条形码序列与数据库进行比对,进而获得环境样品的物种信息。该方法通量较低,操作繁琐,但在早期古环境DNA研究中起到了重要的作用。比较有代表性的工作包括对西伯利亚冻土中的古环境DNA研究,以及对格陵兰深层冰芯的古植物研究等[32, 46]。
2.3.2 宏条形码技术宏条形码技术结合了DNA条形码和高通量测序技术的优势,使用特异性引物对目标生物类群的基因进行扩增,并通过高通量测序技术增加测序深度,从而增加对古环境DNA的覆盖度和识别度[35]。操作上,古环境DNA研究所采用的宏条形码技术对引物的要求比较高,为了获得大量的物种和精确的分类信息需要使用多种引物同时进行扩增来获得主要生物类群的分类信息[55],之后使用特异性引物对样品进行再次扩增进一步揭示比较关注的科或属的物种分类信息[56]。
宏条形码的主要优势是成本低,可以同时扩增并鉴定出指定类群的物种[56]。但也存在一定局限性,如:识别物种范围窄,通过标记物只能扩增并检测到样品中含有的部分物种DNA;分辨率低,较难区分基因序列相似的物种;存在PCR偏向性,扩增过程会导致物种遗传信息的比例发生变化[57~58]。
2.3.3 鸟枪法宏基因组技术全基因组测序方法下的宏基因组技术也被称为鸟枪法宏基因组技术[59~60]。鸟枪法宏基因组技术是用于古环境DNA研究的一种新兴技术,其特点是将环境中的全部生物遗传物质通过高通量技术进行测序,然后通过生物信息学算法将这些遗传信息与标准数据库比对,将基因信息破译为物种组成信息,从而分析古环境生物群落的组成[24, 60]。利用鸟枪法宏基因组技术可以帮助揭示环境样品中的大部分遗传及功能基因信息,目前部分研究已经取得了较好的成果。比较典型的研究包括:通过鸟枪宏基因组的方法恢复西伯利亚东北与北美西北海陆通道上过去12600年来的物种组成[24]和分析牙结石中残留物的基因组成进而揭示古人类的食性等[51]。
相比于传统的宏条形码方法鸟枪法宏基因组不需要对DNA进行扩增,因而排除了引物的限制,但在操作上加入了文库构建的步骤,所以要注意防止建库过程中引入的外源污染。与宏条形码技术相比宏基因组技术可以全面展示DNA片段信息,具有更高的物种分辨率、可量化样品DNA降解水平、直接识别古DNA序列等特点。该技术的出现,对古环境DNA的研究起到极大的促进作用。
2.4 古环境DNA数据分析目前的古环境DNA数据分析主要包括:数据库构建、数据比对鉴定、DNA损伤评估、污染排除、古基因组重建及进化史计算等内容。
(1) 数据库构建。目前常用的数据库主要是GenBank(www.ncbi.nlm.nih.gov/genbank/),同时根据不同物种鉴定的需求也会选取不同的数据库。如:细菌16S rRNA基因序列鉴定选取GREENGENES数据库(http://greengenes.secondgenome.com),真菌内转录间隔区(internal transcribed spacer,ITS)序列鉴定选取UNITE数据库(https://unite.ut.ee),真核生物18S rRNA基因序列鉴定选取SILVA数据库(https://www.arb-silva.de/)等。为了增加物种鉴定的精确度也常常通过本地物种基因序列来重新构建数据库,如: Sønstebø等[61]和Boessenkool等[62]根据842个北极现代植物物种构建的叶绿体trnL(tRNA L基因)序列数据库,在后续的研究中帮助缩减了数据处理流程同时提高了结果的精确度。
(2) 数据比对鉴定。在数据比对鉴定方面,早期DNA条形码和Sanger测序技术获得的数据主要通过直接使用GenBank BLAST进行比对鉴定。随着高通量测序技术的应用,测序深度和数据量的增加,以往手动比对鉴定的方法表现出明显的效率低、准确度差等问题,为了提高数据分析效率,大量的数据处理的软件应运而生。其中应用于metabarcoding测序数据分析的常用软件主要包括: OBITools[63]和DAMe[64]等;而用于shotgun metagenomic数据分析的软件主要包括: Holi pipeline[24]、Kraken pipeline[65]和Centrifuge pipeline[66]等。由于与本文主题无关具体操作流程及使用在这里不做详细介绍。
(3) DNA损伤评估。分析方法的进步也使得古环境DNA的判别成为可能。DNA损伤评估是确定古DNA的有效方法。古DNA一般具有DNA片段化、碱基丢失、交联和错配损伤等特征,其中错配损伤在古环境DNA的检测数据中表现的尤为明显,并且经常表现为高度的碱基胞嘧啶到胸腺嘧啶的替换[22]。而通过DNA损伤模型(DNA damage 2.0)可以良好地揭示古DNA的损伤特征,进而将其与现代DNA相区别[8, 67]。
(4) 污染排除。外源污染排除是古环境DNA研究的重要方面。为了排除外源污染,在古环境DNA的实验过程中需要设置阳性对照来排除取样污染,设置提取空白对照来排除提取污染,设置文库空白对照来排除建库污染,设置层位对比样品来排除采样层位之间的迁移和淋溶等[24]。
(5) 古基因组重建。古基因组重建及进化史计算一般是在获取良好古环境DNA数据基础上进行的深入数据分析内容。要求古环境DNA数据包含足量的古代物种的遗传信息同时具备完整的现代物种参考基因组。如果满足以上两个条件可以从古环境DNA数据集中筛选出目标物种的古DNA序列并根据现代物种的全基因组数据进行补充进而重建古代物种的基因组[51]。
(6) 物种进化史计算。在获取古基因组的基础上,可以进一步对该物种的进化历史进行计算揭示其进化地位和进化分歧时间(进化分子钟)。其中比较有代表性的研究是Weyrich等[51]2017年对尼安德特人牙结石的研究,该研究通过牙结石中的古环境DNA恢复了古细菌Methanobrevibacter oralis subsp. neandertalensis的古基因组同时完成了对其进化分子钟的计算。
3 古环境DNA在环境考古学中的应用随着古环境DNA实验与分析技术的完善,古环境DNA从最初的发现古代生物物种开始逐渐应用到揭示古生态系统组成、人类农业活动特征、人类饮食结构与疾病传播和人类扩散路线等复杂的环境考古学研究领域中。古环境DNA正逐渐拓展其在环境考古学领域的研究范围,并向着环境考古学领域的主要研究手段发展。
3.1 古生态环境重建通过古环境DNA来恢复古生态系统最早开始于Willerslev等[32]2003年对冻土和洞穴沉积物古环境DNA的研究。该研究对距今1万年到40万年的永久冻土和距今600~3000年间的洞穴沉积物进行了古环境DNA提取;通过DNA条形码技术对古植物的130 bp叶绿体rbcL(Rubisco大亚基)基因、脊椎动物的100 bp到280 bp mt DNA(线粒体DNA)16S、12S和cyt b(细胞色素b)基因进行扩增,揭示了西伯利亚动植物物种的变化。
2012年DNA宏条形码方法的出现[35, 68],使得古环境DNA可以进一步应用到无化石生物的鉴定中。Epp等[69]通过宏条形码的方法对来自挪威的湖泊沉积物样品进行了分析,实现了对古环境中多个生物类群的扩增,同时实现了对古环境样品中缺少化石的生物物种的鉴定。研究结果显示古环境样品中可以获取大量的真菌DNA,同时也可以发现微量的苔藓植物、甲虫和鸟类的DNA序列,进而证明了宏条形码技术在古生态学中的潜在应用价值[69]。
随着古环境DNA研究方法的进步,鸟枪法宏基因组技术的应用使获取样品中大部分古代生物遗传信息成为可能。Pederson等[24]通过鸟枪法宏基因组技术良好的恢复了12600年前Charlie和Spring湖泊区域古生态系统中的动植物物种组成。Slon等[26]则通过丹尼索瓦等6个欧洲洞穴沉积物的宏基因组研究重建了古生态系统中哺乳动物的组成,同时在其中没有人类骨骼的Trou Al'Wesse遗址和Chagyrskaya洞穴沉积物中发现了早期人类(尼安德特人和丹尼索瓦人)的古DNA。以上研究证明了古环境DNA在古生态环境重建及人类活动等研究中的重要作用[25]。
3.2 古代农业发展农业起源是人类社会发展的一个重要里程碑。农业物种(包括:家畜和农作物)多数在新旧石器时代过渡阶段被驯化,这些物种在考古遗址中留下大量遗传物质,而古环境DNA则可以通过这些遗传物质恢复古代农业物种的组成,为研究古代农业起源、家畜及作物的驯化历史和人类农业活动变化提供新的证据。Hebsgaard等[70]在2009年最早尝试使用古环境DNA的方法来重建考古遗址的古代人农业活动。研究通过使用DNA结合AMS 14C年代测定揭示了公元1030年到公元1530年间一座荒废的挪威农场的沉积物中的物种组成。研究发现牛和山羊的遗传物质比例随时间发生明显的波动,并且基因含量随着农场的荒废明显减少。该研究显示古环境DNA的研究结果与传统的动物化石形态分析方法获得的结果呈现良好的对应关系,进而证明了古环境DNA在重建古代农业活动研究中的价值[70]。
在揭示农业物种传播方面,Smith等[71]进行了英国怀特岛的一处海底考古遗址文化层的古环境DNA分析,结合微地貌和微体化石的证据重建了该遗址被淹没之前人类生活时期的植物组成。分析表明遗址的生境植被由橡树和草本植物构成。研究结果也通过古环境DNA证明了该地区在距今8000年前就已经有小麦出现,该结果比在英国Hambledon山发现的碳化麦穗遗存早2000年[72];但遗址沉积物中并未发现种植小麦产生的花粉,因而研究人员认为这些DNA证据可能来自小麦贸易,且推测粮食贸易可能促进了北欧农业的开始和发展[71]。
古环境DNA也可以帮助研究家畜的驯化历程。Speller等[73]通过火鸡粪化石的古环境DNA研究揭示了美洲火鸡的驯化历程。对38个考古遗址(时间跨度从公元前200年到公元1800年)中的149块火鸡骨头和29块粪化石的线粒体DNA分析发现在与外界文明接触以前,美洲西南部存在一个不同于其他地区的火鸡驯养品种,并对美洲火鸡的驯化起到重要的作用。美洲西南部火鸡存在的明显基因瓶颈特征也反映了当时强烈的人工选择过程。该研究指出在北美与外来文化接触以前至少有两次火鸡驯化发生,并阐明了新大陆家畜育种实践的强度和复杂性[73]。
3.3 古代人类食性及健康民以食为天,对古代人类饮食结构的揭示可以帮助我们了解古代人类的生计模式、生活条件和对自然环境的适应特征。对古代人健康的研究则可以帮助我们了解古代人的饮食卫生、疾病特征和医疗条件。传统方法主要通过对古代人类牙结石中植硅体和淀粉粒的鉴定来确定古代人类食性[74~76],通过牙齿、骨骼的形态学及粪化石中的寄生虫卵来确定古代人的健康情况[77~80]。最近的研究证明通过牙结石和粪化石中的古环境DNA的研究也可以帮助我们准确的揭示古代人的食性和健康问题。
Weyrich等[51]通过对尼安德特人钙化牙菌斑(牙结石)标本中的古环境DNA的研究揭示了尼安德特人的饮食状况、生活条件和疾病特征。研究显示生活在不同区域的尼安德特人的饮食结构存在明显差别。在比利时的Spy洞,尼安德特人的主要食物是肉类,其中包括:长毛犀牛肉和野生绵羊肉,表现出明显的草原狩猎的生活特征。相比之下,来自西班牙El Sidron洞穴的尼安德特人的饮食成分则主要是蘑菇、松仁和苔藓,反映了当地尼安德特人森林采集的生活特征。对患有牙齿和肠胃疾病的El Sidron穴居人的牙结石的研究发现其食物中含有大量的药用植物基因,说明当时的尼安德特人可能通过食用草药来治疗胃肠及牙龈疾病。在粪便化石与人类健康状况和疾病传播方面,Leles等[48]采用直接从粪化石中提取的古DNA来诊断古代人的蛔虫病,使用细胞色素b基因为靶标灵敏的检测出欧洲粪化石中的寄生虫,从而确定当时古代人的健康状况;Søe等[81]对北欧和中东(公元前500年到公元1700年)古环境DNA研究则进一步揭示了土壤源的寄生虫和肉类来源寄生虫等疾病的传播。
人类饮食结构与疾病的变化与微生物的变化也存在明显的关系[80],传统方法很难发现古代人口腔中的微生物,而古环境DNA则可以在分析古代人食性的同时探索其口腔微生物的特征。研究人员对早期欧洲古代人的钙化牙菌斑(牙结石)的研究显示从狩猎采集到农耕,人类饮食结构的变化导致了古代人类口腔微生物向现代口腔微生物群的变化,同时微生物多样性明显降低[50];Warinner等[80]对从德国St. Petri教堂和Dalheim修道院获取的古代人牙结石的研究发现这些牙结石中含有大量的口腔致病菌和潜在的疾病指示基因,同时研究显示长期以来人类口腔中一直存在着大量的条件致病菌,这些致病菌与人体局部和系统性疾病都有关系,并隐藏着多种可能的抗生素耐药性基因。尽管在过去千年中人类的生活方式、卫生和饮食发生了重大变化,但宿主免疫活动和病原体毒性在牙周发病机制中仍然保持着长期作用。
3.4 人类扩散历史人类历史上发生过多次大规模的人口迁徙和扩散事件,通过不同的迁徙路径人类实现了世界范围的扩散,促进了人类文化的交流和传播[82~83]。已有研究显示冰期时亚洲人群通过白令陆桥进入美洲可能促进了美洲人群的形成与发展,因而对白令陆桥人类迁徙的研究显得格外重要[84]。古环境DNA研究在美洲人定居的历史研究中取得了不错的进展。
末次盛冰期,科迪勒拉冰盖和劳伦冰盖相连将白令陆桥(北美的西北部)与未冻结的北美分隔开来。在大约12600年前,劳伦冰盖消退后在科迪勒拉冰盖和劳伦冰盖之间形成了一条大约1500km长的走廊[24]。植物和动物是何时在这条走廊上定居的,这条走廊在生物学上是否适合人类迁徙等问题受到研究人员的广泛关注。Gilbert等[84]通过从粪化石中提取的线粒体DNA发现在13400年前人类就已经出现在俄勒冈中南部Paisley洞穴中,粪化石的年代比目前公认的最早的克洛维斯文化年代还要早1000年。文化层中的人工制品和粪化石等考古证据显示人类可能在14700年前就已通过白令陆桥到达美洲[84~85],但最近的研究推翻了该假设。该研究从劳伦冰盖退缩形成的Charlie和Spring湖泊沉积物中提取了花粉、大化石以及古环境DNA,结合AMS 14C定年进行分析发现第一批美国人,无论是克洛维斯人还是早期北美族群在12600年前都不可能通过这条路线进入美洲[24]。这主要是由于迁徙需要大量的食物,而古环境DNA的结果显示该通道在12600以前无法为人类利用该通道提供充足的食物来源来实现迁徙[24],但之后生态环境开始逐渐变好促进了人类通过该通道的迁徙活动。该研究采用古环境DNA的手段,通过重建古生态系统更加准确的还原了人类历史上发生的人口迁徙和扩散的生态环境条件,同时也为古人类扩散与环境变化的研究提供了新思路。
3.5 人类活动对环境影响古代人类活动对环境的影响是环境考古研究的主要内容之一[86]。以往的研究显示,人类活动对生物圈的物种组成、丰度、分布和遗传多样性有着重要的影响[41, 87~89]。尤其在新石器时代以来,人类活动对世界物种的分布影响更加明显[90~95]。人类活动不仅将生活区的大片森林变为牧场,荒原变为沃野,也用驯化的农作物取代了多样的植物,用驯养的家畜取代了多样的野生动物,并随着人类的扩散而传播进而产生对世界范围生物分布的影响[87]。
大量的古环境DNA的研究表明新石器时代的农业发展加速了自然物种向农业物种的替换[41, 88~89]。Pansu等[42]通过对湖泊沉积物的古环境DNA研究分析了过去6400年来阿尔卑斯山一个高海拔湖泊流域的植物群落的变化轨迹。研究显示植物群落的显著变异与土壤退化和畜牧活动有关——在大约4500年前的全新世晚期,以农牧结合为主的人类社会生计模式造成该地植被从高山森林和草本植物群落演变为荒野和牧场;Giguet-Covex等[88]对法国北部湖泊的沉积物古环境DNA的研究也显示古代人类畜牧业的发展加速了植被退化;Grealy等[89]对马达加斯加海岸考古遗址混合鱼骨骨粉的古DNA研究则显示古代人类捕鱼业的发展加速了当地鱼类资源的退化。
农牧业的发展促进了粮食产量的增加,充足的食物使得人类迁移和扩散的范围变得更大[90]。随着人类扩散,随之产生的生态问题也更加明显。以往的研究显示人类的扩散不仅会导致生物多样性的改变,疾病的传播,甚至导致大量物种的灭绝[91~95]。随着人类的扩散,许多与人类共生的物种被引入新的区域,这些共生物种往往携带着大量的致病菌。古DNA研究显示结核病、恰加斯病和黑死病等起源于细菌、昆虫和动物的疾病随着人口迁徙与流动出现了大范围的扩散与传播[91~93]。同时人类的迁徙也导致了大量物种的灭绝,研究显示波利尼西亚人殖民后,夏威夷岛出现大量的鸟类灭绝,生态系统结构出现巨大改变[94]。虽然目前还没有通过古环境DNA来直接反映人类入侵导致生物灭绝的研究,但对生物骨骼中的古DNA的相关研究已经取得了不错的进展。Waters等[95]对新西兰海岸生态系统中的古DNA研究显示人类活动驱动了新西兰大陆陆生和海洋大型动物的灭绝。伴随灭绝事件的发生和新西兰南部人口的下降,海洋大型动物开始出现明显的替换,生物多样性发生巨大改变[95]。
人类的活动从古至今都在通过生物圈来影响着整个环境,而这些影响从何时开始,是否在某一历史时期已经作用到了人类自身,古人又通过哪些方式来调整生存策略进而适应或改善周边的生态环境?在当今气候环境剧烈变化的背景下,这些问题都迫切的需要回答。而古环境DNA是揭示人类影响生态系统并作用于整个环境的关键指标。对古环境DNA的研究可以帮助我们了解人类活动对环境的影响,进而帮助我们提出更为有效的应对环境变化的策略。
4 总结与展望 4.1 古环境DNA技术的优势古环境DNA技术作为一种新兴的环境考古学研究技术在古代农业活动、饮食、疾病与扩散等环境考古学研究领域中广泛应用,与传统的动植物遗存、淀粉、植硅体和孢粉等方法相比,古环境DNA技术具有以下显著优势:
(1) 样品用量少,容易获取。以往对古环境中动植物的鉴定需要采集大量的样品来进行浮选及筛查以获取完整的动植物大遗存或淀粉颗粒,如:在陈亭亭等[96]甘青地区齐家文化时期农业结构的研究中,为了获取和鉴定植物大遗存,平均每份样品需要9L的采样量;杨晓燕等[97]关于中国古代淀粉研究的文章也表示受保存条件限制,古代淀粉粒研究经常无法获得足够数量的样本来进行分析。而采用古环境DNA技术的环境考古学研究只需要2~10g的古环境沉积物样品就可以完成对其中包含的古代物种的鉴定[32, 38]。
(2) 应用范围广,获得信息量大。相比于以往孢粉和植硅体等鉴定方法,古环境DNA可以获取更大的生物信息量。对比植物孢粉鉴定我们可以发现,虽然目前通过古环境DNA宏条形码方法检测获得古代植物物种数量仍不及孢粉,但古环境DNA获取的植物中仍然有超过21 %是采用孢粉方法无法检测的[98]。随着宏基因组方法的应用和物种基因数据库的完善,古环境DNA可以检测到的植物物种数量及分辨率得到了进一步提升[99]。而且相比于孢粉和植硅体等古植物的鉴定方法,古环境DNA技术还可以用于揭示脊椎动物、无脊椎动物、藻类和微生物等古代物种组成。
(3) 发展潜力巨大。随着提取技术的发展,宏基因组技术的进步和生物基因组数据库的逐渐完善,通过古环境DNA不仅可以揭示环境样品中物种组成,还可以恢复已灭绝物种的基因组[51]。以往的研究显示孢粉可以良好地保存植物遗传物质,随着DNA提取手段的进步,有望在不久的将来通过提取地层孢粉中的植物古DNA进而实现对孢粉古基因组的重建[100]。
(4) 经济高效。在获取等量的古环境样品中的物种信息时,古环境DNA方法通常比传统方法需要更少的人力及时间。随着实验的完善及高通量测序技术的应用,古环境DNA研究可以实现单人在几周内完成多个古环境样品全部实验操作和检测,并且在一次实验及检测中获得古环境样品中多数目标物种的信息[101]。
(5) 对实验人员要求低。传统的化石形态分析方法要求研究人员具备较高的化石分类和鉴定能力,古环境DNA技术则只需要实验人员掌握分子生物学的一般实验方法即可,后续的数据分析主要由生物信息分析人员完成[102]。
(6) 易于标准化。用计算机程序检测代替人工识别,使得获取的检测结果更容易进行标准化。传统方法主要依靠研究人员的专业素质和经验来获取物种鉴定数据,存在一定的人为误差,而通过计算机程序获得的物种鉴定结果,因处理方法一致从而消除了人为误差,获得的数据更加标准统一。
尽管古环境DNA技术存在许多优势,但以往的研究表明古环境DNA和其他诸如动植物遗存、淀粉、植硅体和孢粉等指标的研究是相互补充的,目前在环境考古学研究中古环境DNA还无法替代传统指标[102~104]。
4.2 古环境DNA技术存在的问题古环境DNA技术与传统遗存形态鉴定方法相比更具优势,但就目前的古环境DNA技术而言,仍存在一些问题与不足。
(1) 古环境DNA含量低,容易被污染。古环境样品中的DNA经过长期的降解只有少量能够保存在环境样品中。虽然目前可以通过空白对照、层位对比和DNA损伤模型分析相结合的方法来区分现代DNA和古代DNA,但如果受到污染,也会导致古环境DNA信号被掩盖,进而无法获得有效的检测结果[82]。沉积物的迁移和淋溶等过程也可能导致古环境DNA发生层位迁移,进而影响对古环境DNA来源的判别。目前主要通过选取无层位迁移的剖面,并采集多个层位的样品进行对比分析来排除存在迁移和淋溶作用的可能,但也因此增加了采样量和测序成本。
(2) 分析方法有待优化。古环境DNA表现出明显的片段化和交联特征,部分情况会使传统的宏条形码技术无法对其进行识别和扩增而导致物种信息丢失;古环境DNA的错配损伤也会导致测序过程中产生错误序列,从而降低对物种的分辨率[20]。目前新兴的鸟枪法宏基因组技术,虽然可以提高古环境DNA的分辨率,但获得的大部分基因信息来自细菌物种,只有不到1 %的动植物数据[51],造成大量测序数据的浪费;而且,海量数据也对后续的分析处理造成了巨大障碍。虽然结合探针富集的鸟枪法宏基因组技术可以高效地从古环境DNA中获取目标类群的古代生物遗传信息,但目前其在古环境DNA研究中的应用还主要集中在对哺乳动物线粒体DNA的富集上[26],其他方向的研究有待进一步拓展。
(3) 标准数据库有待完善。宏条形码和鸟枪宏基因组法对古环境DNA中物种的识别和鉴定主要依赖于标准数据库[102, 105],而目前古环境DNA应用的标准数据库的物种基因组数据还不完善,导致部分物种无法获得准确的比对和鉴定。
4.3 环境DNA技术的前景展望古环境DNA技术作为一种快速、高效的古环境物种检测方法,在环境考古学研究中潜力巨大。虽然目前在国际上古环境DNA研究技术趋于完善,并已经取得了一系列的研究进展,但在中国,该方向的研究还比较缺乏,相关工作亟待开展。在未来的研究中应该主要关注以下几个方面: 1)实现古生态系统的重建,建立古生态系统与古气候环境的关系;2)建立古代农业物种数据库,推进对古代农业物种的传播与驯化的研究;3)加强对考古遗址文化层的古环境DNA研究,增进对古代人类迁徙与演化的了解;4)深化古环境DNA与环境变化的研究,拓展对人类活动造成环境变化的认识。
致谢: 感谢审稿专家和编辑部杨美芳老师的宝贵意见,在此一并感谢!
| [1] |
Willerslev E, Cooper A. Ancient DNA[J]. Proceedings of the Royal Society B:Biological Sciences, 2004, 272(1558): 3-16. |
| [2] |
湖南医学院. 长沙马王堆一号汉墓古尸研究[M]. 北京: 文物出版社, 1980: 184-187. Hunan Medical University. Study on the Ancient Corpse of No.1 Han Tomb in Mawangdui, Changsha[M]. Beijing: Cultural Relics Publishing House, 1980: 184-187. |
| [3] |
Pääbo S. Preservation of DNA in ancient Egyptian mummies[J]. Journal of Archaeological Science, 1985, 12(6): 411-417. DOI:10.1016/0305-4403(85)90002-0 |
| [4] |
Green R E, Krause J, Ptak S E, et al. Analysis of one million base pairs of Neanderthal DNA[J]. Nature, 2006, 444(7117): 330-336. DOI:10.1038/nature05336 |
| [5] |
Krause J, Fu Q, Good J M, et al. The complete mitochondrial DNA genome of an unknown hominin from southern Siberia[J]. Nature, 2010, 464(7290): 894. DOI:10.1038/nature08976 |
| [6] |
Meyer M, Kircher M, Gansauge M T, et al. A high-coverage genome sequence from an archaic Denisovan individual[J]. Science, 2012, 338(6104): 222-226. DOI:10.1126/science.1224344 |
| [7] |
Raghavan M, Skoglund P, Graf K E, et al. Upper Palaeolithic Siberian genome reveals dual ancestry of native Americans[J]. Nature, 2014, 505(7481): 87-91. DOI:10.1038/nature12736 |
| [8] |
Miller W, Drautz D I, Ratan A, et al. Sequencing the nuclear genome of the extinct woolly mammoth[J]. Nature, 2008, 456(7220): 387-392. DOI:10.1038/nature07446 |
| [9] |
Ginolhac A, Rasmussen M, Gilbert M T P, et al. Map damage:Testing for damage patterns in ancient DNA sequences[J]. Bioinformatics, 2011, 27(15): 2153-2155. DOI:10.1093/bioinformatics/btr347 |
| [10] |
杨洪, 程安进, 杨群. 地质体中主要生物分子的研究方法及应用[J]. 地质论评, 1998(1): 44-51. Yang Hong, Cheng Anjin, Yang Qun. Biomolecules in geological record: Recovery and applications[J]. Geological Review, 1998(1): 44-51. DOI:10.3321/j.issn:0371-5736.1998.01.007 |
| [11] |
赖旭龙, 杨洪. 古代生物分子在第四纪研究中的应用[J]. 第四纪研究, 2003, 23(5): 457-470. Lai Xulong, Yang Hong. Ancient biomolecules and their applications in Quaternary sciences[J]. Quaternary Sciences, 2003, 23(5): 457-470. DOI:10.3321/j.issn:1001-7410.2003.05.001 |
| [12] |
周慧, 万成, 朱泓. 古人骨DNA的提取、扩增、测序与研究[J]. 北方文物, 2001(3): 8-11. Zhou Hui, Wan Cheng, Zhu Hong. Extraction, amplification and sequence of ancient DNA[J]. Northern Cultural Relics, 2001(3): 8-11. DOI:10.3969/j.issn.1001-0483.2001.03.002 |
| [13] |
崔银秋, 段然慧, 季朝能, 等. 交河故城古车师人的线粒体DNA分析[J]. 高等学校化学学报, 2002(8): 1510-1514. Cui Yinqiu, Duan Ranhui, Ji Chaoneng, et al. Analysis of mitochondrial DNA from the ancient ruins of Jiao-he[J]. Chemical Journal of Chinese Universities, 2002(8): 1510-1514. DOI:10.3321/j.issn:0251-0790.2002.08.010 |
| [14] |
Fu Q, Meyer M, Gao X, et al. DNA analysis of an early modern human from Tianyuan cave, China[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(6): 2223-2227. DOI:10.1073/pnas.1221359110 |
| [15] |
Yang M A, Gao X, Theunert C, et al. 40, 000-year-old individual from Asia provides insight into early population structure in Eurasia[J]. Current Biology, 2017, 27(20): 3202-3208. DOI:10.1016/j.cub.2017.09.030 |
| [16] |
Hajdinjak M, Fu Q, Hübner A, et al. Reconstructing the genetic history of late Neanderthals[J]. Nature, 2018, 555(7698): 652-656. DOI:10.1038/nature26151 |
| [17] |
Sheng G L, Barlow A, Cooper A, et al. Ancient DNA from giant panda(Ailuropoda melanoleuca)of south-western China reveals genetic diversity loss during the Holocene[J]. Genes, 2018, 9(4): 198. DOI:10.3390/genes9040198 |
| [18] |
Rawlence N J, Lowe D J, Wood J R, et al. Using palaeoenvironmental DNA to reconstruct past environments:Progress and prospects[J]. Journal of Quaternary Science, 2014, 29(7): 610-626. DOI:10.1002/jqs.2740 |
| [19] |
Overballe-Petersen S, Harms K, Orlando L A, et al. Bacterial natural transformation by highly fragmented and damaged DNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(49): 19860-19865. DOI:10.1073/pnas.1315278110 |
| [20] |
Pedersen M W, Overballe-Petersen S, Ermini L, et al. Ancient and modern environmental DNA[J]. Philosophical Transactions of the Royal Society B:Biological Sciences, 2015, 370(1660): 20130383. DOI:10.1098/rstb.2013.0383 |
| [21] |
Poinar H N, Hofreiter M, Spaulding W G, et al. Molecular coproscopy:Dung and diet of the extinct ground sloth Nothrotheriops shastensis[J]. Science, 1998, 281(5375): 402-406. DOI:10.1126/science.281.5375.402 |
| [22] |
Dabney J, Meyer M, Pääbo S. Ancient DNA damage[J]. Cold Spring Harbor Perspectives in Biology, 2013, 5(7): a012567. |
| [23] |
Willerslev E, Cappellini E, Boomsma W, et al. Ancient biomolecules from deep ice cores reveal a forested southern Greenland[J]. Science, 2007, 317(5834): 111-114. DOI:10.1126/science.1141758 |
| [24] |
Pedersen M W, Ruter A, Schweger C, et al. Postglacial viability and colonization in North America's ice-free corridor[J]. Nature, 2016, 537(7618): 45. DOI:10.1038/nature19085 |
| [25] |
Llamas B, Valverde G, Fehren-Schmitz L, et al. From the field to the laboratory:Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era[J]. STAR:Science & Technology of Archaeological Research, 2017, 3(1): 1-14. |
| [26] |
Slon V, Hopfe C, Weiß C L, et al. Neandertal and Denisovan DNA from Pleistocene sediments[J]. Science, 2017, 356(6338): 605-608. DOI:10.1126/science.aam9695 |
| [27] |
Matisoo-Smith E, Roberts K, Welikala N, et al. Recovery of DNA and pollen from New Zealand lake sediments[J]. Quaternary International, 2008, 184(1): 139-149. DOI:10.1016/j.quaint.2007.09.013 |
| [28] |
Haile J, Froese D G, Macphee R D, et al. Ancient DNA reveals late survival of mammoth and horse in interior Alaska[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(52): 22352-22357. DOI:10.1073/pnas.0912510106 |
| [29] |
柳天雄, 罗佳, 黄菊芳, 等. 古DNA提取技术新进展[J]. 现代生物医学进展, 2014, 14(26): 5170-5175. Liu Tianxiong, Luo Jia, Huang Jufang, et al. Progress in ancient DNA extraction technology[J]. Progress in Modern Biomedicine, 2014, 14(26): 5170-5175. |
| [30] |
Anderson-Carpenter L L, Mclachlan J S, Jackson S T, et al. Ancient DNA from lake sediments:Bridging the gap between paleoecology and genetics[J]. BMC Evolutionary Biology, 2011, 11(1): 30. |
| [31] |
Wales N, Andersen K, Cappellini E, et al. Optimization of DNA recovery and amplification from non-carbonized archaeobotanical remains[J]. PLoS One, 2014, 9(1): 1-14. |
| [32] |
Willerslev E, Hansen A J, Binladen J, et al. Diverse plant and animal genetic records from Holocene and Pleistocene sediments[J]. Science, 2003, 300(5620): 791-795. DOI:10.1126/science.1084114 |
| [33] |
Valentini A, Pompanon F, Taberlet P. DNA barcoding for ecologists[J]. Trends in Ecology & Evolution, 2009, 24(2): 110-117. |
| [34] |
Thomsen P F, Willerslev E. Environmental DNA—An emerging tool in conservation for monitoring past and present biodiversity[J]. Biological Conservation, 2015, 183: 4-18. DOI:10.1016/j.biocon.2014.11.019 |
| [35] |
Taberlet P, Coissac E, Pompanon F, et al. Towards next-generation biodiversity assessment using DNA metabarcoding[J]. Molecular Ecology, 2012, 21(8): 2045-2050. DOI:10.1111/j.1365-294X.2012.05470.x |
| [36] |
Key F M, Posth C, Krause J, et al. Mining metagenomic data sets for ancient DNA:Recommended protocols for authentication[J]. Trends in Genetics, 2017, 33(8): 508-520. DOI:10.1016/j.tig.2017.05.005 |
| [37] |
Jørgensen T, Haile J, Möller P, et al. A comparative study of ancient sedimentary DNA, pollen and macrofossils from permafrost sediments of northern Siberia reveals long-term vegetational stability[J]. Molecular Ecology, 2012, 21(8): 1989-2003. DOI:10.1111/j.1365-294X.2011.05287.x |
| [38] |
Willerslev E, Davison J, Moora M, et al. Fifty thousand years of Arctic vegetation and megafaunal diet[J]. Nature, 2014, 506(7486): 47-51. DOI:10.1038/nature12921 |
| [39] |
Parducci L, Jørgensen T, Tollefsrud M M, et al. Glacial survival of boreal trees in Northern Scandinavia[J]. Science, 2012, 335(6072): 1083-1086. DOI:10.1126/science.1216043 |
| [40] |
Giguet-Covex C, Pansu J, Arnaud F, et al. Long livestock farming history and human landscape shaping revealed by lake sediment DNA[J]. Nature Communications, 2014, 5: 3211-3217. DOI:10.1038/ncomms4211 |
| [41] |
Epp L S, Gussarova G, Boessenkool S, et al. Lake sediment multi-taxon DNA from North Greenland records early post-glacial appearance of vascular plants and accurately tracks environmental changes[J]. Quaternary Science Reviews, 2015, 117: 152-163. DOI:10.1016/j.quascirev.2015.03.027 |
| [42] |
Pansu J, Giguet-Covex C, Ficetola G F, et al. Reconstructing long-term human impacts on plant communities:An ecological approach based on lake sediment DNA[J]. Molecular Ecology, 2015, 24(7): 1485-1498. DOI:10.1111/mec.13136 |
| [43] |
Alsos I G, Sjögren P, Edwards M E, et al. Sedimentary ancient DNA from Lake Skartjørna, Svalbard:Assessing the resilience of arctic flora to Holocene climate change[J]. The Holocene, 2016, 26(4): 627-642. DOI:10.1177/0959683615612563 |
| [44] |
Parducci L, Nota K, Wood J. Reconstructing Past Vegetation Communities Using Ancient DNA from Lake Sediments[M]. Cham, Switzerland: Springer, 2018: 163-187.
|
| [45] |
Willerslev E, Hansen A J, Christensen B, et al. Diversity of Holocene life forms in fossil glacier ice[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(14): 8017-8021. DOI:10.1073/pnas.96.14.8017 |
| [46] |
Willerslev E, Hansen A J, Poinar H N. Isolation of nucleic acids and cultures from fossil ice and permafrost[J]. Trends in Ecology & Evolution, 2004, 19(3): 141-147. |
| [47] |
Gould B A, León B, Buffen A M, et al. Evidence of a high-Andean, mid-Holocene plant community:An ancient DNA analysis of glacially preserved remains[J]. American Journal of Botany, 2010, 97(9): 1579-1584. DOI:10.3732/ajb.1000058 |
| [48] |
Leles D, Araújo A, Ferreira L F, et al. Molecular paleoparasitological diagnosis of Ascaris sp. from coprolites:New scenery of ascariasis in pre-Colombian South America times[J]. Memórias do Instituto Oswaldo Cruz, 2008, 103(1): 106-108. DOI:10.1590/S0074-02762008005000004 |
| [49] |
Tito R Y, Knights D, Metcalf J, et al. Insights from characterizing extinct human gut microbiomes[J]. PLoS One, 2012, 7(12): e51146. DOI:10.1371/journal.pone.0051146 |
| [50] |
Adler C J, Dobney K, Weyrich L S, et al. Sequencing ancient calcified dental plaque shows changes in oral microbiota with dietary shifts of the Neolithic and Industrial revolutions[J]. Nature Genetics, 2013, 45(4): 450-455. DOI:10.1038/ng.2536 |
| [51] |
Weyrich L S, Duchene S, Soubrier J, et al. Neanderthal behaviour, diet, and disease inferred from ancient DNA in dental calculus[J]. Nature, 2017, 544(7650): 357-362. DOI:10.1038/nature21674 |
| [52] |
Hagelberg E, Hofreiter M, Keyser C. Ancient DNA:The first three decades[J]. Philosophical Transactions of the Royal Society B:Biological Sciences, 2015, 370: 20130371. DOI:10.1098/rstb.2013.0371 |
| [53] |
Marshall E. Will DNA bar codes breathe life into classification?[J]. Science, 2005, 307(5712): 1037. DOI:10.1126/science.307.5712.1037 |
| [54] |
Taylor H, Harris W. An emergent science on the brink of irrelevance:A review of the past 8 years of DNA barcoding[J]. Molecular Ecology Resources, 2012, 12(3): 377-388. DOI:10.1111/j.1755-0998.2012.03119.x |
| [55] |
Seersholm F V, Cole T L, Grealy A, et al. Subsistence practices, past biodiversity, and anthropogenic impacts revealed by New Zealand-wide ancient DNA survey[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(30): 7771-7776. DOI:10.1073/pnas.1803573115 |
| [56] |
Murray D C, Haile J, Dortch J, et al. Scrapheap challenge:A novel bulk-bone metabarcoding method to investigate ancient DNA in faunal assemblages[J]. Scientific Reports, 2013, 3: 3371. DOI:10.1038/srep03371 |
| [57] |
Furlan E M, Gleeson D, Hardy C M, et al. A framework for estimating the sensitivity of eDNA surveys[J]. Molecular Ecology Resources, 2016, 16(3): 641-654. DOI:10.1111/1755-0998.12483 |
| [58] |
Ficetola G F, Coissac E, Zundel S, et al. An in silico approach for the evaluation of DNA barcodes[J]. BMC Genomics, 2010, 11(1): 434-444. DOI:10.1186/1471-2164-11-434 |
| [59] |
林佳伟, 赵静, 宋晓峰. 宏基因组技术在油藏微生物分析中的应用[J]. 计算机与应用化学, 2018, 35(7): 563-572. Lin Jiawei, Zhao Jing, Song Xiaofeng. Application of metagenomic sequencing in reservoir microbial analysis[J]. Computers and Applied Chemistry, 2018, 35(7): 563-572. |
| [60] |
Quince C, Walker A W, Simpson J T, et al. Shotgun metagenomics, from sampling to analysis[J]. Nature Biotechnology, 2017, 35(9): 833-844. DOI:10.1038/nbt.3935 |
| [61] |
Sønstebø J, Gielly L, Brysting A, et al. Using next-generation sequencing for molecular reconstruction of past Arctic vegetation and climate[J]. Molecular Ecology Resources, 2010, 10(6): 1009-1018. DOI:10.1111/j.1755-0998.2010.02855.x |
| [62] |
Boessenkool S, Epp L S, Haile J, et al. Blocking human contaminant DNA during PCR allows amplification of rare mammal species from sedimentary ancient DNA[J]. Molecular Ecology, 2012, 21(8): 1806-1815. DOI:10.1111/j.1365-294X.2011.05306.x |
| [63] |
Boyer F, Mercier C, Bonin A, et al. Obitools:A unix-inspired software package for DNA metabarcoding[J]. Molecular Ecology Resources, 2016, 16(1): 176-182. DOI:10.1111/1755-0998.12428 |
| [64] |
Zepeda-Mendoza M L, Bohmann K, Baez A C, et al. DAMe:A toolkit for the initial processing of datasets with PCR replicates of double-tagged amplicons for DNA metabarcoding analyses[J]. BMC Research Notes, 2016, 9(1): 255. DOI:10.1186/s13104-016-2064-9 |
| [65] |
Wood D E, Salzberg S L. Kraken: ultrafast metagenomic sequence classification using exact alignments[J]. Genome Biology, 2014, 15(3): R46. DOI:10.1186/gb-2014-15-3-r46 |
| [66] |
Kim D, Song L, Breitwieser F P, et al. Centrifuge:Rapid and sensitive classification of metagenomic sequences[J]. Genome Research, 2016, 26(12): 1721-1729. DOI:10.1101/gr.210641.116 |
| [67] |
Jónsson H, Ginolhac A, Schubert M, et al. mapDamage2.0:Fast approximate Bayesian estimates of ancient DNA damage parameters[J]. Bioinformatics, 2013, 29(13): 1682-1684. DOI:10.1093/bioinformatics/btt193 |
| [68] |
Yu D, Ji Y, Emerson B, et al. Biodiversity soup:Metabarcoding of arthropods for rapid biodiversity assessment and biomonitoring[J]. Methods in Ecology and Evolution, 2012, 3(4): 613-623. DOI:10.1111/j.2041-210X.2012.00198.x |
| [69] |
Epp L S, Boessenkool S, Bellemain E P, et al. New environmental metabarcodes for analysing soil DNA:Potential for studying past and present ecosystems[J]. Molecular Ecology, 2012, 21(8): 1821-1833. DOI:10.1111/j.1365-294X.2012.05537.x |
| [70] |
Hebsgaard M B, Gilbert M T P, Arneborg J, et al. 'The Farm Beneath the Sand'—An archaeological case study on ancient 'dirt'DNA[J]. Antiquity, 2009, 83(320): 430-444. DOI:10.1017/S0003598X00098537 |
| [71] |
Smith O, Momber G, Bates R, et al. Sedimentary DNA from a submerged site reveals wheat in the British Isles 8000 years ago[J]. Science, 2015, 347(6225): 998-1001. DOI:10.1126/science.1261278 |
| [72] |
Stevens C J, Fuller D Q. Did Neolithic farming fail?The case for a Bronze Age agricultural revolution in the British Isles[J]. Antiquity, 2012, 86(333): 707-722. DOI:10.1017/S0003598X00047864 |
| [73] |
Speller C F, Kemp B M, Wyatt S D, et al. Ancient mitochondrial DNA analysis reveals complexity of indigenous North American turkey domestication[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(7): 2807-2812. DOI:10.1073/pnas.0909724107 |
| [74] |
Piperno D R, Dillehay T D. Starch grains on human teeth reveal early broad crop diet in Northern Peru[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(50): 19622-19627. DOI:10.1073/pnas.0808752105 |
| [75] |
Hardy K, Blakeney T, Copeland L, et al. Starch granules, dental calculus and new perspectives on ancient diet[J]. Journal of Archaeological Science, 2009, 36(2): 248-255. DOI:10.1016/j.jas.2008.09.015 |
| [76] |
Henry A G, Brooks A S, Piperno D R. Microfossils in calculus demonstrate consumption of plants and cooked foods in Neanderthal diets(Shanidar Ⅲ, Iraq; Spy Ⅰ and Ⅱ, Belgium)[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(2): 486-491. DOI:10.1073/pnas.1016868108 |
| [77] |
Forshaw R. Dental health and disease in ancient Egypt[J]. British Dental Journal, 2009, 206(8): 421-425. DOI:10.1038/sj.bdj.2009.309 |
| [78] |
Zias J. Current archaeological research in Israel:Death and disease in ancient Israel[J]. The Biblical Archaeologist, 1991, 54(3): 147-159. DOI:10.2307/3210263 |
| [79] |
Shin D H, Oh C S, Chung T, et al. Detection of parasite eggs from a moat encircling the royal palace of Silla, the ancient Korean Kingdom[J]. Journal of Archaeological Science, 2009, 36(11): 2534-2539. DOI:10.1016/j.jas.2009.07.009 |
| [80] |
Warinner C, Rodrigues J F M, Vyas R, et al. Pathogens and host immunity in the ancient human oral cavity[J]. Nature Genetics, 2014, 46(4): 336-344. DOI:10.1038/ng.2906 |
| [81] |
Søe M J, Nejsum P, Seersholm F V, et al. Ancient DNA from latrines in Northern Europe and the Middle East(500 BC-1700 AD)reveals past parasites and diet[J]. PLoS One, 2018, 13(4): e0195481. DOI:10.1371/journal.pone.0195481 |
| [82] |
Diamond J, Bellwood P. Farmers and their languages:The first expansions[J]. Science, 2003, 300(5619): 597-603. DOI:10.1126/science.1078208 |
| [83] |
Timmermann A, Friedrich T. Late Pleistocene climate drivers of early human migration[J]. Nature, 2016, 538(7623): 92-95. DOI:10.1038/nature19365 |
| [84] |
Gilbert M T P, Jenkins D L, Götherstrom A, et al. DNA from pre-Clovis human coprolites in Oregon, North America[J]. Science, 2008, 320(5877): 786-789. DOI:10.1126/science.1154116 |
| [85] |
Dillehay T D, Ramírez C, Pino M, et al. Monte Verde: Seaweed, food, medicine, and the peopling of South America[J]. Science, 2008, 320(5877): 784-786. DOI:10.1126/science.1156533 |
| [86] |
夏正楷, 张俊娜. 中国环境考古学的兴起、发展和展望[J]. 古地理学报, 2019, 21(1): 175-188. Xia Zhengkai, Zhang Junna. The rise, development and outlook of Huanjingkaogu(environmental archaeology in China)[J]. Journal of Palaeogeography, 2019, 21(1): 175-188. |
| [87] |
Boivin N L, Zeder M A, Fuller D Q, et al. Ecological consequences of human niche construction:Examining long-term anthropogenic shaping of global species distributions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(23): 6388-6396. DOI:10.1073/pnas.1525200113 |
| [88] |
Giguet-Covex C, Pansu J, Arnaud F, et al. Long livestock farming history and human landscape shaping revealed by lake sediment DNA[J]. Nature communications, 2014, 5: 3211. DOI:10.1038/ncomms4211 |
| [89] |
Grealy A, Douglass K, Haile J, et al. Tropical ancient DNA from bulk archaeological fish bone reveals the subsistence practices of a historic coastal community in Southwest Madagascar[J]. Journal of Archaeological Science, 2016, 75: 82-88. DOI:10.1016/j.jas.2016.10.001 |
| [90] |
张山佳, 董广辉. 青藏高原东北部青铜时代中晚期人类对不同海拔环境的适应策略探讨[J]. 第四纪研究, 2017, 37(4): 696-708. Zhang Shanjia, Dong Guanghui. Human adaptation strategies to different altitude environment during mid-late Bronze Age in northeast Tibetan Plateau[J]. Quaternary Sciences, 2017, 37(4): 696-708. |
| [91] |
Guhl F, Jaramillo C, Vallejo G A, et al. Chagas disease and human migration[J]. Memórias do Instituto Oswaldo Cruz, 2000, 95(4): 553-555. DOI:10.1590/S0074-02762000000400018 |
| [92] |
Spyrou M A, Tukhbatova R I, Wang C C, et al. Analysis of 3800-year-old Yersinia pestis genomes suggests Bronze Age origin for bubonic plague[J]. Nature Communications, 2018, 9(1): 2234. DOI:10.1038/s41467-018-04550-9 |
| [93] |
Comas I, Coscolla M, Luo T, et al. Out-of-Africa migration and Neolithic coexpansion of Mycobacterium tuberculosis with modern humans[J]. Nature Genetics, 2013, 45(10): 1176. DOI:10.1038/ng.2744 |
| [94] |
Athens J S, Toggle H D, Ward J V, et al. Avifaunal extinctions, vegetation change, and Polynesian impacts in prehistoric Hawai'i[J]. Archaeology in Oceania, 2002, 37(2): 57-78. DOI:10.1002/j.1834-4453.2002.tb00507.x |
| [95] |
Waters J M, Fraser C I, Maxwell J J, et al. Did interaction between human pressure and Little Ice Age drive biological turnover in New Zealand?[J]. Journal of Biogeography, 2017, 44(7): 1481-1490. DOI:10.1111/jbi.12991 |
| [96] |
陈亭亭, 贾鑫, 黎海明, 等. 甘青地区齐家文化时期农业结构的时空变化及其影响因素分析[J]. 第四纪研究, 2019, 39(1): 132-143. Chen Tingting, Jia Xin, Li Haiming, et al. The analysis of spatiotemporal transformations of agricultural and its influence factors during Qijia culture period in Gansu-Qinghai region[J]. Quaternary Sciences, 2019, 39(1): 132-143. |
| [97] |
杨晓燕. 中国古代淀粉研究:进展与问题[J]. 第四纪研究, 2017, 37(1): 196-210. Yang Xiaoyan. Ancient starch research in China:Progress and problems[J]. Quaternary Sciences, 2017, 37(1): 196-210. |
| [98] |
Wood J R, Wilmshurst J M, Wagstaff S J, et al. High-resolution coproecology:Using coprolites to reconstruct the habits and habitats of New Zealand's extinct upland moa(Megalapteryx didinus)[J]. PLoS One, 2012, 7(6): e40025. DOI:10.1371/journal.pone.0040025 |
| [99] |
Wood J R, Wilmshurst J M, Richardson S J, et al. Resolving lost herbivore community structure using coprolites of four sympatric moa species(Aves:Dinornithiformes)[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(42): 16910-16915. DOI:10.1073/pnas.1307700110 |
| [100] |
Parducci L, Bennett K D, Ficetola G F, et al. Ancient plant DNA in lake sediments[J]. New Phytologist, 2017, 214(3): 924-942. DOI:10.1111/nph.14470 |
| [101] |
Pont C, Wagner S, Kremer A, et al. Paleogenomics:Reconstruction of plant evolutionary trajectories from modern and ancient DNA[J]. Genome Biology, 2019, 20(1): 20-29. DOI:10.1186/s13059-019-1628-0 |
| [102] |
Harbert R S. Algorithms and strategies in short-read shotgun metagenomic reconstruction of plant communities[J]. Applications in Plant Sciences, 2018, 6(3): e1034. DOI:10.1002/aps3.1034 |
| [103] |
Parducci L, Matetovici I, Fontana S L, et al. Molecular-and pollen-based vegetation analysis in lake sediments from Central Scandinavia[J]. Molecular Ecology, 2013, 22(13): 3511-3524. DOI:10.1111/mec.12298 |
| [104] |
Pedersen M W, Ginolhac A, Orlando L, et al. A comparative study of ancient environmental DNA to pollen and macrofossils from lake sediments reveals taxonomic overlap and additional plant taxa[J]. Quaternary Science Reviews, 2013, 75: 161-168. DOI:10.1016/j.quascirev.2013.06.006 |
| [105] |
唐敏, 伊廷双, 王欣, 等. Metabarcoding技术在植物鉴定和多样性研究中的应用[J]. 植物分类与资源学报, 2013, 35(6): 769-773. Tang Min, Yin Tingshuang, Wang Xin, et al. The application of metabarcoding technology in identification of plant species diversity[J]. Plant Diversity and Resources, 2013, 35(6): 769-773. |
2 University of Chinese Academy of Science, Beijing 100049;
3 The Globe Institute, University of Copenhagen, Copenhagen, Denmark 1350;
4 CAS Center for Excellence in Tibetan Plateau Earth Sciences, Chinese Academy of Sciences, Beijing 100101;
5 Key Laboratory of Western China's Environment Systems, Ministry of Education, College of Earth and Environmental Sciences, Lanzhou University, Lanzhou 730000, Gansu)
Abstract
Ancient environmental DNA(ancient eDNA)refers to ancient DNA(aDNA)derived from palaeoenvironmental samples. Different from ancient DNA, ancient eDNA contains a mixture of highly fragmented DNA from multiple organisms, and usually attached to humic acids and mineral particles. Ancient eDNA has been found to be preserved in coprolites, dental calculus, gut contents, ice, permafrost, and the sediments from lake, marine, cave and archaeological sites. Ancient eDNA technology started in 1998, and has experienced three stages of development:(1)identifying specific species through DNA barcodes, (2)recovery of certain groups of organisms via DNA metabarcoding, and (3) reconstruction of paleoecosystems using shotgun metagenomics. Compared to the fossil identification methods, the ancient eDNA methods have several advantages such as:less sample consumption, easier operation, no demand for fossils, and more species information revealed from one sample. Therefore, the ancient eDNA methods have been widely applied in many fields of environmental archaeology, including palaeoecological reconstruction, ancient human diet, ancient agricultural development, ancient human migrations, as well as anthropogenic environmental change. In this paper, we review the development, research methods, application directions and existing problems of ancient eDNA technology. We conclude that with the improvement of ancient eDNA technology, its application in environmental archaeology would be more promising. 2020, Vol.40
2020, Vol.40

