近年来,高通量测序已经成为检测基因组结构变异的主要技术手段。尽管像单核苷酸多态性(SNP)和小的插入缺失(indels)这些碱基水平的变异很普遍,但是诸如缺失、插入、重复和翻转等大的结构变异会影响更多的基因序列,多达15%的人类基因都处在拷贝数变异区域[1]。多项研究表明[2],基因插入结构变异与人类表现的特征有关,包括癌症、精神失常、代谢失常和各种不治之症。例如基因NRXN1的杂合变异与自闭症以及精神分裂有关,而先天性心脏缺陷与22q11.2基因区域的缺失密切相关[3]。二代测序技术是迈向理解人类基因结构的重要一步,它解释了基因变异和疾病之间的关系,但是这种解释依赖于精确检测个体基因和参考基因差异的能力。因此,全基因组范围的结构变异检测的精度和敏感度至关重要。
随着测序技术的持续发展以及测序费用的降低,基于二代测序的结构变异检测方法快速发展,主要有双末端比对、分裂片段比对、映射深度以及序列拼接4类。常用的检测工具BreakDancer[4]和VariationHunter[5]等基于双末端比对策略开发,该策略的基本思想是:如果双末端比对的两端距离比平均插入距离小则存在插入变异。另一个著名的检测工具Pindel[6]应用分裂比对策略,其中测序片段对的一端被作为一个锚单独地比对到参考基因上,而后另一端再进行比对,从而能够检测插入变异断点。但上述两种方法要求插入变异完全包含在一个片段(reads)中,而且在最初比对时能够比对正确,所以这类方法用于检测小的插入很有效,但检测比reads长的插入变异时会失败。对于长插入变异来说,这种变异的reads常常因包含能够比对到参考基因的碱基太少,或者因reads只有一部分能够比对到参考基因,另一端在用二代测序比对工具时被剪切掉了,从而导致检测失败[7]。从头拼接已经被用来检测大于片段长度的插入,例如GATK HaplotypeCall、Platypus[8]及Scalpel工具[9]采取局部或微拼接,而FermiKit工具[10]采用全基因组拼接来检测变异。尽管从头拼接理论上能够识别任何尺寸的插入,但是较高的计算成本和内存开销使得这种方法很难应用于实际检测中。
关于插入变异的检测一直以来都是研究的难点,常规的双末端比对和分裂片段检测方法对于插入的检测效果差强人意。插入变异的检测软件有多种,但是多数软件只能检测相对较短的插入序列,而且假阳性变异出现频率较高。将拼接应用到插入检测也有先例,然而使用拼接方法面临的一个重大挑战是,拼接所需的大量计算资源导致拼接效率和结果不理想,进而影响最终的检测效果。基于此,本文提出了一种基于序列拼接的长插入变异(ISALins)检测方法,该方法主要针对长插入变异事件,首先通过融合多个工具的检测结果以达到较好的检测敏感度,然后通过软切片段的分析来保证后续拼接工作的准确性,进而最终实现对长插入变异的有效检测。
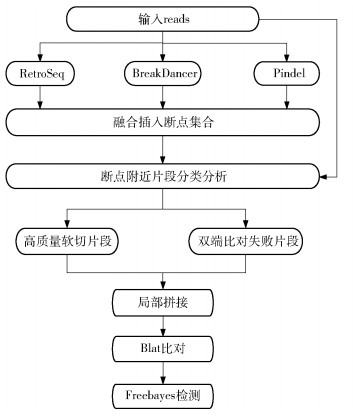
1 插入变异检测方法 1.1 工作流程ISALins方法的检测流程如下:1)将原始测序reads比对到参考基因组,使用多个工具(RetroSeq、BreakDancer、Pindel)检测插入变异断点,再通过一定的断点合并原则得到来自于多个检测工具的插入断点集合;2)在断点集合中每个可疑断点附近对包含插入变异信息的片段进行聚类,包括高质量软切片段和比对失败的片段,并将其作为进一步分析插入变异事件的有利特征;3)使用基于De Bruijn (DB)图的局部拼接技术拼接得到重叠群片段(contigs);4)将拼接得到的contigs比对到参考基因进行长插入的检测。ISALins方法的工作流程如图 1所示。
|
图 1 ISALins的工作流程 Fig.1 The workflow for ISALins |
为了得到更多可疑的断点以便在确保敏感度的同时最大化检测精度,本文使用3种高效的检测工具BreakDancer、Pindel、RetroSeq来获得一个融合的插入变异断点集合。3种工具的检测方法如表 1所示。由于3种工具使用的方法不同,各自的限制因素和检测插入变异的结果也各不相同,本文通过融合3种工具的检测结果可以得到更多的可疑结构变异断点。首先将每个工具的检测结果按照结构变异类型、变异位置以及变异尺寸大小筛选,然后设定一定范围的变异断点进行合并融合,得到一个尽可能少冗余的结构变异初始集合,并作为一个候选SV断点集合。例如,先比较所有来自BreakDancer、Pindel、RetroSeq工具检测的插入变异,如果BreakDancer的插入变异与Pindel的插入变异有重叠,则将该检测结果合并融合,再与RetroSeq的检测结果进行比较来融合其余没有被BreakDancer和Pindel检测到的插入。
| 下载CSV 表 1 不同检测工具方法对比 Table 1 Comparison of different detection tools |
通过将个体基因比对到参考基因上,得到一个保存了比对结果的bam文件。根据比对结果将这些片段分为以下4类[11]。
1) one end anchor(OEA)类。reads片段的一端比对到参考基因组,另一端没有比对到参考基因组;软切片段(soft-clipped reads)也是OEA的一种。
2) Orphan类。reads片段的两端均未比对到参考基因上的片段。
3) Concordant类。reads片段的两端均成功比对到参考基因,比对参考基因的两个位点在[m-3v, m+3v]范围内(m为插入距离,v为标准差);并且两端映射的方向相对,即一端正向另一端反向。
4) Discordant类。reads片段的两端均能成功比对到参考基因,但存在映射距离不一致的情况。
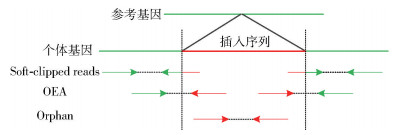
分析以上4类比对结果可知,对于插入变异事件,其碱基信息只可能分布在OEA片段和Orphan片段中,因此主要选择这两类片段信息,其插入片段匹配示意图如图 2所示。因测序序列中可能存在错误的碱基或者人工数据,必须筛选出软切片段来提高检测精度。软切片段是在bam文件中CIGAR[12]字符属性有‘S’标记的片段,其中的高质量软切片段按以下标准定义:①在bam文件中通过MAPQ计算的片段映射质量大于用户指定值;②软切部分的比例大于一端长度的20%;③软切碱基中高质量片段比例大于80%。基于以上过滤条件,排除了因测序质量差而模糊比对被软切的片段,只保留可能包含插入变异的长软切片段。
|
图 2 插入片段匹配示意图 Fig.2 A schematic view of the mapping of insertion reads |
在每个可疑插入位点的上游及下游1 000 bp范围内,从bam文件中提取OEA和Orphan片段,并以片段对的形式保存在fasta文件中,作为序列拼接的输入。因拼接基于DB图方法,在构造DB图时基因序列中存在的重复元素会导致拼接结果出现错误序列。然而重复序列在人类基因组中大量存在,约占基因组的50%,分为简单重复、串联重复、片段重复、分散重复等不同类型,因此为了确保拼接过程得到正确的contigs,有必要在拼接之前屏蔽重复序列。
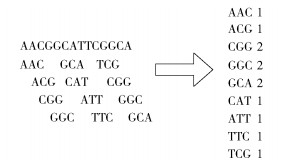
目前常用的重复元素标记工具(如MarkDuplicates)只能标记重复基因库中已知的重复序列,很多未知的重复序列无法处理,为此,本文使用一个基于k-mer频率分析方法来尽可能多地找出重复序列[13]。k-mer是reads序列中长度为k的子字符串序列,在构造DB图之前,输入的reads序列即被随机截取成很多大小为k的k-mer。对于非重复的序列,其k-mer的出现频率等于测序序列的覆盖度,设为x;重复序列的k-mer出现频率会大于等于2x。k-mer频率分析原理如图 3所示。因此可根据k-mer统计频率的分析来标记重复序列,从而保证后续拼接的准确性。
|
图 3 k-mer频率分析示意图 Fig.3 A diagram of k-mer frequency analysis |
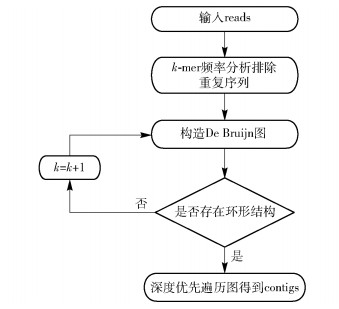
但是频率分析也存在一定的随机性,并不能排除所有的重复序列,所以本文使用一个k值大小变化的k-mer来构造DB图,使k值从一个较小值逐渐变大,同时检测每一个k值构造的DB图中是否包含环形结构,环形结构即为重复序列。若检测到环形结构,则销毁此时的DB图并重新构造;重复这一过程直到新构造的DB图中无环结构或者k值达到某个设定的阈值,停止迭代;最后通过深度优先遍历算法遍历整个图得到拼接的contigs。完整的拼接工作流程如图 4所示。最终只输出拼接之后长度至少比原read长一个碱基的contigs片段。
|
图 4 拼接工作流程 Fig.4 The workflow for assembly |
将局部拼接获得的contigs比对回参考基因的过程为:首先使用bwa-mem软件进行最初的比对,然后使用blat软件[14]将有断点支持的软切片段contigs进行二次比对,来提纯bam文件中的CIGAR字符串。ISALins方法会产生一个bam文件,该文件包含了拼接之后片段比对的所有信息。将初始bam文件排序并建立索引,之后传递给FreeBayes检测工具[15]进行最终的插入变异检测,FreeBayes可以检测出单碱基水平的变异,输出一个变异数据格式(variant call format, VCF)文件。
2 实验验证及结果分析 2.1 评价指标本文选择检测精度(Pre)和检测敏感度(Sen)来评判检测性能,两个指标互为制约,以达到综合平衡为佳。具体公式如下
| $ Pre = \frac{{{C_{{\rm{TP}}}}}}{{{C_{{\rm{TP}}}} + {C_{{\rm{FP}}}}}} $ | (1) |
| $ Sen = \frac{{{C_{{\rm{TP}}}}}}{{{C_{{\rm{TP}}}} + {C_{{\rm{FN}}}}}} $ | (2) |
式中CTP表示真阳性变异数量,CFP表示假阳性变异数量,CFN表示假阴性变异数量。
2.2 仿真数据实验 2.2.1 数据选取本文选取了千人基因组的人类hg19参考基因的21号染色体作为后续处理的参考基因,通过编程生成一个结构变异(SV)信息的参考文件,其中包含结构变异的类型、坐标位置和尺寸大小。选择1 000个插入位置互不重叠的断点坐标,这些插入变异的长度范围为50 bp~1 kb,插入的序列随机产生;然后使用svsim工具包将插入序列添加到参考染色体中,得到目标基因。
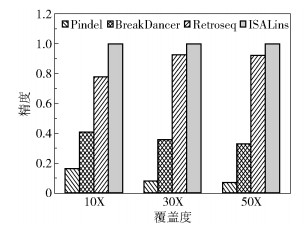
2.2.2 仿真实验结果与讨论利用art_illumina软件通过目标基因仿真得到fastq格式的序列片段文件。仿真参数设置为:reads长度100 bp,插入距离500 bp,标准差50。覆盖度分别设置为10X、30X和50X。使用Pindel、BreakDancer、Retroseq和ISALins 4种工具分别对不同覆盖度的仿真数据进行插入变异的检测(采用各个工具的默认参数),实验结果如表 2和图 5所示。可以看出,无论是高覆盖度还是低覆盖度,ISALins检测方法均能保持稳定的性能,检测精度均达到100%,与此同时敏感度也比单个工具好。可见本文提出的方法在保证不损失敏感度的同时,显著提高了检测的精度。
| 下载CSV 表 2 不同检测工具仿真数据检测结果 Table 2 Summary of detection results for different tools using simulated data |
|
图 5 不同检测工具仿真数据精度对比 Fig.5 Comparison of the precision of simulation data for different tools |
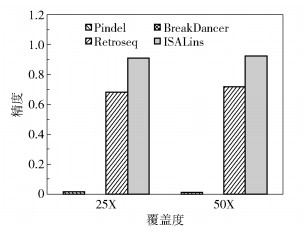
采用人类个体NA12878的测序数据,在全基因组测序上验证ISALins方法的效果,初始的fastq文件从欧洲核苷酸库的访问序号ERA172924(https://www.ebi.ac.uk/ena/data/view/ERA172924)获得。使用bwa-mem比对工具的默认参数将双末端片段比对到hg19人类参考基因上,然后按染色体号将bam文件分解,使每个小的bam文件包含一个染色体的所有比对成功片段和所有比对失败片段,随后使用Pindel、BreakDancer和Retroseq在每个小的bam文件上单独检测,最后将每个染色体的检测结果融合在一个最终的输出文件中。长插入变异的参考标准来源于千人基因组第一阶段Cortex识别的NA12878结果。使用Pindel、BreakDancer、Retroseq和ISALins 4种工具分别对两种覆盖度的数据进行插入变异的检测,实验采用各个工具的默认参数进行,检测结果如表 3、图 6所示。
| 下载CSV 表 3 不同检测工具真实数据检测结果 Table 3 Summary of detection results for different tools using real data |
|
图 6 不同检测工具真实数据精度对比 Fig.6 Comparison of precision for real data accuracy of different tools |
可以看出,公认的有较好检测效果的Pindel工具对真实数据的长插入检测效果很差;BreakDancer工具在检测本文的真实实验数据时完全检测不到长插入,这是由于BreakDancer工具使用的双末端检测方法存在对真实数据插入的检测缺陷。可见与单个检测工具相比,ISALins具有最好的敏感度和精度,分别达到45.7%和92.3%。
3 结论(1) 本文提出了基于序列拼接的基因组长插入变异检测方法ISALins,通过集成多个检测工具的初始检测结果保证了可疑插入断点的可信度,同时通过分析断点附近的比对失败片段和高质量软切片段提高了拼接的适用性和准确性。
(2) 将拼接之前的输入基因序列进行筛选和分析,不仅解决了拼接计算资源问题,也在很大程度上保证了后续检测的准确性。与现有的常用工具相比,在不损失检测敏感度的前提下,本文方法提高了检测长插入变异的精度,不仅对高覆盖数据集有效,对低覆盖度数据集也有较好的结果,而且能够精确到单个碱基。
(3) ISALins方法不限于使用本文中的3个工具,与更多的不同工具都可以结合使用,从而为基于拼接方法检测结构变异提供了一个高效的框架。
| [1] |
STANKIEWICZ P, LUPSKI J R. Structural variation in the human genome and its role in disease[J]. Annual Review of Medicine, 2010, 61: 437-455. DOI:10.1146/annurev-med-100708-204735 |
| [2] |
GENG Y, ZHAO Z M, ZHANG X P, et al. An improved burden-test pipeline for identifying associations from rare germline and somatic variants[J]. BMC Genomics, 2017, 18(S7): 55-62. |
| [3] |
GRAYTON H M, MISSLER M, COLLIER D A, et al. Altered social behaviours in neurexin 1α knockout mice resemble core symptoms in neurodevelopmental disorders[J]. PLoS One, 2013, 8(6): e67114. DOI:10.1371/journal.pone.0067114 |
| [4] |
CHEN K, WALLIS J W, MCLELLAN M D, et al. BreakDancer:an algorithm for high-resolution mapping of genomic structural variation[J]. Nature Methods, 2009, 6(9): 677-681. DOI:10.1038/nmeth.1363 |
| [5] |
HORMOZDIARI F, HAJIRASOULIHA I, DAO P, et al. Next-generation VariationHunter:combinatorial algorithms for transposon insertion discovery[J]. Bioinformatics, 2010, 26(12): i350-i357. DOI:10.1093/bioinformatics/btq216 |
| [6] |
YE K, SCHULZ M H, LONG Q, et al. Pindel:a pattern growth approach to detect break points of large deletions and medium sized insertions from paired-end short reads[J]. Bioinformatics, 2009, 25(21): 2865-2871. DOI:10.1093/bioinformatics/btp394 |
| [7] |
ABEL H J, DUNCAVAGE E J. Detection of structural DNA variation from next generation sequencing data:a review of informatic approaches[J]. Cancer Genetics, 2013, 206(12): 432-440. DOI:10.1016/j.cancergen.2013.11.002 |
| [8] |
RIMMER A, PHAN H, MATHIESON I, et al. Integrating mapping-, assembly-and haplotype-based approaches for calling variants in clinical sequencing applications[J]. Nature Genetics, 2014, 46(8): 912-918. DOI:10.1038/ng.3036 |
| [9] |
NARZISI G, O'RAWE J A, IOSSIFOV I, et al. Accurate de novo and transmitted indel detection in exome-capture data using microassembly[J]. Nature Methods, 2014, 11(10): 1033-1036. DOI:10.1038/nmeth.3069 |
| [10] |
LI H. FermiKit:assembly-based variant calling for Illumina resequencing data[J]. Bioinformatics, 2015, 31(22): 3694-3696. |
| [11] |
HAJIRASOULIHA I, HORMOZDIARI F, ALKAN C, et al. Detection and characterization of novel sequence insertions using paired-end next-generation sequencing[J]. Bioinformatics, 2010, 26(10): 1277-1283. DOI:10.1093/bioinformatics/btq152 |
| [12] |
LI H, HANDSAKER B, WYSOKER A, et al. The sequence alignment/map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078-2079. DOI:10.1093/bioinformatics/btp352 |
| [13] |
PENG Y, LEUNG H C M, YIU S M, et al. IDBA-UD:a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth[J]. Bioinformatics, 2012, 28(11): 1420-1428. DOI:10.1093/bioinformatics/bts174 |
| [14] |
KENT W J. BLAT-the BLAST-like alignment tool[J]. Genome Research, 2002, 12(4): 656-664. DOI:10.1101/gr.229202 |
| [15] |
GARRISON E, MARTH G. Haplotype-based variant detection from short-read sequencing[EB/OL]. (2012-07-20)[2017-06-01]. https://arxiv.org/abs/1207.3907.
|








