文章信息
- 闫爱霞, 康颖, 崔尧, 赵文轩, 李首飞, 王苗, 王园园, 王洛桐, 王凤双, 逄波, 李颖.
- Yan Aixia, Kang Ying, Cui Yao, Zhao Wenxuan, Li Shoufei, Wang Miao, Wang Yuanyuan, Wang Luotong, Wang Fengshuang, Pang Bo, Li Ying
- 一起食源性疾病暴发事件分离的2种血清型沙门菌的病原学分析
- Etiological analysis on a foodborne disease outbreak caused by two serotypes of Salmonella
- 中华流行病学杂志, 2023, 44(9): 1440-1446
- Chinese Journal of Epidemiology, 2023, 44(9): 1440-1446
- http://dx.doi.org/10.3760/cma.j.cn112338-20230306-00126
-
文章历史
收稿日期: 2023-03-06
2. 中国疾病预防控制中心传染病预防控制所, 北京 102206
2. National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China
沙门菌是引起急性胃肠炎的主要病原菌,广泛存在于自然界,可污染肉类、蛋奶及水源等,在人类和动物体内均可寄生[1]。在我国,引起食物中毒暴发事件的病原菌中,沙门菌常居首位[2-3]。沙门菌的传播途径多样且不同物种中沙门菌的分离率差异很大。沙门菌包含超过2 000种血清型,肠炎沙门菌和鼠伤寒沙门菌在我国人感染沙门菌血清型中排在前2位,导致众多暴发事件[2, 4-8]。乌干达沙门菌在我国食品和疾病相关样本中检出率较低,曾有过2次乌干达沙门菌暴发的相关报道[9-10],国外也有该血清型沙门菌导致暴发事件的相关报道[11];伊迪坎沙门菌是全球罕见的血清型,国外在农场动物、动物饲料、农场环境及屠宰场中检出过该菌[12-13]。2020年国内首次在从业人员健康体检中分离到1株伊迪坎沙门菌[9, 14],国内外尚无伊迪坎沙门菌引起暴发的相关报道。本研究基于2022年北京市某区一起由2种血清型沙门菌感染导致的食物中毒事件进行病原识别和特征分析,为此类事件的实验室应对提供基础数据。
材料与方法1. 流行病学调查:暴发事件发生地点为某高校。2022年9月8日多名学生因在同一食堂就餐后出现腹泻、腹痛、发热等症状到医院就诊。当地CDC对学校食堂进行调查并进行病例搜索,病例定义:在该高校食堂就餐后出现腹泻(3次/24 h或粪便性状改变)、腹痛、发热(> 37.3 ℃)任一症状者,共搜集到病例14例(男性11例、女性3例)。所有病例均购买过餐厅的驴肉火烧,按照病例食用驴肉火烧至发病时间计算潜伏期,所有病例潜伏期介于7~48 h之间,平均潜伏期24 h。有11例病例采集到肛拭子(样本编号为P1~P11),每例病例均采集肛拭子2份。同时采集到该食堂9月8日午餐留样13件(样本编号为F1~F13)和环境涂抹样本11件(样本编号为S1~S11)。所有样本采集后2 h内在4 ℃无菌环境下转运至区CDC进行病原学检测。
2. 试剂和仪器:四硫磺酸钠煌绿增菌液(TTB)、亚硒酸盐煌绿增菌液(SBG)、脑心浸液肉汤(BHI)、亚硒酸盐胱氨酸增菌液(SC)、木糖赖氨酸脱氧胆盐琼脂(XLD)、胰酪大豆琼脂(TSA)、Swarm琼脂和细菌全基因组提取试剂盒为北京陆桥生物制品有限公司产品,14种食源性致病菌多重荧光PCR试剂盒与轮状病毒、诺如病毒、札如病毒、腺病毒和星状病毒荧光PCR检测试剂盒为北京卓成惠生公司产品,沙门菌显色平板为法国科马嘉公司产品,沙门菌诊断血清为丹麦SSI公司产品。采用的仪器包括全自动细菌鉴定仪VITEK-2(法国梅里埃公司)、荧光PCR仪(美国伯乐公司CFX96)和全基因组测序仪(美国Illumina HiSeq 2500平台)。
3. 检测方法:
(1)分离培养:病例肛拭子样本接种3 ml SBG 37 ℃增菌24 h,取一环增菌液分别接种沙门显色平板和XLD平板37 ℃培养24 h;可疑污染食品和环境涂抹样本接种缓冲蛋白冻水(BPW)37 ℃增菌18 h,取1 ml增菌液转种分别接种SC和TTB二次增菌24 h,取1环增菌液分别接种沙门显色平板和XLD平板37 ℃培养24 h。所有样本在分离平板上均挑取多个可疑沙门菌菌落转种TSA平板纯培养并进行菌株的全自动细菌生化鉴定,鉴定为沙门菌的菌株进行血清凝集实验。对可疑污染食品检测参照《食品安全国家标准》(GB 4789.4-2016)。
(2)病例肛拭子样本荧光PCR检测:病例肛拭子在接种3 ml SBG的同时同步接种3 ml BHI进行增菌,2种增菌液在增菌0、3、6、12和24 h时,分别取出200 μl增菌液提取细菌DNA,并进行14种常见食源性致病菌多重荧光PCR检测,记录2种增菌液在不同增菌时间点荧光PCR检测的Ct值。
(3)分离沙门菌全基因组测序:使用细菌全基因组提取试剂盒提取分离菌株的DNA进行细菌全基因组测序。使用FastQC 0.11.8软件和Trimmomatic 0.36软件对下机原始数据进行质量评估,去除Q值< 20的数据,使用SPAdes 3.13、GapFiller 2.1.2和MAQ 0.6.6软件PrInSeS-G组件对质控后的数据进行拼接和序列矫正。使用BLAST 2.14.0软件将拼接好的序列与VFDB(Virulence Factors of Pathogenic Bacteria)数据库和CARD(Comprehensive Antibiotic Resistance Database)数据库进行比对得到耐药基因和毒力因子的注释结果,比对使用参数-evalue 1e-5,对比对的结果使用coverage > 95%,identity > 95%标准过滤。使用FastANI 1.33软件比对序列间的平均核苷酸一致性(average nucleotide identity,ANI),使用GATK 3.8软件检测变异位点,使用FastTree 2.1.11软件构建基于核心基因组SNP的系统发育树。
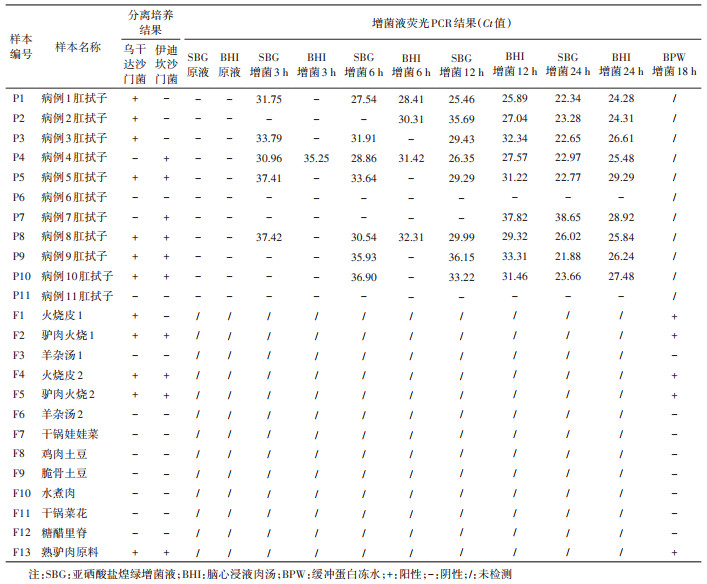
结果1. 分离培养结果:在11例病例肛拭子样本中,沙门菌检出率为9/11,其中样本P1、P2和P3分离沙门菌均为乌干达沙门菌,P4和P7分离沙门菌均为伊迪坎沙门菌,P5、P8、P9和P10均分离到乌干达沙门菌和伊迪坎沙门菌,P6和P11未分离到沙门菌。13件可疑污染食品样本沙门菌检出率为5/13,其中F1分离沙门菌为乌干达沙门菌,F2、F4、F5和F13均同时分离到乌干达沙门菌和伊迪坎沙门菌,其余可疑污染食品样本和所有环境涂抹样本均未分离到沙门菌。见表 1。
2. 增菌液荧光PCR结果:在13件可疑污染食品中,5件增菌液(增菌18 h)沙门菌荧光PCR结果为阳性(F1、F2、F4、F5、F13),检出率为5/13,与分离培养结果一致。11件病例肛拭子样本接种SBG增菌液后,0、3、6、12和24 h沙门菌荧光PCR检出率分别为0/11、5/11、7/11、8/11和9/11,其中培养24 h后荧光PCR结果与分离培养结果一致;11件病例肛拭子样本接种BHI增菌液后,0、3、6、12和24 h沙门菌荧光PCR检出率分别为0/11、1/11、4/11、9/11和9/11(表 1),培养12 h和24 h后荧光PCR结果也与分离培养结果一致。
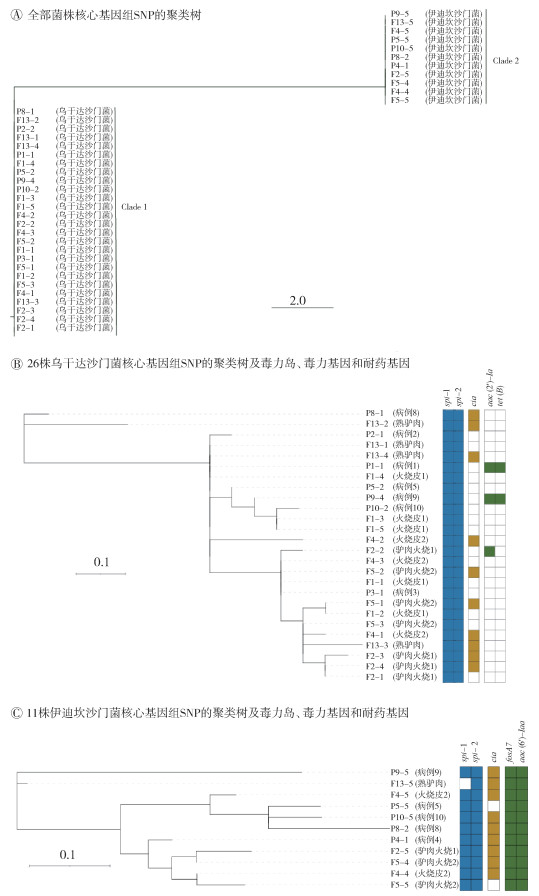
3. 全基因组测序分析:从样本P1、P2、P3、P5、P8、P9、P10、F1、F2、F4、F5和F13中分离到26个乌干达沙门菌单菌落,从P4、P5、P7、P8、P9、P10、F2、F4、F5和F13中分离到11个伊迪坎沙门菌单菌落,这37个沙门菌单菌落基于核心基因组SNP构建聚类树形成2个相互独立且遗传距离较远的分支,遗传分支1(Clade 1)上均为乌干达沙门菌,Clade 2上均为伊迪坎沙门菌(图 1A)。基于单一血清型沙门菌核心基因组SNP构建聚类树,26株乌干达沙门菌形成5个分支,菌株之间SNP差异个数介于0~14个(图 1B);11株伊迪坎沙门菌SNP差异介于0~23个,无高度克隆化菌株分布(图 1C)。
|
| 注:彩色色块:可检测到相应的毒力岛、毒力基因或者耐药基因;白色色块:未检测到相应的成分 图 1 基于核心基因组单核苷酸多态性(SNP)构建聚类树讨论 |
26株乌干达沙门菌均检测到SPI-1和SPI-2毒力岛,10株仅检测到毒力基因cia;耐药基因方面,2株同时检测到氨基糖甙类耐药基因aac(2')-Ia和四环素类耐药基因tet(B),1株仅检测到氨基糖甙类耐药基因aac(2')-Ia(图 1B)。在11株伊迪坎沙门菌中,F13-5仅检测到SPI-2毒力岛,另外10株均同时检测携带SPI-1和SPI-2毒力岛,9株检测到毒力基因cia,耐药基因方面,11株菌均能检测到氨基糖甙类耐药基因aac(6')-Iaa和磷酸类耐药基因fosA7(图 1C)。
本次暴发事件中,9例病例和5件可疑污染食品均分离到沙门菌,其中4例病例和4件可疑污染食品同时分离到乌干达沙门菌和伊迪坎沙门菌,其余病例和食品样本分离到上述2种血清型中的1种。基于本次暴发流行病学调查及分离菌株病原学分析结果,参照“由可疑食品、呕吐物或腹泻粪便中检出血清型相同的沙门菌,或从不同病例呕吐物或粪便中检出血清型别相同的沙门菌”[15],本次事件可判定为由乌干达沙门菌和伊迪坎沙门菌共同导致的食源性疾病暴发事件。
伊迪坎沙门菌目前尚无从腹泻病例或暴发事件中分离的相关报告,国内仅有从健康体检者样本中分离到的报道[16]。国外的伊迪坎沙门菌大多分离自动物及动物相关环境。乍得的研究发现,伊迪坎沙门菌在鸡场(包括蛋鸡、肉鸡、鸡场环境及饲料)的检出率为3.6%[14];南非的研究发现,伊迪坎沙门菌的检出率为4.0%[13]。乌干达沙门菌在我国腹泻病例、动物宿主和食品等沙门菌相关监测中均处于较低检出水平,由其导致的暴发事件十分罕见[17-18]。1998年我国绍兴市报道了乌干达沙门菌导致的食物中毒,可疑食品为烧鸡和麻油鸭[19];美国纽约和芝加哥也分别报道过乌干达沙门菌导致食物中毒,可疑污染食品均为熟肉相关制品[9]。本次事件中4件可疑食品均分离到上述2种血清型沙门菌,且所有被沙门菌污染的食品均为驴肉火烧成品或相关半成品,作为动物性食品的驴肉极有可能是2种血清型沙门菌最初的污染来源。畜禽携带的沙门菌很容易通过动物制品感染人类,2018年刘英玉等[20]抽检我国新疆维吾尔自治区某驴肉销售点发现沙门菌检出率为10%;河北省廊坊市2015-2017年食品中沙门菌污染分析发现驴肉火烧沙门菌检出率较高[21];南京市和甘肃省靖远市等均有食用污染的驴肉食物中毒的报道[10, 22]。我国食品风险监测工作中,应关注乌干达沙门菌和伊迪坎沙门菌在预加工食品中的分布情况、造成成年人感染的主要传播途径,在腹泻监测和暴发监测中加强这2种血清型的识别和监测。
基于2种血清型37株沙门菌核心基因组SNP构建聚类树,2种血清型沙门菌形成独立分支,且2分支遗传关系较远。基于26株乌干达沙门菌核心基因组SNP构建的聚类树可见5个克隆化分支。2种血清型沙门菌菌株分别构建的聚类树中均存在多个遗传分支,尤其11株伊迪坎沙门菌分为11条独立分支,即使分离自同一食品样本、同一血清型的不同菌株也可存在不同遗传分支,可见2种血清型沙门菌呈现了一定的遗传多样性。2种血清型沙门菌均携带与人致病性具有重要关联的SPI-1和SPI-2毒力岛。2种血清型沙门菌耐药基因携带存在差异,11株伊迪坎沙门菌均携带氨基糖甙类耐药基因aac(6')-Iaa和磷酸类耐药基因fosA7,少量乌干达沙门菌则携带氨基糖甙类耐药基因aac(2')-Ia和四环素类耐药基因tet(B);伊迪坎沙门菌携带毒力基因cia的比例也高于乌干达沙门菌。
荧光PCR作为食源性疾病暴发应对中重要的病原筛查手段,可以弥补培养法周期长、各病原培养路线差异大等缺点,提升食源性疾病暴发事件应对效率。病例粪便样本采集通常是食物中毒处理的一个难题,所以常以肛拭子作为替代,但肛拭子样本中病原载量低,对多病原筛查和多病原培养都会造成困难。本次暴发事件中,共采集到11份病例样本,全部为肛拭子样本。本研究中对病例同步采集了2件肛拭子样本,分别接种沙门菌特异性增菌液SBG,以及可以让大部分需氧食源性致病菌普遍快速增长和保存的BHI,并在增菌的不同时间使用等量增菌液提取DNA进行沙门菌荧光PCR检测。对比不同增菌时间2种增菌液的检测结果,0 h 2种增菌液荧光PCR检测结果均为阴性,说明肛拭子的原始样本载量低,不能达到沙门菌荧光PCR检测方法的检出限;SBG增菌3 h和6 h后沙门菌荧光PCR检出率分别高于BHI,可见SBG增菌效果优于非特异性增菌液BHI,可让更多肛拭子样本增菌量达到沙门菌荧光PCR检出限;在增菌12 h后,基于BHI增菌的荧光PCR检出率反而超过SBG,与培养法阳性结果一致。可见BHI增菌12 h是可以弥补肛拭子样本因沙门菌载量低不能达到荧光PCR检出限这一问题的。相比沙门菌特异性增菌液SBG,BHI还能兼顾弧菌和其他肠杆菌等食源性病原的增菌,因此在缺少粪便样本的暴发处理中,同步采集一支保存于BHI的肛拭子对多病原筛查有较大帮助。特别是本研究中的P7样本基于SBG增菌12 h未检测到沙门菌,基于SBG增菌24 h沙门菌荧光PCR的Ct值仅为38.65,仍处于低载量无法培养状态(样本P7基于SBG增菌24 h分离沙门菌结果为阴性,基于BHI增菌24 h分离沙门菌结果为阳性),这可能与BHI更利于细菌修复,而恰好P7样本中的沙门菌致伤较为严重,因此BHI增菌效果反而优于SBG。
综上所述,此次事件中分离的乌干达沙门菌和伊迪坎沙门菌各自均呈现了一定的遗传多样性,虽然美国食品药品监督管理局认为变异小于20个SNP具有共同污染来源,但是,也有其他研究的结果与之不一致。因此本研究无法通过基因组数据来判定此次突发事件为暴发事件。从流行病学方面来说,这些病例又在相同的时间有共同进餐史;从菌株的血清型角度来说,病例肛拭子样本之间、病例与食品样本之间均分离到2种血清型沙门菌,支持该事件是由2种沙门菌共同导致食源性疾病暴发事件。另外,BHI用作沙门菌的初筛增菌液与SBG的效果接近,同时能兼顾其他病原菌的筛查以弥补肛拭子原始样本载量低的问题。因此,在暴发事件中可以应用基于BHI对病例肛拭子增菌并进行沙门菌荧光PCR检测的方案。
利益冲突 所有作者声明无利益冲突
作者贡献声明 闫爱霞:论文撰写、实施研究;康颖、崔尧、赵文轩:整理分析数据;李首飞、王苗、王园园、王洛桐:设计实验、实施研究;王凤双:收集数据;逄波、李颖:论文修改、研究指导
| [1] |
Wang BX, Butler DS, Hamblin M, et al. One species, different diseases: the unique molecular mechanisms that underlie the pathogenesis of typhoidal Salmonella infections[J]. Curr Opin Microbiol, 2023, 72: 102262. DOI:10.1016/j.mib.2022.102262 |
| [2] |
白莉, 刘丽莎, 李亮亮, 等. 2011-2017年中美两国食源性疾病暴发监测资料比较及对我国监测体系建设的启示[J]. 中国食品卫生杂志, 2022, 34(5): 863-870. DOI:10.13590/j.cjfh.2022.05.001 Bai L, Liu LS, Li LL, et al. Comparison of foodborne disease outbreaks data in China and the United States from 2011 to 2017 and its inspiration for the construction of monitoring system in China[J]. Chin J Food Hyg, 2022, 34(5): 863-870. DOI:10.13590/j.cjfh.2022.05.001 |
| [3] |
张雁雁. 基层疾控部门细菌性食物中毒检验分析[J]. 临床研究, 2021, 29(7): 137-139. Zhang YY. Analysis of bacterial food poisoning in grass-roots disease control department[J]. Clin Res, 2021, 29(7): 137-139. |
| [4] |
汤全英, 崔家瑞, 张梦寒, 等. 苏州市3起疑似肠炎沙门菌食物中毒事件的病原学及耐药分析[J]. 江苏预防医学, 2022, 33(2): 192-193, 196. DOI:10.13668/j.issn.1006-9070.2022.02.022 Tang QY, Cui JR, Zhang MH, et al. Etiology and drug resistance analysis of 3 suspected Salmonella enteritidis food poisoning cases in Suzhou[J]. Jiangsu J Prev Med, 2022, 33(2): 192-193, 196. DOI:10.13668/j.issn.1006-9070.2022.02.022 |
| [5] |
欧秀华, 邱艺燕. 一起肠炎沙门菌食物中毒的实验室检测分析[J]. 热带病与寄生虫学, 2022, 20(2): 89-90, 98. DOI:10.3969/j.issn.1672-2302.2022.02.008 Ou XH, Qiu YY. Laboratory study and analysis of one case of food poisoning caused by Salmonella enteritidis[J]. J Trop Dis Parasitol, 2022, 20(2): 89-90, 98. DOI:10.3969/j.issn.1672-2302.2022.02.008 |
| [6] |
刘晓强, 古文鹏, 黄达峰, 等. 一起鼠伤寒沙门菌引起的食物中毒暴发调查[J]. 疾病监测, 2014, 29(11): 920-924. DOI:10.3784/j.issn.1003-9961.2014.11.019 Liu XQ, Gu WP, Huang DF, et al. Survey of a food poisoning outbreak caused by Salmonella typhimurium[J]. Dis Surveill, 2014, 29(11): 920-924. DOI:10.3784/j.issn.1003-9961.2014.11.019 |
| [7] |
付秀影, 黄露. 一起鼠伤寒沙门氏菌食物中毒的流行病学调查[J]. 首都公共卫生, 2016, 10(2): 86-89. DOI:10.16760/j.cnki.sdggws.2016.02.012 Fu XY, Huang L. Epidemiological investigation on a food poisoning caused by Salmonella typhimurium[J]. Cap J Public Health, 2016, 10(2): 86-89. DOI:10.16760/j.cnki.sdggws.2016.02.012 |
| [8] |
苏靖华, 施建芳, 王闻卿, 等. 腹泻病监测中非伤寒沙门菌血清型及耐药性分析[J]. 职业与健康, 2016, 32(1): 52-54, 58. DOI:10.13329/j.cnki.zyyjk.2016.0062 Su JH, Shi JF, Wang WQ, et al. Analysis on serotype and antibiotic resistance of non-typhoidal Salmonella from diarrheal patients[J]. Occup Health, 2016, 32(1): 52-54, 58. DOI:10.13329/j.cnki.zyyjk.2016.0062 |
| [9] |
杨树博, 王力, 马源, 等. 一起农村家庭自办宴席引起的食物中毒调查[J]. 医学动物防制, 2016, 32(7): 799-800. DOI:10.7629/yxdwfz201607031 Yang SB, Wang L, Ma Y, et al. Investigation of food poisoning caused by a rural family self-banquet[J]. J Med Pest Control, 2016, 32(7): 799-800. DOI:10.7629/yxdwfz201607031 |
| [10] |
钱瑶, 严涛. 一起食物中毒分离出二种沙门菌[J]. 中国卫生检验杂志, 2001, 11(6): 760. DOI:10.3969/j.issn.1004-8685.2001.06.090 Qian Y, Yan T. Two serotypes Salmonella isolated from a food poisoning[J]. Chin J Health Lab Technol, 2001, 11(6): 760. DOI:10.3969/j.issn.1004-8685.2001.06.090 |
| [11] |
Jones RC, Reddy V, Kornstein L, et al. Salmonella enterica serotype Uganda infection in New York City and Chicago[J]. Emerg Infect Dis, 2004, 10(9): 1665-1667. DOI:10.3201/eid1009.030713 |
| [12] |
Monte DFM, Nethery MA, Barrangou R, et al. Whole-genome sequencing analysis and CRISPR genotyping of rare antibiotic-resistant Salmonella enterica serovars isolated from food and related sources[J]. Food Microbiol, 2021, 93: 103601. DOI:10.1016/j.fm.2020.103601 |
| [13] |
Magwedere K, Rauff D, De Klerk G, et al. Incidence of nontyphoidal Salmonella in food-producing animals, animal feed, and the associated environment in South Africa, 2012-2014[J]. Clin Infect Dis, 2015, 61 Suppl 4: S283-289. DOI: 10.1093/cid/civ663.
|
| [14] |
Tabo DA, Diguimbaye CD, Granier SA, et al. Prevalence and antimicrobial resistance of non-typhoidal Salmonella serotypes isolated from laying hens and broiler chicken farms in N'Djamena, Chad[J]. Vet Microbiol, 2013, 166(1/2): 293-298. DOI:10.1016/j.vetmic.2013.05.010 |
| [15] |
中华人民共和国卫生部. WS/T 13-1996沙门氏菌食物中毒诊断标准及处理原则[S]. 北京: 中国标准出版社, 2000. Ministry of the Health P. R. China. WS/T 13-1996 Diagnostic criteria and principles of management for food poisoning ofSalmonella[S]. Beijing: Standards Press of China, 2000. |
| [16] |
李映霞, 许少洪, 郭凯纯, 等. 国内首次发现的伊迪坎沙门菌的鉴定与探讨[J]. 中国卫生检验杂志, 2020, 30(12): 1450-1452. Li YX, Xu SH, Guo KC, et al. Identification and discussion of the first-found S. Idikan in China[J]. Chin J Health Lab Technol, 2020, 30(12): 1450-1452. |
| [17] |
钟宇婧, 周倩, 黄靖宇, 等. 贵州省动物性食品中沙门菌耐药及分子特征相关性研究[J]. 现代预防医学, 2023, 50(1): 163-168. DOI:10.20043/j.cnki.MPM.202206568 Zhong YJ, Zhou Q, Huang JY, et al. Correlation of drug resistance and molecular characteristics of Salmonella in animal food, Guizhou[J]. Mod Prev Med, 2023, 50(1): 163-168. DOI:10.20043/j.cnki.MPM.202206568 |
| [18] |
曲梅, 田祎, 黄瑛, 等. 2018-2021年北京市沙门菌血清型及喹诺酮类耐药表型和基因型分析[J]. 现代预防医学, 2022, 49(13): 2413-2419. DOI:10.20043/j.cnki.MPM.202201164 Qu M, Tian Y, Huang Y, et al. Serotypes and quinolone resistant phenotypes and genotypes of Salmonella strains in Beijing, 2018-2021[J]. Mod Prev Med, 2022, 49(13): 2413-2419. DOI:10.20043/j.cnki.MPM.202201164 |
| [19] |
徐来潮, 徐阿良, 叶群. 一起乌干达沙门氏菌食物中毒调查报告[J]. 浙江预防医学, 1998(3): 146-147. DOI:10.19485/j.cnki.issn1007-0931.1998.03.014 Xu LC, Xu AL, Ye Q. An investigation report of a Salmonella Uganda food poisoning[J]. Zhejiang Prev Med, 1998(3): 146-147. DOI:10.19485/j.cnki.issn1007-0931.1998.03.014 |
| [20] |
刘英玉, 苏月, 王春梅, 等. 新疆某驴肉销售点五种常见致病菌的检测分析[J]. 新疆畜牧业, 2019, 34(6): 22-25. DOI:10.16795/j.cnki.xjxmy.2019.6.005 Liu YY, Su Y, Wang CM, et al. Detection and analysis of five common pathogenic bacteria in a donkey meat selling point in Xinjiang[J]. Xinjiang Xumuye, 2019, 34(6): 22-25. DOI:10.16795/j.cnki.xjxmy.2019.6.005 |
| [21] |
邱颖, 王宇卓, 张丽敏, 等. 2015-2017年河北省廊坊市食品中沙门氏菌污染状况及血清型监测分析[J]. 医学动物防制, 2019, 35(2): 134-136. DOI:10.7629/yxdwfz201902008 Qiu Y, Wang YZ, Zhang LM, et al. Analysis of Salmonella contamination and serotype monitoring in foods in Langfang city, Hebei province, 2015-2017[J]. J Med Pest Control, 2019, 35(2): 134-136. DOI:10.7629/yxdwfz201902008 |
| [22] |
叶艳华, 江晓, 史小超, 等. 南京市一起斯坦利沙门氏菌食物中毒的病原学检测及溯源分析[J]. 现代预防医学, 2019, 46(9): 1711-1714. Ye YH, Jiang X, Shi XC, et al. Pathogen detection and traceability analysis of a Stanley Salmonella food poisoning, Nanjing[J]. Mod Prev Med, 2019, 46(9): 1711-1714. |
 2023, Vol. 44
2023, Vol. 44