文章信息
- 杨玉花, 库鑫, 宫雅楠, 孟凡亮, 卜东波, 郭亚慧, 魏销玥, 龙丽瑾, 范佳铭, 张茂俊, 张建中, 闫笑梅.
- Yang Yuhua, Ku Xin, Gong Yanan, Meng Fanliang, Bu Dongbo, Guo Yahui, Wei Xiaoyue, Long Lijin, Fan Jiaming, Zhang Maojun, Zhang Jianzhong, Yan Xiaomei
- 金黄色葡萄球菌类肠毒素W的超抗原活性位点预测与克隆表达
- Prediction of superantigen active sites and clonal expression of staphylococcal enterotoxin-like W
- 中华流行病学杂志, 2023, 44(4): 629-635
- Chinese Journal of Epidemiology, 2023, 44(4): 629-635
- http://dx.doi.org/10.3760/cma.j.cn112338-20220822-00725
-
文章历史
收稿日期: 2022-08-22
2. 中国科学院计算技术研究所智能信息处理重点实验室, 北京 100190;
3. 中国科学院大学, 北京 100049;
4. 中科大数据研究院, 郑州 450046;
5. 内蒙古科技大学包头医学院, 包头 014040;
6. 中国医科大学, 沈阳 110122
2. Key Lab of Intelligent Information Processing, Institute of Computing Technology, Chinese Academy of Sciences, Beijing 100190, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Big Data Academy, Zhongke, Zhengzhou 450046, China;
5. Baotou Medical College, Inner Mongolia University of Science and Technology, Baotou 014040, China;
6. China Medical University, Shenyang 110122, China
金黄色葡萄球菌(Staphylococcus aureus),是一种重要的病原体,可引起一系列临床综合征,从皮肤和软组织感染等非侵入性感染到心内膜炎、骨髓炎和菌血症等侵入性感染[1-2]。葡萄球菌超抗原(SAgs)是金黄色葡萄球菌产生的典型的毒力因子,可绕过抗原加工和呈递,直接结合抗原提呈细胞上的主要组织相容性复合体Ⅱ类分子与T细胞受体(TCR)上的Vβ链并形成桥梁,诱导T淋巴细胞的异常快速增殖并释放大量的促炎细胞因子[3-5]。葡萄球菌肠毒素(SE)是一类低分子量的单链碱性球状蛋白,属于SAgs,是引起葡萄球菌食物中毒的重要毒力因子[6-7]。金黄色葡萄球菌类肠毒素W(SElW)是近几年发现的新型类肠毒素。最初被认为是金黄色葡萄球菌类肠毒素U(SElU)的等位基因突变体,命名为SElU2。后续研究证明SElW是由核心基因编码的新型类肠毒素,与金黄色葡萄球菌肠毒素A(SEA)氨基酸序列有36%的同源性[8],在金黄色葡萄球菌分离株中的携带率达到了90%以上[3, 9]。研究表明SElW能诱导Vβ-T细胞大量增殖,具有典型的超抗原特征;菌血症动物模型证明了SElW能增加肝脏的细菌载量,有助于金黄色葡萄球菌的致病[3]。目前对于该蛋白的研究相对较少,为了进一步了解SElW的超抗原活性位点,本研究利用AlphaFold和ZDOCK服务器模拟SElW与TCR分子的对接,并结合SElW和其他肠毒素的氨基酸多序列比对共同预测重要的可能的超抗原活性位点;利用大肠埃希菌重组表达系统在体外表达SElW融合蛋白,为进一步运用定点突变技术研究SElW超抗原活性位点提供理论依据。
材料与方法1. 菌株和载体:
(1)菌株:DC51619菌株(中国CDC传染病预防控制所感染性疾病诊治协同创新中心保存)、感受态细胞BL21(DE3)(中国江苏康为世纪生物科技股份有限公司)
(2)载体:原核表达载体pET-28a(+)(中国CDC传染病预防控制所感染性疾病诊治协同创新中心保存)、pMD18-T载体(日本TaKaRa公司)
2. 试剂:LB琼脂、LB肉汤(中国北京路桥技术股份有限公司);超保真酶、T4连接酶、卡那霉素、异丙基-β-D-硫代半乳糖苷(IPTG)试剂(中国北京全式金生物技术股份有限公司);限制性内切酶BamHⅠ和Hind Ⅲ(日本TaKaRa公司);SDS-PAGE胶配置试剂盒、10×SDS-PAGE电泳缓冲液、蛋白酶抑制剂混合物(中国江苏康为世纪生物科技股份公司);PCR产物纯化试剂盒、胶回收DNA试剂盒(美国Omega公司);质粒小提试剂盒(德国Qiagen公司)、蛋白定量试剂盒[赛默飞世尔科技(中国)有限公司]。
3. 仪器设备:梯度PCR仪(德国Sensoquest公司)、5804R型冷冻离心机(德国Eppendorf公司)、超声破碎仪(德国Bandelin公司)、DYY-6C型核酸电泳仪(中国北京六一仪器厂)、低温空气浴摇床(瑞士INFORS公司)、蛋白纯化仪(瑞典GE公司)、SDS-PAGE电泳槽(美国伯乐公司)
4. SElW蛋白三维结构的预测及评估:将fasta格式的SElW氨基酸序列输入至AlphaFold软件中[10],设置参数evoformer block的层数为48,structure module的层数为8,循环3次,用5个模型预测得到5个结果,选择预测局部距离差检验(pLDDT)最高的模型作为最终选用的蛋白模型。利用UCLA-DOE的SAVES在线服务器(https://saves.mbi.ucla.edu/),使用ERRAT、拉氏图(Ramachandran Plot)和Verify_3D等软件对SElW蛋白模型预测结构进行评估。采用ERRAT软件计算0.35 nm范围之内不同的原子类型对之间形成的非键相互作用的数目(侧链),当得分 > 85时,说明预测结果准确度较高;PROCHECK软件中的拉氏图利用蛋白中非键合原子间的最小接触距离,体现氨基酸残基理论上可以出现的构象,表示蛋白中允许和不允许的构象,要求位于不允许区的氨基酸数目≤5%。Verify_3D用来检查评估模型的三维结构和氨基酸一级序列的匹配度,当超过80%的氨基酸残基的得分≥0.2时,说明构象合理。使用ZDOCK 3.0.2服务器(https://zdock.umassmed.edu/)进行蛋白单体和TCR(PDB ID:4udt)的对接,ZDOCK自动默认参数设定(设置对接网格大小为128,间距为1.2 Å),输出10个打分最高的对接构象,选择打分最高的对接构象作为分析对象,获得的结果用PyMOL软件进行分析。
5. SElW与其他肠毒素氨基酸序列比对:从美国国立生物技术信息中心数据库里下载SEA、SEB、SEC3、SED、SEE、SEN的氨基酸序列,利用Geneious软件中的Align对SElW与上述肠毒素的氨基酸序列比对。
6. 重组蛋白的克隆表达:
(1)目的基因的扩增:根据selw序列设计引物。正向引物(F):5'-CCC CGG ATC CAT CGA ATA TTC AGA CTT ACA TC-3',反向引物(R):5'-CCC CAA GCT TTT ATG ATT TGA ATA AAT AGA TAT CT-3'。正反向引物中分别引入保护碱基和BamHⅠ、Hind Ⅲ的酶切位点(画横线处)。
(2)重组质粒表达载体的构建:PCR产物纯化后连接到pMD-18-T载体上,进行蓝白斑筛选后,将菌落PCR鉴定阳性的菌落进行测序。将测序正确的产物-T载体和表达载体pET-28a(+)同时用BamH Ⅰ和Hind Ⅲ进行酶切,酶切成功的目的片段连接到表达载体上后转化至感受态细胞BL21(DE3)中,在含卡那霉素的LB固体培养基上进行菌落PCR和重组质粒双酶切鉴定。
(3)重组蛋白的诱导表达、纯化及蛋白定量:将鉴定正确的重组质粒pET-28a(+)-selw接种在含有卡那霉素抗性的LB固体培养基上,挑取单克隆过夜培养种子液,按1∶100的比例扩大培养至含有终浓度为50 μg/ml卡那霉素抗性的LB液体培养基中,待A600的值升高至0.6~0.8时,加入终浓度为1 mmol/L的IPTG诱导剂,16 ℃过夜诱导表达。将表达后的菌液离心洗涤、超声破碎后再次离心,上清用0.45 μm滤膜过滤后,用AKTA蛋白纯化仪-镍亲和层析柱纯化带有His标签的重组蛋白,纯化完后进行SDS-PAGE观察蛋白的纯化效果,并利用BCA蛋白定量试剂盒对重组蛋白SElW进行定量。
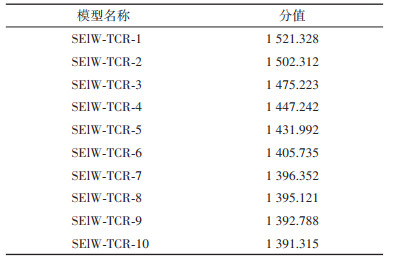
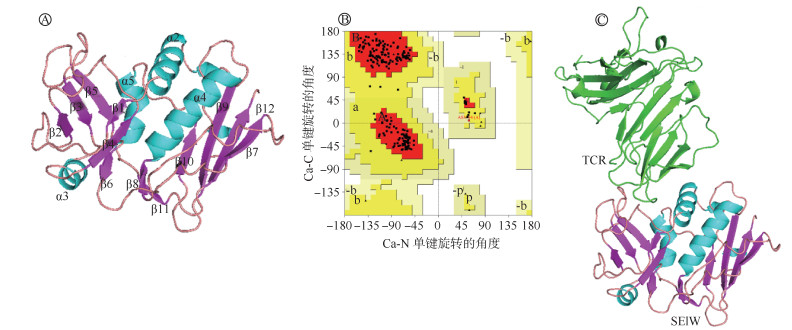
结果1. SElW与TCR对接模型预测:AlphaFold共预测5个SElW单体结构模型,5个模型的pLDDT的值分别为95.13、95.33、95.03、94.56、94.98。最终选择模型2为对接蛋白单体(图 1A)。SElW的空间结构与已知其他肠毒素的结构相似,具有两个结构域(氨基末端结构域和羧基末端结构域)。氨基末端由与“寡糖/寡核苷酸折叠”类似的β-基序组成,包含3个α-螺旋(α1~α3)及6个β-片层(β1~β6);羧基末端的β-grasp结构域由2个α-螺旋(α4、α5)和反向平行的β-片层(β7~β13)组成。氨基末端的前17个氨基酸与羧基末端相邻。ERROT评估结果显示,SElW蛋白预测模型的整体质量因素得分为98.08,表明模型的准确度高;拉氏图的评估结果见图 1B,优化后的最合适区氨基酸数目为94.1%,无氨基酸位于不允许区,表明模型构架结构合理。Verify_3D的结果显示,93.24%的氨基酸的评分≥0.2,说明模型和氨基酸序列的匹配度高。利用ZDOCK服务器对SElW与TCR进行对接后,输出了10个评分高的对接构象,选择评分最高的对接构象作为分析对象(图 1C),其评分值为1 521.328,符合ZDOCK筛选相互作用蛋白的要求(具体打分见表 1)。使用PyMOL软件分析SElW与TCR相应的氨基酸残基之间形成的19个氢键,氢键信息见表 2。模型显示,TCR与SElW蛋白的氨基末端结构域相邻。
|
| 注:A为SElW蛋白单体,蓝色为SElW蛋白的α-螺旋,桃红色为β-片层,棕色为不规则卷曲;B为SElW蛋白模型评估的拉氏图,红色区域为完全允许区,黄色区域为允许区,白色为不允许区;C为SElW与TCR对接模型,绿色代表TCR 图 1 金黄色葡萄球菌类肠毒素W(SElW)单体预测、评估及SElW与T细胞受体(TCR)对接预测 |
2. SElW与TCR对接的重要位点汇总分析:序列比对结果显示,SElW与SEA、SEB、SEC3、SED、SEE、SEN在羧基末端相对保守(图 2),既往文献实验验证的与TCR相互作用的肠毒素位点主要集中在氨基端,如SEA的位点N25、C96、C106、S206和N207,SED的位点N23和F46。结合既往文献报道的活性位点和不同肠毒素序列间同一位点的保守性,总结出SElW可能的重要位点:Y18、W55、N19、C88和C98(表 3),并且这些位点与分子对接预测的可能形成氢键的氨基酸残基位点重合。

|
注:红色方框表示SEA与TCR的作用位点;绿色方框表示SED与TCR的作用位点; 表示α-螺旋, 表示α-螺旋, 表示β-片层,剩余未标注的位点表示不规则卷曲;*表示SElW与TCR的预测位点
图 2 金黄色葡萄球菌类肠毒素W(SElW)与金黄色葡萄球菌肠毒素(SEA、SED、SEE、SEN、SEB、SEC3)的氨基酸序列比对 表示β-片层,剩余未标注的位点表示不规则卷曲;*表示SElW与TCR的预测位点
图 2 金黄色葡萄球菌类肠毒素W(SElW)与金黄色葡萄球菌肠毒素(SEA、SED、SEE、SEN、SEB、SEC3)的氨基酸序列比对
|
3. 重组蛋白的克隆表达:
(1)pET-28a(+)-selw重组质粒的构建:selw基因PCR扩增以菌株DC51619为模板,PCR产物与T载体连接成功后基因测序,结果显示selw基因全长669 bp(图 3A),无碱基突变。将测序正确的目的片段与表达载体pET-28a(+)连接成功后,提取质粒进行双酶切鉴定,结果显示除质粒条带外,在接近700 bp有一条目标条带(图 3B),提示重组质粒表达载体pET-28a(+)-selw构建成功。

|
| 注:A:selw基因PCR扩增:1:Marker;2:阴性对照;3:目标条带;B:重组质粒双酶切鉴定:1:Marker;2:空质粒;3:重组质粒 图 3 金黄色葡萄球菌类肠毒素W(SElW)PCR扩增及重组质粒双酶切鉴定 |
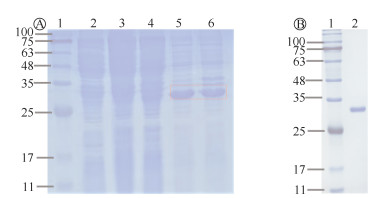
(2)重组蛋白的诱导表达、纯化及定量:经过对诱导表达条件的摸索,发现在IPTG终浓度为1 mmol/L、16 ℃诱导14 h时更有利于selw基因的表达。收集表达好的菌体,超声破碎后进行SDS-PAGE检测,结果显示重组表达的SElW蛋白在上清和沉淀里均有表达(图 4A)。离心收集上清后用AKTA纯化仪进行纯化,纯化效果见图 4B。对纯化的SElW蛋白进行定量,得到重组蛋白的浓度为0.622 mg/ml。
|
| 注:A:重组pET-28a(+)-selw诱导表达:1:Marker;2:未诱导空质粒;3:诱导空质粒;4:未诱导重组质粒;5:破碎后上清;6:破碎后沉淀;B:SElW蛋白纯化:1:Marker;2:纯化后蛋白 图 4 金黄色葡萄球菌类肠毒素W(SElW)诱导表达及纯化 |
SElW是由核心基因编码的新型超抗原,几乎分布于金黄色葡萄球菌所有的流行克隆系,如CC1、CC5、CC8、CC45、CC121和CC398等,现已发现14个主要的等位基因变异体,氨基酸序列一致性高达92%[3, 9]。SElW是目前预测的,存在于人类和家畜重要流行克隆CC398的唯一超抗原。体外实验表明,该蛋白的缺失可以导致CC398菌株失去促进T细胞增殖活性,表明该蛋白在致病性方面具有重要的功能[11-12]。
AlphaFold是一种新型的、准确性好、精密度高的预测蛋白3D结构的程序,甚至能分析X-射线晶体学很难解决的楔入细胞膜中的蛋白质结构[10],现已得到广泛的应用。ZDOCK使用快速傅里叶变换算法在3D网格上实现高效的全局对接搜索,并利用形状互补性、静电学和统计势项的组合进行评分,在蛋白质-蛋白质刚性对接方面有很高的预测准确性[13]。本研究利用AlphaFold软件对SElW蛋白进行三维结构预测,并借助SAVES在线服务器中的ERRAT、拉氏图和Verify_3D对预测模型进行评估,然后通过ZDOCK对SElW和TCR进行对接,以较高的准确性成功模拟了SElW与TCR的结合方式,并结合SElW和其他肠毒素的氨基酸多序列比对共同预测重要的可能的氨基酸位点。
根据核苷酸或氨基酸序列的相似性,可将超抗原分成5个组[14],本研究的SElW与SEA共同属于第3组[8]。单体预测模型显示,SElW由两个结构域组成,氨基末端的寡糖/寡核苷酸-折叠结构域和羧基末端的β-grasp结构域,相对保守的TCR结合位点位于分隔这两个结构域的浅槽中;两个结构域之间由一个横跨分子中心的结构保守的α-螺旋分割,这与已经报道的超抗原的结构相似[4, 15-17]。SElW的空间结构高度保守,α-螺旋和β-折叠多数位于氨基酸序列的保守区,而不规则卷曲多位于非保守区。
有研究报道,位点Y18和W55可能是SElW与TCR相互作用的重要免疫活性位点[3],本研究的对接结果也显示Y18、W55可与TCR形成氢键。对接构象同时显示位于α-螺旋内的N19可形成氢键,可能影响SElW与TCR的结合,此位点与研究报道的SEA、SEB和SEC3(编号分别为N25、N23、N23)共同超抗原锚点高度保守[18-20],说明此位点对SElW和TCR的相互作用可能具有重要影响。前期的突变研究显示,N25、C96、C106、S206和N207五个位点是SEA与TCR相互作用位点[18, 21-22];N23、F46是SED与TCR作用位点[23]。这些残基的共同特点是单个氨基酸残基的理化性质的变化,即可影响凹槽与抗原肽的结合,如当某氨基酸突变为丙氨酸时,能显著影响T淋巴细胞的增殖和细胞因子的释放。经报道,位于环区上的位点通过氢键和范德华力能影响肠毒素与TCR之间的相互作用[22],而TCR位点中W55和C88均位于环区,并同时与已被报道的SEA相对应的位点保持一致,值得重点关注。根据序列比对后氨基酸位点的保守性及汇总文献数据,本研究预测以下5个位点为重要的可能的TCR结合位点:Y18、N19、W55、C88和C98。但是序列比对发现Y18和W55两个位点与其他肠毒素相对应的位点保守性较低,因此后续还需对不同来源的SElW氨基酸序列进行多态性分析进一步验证Y18和W55的保守性。
综上所述,本研究对SElW与TCR进行分子对接模型预测,结合多序列比对分析总结出了5个需要重点关注的可能的超抗原活性位点,为进一步探索SElW免疫识别机制提供了理论依据;同时运用体外重组技术成功构建并表达了SElW蛋白,为后续构建突变体奠定了基础。
利益冲突 所有作者声明无利益冲突
作者贡献声明 杨玉花:实验设计、实施研究、分析数据、论文撰写;库鑫、宫雅楠、孟凡亮、卜东波:实施研究、采集数据、分析/解释数据;郭亚慧、龙丽瑾、范佳铭:采集数据、分析/解释数据;魏销玥:采集数据、分析/解释数据、技术或材料支持;张茂俊、张建中:课题讨论、论文指导和修改;闫笑梅:课题总体设计、课题讨论、论文指导和修改
| [1] |
Lowy FD. Staphylococcus aureus infections[J]. N Engl J Med, 1998, 339(8): 520-532. DOI:10.1056/nejm199808203390806 |
| [2] |
Hanawa T, Shimoda-Komatsu Y, Araki K, et al. Skin and soft tissue infections caused by different genotypes of PVL-positive community-acquired methicillin-resistant Staphylococcus aureus strains[J]. Jpn J Infect Dis, 2020, 73(1): 72-75. DOI:10.7883/yoken.JJID.2019.162 |
| [3] |
Vrieling M, Tuffs SW, Yebra G, et al. Population analysis of Staphylococcus aureus reveals a cryptic, highly prevalent superantigen SElW that contributes to the pathogenesis of bacteremia[J]. mBio, 2020, 11(5): e02082-20. DOI:10.1128/mBio.02082-20 |
| [4] |
Fraser JD, Proft T. The bacterial superantigen and superantigen-like proteins[J]. Immunol Rev, 2008, 225(1): 226-243. DOI:10.1111/j.1600-065X.2008.00681.x |
| [5] |
Krakauer T. Staphylococcal superantigens: pyrogenic toxins induce toxic shock[J]. Toxins (Basel), 2019, 11(3): 178. DOI:10.3390/toxins11030178 |
| [6] |
Liu YX, Chen W, Ali T, et al. Staphylococcal enterotoxin H induced apoptosis of bovine mammary epithelial cells in vitro[J]. Toxins (Basel), 2014, 6(12): 3552-3567. DOI:10.3390/toxins6123552 |
| [7] |
McCormick JK, Yarwood JM, Schlievert PM. Toxic shock syndrome and bacterial superantigens: an update[J]. Annu Rev Microbiol, 2001, 55: 77-104. DOI:10.1146/annurev.micro.55.1.77 |
| [8] |
Okumura K, Shimomura Y, Murayama SY, et al. Evolutionary paths of streptococcal and staphylococcal superantigens[J]. BMC Genomics, 2012, 13: 404. DOI:10.1186/1471-2164-13-404 |
| [9] |
Aung MS, Urushibara N, Kawaguchiya M, et al. Prevalence and genetic diversity of staphylococcal enterotoxin (-like) genes sey, selw, selx, selz, sel26 and sel27 in community-acquired methicillin-resistant Staphylococcus aureus[J]. Toxins (Basel), 2020, 12(5): 347. DOI:10.3390/toxins12050347 |
| [10] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold[J]. Nature, 2021, 596(7873): 583-589. DOI:10.1038/s41586-021-03819-2 |
| [11] |
Uhlemann AC, Porcella SF, Trivedi S, et al. Identification of a highly transmissible animal-independent Staphylococcus aureus ST398 clone with distinct genomic and cell adhesion properties[J]. mBio, 2012, 3(2): e00027-12. DOI:10.1128/mBio.00027-12 |
| [12] |
Uhlemann AC, Mcadam PR, Sullivan SB, et al. Evolutionary dynamics of pandemic methicillin-sensitive Staphylococcus aureus ST398 and its international spread via routes of human migration[J]. mBio, 2017, 8(1): e01375-16. DOI:10.1128/mBio.01375-16 |
| [13] |
Pierce BG, Wiehe K, Hwang H, et al. ZDOCK server: interactive docking prediction of protein-protein complexes and symmetric multimers[J]. Bioinformatics, 2014, 30(12): 1771-1773. DOI:10.1093/bioinformatics/btu097 |
| [14] |
Xu SX, McCormick JK. Staphylococcal superantigens in colonization and disease[J]. Front Cell Infect Microbiol, 2012, 2: 52. DOI:10.3389/fcimb.2012.00052 |
| [15] |
Proft T, Fraser JD. Bacterial superantigens[J]. Clin Exp Immunol, 2003, 133(3): 299-306. DOI:10.1046/j.1365-2249.2003.02203.x |
| [16] |
Rödström KEJ, Regenthal P, Lindkvist-Petersson K. Structure of staphylococcal enterotoxin E in complex with TCR defines the role of TCR loop positioning in superantigen recognition[J]. PLoS One, 2015, 10(7): e0131988. DOI:10.1371/journal.pone.0131988 |
| [17] |
Zeng C, Liu Z, Han Z. Structure of Staphylococcal Enterotoxin N: Implications for Binding Properties to Its Cellular Proteins [J/OL]. Int J Mol Sci, 2019, 20(23): 5921. DOI: 10.3390/ijms20235921.
|
| [18] |
Rödström KEJ, Regenthal P, Bahl C, et al. Two common structural motifs for TCR recognition by staphylococcal enterotoxins[J]. Sci Rep, 2016, 6: 25796. DOI:10.1038/srep25796 |
| [19] |
Fields BA, Malchiodi EL, Li HM, et al. Crystal structure of a T-cell receptor β-chain complexed with a superantigen[J]. Nature, 1996, 384(6605): 188-192. DOI:10.1038/384188a0 |
| [20] |
Li HM, Llera A, Tsuchiya D, et al. Three-dimensional structure of the complex between a T cell receptor β chain and the superantigen staphylococcal enterotoxin B[J]. Immunity, 1998, 9(6): 807-816. DOI:10.1016/s1074-7613(00)80646-9 |
| [21] |
Grossman D, van M, Mollick JA, et al. Mutation of the disulfide loop in staphylococcal enterotoxin A. Consequences for T cell recognition[J]. J Immunol, 1991, 147(10): 3274-3281. DOI:10.4049/jimmunol.147.10.3274 |
| [22] |
Benkerroum N. Staphylococcal enterotoxins and enterotoxin-like toxins with special reference to dairy products: An overview[J]. Crit Rev Food Sci Nutr, 2018, 58(12): 1943-1970. DOI:10.1080/10408398.2017.1289149 |
| [23] |
Li YF, Zhu XH, Huang YH, et al. Mutational analysis of the binding of staphylococcal enterotoxin D to the T cell receptor Vβ chain and major histocompatibility complex class Ⅱ[J]. Immunol Lett, 2006, 105(1): 55-60. DOI:10.1016/j.imlet.2005.12.005 |
 2023, Vol. 44
2023, Vol. 44