文章信息
- 王玉琢, 沈洪兵.
- Wang Yuzhuo, Shen Hongbing
- 基于多组学的肿瘤病因学研究:从基因组学、暴露组学、代谢组学到系统流行病学
- Multi-omics approaches for revealing the etiology of cancer: from genomics, exposomics, metabolomics to system epidemiology
- 中华流行病学杂志, 2023, 44(4): 521-528
- Chinese Journal of Epidemiology, 2023, 44(4): 521-528
- http://dx.doi.org/10.3760/cma.j.cn112338-20221201-01026
-
文章历史
收稿日期: 2022-12-01
2. 中国疾病预防控制中心, 北京 102206
2. Chinese Center for Disease Control and Prevention, Beijing 102206, China
肿瘤的发生受到遗传和环境因素的共同影响。近年来,随着基因组、暴露组、转录组、蛋白质组、代谢组等组学技术的发展,肿瘤病因学研究进入系统流行病学研究阶段,即基于基因组学、暴露组学和代谢组学等为代表的多组学肿瘤病因学研究。从遗传方面,基因组学研究阐明肿瘤易感位点及其致病机制。从环境方面,暴露组学研究探索环境因素对机体生物学过程的影响及其对肿瘤发病风险的效应。遗传和环境因素作用于机体后,可引起表观基因组、转录组、蛋白质组、代谢组等组学分子标志物的改变。其中代谢组处于生物调控网络的下游,反映了基因、环境及其交互作用所产生的总体生物学效应,是对机体生理或疾病状况的直接评估(图 1)。基因组、转录组、蛋白质组等层面的功能性微小变化可以在代谢组水平上进一步放大,引起代谢物水平的改变[1]。因此,代谢组学研究不仅能够有效识别与肿瘤发生密切相关的生物标志物,还可以全面揭示肿瘤相关的代谢网络失调,有助于深入理解肿瘤的发病机制[2-3]。本文介绍基因组学、暴露组学和代谢组学在肿瘤病因学研究中的应用和进展,总结多组学和系统流行病学在肿瘤病因学研究中的意义和价值,展望未来发展方向。
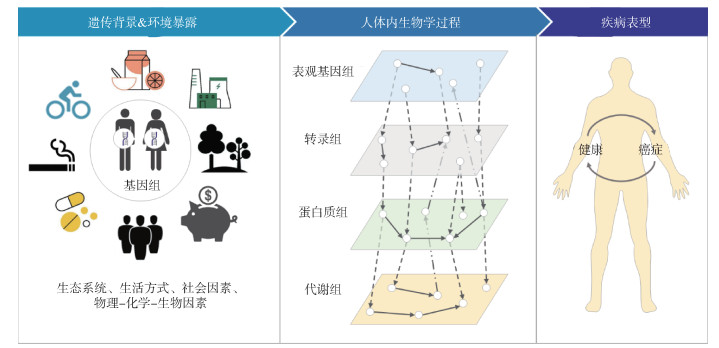
|
| 图 1 多组学与系统流行病学分析框架 |
基因组学是对生物体全基因组的研究[4]。基因组变异分为单核苷酸变异和结构变异,结构变异又分为染色体倒位或异位、小片段插入/丢失和拷贝数变异。本文关注基因组遗传变异对肿瘤发病风险的影响。遗传因素在肿瘤发生过程中具有重要作用[5]。21世纪以来,伴随着人类基因组计划的完成和高通量基因分型技术的发展,全基因组关联研究(genome-wide association study,GWAS)应运而生,使研究者能够从基因组学层面探讨恶性肿瘤的遗传易感机制。迄今为止,GWAS报道了超过3 000个与肿瘤发病风险相关(P < 5×10-8)的遗传变异,表明肿瘤遗传易感性与基因组范围内数量众多的低外显率常见变异有关[6]。GWAS发现的易感位点可用于肿瘤风险预测和致病机制探索。
联合多个遗传易感位点构建多基因风险评分(polygenic risk scores,PRSs)是量化个体肿瘤遗传风险的有效方式,也是现阶段肿瘤流行病学研究的热点之一[7]。近年来,一系列研究利用肿瘤GWAS和大规模人群队列,成功构建并评价了多种常见恶性肿瘤的PRSs,发现PRSs有利于肿瘤风险分层。结合PRSs与传统危险因素能够优化肿瘤风险预测模型的预测效能。在肿瘤精准预防领域,PRSs可优化高危人群筛选标准,提高肿瘤筛查的成本效益;同时有助于引导个体采用健康生活方式,预防或延缓肿瘤的发生[8]。
GWAS发现的易感位点可帮助识别潜在的肿瘤发病机制。在GWAS报道的肿瘤易感位点中,少数位于外显子区、剪接位点和3'非翻译区(3' untranslated region,3'UTR)。此类遗传变异的生物学作用机制相对明确,可能通过影响蛋白质结构及功能、调控基因剪接和RNA转录后加工来改变肿瘤发病风险,例如位于外显子区的肺癌易感位点rs11571833 (BRCA2 p.Lys3326X) 和rs17879961 (CHEK2 p.Ile157Thr)[9]、位于剪接位点的乳腺癌和卵巢癌易感位点rs10069690(TERT)[10]以及位于3'UTR的前列腺癌和神经胶质瘤易感位点rs78378222(TP53)[11-12]。然而,大多数肿瘤易感位点位于非编码区或基因间区,这对易感位点的功能解读和转化应用构成了挑战。
值得关注的是,GWAS发现的易感位点富集于启动子、增强子和转录因子结合区等基因组调控元件[13],可通过调控特定组织或细胞的DNA甲基化、组蛋白修饰、基因表达、蛋白质丰度、代谢物水平等“组学”特征来影响肿瘤发病风险[14-15]。因此,整合“遗传变异-组学特征”与“遗传变异-肿瘤风险”的关联信息有助于推断肿瘤易感位点调控的分子靶标,破译肿瘤发生的生物学机制,这依赖于组学数据的累积和新兴统计方法的开发。近年来,一系列研究系统评估了人类基因组遗传变异对组学特征的影响,包括表观基因组学[16-17]、转录组学[18]、蛋白质组学[19-20]和代谢组学[21-22]等,发现了一批与组学特征有关的数量性状位点(quantitative trait loci,QTLs)。
孟德尔随机化(Mendelian randomization,MR)、共定位分析和全转录组关联研究(transcriptome-wide association study,TWAS)等方法学的出现则进一步推动了多组学与GWAS数据的整合。例如,Prince等[23]利用外周血表达数量性状位点(expression quantitative trait loci,eQTLs)和肿瘤GWAS(包括乳腺癌、卵巢癌和前列腺癌)对蛋白质编码基因开展全转录组MR研究,发现了一系列肿瘤易感基因。Beesley等[24]基于癌症基因组图谱(the Cancer Genome Atlas,TCGA)项目、基因型组织表达项目(the Genotype-Tissue Expression project,GTEx)eQTL数据以及乳腺癌GWAS,采用共定位分析发现了17个潜在的乳腺癌易感基因;其中NTN4基因中的乳腺癌易感位点rs61938093位于增强子区,通过增强子-启动子相互作用调控NTN4基因的表达水平,进而影响乳腺癌发病风险。Guo等[25]利用TCGA和GTEx结肠组织的基因组、转录组数据以及结直肠癌GWAS开展TWAS,发现了25个结直肠癌易感基因,其中包含4个新发现的结直肠癌易感位点;功能实验显示易感位点rs1741640通过影响CABLES2基因启动子活性调控该基因的表达,细胞和动物实验证实CABLES2基因在结直肠癌发生过程中具有重要作用。此外,Tian等[26]系统整合了TCGA项目eQTLs和结直肠癌GWAS数据,鉴定出染色体11q12.2区域内的潜在致病变异rs174575;功能实验显示rs174575可在转录因子E2F1的协助下通过远程增强子-启动子相互作用调控FADS2基因和长链非编码RNA AP002754.2的表达水平,AP002754.2可激活转录因子并进一步上调FADS2基因表达水平;FADS2基因高表达于结直肠癌肿瘤组织,通过促进致癌分子PGE2的代谢增强结直肠癌细胞的增殖能力;该研究系统整合基因组和转录组,成功揭示出易感位点rs174575的靶基因及其分子调控网络,为理解结直肠癌的发病机制提供了新见解。肿瘤GWAS和组织特异性QTL数据的不断累积和开放共享将为组学数据的整合分析提供宝贵的资源和机遇,有利于鉴定肿瘤发生过程中的关键分子事件、阐明肿瘤发病机制,为肿瘤的精准预防和精准治疗提供靶点。
二、肿瘤暴露组学研究环境因素可影响肿瘤发病风险。据估计,2014年中国20岁以上人群中45.2%的恶性肿瘤死亡病例归因于可改变的环境危险因素[27]。鉴定肿瘤环境危险因素,进而减少或消除有害暴露,对于肿瘤防治具有战略性意义。大多数环境暴露与肿瘤的关联研究仅考虑一种或一类暴露,研究效率较低。暴露组学概念的提出、新兴环境暴露检测技术的涌现、统计学和生物信息分析技术的发展使从组学层面探索环境暴露对肿瘤发病风险的影响成为可能。
近年来,肿瘤暴露组学研究日益增多,并取得了一定的进展。Saberi等[28]在欧洲癌症和营养前瞻性调查队列(475 426名,中位随访时间14.86年,2 402名发生B细胞淋巴瘤)中通过体格检查和问卷调查获取人体测量学、生活方式、社会经济状况和既往病史等外暴露组信息,在此基础上开展全暴露组关联研究以系统探讨B细胞淋巴瘤的危险因素。其中LASSO-Cox模型发现身高、体重、臀围、乳制品、糖和动物脂肪摄入与B细胞淋巴瘤发病风险增高有关,而摄入水果、坚果、多不饱和脂肪酸和视黄醇则能降低B细胞淋巴瘤发病风险[28]。除了表征外暴露组以外,一系列研究通过高通量组学技术测量内暴露组,评估组学特征与恶性肿瘤发病风险之间的关联[29-30]。其他研究则对外暴露和内暴露进行整合,分析外暴露对内暴露组学标志物的影响及其致病机制[31-33]。例如,苯并芘是一种可导致肺癌发病风险增高的致癌物,但其致病机制尚不清楚。Meng等[33]在2项肺癌病例对照研究(231对肺癌病例和对照)中,分别使用ELISA和全基因组甲基化芯片测量血浆苯并芘二氢二醇环氧化物-白蛋白(BPDE-Alb)加合物和外周血DNA甲基化水平,发现15个CpGs位点的甲基化水平与BPDE-Alb加合物有关,其中位于UBE2O、SAMD4A、ACBD6、DGKZ和SLFN13基因的5个CpGs位点甲基化水平能够影响肺癌发病风险并共同介导了58.2%的BPDE-Alb加合物对肺癌发病风险的效应,提示血浆DNA甲基化水平改变是苯并芘暴露诱导肺癌发生的潜在生物学机制。
目前,欧美国家已经建立了多个暴露组学研究项目,例如欧盟人类生命早期暴露组项目[34]、EXPOsOMICS研究[35]、人类生物监测项目[36]、美国人类暴露组研究中心[37]和人类健康暴露分析资源项目[38],探讨人群外暴露、内暴露特征及其与肿瘤等健康结局之间的关系。我国暴露组学研究仍鲜有报道。与国外相比,我国环境污染来源、暴露物组分、个人生活习惯以及个体对环境暴露的敏感性均具有一定差异,因此亟需进行我国人群暴露组及其健康效应的评估工作[39]。
三、肿瘤代谢组学研究代谢异常是恶性肿瘤的核心标志之一,能够有效支持肿瘤细胞增殖,在肿瘤发生过程中发挥重要作用[40]。代谢组学研究不仅能够有效识别与肿瘤密切相关的生物标志物,还可以全面揭示肿瘤相关的代谢网络失调,有助于深入理解肿瘤的发病机制[2-3]。近年来,国内外学者开展了多项肿瘤代谢组学研究。例如,Seow等[41]在中国上海女性健康研究队列的非吸烟人群中,采用超高效液相色谱-质谱(liquid chromatography-mass spectrometry,LC-MS)和核磁共振(nuclear magnetic resonance,NMR)技术对275例肺癌病例和289例对照的尿液代谢组进行非靶向检测,发现5-甲基-2-糠酸与非吸烟女性肺癌发病风险降低有关;通路分析提示一碳代谢、核苷酸代谢、氧化应激和炎症等可能参与非吸烟女性肺癌的发生。一项基于法国人群队列的巢式病例对照研究采用非靶向NMR技术检测了791对乳腺癌病例和对照的基线血浆代谢组,发现在绝经前的女性人群中血浆N-乙酰糖蛋白、乙醇、组氨酸、甘油、鸟氨酸、亮氨酸、白蛋白、谷氨酰胺、谷氨酸、丙酮酸浓度与乳腺癌发病风险呈正相关;而在总人群和绝经后女性人群中则未发现与乳腺癌发病风险显著相关的代谢物[42]。另一项基于α-生育酚、β-胡萝卜素癌症预防队列的巢式病例对照研究(523对前列腺癌病例和对照)采用LC-MS平台对空腹血清中的860种已知代谢物进行检测和分析,发现参与氧化还原代谢的硫代脯氨酸、胱氨酸和半胱氨酸能够降低前列腺癌的发病风险;相反地,亮氨酰甘氨酸和γ-谷氨酰氨基酸则与前列腺癌风险增加有关[43]。Hang等[44]在2项巢式病例对照研究(包括南通队列108对肝癌病例对照和常州队列55对肝癌病例对照)中,采用非靶向LC-MS技术对受试者的血浆代谢组进行检测,发现12种雄激素或孕激素、8种胆汁酸、10种氨基酸、6种磷脂和8种其他类型的代谢物与肝癌发病风险有关;利用肝癌发病风险独立相关的血浆代谢物构建风险预测模型,模型在训练集(南通队列)和验证集(常州队列)的受试者工作特征曲线下面积(area under the curve,AUC)分别为0.87(95%CI:0.82~0.92)和0.86(95%CI:0.80~0.93),提示血浆代谢物在恶性肿瘤风险预测方面具有潜在应用价值。其他恶性肿瘤如结直肠癌[45]、胰腺癌[46-47]、卵巢癌[48]、胃癌[49]等,国际上也有相应的代谢组学研究报道。
四、肿瘤基因组学、暴露组学与代谢组学的整合肿瘤的发生是多因素、多阶段的复杂生物学过程。单个组学研究只能在单一水平上识别肿瘤相关生物标志物。若要理解肿瘤发生过程的全貌,需要采取多组学整合研究策略[50]。近年来,在肿瘤病因学研究领域,国内外学者开展了一系列多组学整合研究。
Bar等[51]基于一项前瞻性队列研究进行基因组学、暴露组学与代谢组学的整合分析。该队列招募了491名健康成年人,采用基因芯片对血液DNA进行全基因组基因型检测,通过非靶向LC-MS对血清标本进行代谢组检测,利用宏基因组测序和16S rRNA基因测序对粪便标本进行肠道微生物组检测,并采用问卷调查、智能手机应用程序和体格检查的方式收集队列成员生活方式、临床特征和人体测量学信息。研究者通过梯度提升决策树算法构建了基于基因组、暴露组和肠道微生物组的代谢物预测模型,分析各因素对血清代谢物浓度的影响程度,发现饮食因素对血清代谢物浓度的影响最大,共计335种代谢物与饮食有关,例如饮用咖啡能够影响血清副黄嘌呤、咖啡因等黄嘌呤代谢通路成员的浓度,长期食用鱼类则能够准确预测3-羧基-4-甲基-5-丙基-2-呋喃丙酸等血清脂质水平[51]。
另一项研究招募了6 136名芬兰人,采用Metabolon Discovery HD4质谱平台测定血浆代谢组,同时利用基因芯片结合基因型填补检测基因组遗传变异[52]。该研究针对1 391种血浆代谢物分别进行GWAS分析,进而整合FinnGen GWAS数据库(包含17.7万名芬兰人),通过共定位分析发现248种血浆代谢物相关遗传变异能够影响105种疾病的易感性,其中位于羟基赖氨酸激酶基因外显子区的错义突变rs201135688可能通过调控血浆5-羟基赖氨酸浓度而影响肺癌发病风险[52]。
Bai等[53]基于代谢组学对环境-代谢-肿瘤病因链进行了探索。血浆锌浓度升高与肺癌发病风险降低有关,为了探索锌暴露影响肺癌发病风险的潜在生物学机制,该研究设计了一项巢式病例对照研究,包含来自东风-同济队列的肺癌病例101名和与之匹配的健康对照202名。研究者使用电感耦合等离子体质谱和非靶向超高效LC-MS技术分别测定血浆锌元素和代谢物浓度,应用广义线性模型发现55种血浆代谢物与锌元素浓度呈线性剂量反应关系,其中血浆鞘磷脂浓度与肺癌发病风险呈负相关,并且锌元素对肺癌发病风险效应的41.7%是由血浆鞘磷脂所介导,这为锌元素与肺癌发病风险之间的关联机制提供了新见解。此外,与传统因素(包括年龄、性别、BMI、吸烟包年、饮酒和恶性肿瘤家族史)构成的风险预测模型相比,增加血浆鞘磷脂浓度可改善模型的预测效能,使AUC从0.663提高到0.714[53]。
因此,整合基因组学、暴露组学与代谢组学有望发现遗传、环境因素所导致的下游代谢网络的异常,进而揭示肿瘤发病机制。然而,目前的研究仅限于对“遗传/环境因素-组学标志物-肿瘤发生”病因链的探索,而未能揭示遗传、环境以及下游代谢物在肿瘤发病过程中的复杂相互作用和病因网络。因此,亟需采用系统流行病学研究策略来开展基于多组学的肿瘤病因学研究。
五、展望 (1) 多组学和系统流行病学研究将进一步揭示肿瘤发生发展的全貌随着高通量组学技术、机器学习算法和计算机科学的发展,组学研究障碍逐渐消除,为肿瘤病因学探索提供了更广阔的空间。大多数恶性肿瘤的发生是多因素、多阶段长期相互作用的结果,受到遗传和环境因素的共同影响,其作用于机体后会引起表观基因组、转录组、蛋白质组、代谢组等组学层面的改变,因此需要收集疾病相关组织在多个时间点的多组学数据进行整合分析才能洞悉肿瘤发病机制。多组学整合分析将来自不同组学的数据进行归一化处理和整合分析,对生物过程从基因、转录、蛋白质和代谢水平进行深入探究,能够更清晰地了解从致病因素到肿瘤结局之间多个组学层级的信息流变化[50]。这与系统流行病学的思想一致。系统流行病学利用系统生物学、流行病学、计算数学等技术,依托人群队列研究,将人体从暴露组、基因组、表观基因组、转录组、蛋白质组、代谢组等,再到临床表型组的各个层次有机地整合在一起,在分子、细胞、组织、人群社会行为和生态环境等多水平、多组学上深入研究多层次因素间复杂的相互作用及其网络关系,实现人群层面“遗传/环境因素-组学标志物-肿瘤发生”的病因学推断,并构建以病因网络为基础的疾病风险预测模型[54-55]。多组学和系统流行病学研究有助于进一步揭示肿瘤发生发展的全貌,推动肿瘤的精准预防。
(2) 基于多组学的肿瘤病因学研究需要队列资源的累积与数据共享多组学和系统流行病学研究依赖于多层次、全组学设计的前瞻性人群队列[54]。使用先进的技术收集队列成员的暴露组和表型组信息,进行多次动态随访追踪队列成员的健康结局,同时采集和储存多时点生物样本,开展多组学检测,通过前瞻性观察比较具有不同遗传背景和暴露水平的人群疾病发生率的差异,可以获得真实世界中基因、环境及其交互作用对疾病发生的影响并阐释其作用机制[56]。
随着各国政府的重视以及现代信息化技术的发展,国际上已经建立了一批具有生物样本库的超大规模人群队列,例如英国生物样本库(UK Biobank)、美国百万老兵项目(Million Veteran Program)和美国“All of Us”计划[57]。我国的代表性大型人群队列包括中国慢性病前瞻性研究(China Kadoorie Biobank)、泰州人群健康跟踪调查(Taizhou Longitudinal Study)队列,以及国家重点研发计划支持下的百万级自然人群健康队列和重大疾病专病队列[55]。人群队列资源的累积和数据共享为将来开展全组学流行病学研究创造了条件。
(3) 多组学和系统流行病学研究需要创新性地开发组学分析工具传统的多组学整合统计方法包括相关性分析(如Pearson和Spearman相关分析)、多元分析(如偏最小二乘法、主成分分析)和通路分析[50]。网络分析法是系统流行病学研究的经典方法,蛋白质-蛋白质相互作用网络、共表达网络、代谢网络、基于Hi-C的基因组相互作用网络等网络模型可用于识别致病因素和通路。网络模型还可以跨越不同的组学层级,例如染色质免疫共沉淀测序技术能够检测并量化特定蛋白质与DNA的结合[58]。在肿瘤研究领域,通过比较患者和非患者的分子网络架构能够发现与肿瘤发生有关的网络节点,识别关键生物标志物[58]。
随着计算资源持续增长以及数学、统计学和计算科学的发展,新的多组学集成算法不断被开发出来。以机器学习和深度学习为代表的人工智能技术在肿瘤多组学研究中的应用日益普及。组学数据通常样本量远低于分子特征数,并且某些特征之间存在相关性,这对算法训练构成了挑战。机器学习算法可以通过特征提取(如主成分分析、线性判别分析、多维尺度分析)和特征选择(如随机森林、支持向量机、最小绝对收缩与选择算子)来实现组学数据降维,从中筛选出重要特征。深度学习使用由执行不同操作的隐藏层构成的神经网络从复杂数据中寻找代表性特征,其性能可超越传统机器学习算法,特别是在高维度、大规模数据分析方面性能优越,为多组学数据整合提供了新方法[59-60]。事实上,深度学习已经被应用于肿瘤组学大数据分析,识别与肿瘤发病相关的生物标志物、揭示肿瘤的发病机制并预测肿瘤发病风险,在肿瘤病因学研究中具有重大的应用价值[61-63]。
(4) 基于多组学的肿瘤病因学研究需要创新结果解读与因果推断方法每个组学数据都存在一定的偏倚和变异度,且多个组学特征之间可能存在高度的相关性,从大量的相关性信号中区分出真实的致病信号已成为一大挑战。研发更先进的数据分析方法、开展大规模人群队列研究、收集多组学数据进行整合分析以及使用细胞和动物实验验证潜在致病因素的功能均有助于识别致病性生物标志物。此外,通过整合多项独立研究的结果获取“综合证据”是评价结果可靠性的重要方法,多项研究结果的一致性程度越高,研究结论就越可靠[55]。
肿瘤多组学研究大多属于观察性流行病学研究范畴,易受残余混杂和反向因果的影响,导致所发现的关联并非因果关系。MR法使用与组学特征具有强相关关系的遗传变异作为工具变量,推断组学特征与结局之间的因果关系,能够克服观察性研究中混杂和反向因果的影响,为因果推断提供有力证据。随着组学特征QTLs数据的累积,MR法有望在多组学研究的因果推断中发挥重要作用[64]。
六、结论肿瘤的发生受到遗传和环境因素的共同影响。肿瘤基因组学和暴露组学研究能够识别基因、环境及其相互作用对肿瘤发病风险的影响。代谢组处于生物调控网络的下游,反映了基因、环境及其交互作用产生的总生物学效应。代谢组学研究有利于发现新的生物标志物,揭示遗传和环境因素在肿瘤发生中的生物学机制。然而,若要全面洞悉肿瘤的发病机制,必须开展肿瘤多组学数据的整合分析和系统流行病学研究,深入探讨多层次因素间复杂的相互作用及其网络关系,实现人群层面“遗传/环境因素-组学标志物-肿瘤发生”的病因学推断,揭示肿瘤发生发展的全貌,推动肿瘤的精准预防。
利益冲突 所有作者声明无利益冲突
| [1] |
Johnson CH, Ivanisevic J, Siuzdak G. Metabolomics: beyond biomarkers and towards mechanisms[J]. Nat Rev Mol Cell Biol, 2016, 17(7): 451-459. DOI:10.1038/nrm.2016.25 |
| [2] |
Schmidt DR, Patel R, Kirsch DG, et al. Metabolomics in cancer research and emerging applications in clinical oncology[J]. CA A Cancer J Clin, 2021, 71(4): 333-358. DOI:10.3322/caac.21670 |
| [3] |
杭栋, 沈洪兵. 代谢组流行病学研究进展[J]. 中华流行病学杂志, 2021, 42(7): 1148-1153. Hang D, Shen HB. Progress in metabolomics epidemiology[J]. Chin J Epidemiol, 2021, 42(7): 1148-1153. DOI:10.3760/cma.j.cn112338-20210413-00310 |
| [4] |
庞元捷, 吕筠, 余灿清, 等. 多组学在慢性病病因学研究中的应用及其进展[J]. 中华流行病学杂志, 2021, 42(1): 1-9. Pang YJ, Lyu J, Yu CQ, et al. A multi-omics approach to investigate the etiology of non-communicable diseases: recent advance and applications[J]. Chin J Epidemiol, 2021, 42(1): 1-9. DOI:10.3760/cma.j.cn112338-20201201-01370 |
| [5] |
Sud A, Kinnersley B, Houlston RS. Genome-wide association studies of cancer: current insights and future perspectives[J]. Nat Rev Cancer, 2017, 17(11): 692-704. DOI:10.1038/nrc.2017.82 |
| [6] |
Buniello A, MacArthur JAL, Cerezo M, et al. The NHGRI-EBI GWAS Catalog of published genome-wide association studies, targeted arrays and summary statistics 2019[J]. Nucleic Acids Res, 2019, 47(D1): D1005-1012. DOI:10.1093/nar/gky1120 |
| [7] |
Ma Y, Zhou X. Genetic prediction of complex traits with polygenic scores: a statistical review[J]. Trends Genet, 2021, 37(11): 995-1011. DOI:10.1016/j.tig.2021.06.004 |
| [8] |
Wang YZ, Zhu M, Ma HX, et al. Polygenic risk scores: the future of cancer risk prediction, screening, and precision prevention[J]. Med Rev, 2021, 1(2): 129-149. DOI:10.1515/mr-2021-0025 |
| [9] |
Wang YF, McKay JD, Rafnar T, et al. Rare variants of large effect in BRCA2 and CHEK2 affect risk of lung cancer[J]. Nat Genet, 2014, 46(7): 736-741. DOI:10.1038/ng.3002 |
| [10] |
Killedar A, Stutz MD, Sobinoff AP, et al. A common cancer risk-associated allele in the hTERT locus encodes a dominant negative inhibitor of telomerase[J]. PLoS Genet, 2015, 11(6): e1005286. DOI:10.1371/journal.pgen.1005286 |
| [11] |
Stacey SN, Sulem P, Jonasdottir A, et al. A germline variant in the TP53 polyadenylation signal confers cancer susceptibility[J]. Nat Genet, 2011, 43(11): 1098-1103. DOI:10.1038/ng.926 |
| [12] |
Enciso-Mora V, Hosking FJ, di Stefano AL, et al. Low penetrance susceptibility to glioma is caused by the TP53 variant rs78378222[J]. Br J Cancer, 2013, 108(10): 2178-2185. DOI:10.1038/bjc.2013.155 |
| [13] |
The ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome[J]. Nature, 2012, 489(7414): 57-74. DOI:10.1038/nature11247 |
| [14] |
Shirai Y, Okada Y. Elucidation of disease etiology by trans-layer omics analysis[J]. Inflamm Regen, 2021, 41(1): 6. DOI:10.1186/s41232-021-00155-w |
| [15] |
Rau CD, Lusis AJ, Wang YB. Systems genetics for mechanistic discovery in heart diseases[J]. Circ Res, 2020, 126(12): 1795-1815. DOI:10.1161/CIRCRESAHA.119.315863 |
| [16] |
Houlahan KE, Shiah YJ, Gusev A, et al. Genome-wide germline correlates of the epigenetic landscape of prostate cancer[J]. Nat Med, 2019, 25(10): 1615-1626. DOI:10.1038/s41591-019-0579-z |
| [17] |
Gong J, Wan H, Mei SF, et al. Pancan-meQTL: a database to systematically evaluate the effects of genetic variants on methylation in human cancer[J]. Nucleic Acids Res, 2019, 47(D1): D1066-1072. DOI:10.1093/nar/gky814 |
| [18] |
The GTEx Consortium. The GTEx Consortium atlas of genetic regulatory effects across human tissues[J]. Science, 2020, 369(6509): 1318-1330. DOI:10.1126/science.aaz1776 |
| [19] |
Sun BB, Maranville JC, Peters JE, et al. Genomic atlas of the human plasma proteome[J]. Nature, 2018, 558(7708): 73-79. DOI:10.1038/s41586-018-0175-2 |
| [20] |
Wu L, Shu X, Bao JD, et al. Analysis of Over 140 000 European descendants identifies genetically predicted blood protein biomarkers associated with prostate cancer risk[J]. Cancer Res, 2019, 79(18): 4592-4598. DOI:10.1158/0008-5472.can-18-3997 |
| [21] |
Di'Narzo AF, Houten SM, Kosoy R, et al. Integrative analysis of the inflammatory bowel disease serum metabolome improves our understanding of genetic etiology and points to novel putative therapeutic targets[J]. Gastroenterology, 2022, 162(3): 828-843.e11. DOI:10.1053/j.gastro.2021.11.015 |
| [22] |
Long T, Hicks M, Yu HC, et al. Whole-genome sequencing identifies common-to-rare variants associated with human blood metabolites[J]. Nat Genet, 2017, 49(4): 568-578. DOI:10.1038/ng.3809 |
| [23] |
Prince C, Mitchell RE, Richardson TG. Integrative multiomics analysis highlights immune-cell regulatory mechanisms and shared genetic architecture for 14 immune-associated diseases and cancer outcomes[J]. Am J Hum Genet, 2021, 108(12): 2259-2270. DOI:10.1016/j.ajhg.2021.10.003 |
| [24] |
Beesley J, Sivakumaran H, Marjaneh MM, et al. eQTL colocalization analyses identify NTN4 as a candidate breast cancer risk gene[J]. Am J Hum Genet, 2020, 107(4): 778-787. DOI:10.1016/j.ajhg.2020.08.006 |
| [25] |
Guo XY, Lin WQ, Wen WQ, et al. Identifying novel susceptibility genes for colorectal cancer risk from a transcriptome-wide association study of 125, 478 subjects[J]. Gastroenterology, 2021, 160(4): 1164-1178.e6. DOI:10.1053/j.gastro.2020.08.062 |
| [26] |
Tian JB, Lou J, Cai YM, et al. Risk SNP-mediated enhancer-promoter interaction drives colorectal cancer through both FADS2 and AP002754.2[J]. Cancer Res, 2020, 80(9): 1804-1818. DOI:10.1158/0008-5472.CAN-19-2389 |
| [27] |
Chen WQ, Xia CF, Zheng RS, et al. Disparities by province, age, and sex in site-specific cancer burden attributable to 23 potentially modifiable risk factors in China: a comparative risk assessment[J]. Lancet Glob Health, 2019, 7(2): e257-269. DOI:10.1016/S2214-109X(18)30488-1 |
| [28] |
Saberi Hosnijeh F, Casabonne D, Nieters A, et al. Association between anthropometry and lifestyle factors and risk of B-cell lymphoma: An exposome-wide analysis[J]. Int J Cancer, 2021, 148(9): 2115-2128. DOI:10.1002/ijc.33369 |
| [29] |
Xu ZL, Sandler DP, Taylor JA. Blood DNA methylation and breast cancer: a prospective case-cohort analysis in the sister study[J]. J Natl Cancer Inst, 2020, 112(1): 87-94. DOI:10.1093/jnci/djz065 |
| [30] |
Kartsonaki C, Pang YJ, Millwood I, et al. Circulating proteins and risk of pancreatic cancer: a case-subcohort study among Chinese adults[J]. Int J Epidemiol, 2022, 51(3): 817-829. DOI:10.1093/ije/dyab274 |
| [31] |
Meng H, Wei W, Li GYN, et al. Epigenome-wide DNA methylation signature of plasma zinc and their mediation roles in the association of zinc with lung cancer risk[J]. Environ Pollut, 2022, 307: 119563. DOI:10.1016/j.envpol.2022.119563 |
| [32] |
Zhou X, Wang LJ, Xiao JR, et al. Alcohol consumption, DNA methylation and colorectal cancer risk: Results from pooled cohort studies and Mendelian randomization analysis[J]. Int J Cancer, 2022, 151(1): 83-94. DOI:10.1002/ijc.33945 |
| [33] |
Meng H, Li GYN, Wei W, et al. Epigenome-wide DNA methylation signature of benzo[a]pyrene exposure and their mediation roles in benzo[a]pyrene-associated lung cancer development[J]. J Hazard Mater, 2021, 416: 125839. DOI:10.1016/j.jhazmat.2021.125839 |
| [34] |
Vrijheid M, Slama R, Robinson O, et al. The human early-life exposome (HELIX): project rationale and design[J]. Environ Health Perspect, 2014, 122(6): 535-544. DOI:10.1289/ehp.1307204 |
| [35] |
Vineis P, Chadeau-Hyam M, Gmuender H, et al. The exposome in practice: Design of the EXPOsOMICS project[J]. Int J Hyg Environ Health, 2017, 220(2 Pt A): 142-151. DOI:10.1016/j.ijheh.2016.08.001 |
| [36] |
Apel P, Rousselle C, Lange R, et al. Human biomonitoring initiative (HBM4EU) -Strategy to derive human biomonitoring guidance values (HBM-GVs) for health risk assessment[J]. Int J Hyg Environ Health, 2020, 230: 113622. DOI:10.1016/j.ijheh.2020.113622 |
| [37] |
Niedzwiecki MM, Miller GW. The exposome paradigm in human health: lessons from the emory Exposome Summer course[J]. Environ Health Perspect, 2017, 125(6): 064502. DOI:10.1289/EHP1712 |
| [38] |
Viet SM, Falman JC, Merrill LS, et al. Human Health Exposure Analysis Resource (HHEAR): A model for incorporating the exposome into health studies[J]. Int J Hyg Environ Health, 2021, 235: 113768. DOI:10.1016/j.ijheh.2021.113768 |
| [39] |
白志鹏, 陈莉, 韩斌. 暴露组学的概念与应用[J]. 环境与健康杂志, 2015, 32(1): 1-9. Bai ZP, Chen L, Han B. Exposome and exposomics: from concepts to application[J]. J Environ Health, 2015, 32(1): 1-9. DOI:10.16241/j.cnki.1001-5914.2015.01.001 |
| [40] |
Ward PS, Thompson CB. Metabolic reprogramming: a cancer hallmark even warburg did not anticipate[J]. Cancer Cell, 2012, 21(3): 297-308. DOI:10.1016/j.ccr.2012.02.014 |
| [41] |
Seow WJ, Shu XO, Nicholson JK, et al. Association of untargeted urinary metabolomics and lung cancer risk among never-smoking Women in China[J]. JAMA Netw Open, 2019, 2(9): e1911970. DOI:10.1001/jamanetworkopen.2019.11970 |
| [42] |
Jobard E, Dossus L, Baglietto L, et al. Investigation of circulating metabolites associated with breast cancer risk by untargeted metabolomics: a case-control study nested within the French E3 N cohort[J]. Br J Cancer, 2021, 124(10): 1734-1743. DOI:10.1038/s41416-021-01304-1 |
| [43] |
Huang JQ, Mondul AM, Weinstein SJ, et al. Prospective serum metabolomic profiling of lethal prostate cancer[J]. Int J Cancer, 2019, 145(12): 3231-3243. DOI:10.1002/ijc.32218 |
| [44] |
Hang D, Yang XL, Lu JY, et al. Untargeted plasma metabolomics for risk prediction of hepatocellular carcinoma: A prospective study in two Chinese cohorts[J]. Int J Cancer, 2022, 151(12): 2144-2154. DOI:10.1002/ijc.34229 |
| [45] |
Rothwell JA, Murphy N, Bešević J, et al. Metabolic signatures of healthy lifestyle patterns and colorectal cancer risk in a European cohort[J]. Clin Gastroenterol Hepatol, 2022, 20(5): e1061-1082. DOI:10.1016/j.cgh.2020.11.045 |
| [46] |
Wang SY, Li M, Yan L, et al. Metabolomics study reveals systematic metabolic dysregulation and early detection markers associated with incident pancreatic cancer[J]. Int J Cancer, 2022, 150(7): 1091-1100. DOI:10.1002/ijc.33877 |
| [47] |
Shishavan NG, Mohamadkhani A, Sepanlou SG, et al. Circulating plasma fatty acids and risk of pancreatic cancer: Results from the Golestan Cohort Study[J]. Clin Nutr, 2021, 40(4): 1897-1904. DOI:10.1016/j.clnu.2020.09.002 |
| [48] |
Zeleznik OA, Eliassen AH, Kraft P, et al. A prospective analysis of circulating Plasma metabolites associated with Ovarian cancer risk[J]. Cancer Res, 2020, 80(6): 1357-1367. DOI:10.1158/0008-5472.CAN-19-2567 |
| [49] |
Huang S, Guo Y, Li ZW, et al. Identification and validation of plasma metabolomic signatures in precancerous gastric lesions that progress to cancer[J]. JAMA Netw Open, 2021, 4(6): e2114186. DOI:10.1001/jamanetworkopen.2021.14186 |
| [50] |
龙智平, 王帆. 多组学整合分析的设计及统计方法在肿瘤流行病学研究中的应用[J]. 中华流行病学杂志, 2020, 41(5): 788-793. Long ZP, Wang F. Study design and statistical methods used for integrative analysis on multi-omics in cancer epidemiology[J]. Chin J Epidemiol, 2020, 41(5): 788-793. DOI:10.3760/cma.j.cn112338-20190624-00461 |
| [51] |
Bar N, Korem T, Weissbrod O, et al. A reference map of potential determinants for the human serum metabolome[J]. Nature, 2020, 588(7836): 135-140. DOI:10.1038/s41586-020-2896-2 |
| [52] |
Yin XY, Chan LS, Bose D, et al. Genome-wide association studies of metabolites in Finnish men identify disease-relevant loci[J]. Nat Commun, 2022, 13(1): 1644. DOI:10.1038/s41467-022-29143-5 |
| [53] |
Bai YS, Cao Q, Guan X, et al. Metabolic linkages between zinc exposure and lung cancer risk: A nested case-control study[J]. Sci Total Environ, 2022, 837: 155796. DOI:10.1016/j.scitotenv.2022.155796 |
| [54] |
黄涛, 李立明. 系统流行病学[J]. 中华流行病学杂志, 2018, 39(5): 694-699. Huang T, Li LM. Systems epidemiology[J]. Chin J Epidemiol, 2018, 39(5): 694-699. DOI:10.3760/cma.j.issn.0254-6450.2018.05.031 |
| [55] |
王玉琢, 马红霞, 靳光付, 等. 大数据时代的流行病学研究: 机遇、挑战与展望[J]. 中华流行病学杂志, 2021, 42(1): 10-14. Wang YZ, Ma HX, Jin GF, et al. Epidemiological research in the big data era: opportunities, challenges and prospectives[J]. Chin J Epidemiol, 2021, 42(1): 10-14. DOI:10.3760/cma.j.cn112338-20201203-01377 |
| [56] |
Shilo S, Rossman H, Segal E. Axes of a revolution: challenges and promises of big data in healthcare[J]. Nat Med, 2020, 26(1): 29-38. DOI:10.1038/s41591-019-0727-5 |
| [57] |
Karczewski KJ, Snyder MP. Integrative omics for health and disease[J]. Nat Rev Genet, 2018, 19(5): 299-310. DOI:10.1038/nrg.2018.4 |
| [58] |
Hasin Y, Seldin M, Lusis A. Multi-omics approaches to disease[J]. Genome Biol, 2017, 18(1): 83. DOI:10.1186/s13059-017-1215-1 |
| [59] |
Reel PS, Reel S, Pearson E, et al. Using machine learning approaches for multi-omics data analysis: A review[J]. Biotechnol Adv, 2021, 49: 107739. DOI:10.1016/j.biotechadv.2021.107739 |
| [60] |
Oh M, Park S, Kim S, et al. Machine learning-based analysis of multi-omics data on the cloud for investigating gene regulations[J]. Brief Bioinform, 2021, 22(1): 66-76. DOI:10.1093/bib/bbaa032 |
| [61] |
Jha A, Quesnel-Vallières M, Wang D, et al. Identifying common transcriptome signatures of cancer by interpreting deep learning models[J]. Genome Biol, 2022, 23(1): 117. DOI:10.1186/s13059-022-02681-3 |
| [62] |
Elmarakeby HA, Hwang J, Arafeh R, et al. Biologically informed deep neural network for prostate cancer discovery[J]. Nature, 2021, 598(7880): 348-352. DOI:10.1038/s41586-021-03922-4 |
| [63] |
Arslan E, Schulz J, Rai K. Machine learning in epigenomics: insights into cancer biology and medicine[J]. Biochim Biophys Acta Rev Cancer, 2021, 1876(2): 188588. DOI:10.1016/j.bbcan.2021.188588 |
| [64] |
王玉琢, 沈洪兵. 孟德尔随机化研究应用于因果推断的影响因素及其结果解读面临的挑战[J]. 中华流行病学杂志, 2020, 41(8): 1231-1236. Wang YZ, Shen HB. Challenges and factors that influencing causal inference and interpretation, based on Mendelian randomization studies[J]. Chin J Epidemiol, 2020, 41(8): 1231-1236. DOI:10.3760/cma.j.cn112338-20200521-00749 |
 2023, Vol. 44
2023, Vol. 44


