文章信息
- 赵振平, 李艳, 王丽敏, 张梅, 黄正京, 张德韬, 刘江美, 毛凡, 周宇畅, 刘亚宁, 聂超, 周脉耕.
- Zhao Zhenping, Li Yan, Wang Limin, Zhang Mei, Huang Zhengjing, Zhang Detao, Liu Jiangmei, Mao Fan, Zhou Yuchang, Liu Yaning, Nie Chao, Zhou Maigeng
- 自组织神经网络在长寿基因研究设计中的应用:巢式病例对照研究样本选择
- Application of self-organizing maps in the design of longevity genetic research: sample selection in a nested case-control study
- 中华流行病学杂志, 2023, 44(2): 326-334
- Chinese Journal of Epidemiology, 2023, 44(2): 326-334
- http://dx.doi.org/10.3760/cma.j.cn112338-20220616-00536
-
文章历史
收稿日期: 2022-06-16
2. 深圳华大生命科学研究院, 深圳 518083
2. BGI Shenzhen, Shenzhen 518083, China
长寿受遗传、环境因素和社会行为等多因素影响[1]。近年来,各国开展的长寿全基因组关联研究(genome-wide association studies,GWAS)报告了诸多长寿相关基因位点,然而迄今为止,除了载脂蛋白E,其他候选基因均未得到稳定重复验证[2-5]。我国已建立十余个百岁或长寿老人队列,但其中GWAS较少[5]。目前已开展的长寿基因研究,由于缺少对照组的真实死亡年龄,通常以采样时的生存年龄作为高龄对照[6-7]。
自组织神经网络(self-organized map,SOM)是一种无监督机器学习技术。该技术通过非参数递归回归,将多因子高维数据项之间复杂的非线性统计关系聚类可视化为二维关系,最大限度地满足分区内部相似性和彼此差异性原则[8-9]。近年来,有研究采用SOM模型识别心血管疾病、抑郁症等人群特征[10-11]。本研究基于2013年中国慢性病及其危险因素监测调查自然队列人群,采用SOM模型综合考虑人口学特征、慢性病患病、生活习惯、社会行为、精神心理等多维度变量,创新筛选样本,组建巢式病例对照设计的GWAS,为探索长寿候选基因和遗传交互作用研究提供参考范式。
对象与方法1. 研究对象:选自2013年中国慢性病及其危险因素监测的调查对象。2013年中国慢性病及其危险因素监测在全国31个省(自治区、直辖市)的298个监测点,纳入了171 567名≥18岁居民。所有调查对象签署知情同意书。调查对象的选取具有全国代表性及分省的代表性[12]。2013年中国慢性病及其危险因素监测采用多阶段分层整群抽样的方法,包括问卷调查、体格测量和实验室检测,获取个体慢性病主要危险因素、主要慢性病患病及控制情况,以及身高、体重、腰围、血压和心率等体格测量指标,血糖、血脂、胰岛素、糖化血红蛋白、血尿酸等生化指标[13]。2013年中国慢性病及其危险因素监测数据与全国死因监测数据关联,获取个体死因和死亡时间,形成了具有全国代表性的自然人群队列。本研究在截至2020年已死亡的研究对象中,采用SOM选取对照个案,对选中的个案开展全基因组测序,测序由项目合作方深圳华大生命科学研究院完成,研究的人类遗传资源采集审批号为国科遗办审字[2021]CJ1519号。
2. 表型变量:①社会经济状况:文化程度和家庭经济状况。②行为习惯和膳食:累计吸烟量定义为吸烟者每天吸烟的包数乘以吸烟的年数;饮酒频率、蔬果摄入、红肉摄入量均基于食物频率调查表测算;身体活动询问纳入工作、农活及家务劳动活动中的高强度和中等强度时间,持续至少10 min,引起呼吸、心率显著或轻度增加的运动时间,看电视、使用电脑和手机的静态行为时间,以及外出步行或骑自行车的交通时间。③自报健康状态和睡眠:自评健康状态为调查对象对健康状况的自我评价;自评口腔健康状况定义为调查对象对口腔健康状况自我评价;睡眠时间为自报一天内睡觉累计时间。④患病综合情况:高血压、糖尿病、急性心肌梗死、卒中、哮喘、恶性肿瘤的患病情况作为综合评分纳入。⑤身体测量:身高测量采用长度为2.0 m、精确度为0.1 cm的机械式身高坐高计(TZG型,中国恒盛体检设备有限公司);体重测量采用最大称重为150 kg、精确度为0.1 kg的杠杆秤(RGT-140型,中国恒盛体检设备有限公司)。BMI为体重除以身高的平方(kg/m2)。⑥实验室检测:采用全自动生化分析仪(COBASc702型,瑞士罗氏公司)检测TC水平和LDL-C(胆固醇氧化酶法)。
3. 结局变量:调查对象的死亡年龄,≥90岁定义为长寿组。
4. SOM模型简介:常用的SOM又被称为Kohonen网络[14-15]。该方法属于人工神经网络的一种,被广泛用作聚类探索性数据分析中的可视化工具。SOM的主要目标是将复杂的高维输入转换为更简单的低维离散输出空间,保留数据中的关系但不保留实际距离[16]。SOM输出空间中节点的位置(即坐标)表示输入空间中包含的固有统计特征。
5. SOM聚类分析:分为数据预处理以及SOM聚类分析两个阶段。预处理阶段,为获取完整并且连续的数据矩阵,首先针对连续型变量处理数据缺失的情况,利用链式方程进行多元插补[17],插补时不纳入年龄。其次,采用多元线性回归建模,将表型变量中的多项分类变量作为固定效应预测年龄,估测多项分类变量合并后对年龄的预测值,实现分类变量转换成连续型变量。聚类分析阶段,将预处理后的数据矩阵输入自组织神经映射的双层神经网络中,该网络通过学习输入数据的特征,逐个将输入的样本分配到竞争层的神经元中,依靠神经元之间的相互竞争,将特征相似的样本纳入相同的神经元,并更新获胜神经元及其邻接神经元的拓扑位置,不断迭代使样本映射到二维的地图上,实现在平面地图中几何距离较近的样本表型信息更相似,从而实现样本的聚类,挑选长寿组和对照组相似表型。
6. 全基因组测序:受试者的全血储存在乙二胺四乙酸抗凝管中,血浆通过离心(1 500 g,10 min)获得,并在-80 ℃下保存。全血提取DNA后,15× 全基因组测序在MGI测序仪上进行。测序后对数据进行质控,利用Picard/BWA61/GATK62流程进行序列比对和变异检测。每个样本生成GVCF文件后,再合并整合进行变异检测。生成初始的变异检测数据后,利用VQSR模块对变异进行质量值预测,单核苷酸多态性(SNPs)与InDel的敏感度阈值分别设置为99.5和95.0。研究保留敏感度低于该阈值,且比对质量 > 40、测序深度 > 2、变异质量 > 2.0、等位基因比例正常(Fisher检验P值的Phred分数 < 60.0)、单倍型分数 < 13.0、突变位置与测序片段末端的距离 > 8.0的SNPs及InDel多态性,供后续分析。
7. GWAS质控:从数据的样本和变异位点两个维度开展。评估和过滤均使用PLINK 1.9软件进行。排除以下样本及位点:①基因型SNPs缺失超过2%;②与表型数据库中记录的遗传性别不同;③常染色体杂合度异常;④具有三级以内亲缘关系的样本;⑤SNPs具有较高的基因型缺失率(> 2%),且偏离了Hardy-Weinberg平衡检验(P≤1e-5)以及性染色体和线粒体上的SNPs;⑥次等位基因频率 < 1%的位点。同时采用PLINK 1.9软件对独立的常染色体上的SNPs进行主成分分析,采用logistic回归分析添加了前3个主成分以及性别作为关联分析的协变量,关联分析的结果P值,使用R 4.1.2软件绘制Q-Q图,计算基因组膨胀系数,以查看人群分层情况。
结果1. 样本特征与纳入流程:从2013年中国慢性病及其危险因素监测选取样本的流程见图 1。纳入巢式病例对照研究的样本共1 019例,年龄M为90.1岁,女性占50.7%,城市居民占46.4%。见表 1。
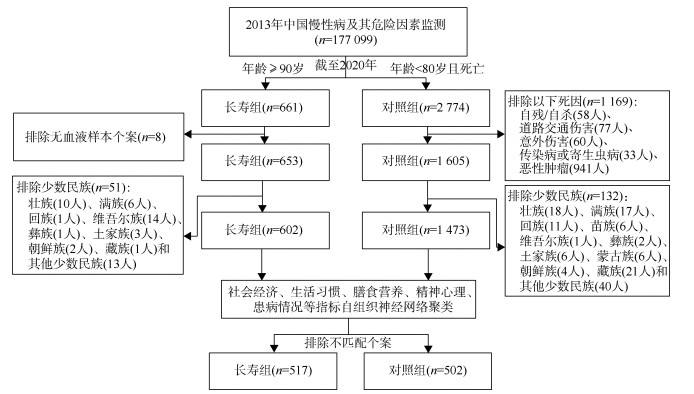
|
| 图 1 巢式病例对照研究的样本筛选流程 |
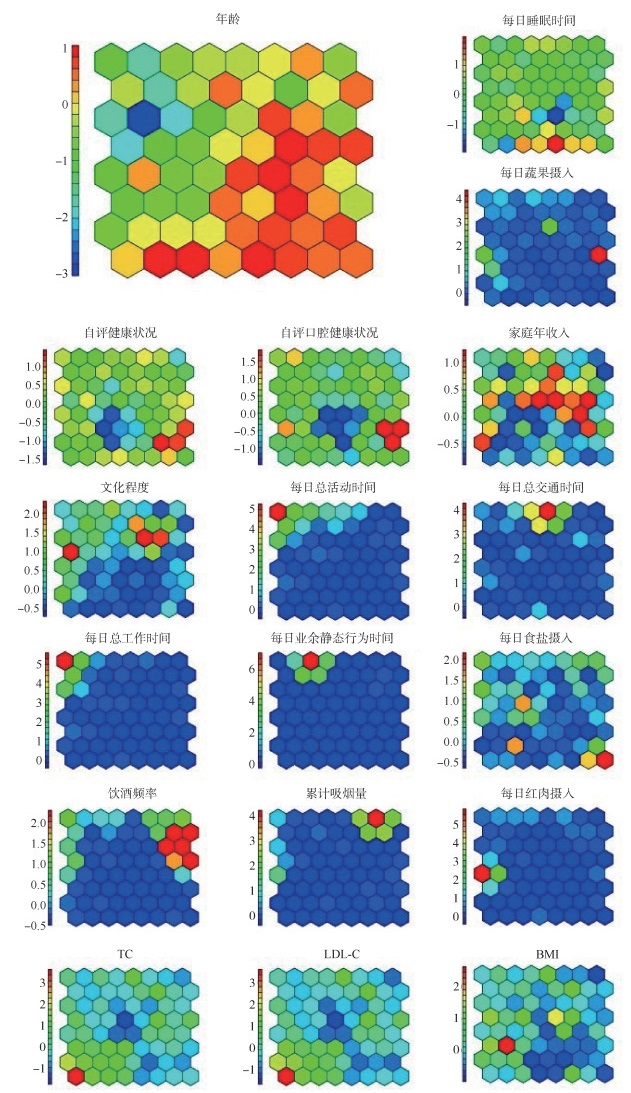
基于SOM的人群分类:用于训练SOM模型的变量及定义见表 2,聚类结果见图 2。不同蜂窝单元中人群的表型指标存在明显的不同,蜂窝图的右侧方位聚集了生活习惯较好的人群,这些位置的柱形图偏向绿色。蜂窝图的中下方位聚集了更多红色柱形图,指示了这些方位的人群具有较差的生活习惯。此种方法实现了人群的精细分类,生活习惯相匹配的人群聚集在同一个蜂窝中。研究逐级针对对照样本进行筛选。首先统计了每个蜂窝中的长寿及早死人群的比例,其次优先排除没有长寿人群匹配只包含早死人群的类别。再次,在筛选后的人群中,挑选表型指标缺失数量 < 2个的数据记录齐全的样本。最终从1 473例早死人群中获得生活习惯与长寿人群匹配的502例年龄 < 80岁的早死对照人群,对照组人群在生活习惯、饮食健康程度以及活动时间方面与长寿人群完全匹配。

|
| 注:背景颜色深浅显示了聚类后的每个蜂窝的人数,黄色越深则该蜂窝人数越多;每个蜂窝图内部包含一张环形柱状图,其长短代表指标的数值大小或严重程度,其中人群自评口腔健康状况、自评健康状况、每日睡眠时间及每日蔬果摄入变量标记为绿色系;每日总运动时间、家庭年收入、文化程度标记为蓝色系;不良生活习惯如累计吸烟量、饮酒频率,每日食盐摄入、每日红肉摄入过多以及高血脂和肥胖等患病情况整合指标标记为红色系;绿色柱越高,代表健康状况及睡眠、饮食状况越好;蓝色柱越高,代表社会经济状况与文化程度越好;红色柱越高,代表不良生活习惯越严重 图 2 自组织神经网络人群分层聚类分析示意图 |
完成样本选择后,进一步对选出样本进行SOM聚类,并详细观测了每个类别中的生活习惯特征(图 3)。本次聚类加入了年龄信息,并针对同一聚类结果,只用每一个表型信息进行染色,可视化人群的表型特征。研究发现长寿与对照人群的吸烟、家庭收入、文化程度、食盐摄入、活动时间、BMI、自评健康等指标呈现一定特征。虽然长寿组吸烟量明显少于对照组,但由于对照组吸烟量变异较大,分析结果显示两组吸烟量差异无统计学意义。与对照组相比,长寿组家庭年收入较低的人群更少,但文化程度较低的人群更多。大部分长寿与对照人群的每日食盐摄入量均值差异无统计学意义,但个别长寿亚组的每日食盐摄入量高于对照组。与对照组相比,长寿组每日总运动时间、每日总交通时间和每日总工作时间更长,但每日业余静态行为时间更短。本研究未发现每日蔬果摄入量、每日红肉摄入量、饮酒频率和累计吸烟量在长寿组和对照组间的关联关系。此外,尽管研究选择了生活习惯、饮食习惯、生理生化指标相对比较接近的长寿组及对照组,但从单一指标来看两组仍有一些差异,因此在基因研究分析中以主成分方法纳入校正。
|
| 注:不同指标蜂窝图中相同位置的蜂窝代表同一个亚组人群,红色、黄色、绿色、青色和蓝色蜂窝分别代表相关指标相对值从高到低;对照年龄分布蜂窝图可解读相关亚组的其他指标分布情况 图 3 样本自组织神经网络聚类指标分层信息可视化示意图 |
2. 全基因组测序质控情况:研究对SOM选择的长寿及对照样本进行了15×全基因组测序,测序数据利用BWA/GATK经典流程进行分析。经质控,4 618 216个SNPs(MAF > 0.01)进入GWAS分析。根据主成分分析结果,排除6个离群样本后,Q-Q图结果见图 4。研究将数据分析结果的Q-Q图与目前已发表的规模最大的长寿全基因组测序研究的Q-Q图进行了对比[18],以验证实验设计优化方法是否可以提升GWAS分析的统计学效力。509例长寿组及475例早死对照组的GWAS的膨胀系数为1.005。结果显示,存在一个全基因组显著(P < 5e-8)的信号,和一些P < 1e-7的遗传位点。同时,本研究结果中,P值在1e-4的区域有明显的小于预期P值的富集,这表明本研究也侦测到了一些有统计学意义的关联信号。
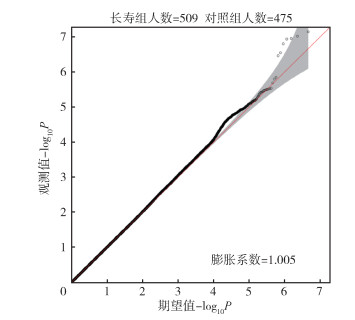
|
| 注:横坐标为假设性状与基因间没有关联情况下的期望P值的负对数,纵坐标为本次全基因组关联分析实际观测到的P值的负对数 图 4 自组织神经网络选择样本进行全基因组测序后长寿与非长寿人群全基因组关联研究的Q-Q图 |
本研究为GWAS提供了创新的对照筛选和匹配策略。本研究基于2013年中国慢性病及其危险因素监测人群,利用非监督机器学习SOM模型,提供了一个利用错综复杂的环境因素、生活习惯特征以及社会经济学因素对长寿人群及早死人群进行聚类匹配的框架,研究为挖掘长寿易感性基因的样本选择提供设计思路。
大规模自然人群队列为长寿基因研究奠定了基础和条件。我国已构建中国慢性病及其危险因素监测创新监测技术体系,可动态掌握我国人群慢性病及其危险因素状况[19]。将大规模自然人群队列拓展基因维度,可以解决流行病学、预防医学等领域长期被忽视的遗传异质性问题,也可以突破仅从基因单一角度研究健康长寿[20]。流行病学、预防医学与基因研究跨领域协同发展,将极大地提高疾病预防和健康干预效益,也为病因学研究提供了新思路。
本研究为从大规模自然人群队列中筛选样本开展GWAS提供了参考范式。近年来,机器学习、深度学习等算法逐步应用至流行病学研究设计或临床试验设计中,有望提高研究的效率和质量[21-22]。本研究通过在高维参数空间中不断迭代匹配样本,最终获得与长寿样本匹配了生活习惯、饮食习惯、生理生化指标最为相似的早死对照样本。通过与已发表的同等样本量水平的长寿基因测序研究进行对比,本研究优化设计后,全基因组关联分析的膨胀系数(1.005)明显优于该项研究(0.98)[18]。同时,本研究中最显著的信号达到全基因组显著水平(P < 5e-8),进一步佐证了SOM方法可以有效地降低混淆因素对后续遗传关联分析的影响,筛选出更有可能具有遗传差异的两组人群,从而在样本量有限的情况下,增加遗传关联分析的统计学效力。
与传统病例对照匹配策略相比,SOM有很多优势。第一,不对变量的分布作出假设,也无需考虑变量间的独立性关系。SOM作为一种无模型聚类分析技术,其优势在于采用数据驱动的方法,即通过学习输入数据的规律性和相关性,对特征相似数据聚类为一组。倾向性评分的测算所纳入自变量与因变量需满足logit回归模型假设,预测概率也需综合考虑协变量交互作用和模型拟合优度。与倾向性评分筛选对照样本策略相比,SOM的优势是不需要考虑纳入变量与因变量的线性关系,也不需要考虑变量之间的交互作用,SOM可根据纳入考虑变量的数据情况,匹配与长寿组关联的对照数量。第二,更容易实现并且能够解决非常复杂的非线性问题。第三,更有效地处理嘈杂和缺失的数据、非常小的维度和无限大小的样本。第四,与传统的聚类方法相比,SOM具有解释度更高,可视化直观效果好等优势。如主成分分析法只能可视化聚类降维后的散点图,而散点图的横、纵坐标并不具备生物学含义,因此很难辨别或描述图中的类别具有的表型特征[23]。SOM算法强制将高维数据映射到二维平面图上,并提供良好的表型可视化方案,实现了数据的分类、筛选以及可视化解读完整的目的,可以作为大规模人群队列筛选巢式病例对照样本的有效工具。
本研究的优势在于考虑了长寿对照人群的真实死亡年龄和死亡原因。国内外已广泛开展长寿基因研究,但长寿研究对照样本多为未死亡的健康人群,且未考虑社会经济和生活行为方式等混杂因素对长寿差异的影响。此种设计方法导致高龄老人和对照组内的异质性较高,潜在的错分偏倚降低了基因组关联分析的效能。目前已发表的长寿队列GWAS分析对照组的设计,导致环境与基因的交互作用引入的噪声太多难以找到目标基因。此外,在众多长寿的遗传研究中,目前对长寿的定义都仅考虑了年龄单一因素,且不同研究对长寿年龄的判定不同。因此,在开展GWAS分析前,应综合考虑社会经济、生活行为方式以细分样本人群,选取最有可能受基因影响的长寿组及早死对照组开展遗传学研究,以提高基因研究效能。
本研究存在局限性。第一,研究只纳入汉族人群,无法考虑少数民族的长寿基因特性。因筛选时发现少数民族样本过少,不具有足够的统计学检验效力,因此在样本筛选过程中只保留汉族人群开展基因研究。第二,研究只考虑基线调查时的社会经济、精神心理和行为习惯,无法考虑随访过程中的相关指标随时间的变化情况。第三,在SOM聚类之后,虽然实现了多变量整体匹配,但是仍然存在少数单个指标在长寿组及早死组中差异显著。其中差异较大的为样本地区分布情况、每日总活动时间和文化程度。由于目前我国各地的流动人口较多,调查地点并不能很好地反映研究对象的祖籍。因此,调查地点的差异不会影响遗传学分析,在后续基因分析中会使用基因型数据完成祖籍的追溯。身体活动时间和文化程度等的差异,主要是由于研究对象的长寿组和对照组的年龄相差一代人,这两代人经历了我国飞速发展的时间段,社会整体变迁导致了两代人之间的生活方式和文化程度的改变。对于组间差别较大的变量,在遗传学研究中会加以校正。
综上所述,本研究为大规模自然人群队列筛选样本开展巢式病例研究提供了方法学参考。采用SOM方法,有利于直观展示影响长寿的环境和社会行为等多维因素,降低高龄老人和早死对照组的组内异质性,提高基因研究效能。该方法也可应用于基于大规模队列研究巢式病例选取样本的其他研究。
利益冲突 所有作者声明无利益冲突
作者贡献声明 赵振平、李艳、张德韬:数据整理、统计学分析、结果解释、论文撰写;王丽敏、张梅、黄正京:监测数据收集与质控;刘江美:多源数据融合整理;毛凡、周宇畅、刘亚宁:实施研究;聂超、周脉耕:项目设计、监督项目实施、审阅文章
志谢 感谢所有参加中国慢性病及其危险因素监测项目的成员和现场工作人员;感谢国家项目组指导;感谢国家卫生健康委员会科学技术研究所的大力支持
| [1] |
Christensen K, Vaupel JW. Determinants of longevity: genetic, environmental and medical factors[J]. J Intern Med, 1996, 240(6): 333-341. DOI:10.1046/j.1365-2796.1996.d01-2853.x |
| [2] |
曾毅. 老龄健康影响因素的跨学科研究国际动态[J]. 科学通报, 2011, 56(35): 2929-2940. Zeng Y. A review on international trends in interdisciplinary research of factors affecting healthy aging[J]. Chin Sci Bull, 2011, 56(35): 2929-2940. |
| [3] |
Deelen J, Evans DS, Arking DE, et al. A meta-analysis of genome-wide association studies identifies multiple longevity genes[J]. Nat Commun, 2019, 10(1): 3669. DOI:10.1038/s41467-019-11558-2 |
| [4] |
Zeng Y, Feng QS, Gu DN, et al. Demographics, phenotypic health characteristics and genetic analysis of centenarians in China[J]. Mech Ageing Dev, 2017, 165: 86-97. DOI:10.1016/j.mad.2016.12.010 |
| [5] |
Deelen J, Beekman M, Uh HW, et al. Genome-wide association meta-analysis of human longevity identifies a novel locus conferring survival beyond 90 years of age[J]. Hum Mol Genet, 2014, 23(16): 4420-4432. DOI:10.1093/hmg/ddu139 |
| [6] |
Zeng Y, Nie C, Min JX, et al. Novel loci and pathways significantly associated with longevity[J]. Sci Rep, 2016, 6(1): 21243. DOI:10.1038/srep21243 |
| [7] |
Zeng Y, Feng QS, Hesketh T, et al. Survival, disabilities in activities of daily living, and physical and cognitive functioning among the oldest-old in China: a cohort study[J]. Lancet, 2017, 389(10079): 1619-1629. DOI:10.1016/S0140-6736(17)30548-2 |
| [8] |
Kohonen T, Hynninen J, Kangas J, et al. SOM_PAK: The self-organizing map program package. Report A31. Helsinki University of Technology, Laboratory of Computer and Information Science[R]. Espoo, 1996.
|
| [9] |
Kohonen T. Self-organizing maps[M]. Berlin: Springer, 1995.
|
| [10] |
Ramezankhani A, Azizi F, Hadaegh F, et al. Sex-specific clustering of metabolic risk factors and their association with incident cardiovascular diseases: a population-based prospective study[J]. Atherosclerosis, 2017, 263: 249-256. DOI:10.1016/j.atherosclerosis.2017.06.921 |
| [11] |
Galkin F, Kochetov K, Keller M, et al. Optimizing future well-being with artificial intelligence: self-organizing maps (SOMs) for the identification of islands of emotional stability[J]. Aging, 2022, 14(12): 4935-4958. DOI:10.18632/aging.204061 |
| [12] |
赵振平, 王丽敏, 李镒冲, 等. 2013年中国慢性病及其危险因素监测系统省级代表性评价[J]. 中华预防医学杂志, 2018, 52(2): 165-169. Zhao ZP, Wang LM, Li YC, et al. Provincial representativeness assessment of China Non-communicable and Chronic Disease Risk Factor Surveillance System in 2013[J]. Chin J Prev Med, 2018, 52(2): 165-169. DOI:10.3760/cma.j.issn.0253-9624.2018.02.009 |
| [13] |
王丽敏, 张梅, 李镒冲, 等. 2013年中国慢性病及其危险因素监测总体方案[J]. 中华预防医学杂志, 2018, 52(2): 191-194. Wang LM, Zhang M, Li YC, et al. Scheme of the Chinese chronic non-communicable disease and risk factor surveillance[J]. Chin J Prev Med, 2018, 52(2): 191-194. DOI:10.3760/cma.j.issn.0253-9624.2018.02.015 |
| [14] |
Kohonen T. Self-organized formation of topologically correct feature maps[J]. Biol Cybern, 1982, 43(1): 59-69. |
| [15] |
Kohonen T. Self-organizing maps[M]. 3rd ed. Berlin: Springer, 2001.
|
| [16] |
Larose DT. Discovering knowledge in data[M]. Hoboken: John Wiley & Sons, 2005.
|
| [17] |
Azur MJ, Stuart EA, Frangakis C, et al. Multiple imputation by chained equations: what is it and how does it work?[J]. Int J Methods Psychiatr Res, 2011, 20(1): 40-49. DOI:10.1002/mpr.329 |
| [18] |
Erikson GA, Bodian DL, Rueda M, et al. Whole-genome sequencing of a healthy aging cohort[J]. Cell, 2016, 165(4): 1002-1011. DOI:10.1016/j.cell.2016.03.022 |
| [19] |
王丽敏, 张梅, 周脉耕, 等. 中国慢性病及危险因素监测创新技术体系构建与应用研究[J]. 中华流行病学杂志, 2021, 42(7): 1154-1159. Wang LM, Zhang M, Zhou MG, et al. Study on construction and application of technology system of chronic diseases and risk factor surveillance in China[J]. Chin J Epidemiol, 2021, 42(7): 1154-1159. DOI:10.3760/cma.j.cn112338-20210104-00002 |
| [20] |
曾毅. 老龄健康的跨学科研究: 社会、行为、环境、遗传因素及其交互作用[J]. 中国卫生政策研究, 2012, 5(2): 5-11. Zeng Y. Interdisciplinary research on healthy aging: Social, behavioral, environmental, genetic factors and their interactions[J]. Chin J Health Policy, 2012, 5(2): 5-11. DOI:10.3969/j.issn.1674-2982.2012.02.002 |
| [21] |
Harrer S, Shah P, Antony B, et al. Artificial intelligence for clinical trial design[J]. Trends Pharmacol Sci, 2019, 40(8): 577-591. DOI:10.1016/j.tips.2019.05.005 |
| [22] |
Weissler EH, Naumann T, Andersson T, et al. The role of machine learning in clinical research: transforming the future of evidence generation[J]. Trials, 2021, 22(1): 537. DOI:10.1186/s13063-021-05489-x |
| [23] |
Chattopadhyay M, Dan PK, Majumdar S. Principal component analysis and self organizing map for visual clustering of machine-part cell formation in cellular manufacturing system[J]. arXiv: 1201.5524, 2012. DOI: |
 2023, Vol. 44
2023, Vol. 44




