文章信息
- 郭翀宇, 王金桃, 冉朝霞, 巩璐, 朱京京, 李德铖, 丁玲.
- Guo Chongyu, Wang Jintao, Ran Zhaoxia, Gong Lu, Zhu Jingjing, Li Decheng, Ding Ling
- HPV16长控制区甲基化与宫颈上皮内瘤变2级及以上病变关系的Meta分析
- The correlation between methylation in HPV16 long control region and cervical intraepithelial neoplasia grade 2 or more: a Meta-analysis
- 中华流行病学杂志, 2022, 43(11): 1821-1827
- Chinese Journal of Epidemiology, 2022, 43(11): 1821-1827
- http://dx.doi.org/10.3760/cma.j.cn112338-20220307-00172
-
文章历史
收稿日期: 2022-03-07
2020年全球癌症统计数据显示,宫颈癌是世界上第四大女性癌症,发病率在中国女性癌症中排第六位,死亡率位列第七位[1],严重威胁着女性的身心健康。高危型HPV的持续感染是宫颈癌及癌前病变的主要致病因素[2],其中,16型是主要致癌型别,在宫颈癌患者中检出率超过60%[3]。HPV16是一种感染上皮细胞的DNA病毒,包含早期编码区(E区,包含E1、E2、E4、E5、E6和E7基因)、晚期编码区(L区,包含L1和L2基因)和长控制区(LCR)[4]。早期基因E2、E6和E7在HPV致癌过程中起着至关重要的作用,其中,E2调控关键致癌基因E6和E7的转录,从而影响宫颈癌的发生发展[5]。晚期基因编码主要衣壳蛋白L1和次要衣壳蛋白L2,参与病毒颗粒的组装,在病毒感染过程中发挥重要作用[4]。LCR是非编码区,对HPV基因复制和表达发挥调控作用,包含5'端、增强子区和启动子区3个功能独立的片段,其中所含4个E2结合位点(E2BS)可与E2进行结合并保证E2正常发挥功能[6]。DNA甲基化作为表观遗传学的主要分子机制,对癌症的发生发展起重要作用[7]。目前,关于HPV16 LCR甲基化与宫颈癌及癌前病变的相关研究较多,但由于宫颈病变病理分期、生物样本类型、人群来源、甲基化检测方法等的不同,研究结果存在差异[8-9]。宫颈上皮内瘤变1级(CIN1)反转率约为60%,大多数患者无需治疗即可自愈,而宫颈上皮内瘤变2级(CIN2)属于高度宫颈癌前病变,发生癌变的风险较高[10],因此,CIN2及以上病变(CIN2+)的发生机制值得深入和系统地探讨。本研究旨在分析HPV16 LCR甲基化与CIN2+发病风险的关系,以期为宫颈癌的发生机制提供进一步的科学依据。
资料与方法1. 文献检索:检索英文数据库(PubMed、Web of Science、Cochrane Library、Medline及Embase),以及中文数据库(中国知网、万方数据知识服务平台、中国生物医学文献数据库和维普数据库),检索期限均为自建库至2022年2月27日。英文检索词包括“HPV 16”“HPV-16”“Human papillomavirus 16”“LCR”“Long Control Region”“URR”“upstream regulatory region”“methylation”“DNA methylation”“Uterine Cervical Neoplasm”“Uterine Cervical Cancer”“Cervical Cancer”“Cervical Neoplasm”“Cancer of the Cervix”“Cervix Cancer”“Cervix Neoplasm”“Cervical Intraepithelial Neoplasia”,中文检索词包括“人乳头瘤病毒”“HPV16”“人乳头瘤病毒16”“LCR”“长控制区”“长调控区”“上游控制区”“上游调控区”“甲基化”“DNA甲基化”“宫颈癌”“宫颈肿瘤”“子宫颈癌”“宫颈上皮内瘤变”。此外,手动检索纳入文献的参考文献,尽可能查找全面。
2. 纳入与排除标准:纳入标准:①公开发表的HPV16 LCR甲基化与宫颈癌变发生发展关系的相关中英文文献;②研究对象为组织或细胞学确诊的正常及HPV16阳性(含单一和多重感染)人群;③研究类型为病例对照研究;④所纳入文献的病例组为CIN2+病变的患者,对照组为宫颈上皮内瘤变1级及以下病变(< CIN2),包括无症状HPV16感染者和(或)CIN1的患者;⑤能从原始文献中直接或间接获得发生甲基化的人数;对同一研究重复报道的文献选择数据最完整的文献。排除标准:①重复报道、资料不完整、数据不全的文献;②仅包含细胞系的研究;③可信度低的研究、个案报告、会议、综述和学位论文等。
3. 文献筛选与文献质量评价:由2名研究人员根据文献纳入排除标准对检索到的所有文献进行独立筛选,对于存在疑问不能确定的文献,经由2人讨论并向第三人咨询决定是否纳入。对于初筛纳入的文献通过纽卡斯尔-渥太华量表(NOS量表)文献质量评价量表进行质量评估[11],评估标准包括研究人群的选择、组间可比性和暴露因素的测量。文献质量评分满分为9分,0~3分为低质量研究,4~6分为中等质量研究,≥7分为高质量研究,本研究只纳入中高质量研究。
4. 资料提取:提取文献资料中的相关数据,数据提取内容包括文献的一般资料(如题目、第一作者、国家、杂志名称、发表时间)、标本类型、甲基化检测方法、病例组和对照组人数以及发生HPV16 LCR甲基化的人数。
5. 统计学分析:采用RevMan 5.3、Stata 15.1软件对数据进行分析。对各研究进行Q检验估计异质性,以OR值及其95%CI作为统计量分析HPV16 LCR的甲基化水平和CIN2+患者之间的关系。若P > 0.10且I2≤50%,提示各研究结果同质性较好,需采用固定效应模型,否则采用随机效应模型。进行亚组分析寻找异质性来源;进行漏斗图制作及Egger's检验来评估发表偏倚;使用敏感性分析测量合并后结果的稳定程度,观察单个研究对总体效应是否存在影响。检验水准α=0.05。
结果1. 文献筛选:根据检索策略和纳入排除标准,对中英文数据库进行全面检索,并追溯研究中包含的参考文献,共检索到180篇文献,经过筛选后,最终纳入17篇。筛选流程见图 1。
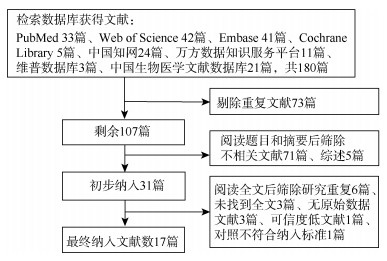
|
| 图 1 文献筛选流程 |
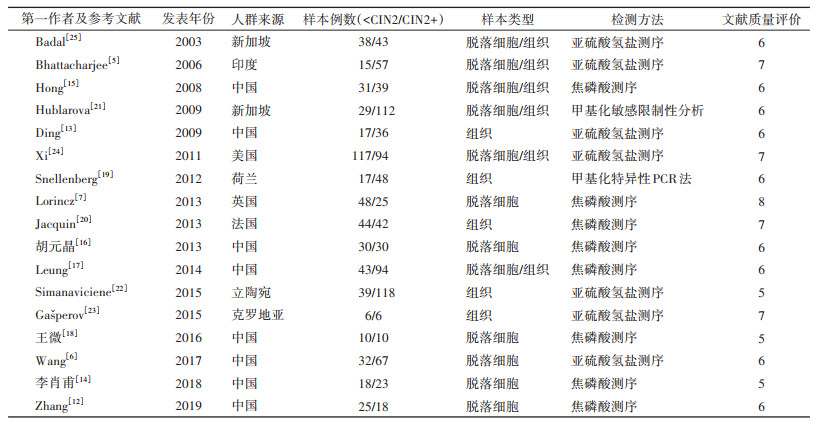
2. 文献的基本特征及质量评价:纳入研究共1 421例研究对象,发表年份为2003-2019年,8项研究人群来自中国[6, 12-18],9项研究人群来自其他国家[5, 7, 19-25]。7项研究采用了亚硫酸氢盐测序的方法[5-6, 13, 22-25],8项研究采用了焦磷酸测序的方法[7, 12, 14-18, 20],1项研究采用了甲基化特异性PCR法[19],1项研究采用了甲基化敏感限制性分析的方法[21]。6项研究样本类型为脱落细胞[6-7, 12, 14, 16, 18],11项研究样本类型为宫颈组织[5, 13, 15, 17, 19-25]。6项研究探讨了HPV16 LCR E2BS的甲基化和宫颈癌变的关系[5, 12, 14, 17, 19-20]。9项研究探讨了HPV16 LCR 5'端、增强子区和启动子区的甲基化和宫颈癌变的关系[5-7, 15-16, 18, 20, 22, 24]。对符合纳入排除标准的文献采用NOS量表对每篇文献的研究质量进行评估。经文献质量评价,共纳入中等质量文献12篇、高质量文献5篇,基本特征及文献质量评价见表 1。
3. Meta分析结果:
(1)HPV16 LCR甲基化与宫颈癌变的关系:将HPV16 LCR甲基化作为有效结局指标,共有17篇文献纳入研究,研究对象共1 421例,其中CIN2+组862例,< CIN2组559例。对所纳入文献进行异质性分析。见图 2。I2=80%,提示纳入的文献存在异质性,采用随机效应模型进行分析,HPV16 LCR甲基化在CIN2+组和 < CIN2组间(OR=1.56,95%CI:0.70~3.47,P=0.280)差异无统计学意义。
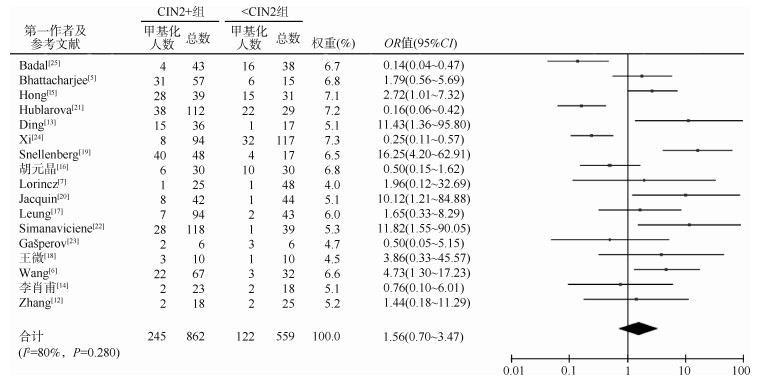
|
| 图 2 HPV16长控制区甲基化与宫颈癌变关系的异质性分析 |
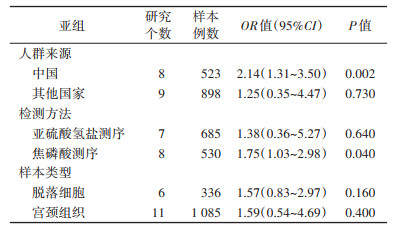
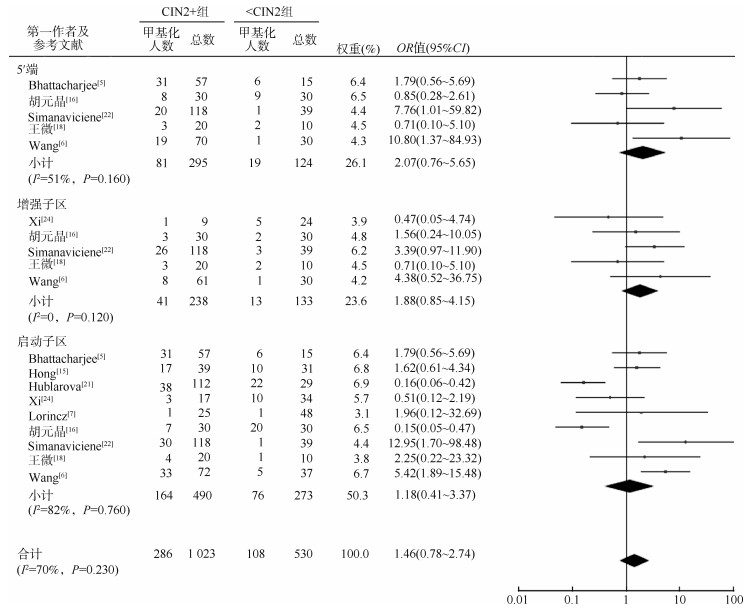
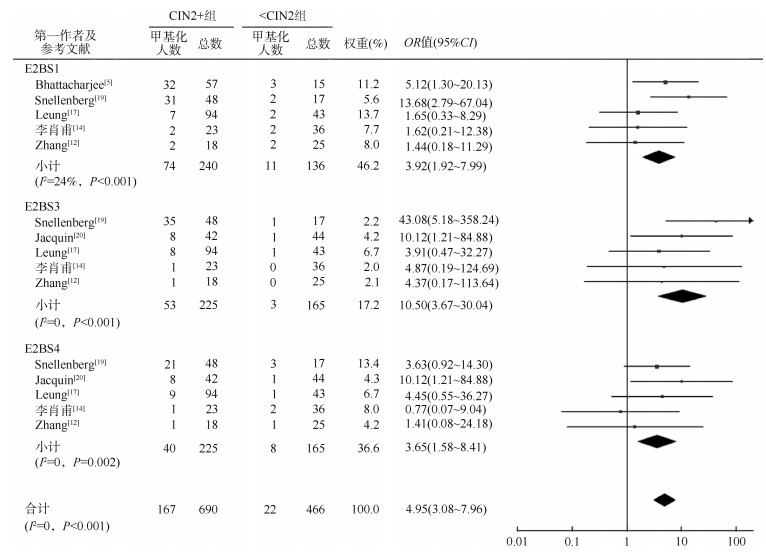
(2)亚组分析:为寻找异质性来源,对LCR 5'端、增强子区和启动子区3个分区进行了亚组分析。由于5'端和启动子区I2 > 50%,故3个分区均采用随机效应模型,结果显示,5'端(OR=2.07,95%CI:0.76~5.65,P=0.160)、增强子区(OR=1.88,95%CI:0.85~4.15,P=0.120)和启动子区(OR=1.18,95%CI:0.41~3.37,P=0.760)在CIN2+组和 < CIN2组间的差异均无统计学意义。见图 3。进一步对LCR内分布的E2BS进行亚组分析,各亚组I2 < 50%,故采用固定效应模型,结果显示,E2BS1(OR=3.92,95%CI:1.92~7.99,P < 0.001)、E2BS3(OR=10.50,95%CI:3.67~30.04,P < 0.001)和E2BS4(OR=3.65,95%CI:1.58~8.41,P=0.002)在CIN2+组和 < CIN2组间的差异均有统计学意义。见图 4。对人群来源进行亚组分析,中国人群CIN2+组的HPV16 LCR甲基化大于 < CIN2组,差异有统计学意义(P=0.002);其他国家人群CIN2+组和 < CIN2组间甲基化差异无统计学意义(P=0.730)。按甲基化检测方法进行亚组分析,采用亚硫酸氢盐测序方法的人群CIN2+组和 < CIN2组间甲基化差异无统计学意义(P=0.640);采用焦磷酸测序方法的人群CIN2+组的HPV16 LCR甲基化大于 < CIN2组,差异有统计学意义(P=0.040)。按样本类型进行亚组分析,样本类型为脱落细胞的人群CIN2+组和 < CIN2组间甲基化差异无统计学意义(P=0.160);样本类型为宫颈组织的人群CIN2+组和 < CIN2组间甲基化差异无统计学意义(P=0.400)。见表 2。
|
| 图 3 HPV16长控制区甲基化与宫颈癌变相关性的异质性分析 |
|
| 图 4 HPV16长控制区E2结合位点甲基化与宫颈癌变相关性的异质性分析 |
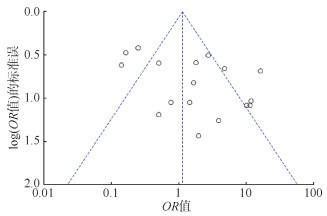
(3)发表偏倚评估:对所纳入研究进行发表偏倚的评估,Egger's检验结果P=0.143,且漏斗图中代表纳入研究发表情况的散点基本对称,表明各研究间无明显的发表偏倚。见图 5。
|
| 图 5 17篇文献的发表偏倚 |
(4)敏感性分析:对所纳入研究进行敏感性分析,未发现存在导致总体效应值明显波动的单个研究。见图 6。鉴于样本量对研究结果的影响,分别去掉小样本原始研究(样本量 < 30人[18, 23]或样本量 < 50人[12, 14, 18, 23])后再次进行敏感性分析,仍未发现存在导致总体效应值明显波动的单个研究,提示本次Meta分析稳健性较好。

|
| 图 6 纳入研究的敏感性分析 |
本研究纳入17篇文献,探讨了HPV16 LCR甲基化与CIN2+发生风险的相关性。LCR的3个功能分区包含基因表达和HPV复制所需的序列基序,其中启动子区可调节病毒癌基因的表达[4]。Meta分析结果显示,HPV16 LCR甲基化与CIN2+的发生风险未显示相关性。Bowden等[26]的Meta分析研究结果与本研究一致,本研究较之进一步纳入了中文文献和年份更近的英文文献,再次证实尚不能认为HPV16 LCR的甲基化程度与CIN2+的发生风险有关。DNA甲基化可能是HPV逃避宿主对外来细胞内因子免疫识别的一种方法,而病毒抵抗基因组的任何表观遗传变化可能是一种自然保护,所以只有少数胞嘧啶-磷酸-鸟嘌呤(CpG)位点具有显著预测疾病的能力[27]。进一步对位于LCR的E2BS进行了亚组分析,结果显示E2BS甲基化可增加CIN2+的发生风险。E2BS含有可能发生甲基化的CpG位点,其发生甲基化会影响E2与E2BS的结合,E2不能正常发挥对关键病毒癌基因E6和E7的转录抑制功能,使得E6和E7转录增强,促进病毒的复制和转录[28-29],最终导致癌变的发生[30]。Cheung等[31]的研究发现,E2BS在宫颈癌中的甲基化率高于低级别宫颈病变,与本研究结果一致。针对E2BS2甲基化的相关研究仅2篇,故未对其进行亚组分析。对人群来源进行的亚组分析结果显示,仅中国人群CIN2+的发生风险与HPV16 LCR甲基化相关,可能是种族、生存环境和生活习惯等原因引起的[32]。对甲基化检测方法进行的亚组分析结果显示,采用焦磷酸测序法进行检测的人群CIN2+的发生风险可能与HPV16 LCR甲基化相关。焦磷酸测序可直接得到目的核酸片段的序列,对多个CpG位点的甲基化状态进行定量评估,更为准确地对具体位点进行定量检测,并且特异性高,是分子诊断的“金标准”[9],而亚硫酸氢盐测序能直接检测分析基因组DNA的甲基化模式,可以检测到甲基化的程度并且明确甲基化的确切位置,但只能定性分析位点的甲基化情况,敏感性较低[33]。
本研究采用NOS量表从研究人群的选择、组间可比性和暴露因素的测量3个方面入手进行文献质量评价,其中5项研究为高质量,其余12项为中等质量,多数研究的组间可比性评分较低,根据评分标准:①研究控制了最重要的混杂因素;②研究控制了任何其他混杂因素,判定所纳入研究是否在设计和统计分析阶段体现病例和对照的可比性。虽然总体评分受到组间可比性这一指标的影响,12项研究评分属于中等质量,但是评分接近中等质量的高线,而且其他指标分数很高,所以研究结果仍具有较高的可信度。
本研究存在局限性。本次纳入的研究限定为病例对照研究,可能存在一定的选择偏倚;且本研究纳入的研究均为中英文,未将其他语言的研究纳入分析,具有一定的片面性;再者,由于检索到的文献大多数呈现的是位点的甲基化率,数据多是间接计算得到,可能是本次Meta分析异质性较大的原因之一,本研究分析异质性来源仅限于LCR各分区、人群来源、检测方法和样本类型,可能还有其他因素对HPV16 LCR甲基化与CIN2+发生风险的关系有影响,尚有待进一步的研究和拓展。
综上所述,本研究结果表明,CIN2+的发生风险与HPV16 LCR整体的甲基化未显示相关性,但可能与E2BS的甲基化有关,中国人群和经焦磷酸测序方法检测的人群CIN2+的发生风险与HPV16 LCR的甲基化具有相关性。本研究探讨了CIN2+的发生风险与HPV16 LCR甲基化的相关性,尚有待纳入更多高质量、大样本的相关研究以得到更准确的结论。
利益冲突 所有作者声明无利益冲突
| [1] |
Cao W, Chen HD, Yu YW, et al. Changing profiles of cancer burden worldwide and in China: a secondary analysis of the global cancer statistics 2020[J]. Chin Med J, 2021, 134(7): 783-791. DOI:10.1097/CM9.0000000000001474 |
| [2] |
Olusola P, Banerjee HN, Philley JV, et al. Human papilloma virus-associated cervical cancer and health disparities[J]. Cells, 2019, 8(6): 622. DOI:10.3390/cells8060622 |
| [3] |
Schiffman M, Solomon D. Cervical-cancer screening with human papillomavirus and cytologic cotesting[J]. N Engl J Med, 2013, 369(24): 2324-2331. DOI:10.1056/NEJMcp1210379 |
| [4] |
Amaro-Filho SM, Chaves CBP, Felix SP, et al. HPV DNA methylation at the early promoter and E1/E2 integrity: a comparison between HPV16, HPV18 and HPV45 in cervical cancer[J]. Papillomavirus Res, 2018, 5: 172-179. DOI:10.1016/j.pvr.2018.04.002 |
| [5] |
Bhattacharjee B, Sengupta S. CpG methylation of HPV16 LCR at E2 binding site proximal to P97 is associated with cervical cancer in presence of intact E2[J]. Virology, 2006, 354(2): 280-285. DOI:10.1016/j.virol.2006.06.018 |
| [6] |
Wang W, Sun ZR, Liu JH, et al. Increased methylation of human papillomavirus type 16 DNA is associated with the severity of cervical lesions in infected females from northeast China[J]. Oncol Lett, 2017, 13(5): 3809-3816. DOI:10.3892/ol.2017.5903 |
| [7] |
Lorincz AT, Brentnall AR, Vasiljević N, et al. HPV16 L1 and L2 DNA methylation predicts high-grade cervical intraepithelial neoplasia in women with mildly abnormal cervical cytology[J]. Int J Cancer, 2013, 133(3): 637-644. DOI:10.1002/ijc.28050 |
| [8] |
Senapati R, Senapati NN, Dwibedi B. Molecular mechanisms of HPV mediated neoplastic progression[J]. Infect Agents Cancer, 2016, 11(1): 59. DOI:10.1186/s13027-016-0107-4 |
| [9] |
Clarke MA, Wentzensen N, Mirabello L, et al. Human papillomavirus DNA methylation as a potential biomarker for cervical cancer[J]. Cancer Epidemiol Biomarkers Prev, 2012, 21(12): 2125-2137. DOI:10.1158/1055-9965.EPI-12-0905 |
| [10] |
Moore LD, Le T, Fan GP. DNA methylation and its basic function[J]. Neuropsychopharmacology, 2013, 38(1): 23-38. DOI:10.1038/npp.2012.112 |
| [11] |
Stang A. Critical evaluation of the Newcastle-Ottawa scale for the assessment of the quality of nonrandomized studies in meta-analyses[J]. Eur J Epidemiol, 2010, 25(9): 603-605. DOI:10.1007/s10654-010-9491-z |
| [12] |
Zhang XA, Zhi YF, Li Y, et al. Study on the relationship between methylation status of HPV16 E2 binding sites and cervical lesions[J]. Clin Chim Acta, 2019, 493: 98-103. DOI:10.1016/j.cca.2019.02.027 |
| [13] |
Ding DC, Chiang MH, Lai HC, et al. Methylation of the long control region of HPV16 is related to the severity of cervical neoplasia[J]. Eur J Obstet Gynecol Reprod Biol, 2009, 147(2): 215-220. DOI:10.1016/j.ejogrb.2009.08.023 |
| [14] |
李肖甫, 樊婷婷, 智艳芳, 等. HPV-16 E2结合位点甲基化水平与宫颈病变的相关性研究[J]. 现代妇产科进展, 2018, 27(4): 259-262, 267. Li XF, Fan TT, Zhi YF, et al. Study on the correlation between methylation status of HPV-16 E2 binding sites and cervical lesions[J]. Prog Obstet Gynecol, 2018, 27(4): 259-262, 267. DOI:10.13283/j.cnki.xdfckjz.2018.04.004 |
| [15] |
Hong D, Ye F, Lu WG, et al. Methylation status of the long control region of HPV16 in clinical cervical specimens[J]. Mol Med Rep, 2008, 1(4): 555-560. DOI:10.3892/mmr.1.4.555 |
| [16] |
胡元晶, 曲芃芃. HPV16 L1基因3'端和LCR基因甲基化与其致病性的关系[J]. 中华妇产科杂志, 2013, 48(8): 607-610. Hu YJ, Qu PP. Relationship between the methylation of L1 3' and long control region gene of HPV16 DNA and it's pathogenicity[J]. Chin J Obstet Gynecol, 2013, 48(8): 607-610. DOI:10.3760/cma.j.issn.0529-567x.2013.08.010 |
| [17] |
Leung TW, Liu SS, Leung RCY, et al. HPV16 E2 binding sites 1 and 2 become more methylated than E2 binding site 4 during cervical carcinogenesis[J]. J Med Virol, 2015, 87(6): 1022-1033. DOI:10.1002/jmv.24129 |
| [18] |
王微, 刘建华, 王桂丽, 等. 人乳头瘤病毒16型甲基化水平与子宫颈病变程度关系的研究[J]. 中国医科大学学报, 2016, 45(4): 293-297. Wang W, Liu JH, Wang GL, et al. Correlation between severity of cervical lesions and methylation of human papilloma virus type 16 DNA[J]. J China Med Univ, 2016, 45(4): 293-297. DOI:10.12007/j.issn.0258-4646.2016.04.002 |
| [19] |
Snellenberg S, Schütze DM, Claassen-Kramer D, et al. Methylation status of the E2 binding sites of HPV16 in cervical lesions determined with the Luminex® xMAP™ system[J]. Virology, 2012, 422(2): 357-365. DOI:10.1016/j.virol.2011.11.006 |
| [20] |
Jacquin E, Baraquin A, Ramanah R, et al. Methylation of human papillomavirus Type 16 CpG sites at E2-binding site 1 (E2BS1), E2BS2, and the Sp1-binding site in cervical cancer samples as determined by high-resolution melting analysis-PCR[J]. J Clin Microbiol, 2013, 51(10): 3207-3215. DOI:10.1128/JCM.01106-13 |
| [21] |
Hublarova P, Hrstka R, Rotterova P, et al. Prediction of human papillomavirus 16 E6 gene expression and cervical intraepithelial neoplasia progression by methylation status[J]. Int J Gynecol Cancer, 2009, 19(3): 321-325. DOI:10.1111/IGC.0b013e31819d8a5c |
| [22] |
Simanaviciene V, Popendikyte V, Gudleviciene Z, et al. Different DNA methylation pattern of HPV16, HPV18 and HPV51 genomes in asymptomatic HPV infection as compared to cervical neoplasia[J]. Virology, 2015, 484: 227-233. DOI:10.1016/j.virol.2015.06.008 |
| [23] |
Gašperov NM, Sabol I, Planinić P, et al. Methylated host cell gene promoters and human papillomavirus type 16 and 18 predicting cervical lesions and cancer[J]. PLoS One, 2015, 10(6): e0129452. DOI:10.1371/journal.pone.0129452 |
| [24] |
Xi LF, Jiang MJ, Shen ZP, et al. Inverse association between methylation of human papillomavirus type 16 DNA and risk of cervical intraepithelial neoplasia grades 2 or 3[J]. PLoS One, 2011, 6(8): e23897. DOI:10.1371/journal.pone.0023897 |
| [25] |
Badal V, Chuang LSH, Tan EHH, et al. CpG methylation of human papillomavirus type 16 DNA in cervical cancer cell lines and in clinical specimens: genomic hypomethylation correlates with carcinogenic progression[J]. J Virol, 2003, 77(11): 6227-6234. DOI:10.1128/jvi.77.11.6227-6234.2003 |
| [26] |
Bowden SJ, Kalliala I, Veroniki AA, et al. The use of human papillomavirus DNA methylation in cervical intraepithelial neoplasia: a systematic review and meta-analysis[J]. eBioMedicine, 2019, 50: 246-259. DOI:10.1016/j.ebiom.2019.10.053 |
| [27] |
Dong L, Zhang L, Hu SY, et al. Risk stratification of HPV16 DNA methylation combined with E6 oncoprotein in cervical cancer screening: a 10-year prospective cohort study[J]. Clin Epigenet, 2020, 12(1): 62. DOI:10.1186/s13148-020-00853-1 |
| [28] |
Thierry F. Transcriptional regulation of the papillomavirus oncogenes by cellular and viral transcription factors in cervical carcinoma[J]. Virology, 2009, 384(2): 375-379. DOI:10.1016/j.virol.2008.11.014 |
| [29] |
Yang HJ. Aberrant DNA methylation in cervical carcinogenesis[J]. Chin J Cancer, 2013, 32(1): 42-48. DOI:10.5732/cjc.012.10033 |
| [30] |
Reuschenbach M, Huebbers CU, Prigge ES, et al. Methylation status of HPV16 E2-binding sites classifies subtypes of HPV-associated oropharyngeal cancers[J]. Cancer, 2015, 121(12): 1966-1976. DOI:10.1002/cncr.29315 |
| [31] |
Cheung JLK, Cheung TH, Yu MY, et al. Virological characteristics of cervical cancers carrying pure episomal form of HPV16 genome[J]. Gynecol Oncol, 2013, 131(2): 374-379. DOI:10.1016/j.ygyno.2013.08.026 |
| [32] |
Mix J, Saraiya M, Hallowell BD, et al. Cervical precancers and cancers attributed to HPV types by race and ethnicity: implications for vaccination, screening, and management[J]. J Natl Cancer Inst, 2022, 114(6): 845-853. DOI:10.1093/jnci/djac034 |
| [33] |
晏建国, 付海英, 沈建箴, 等. 重亚硫酸氢盐测序法检测Wnt信号通路抑制基因在急性早幼粒细胞白血病细胞中甲基化变化[J]. 中国实验血液学杂志, 2016, 24(5): 1299-1304. Yan JG, Fu HY, Shen JZ, et al. Application of bisulfite sequencing PCR in detecting the abnormal methylation of suppressor gene of Wnt signaling pathway in acute promyelocytic leukemia[J]. J Exp Hematol, 2016, 24(5): 1299-1304. |
 2022, Vol. 43
2022, Vol. 43