文章信息
- 廖春晓, 王波, 吕筠, 李立明.
- Liao Chunxiao, Wang Bo, Lyu Jun, Li Liming
- 新型冠状病毒Omicron变异株病原学及流行病学研究进展
- Progress in research of etiology and epidemiology of 2019-nCoV Omicron variant
- 中华流行病学杂志, 2022, 43(11): 1691-1698
- Chinese Journal of Epidemiology, 2022, 43(11): 1691-1698
- http://dx.doi.org/10.3760/cma.j.cn112338-20220929-00829
-
文章历史
收稿日期: 2022-09-29
2. 北京大学医学部美年公众健康研究院, 北京 100191;
3. 北京大学公众健康与重大疫情防控战略研究中心, 北京 100191
2. Peking University Health Science Center Meinian Public Health Institute, Beijing 100191, China;
3. Peking University Center for Public Health and Epidemic Preparedness & Response, Beijing 100191, China
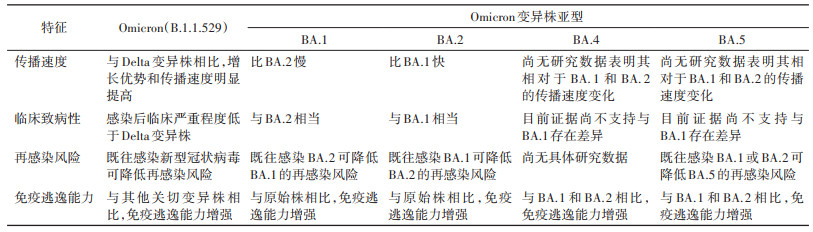
新型冠状病毒(新冠病毒)引发的新冠病毒肺炎(新冠肺炎)大流行仍在全球蔓延。截至2022年10月2日,全球累计感染人数超6亿,死亡人数超650万[1]。新冠病毒持续进化和变异,先后出现Alpha(B.1.1.7)、Beta(B.1.351)、Gamma(P.1)、Delta(B.1.617.2)及Omicron(B.1.1.529)变异株。B.1.1.529变异株最早于2021年11月在南非检出,该变异株具有大量的突变,传播速度快,WHO根据新冠病毒进化技术咨询小组的建议,将B.1.1.529列为值得关切的变异株(variants of concern,VOC),并命名为Omicron变异株[2]。Omicron持续变异,不断出现新的亚型,主要包括BA.1,BA.2,BA.3,BA.4和BA.5亚型及其后代谱系。Omicron变异株不同亚型特征的比较见表 1。本文对Omicron变异株及其亚型的病原学特点、感染的致病特征和流行病学特征及疫苗的保护效果方面进行综述,以期为Omicron变异株的科学防控提供参考。
1. 遗传变异:Omicron(B.1.1.529)变异株来自B.1.1谱系,为新冠病毒的进化支系之一。从新冠病毒基因序列的进化树来看,BA.1、BA.2和BA.3属于Omicron变异株不同的进化分支,而BA.4和BA.5则是在BA.2的基础上进化而来,由于其遗传变异的相似性,经常把BA.4和BA.5合并称为BA.4/5[3]。
新冠病毒入侵人体后,主要依靠其表面的刺突蛋白上的受体结合域(receptor binding domain,RBD)识别宿主细胞受体血管紧张素转化酶2(angiotensin converting enzyme 2,ACE2),并与之结合感染宿主细胞。刺突蛋白是唯一修饰病毒粒子表面的病毒蛋白,也是自然感染和疫苗设计的主要抗原靶标[4]。与原始株相比,BA.1在刺突蛋白中含有34个突变,这些刺突蛋白突变会引起其病毒学特征的改变,如复制能力和毒性降低,传染力增强[5]。随后的Omicron变异株亚型均保留这些突变。研究显示,BA.1和BA.2由于E484A和H655Y突变,导致利用跨膜丝氨酸蛋白酶2(TMPRSS2)的能力下降;S375F和H655Y突变降低了刺突蛋白介导的细胞融合性,影响病毒入侵能力从而导致其复制能力减弱[5]。BA.4/5与BA.2相比增加了L452R、F486V和R493Q突变,其中L452R突变是此前Delta变异株的重要突变位点,有利于病毒躲避针对RBD 2类和3类区域的抗体,是导致高免疫逃逸能力的关键突变位点之一[3]。此外,BA.4/5携带的F486V突变还有利于逃逸针对RBD的1类和2类抗体,R493Q回复突变又增加了RBD与受体结合的亲和力,进而提高了BA.4/5的适应性[3]。
2. 致病力和毒力:动物模型研究结果显示,BA.1和BA.2与原始株和VOC相比,复制能力降低、致病性减弱[6-7]。在相似的感染剂量下,感染BA.2和BA.1小鼠肺和鼻中感染性病毒的滴度和病毒载量无显著差异,且与原始株相比显著降低。当使用仓鼠模型时,在相似的感染剂量下,感染BA.1仓鼠鼻中感染性病毒的滴度和病毒载量显著高于BA.2,且BA.1在仓鼠鼻部复制效率高于BA.2;当处于“体重减轻10%,5 d后100%死亡”的原始株感染剂量下,感染BA.1和BA.2的仓鼠存活率分别为75%和100%[7]。由此可见,与BA.1相比,BA.2的致病性和毒力较低。目前关于BA.4和BA.5的动物模型研究较少。发表于bioRxiv预印本上一项研究显示,用BA.5感染仓鼠3 d后,其肺部病毒复制能力是BA.2感染仓鼠的5.7倍。一周内,感染BA.5的仓鼠在上呼吸道和肺部的症状更为严重和明显[6]。
3. 免疫逃逸:是指病原体通过不同机制逃避机体的免疫识别和攻击。Omicron变异株携带多种不同寻常的突变,特别是在刺突蛋白的RBD,导致其具有极强的免疫逃逸能力,使得疫苗和治疗性单克隆抗体的中和活性大大降低[8]。其中BA.4/5的免疫逃逸能力最强,是BA.2的4.2倍,是原始株的19.2倍[3]。BA.4/5不仅可逃逸BA.1感染诱导的中和抗体,还能够逃逸疫苗接种和BA.1或BA.2感染联合诱导的中和抗体。南非研究显示,在未接种过疫苗的情况下,即使之前感染过BA.1,对BA.4/5亚型的中和能力也仅为BA.1的1/7.5左右;接种过疫苗且感染过BA.1者,对BA.4/5的中和能力与BA.1相比下降2.6~3.2倍[9]。上述研究表明BA.4/5的免疫逃逸能力强于BA.1和BA.2,会影响既往接种疫苗或感染产生的保护效果,可能是造成其感染数量增加的主要原因。
二、Omicron变异株感染的致病特征1. 感染特点:Omicron变异株强大的免疫逃逸能力使得再感染和突破性感染(完全接种疫苗后发生的感染)风险陡增。研究显示,与Delta变异株相比,Omicron变异株再感染风险高5.41倍[10]。大型人群血清学研究提示,在Omicron变异株流行之前,全球已有43.9%的人群感染过新冠病毒[11]。通过自然感染或者疫苗接种,美国人群新冠病毒抗体血清阳性率已经达到94.7%(2021年5-12月)[12]。尽管如此,随着2022年初Omicron变异株占据主导地位,全球出现新一轮的确诊病例高峰。丹麦针对早期785例确诊的Omicron变异株感染者的分析显示,完全接种或者加强接种疫苗的比例为83.1%[13];我国浙江省163例确诊的Omicron变异株感染者(≥60岁)中,57.06%的感染者加强接种了疫苗[14]。高感染率可能与Omicron变异株免疫逃逸增强有关,同时来自疫苗接种和既往感染的保护也在减弱[12]。
2. 临床表现:人群流行病学研究表明,Omicron变异株感染后轻症和无症状感染者居多,症状持续时间短,以上呼吸道症状为主。丹麦研究中,20%自报为无症状感染[13]。我国浙江省宁波市61例确诊的Omicron变异株感染者中,54.1%为无症状感染者[15]。我国广东省珠海市38例确诊的Omicron变异株感染者中,76%为轻症或无症状感染者[16]。英国的一项纵向研究,分别纳入Delta和Omicron变异株感染者各4 990例,结果显示,Omicron变异株感染者症状持续6.87 d,明显小于Delta变异株(8.89 d);Omicron变异株感染者的嗅觉丧失比例降低(16.7% vs. 52.7%),脑雾、眩晕和发热等使人虚弱的症状也明显减少,但咽喉疼痛风险增加(70.5% vs. 60.8%)[17]。挪威基于81例确诊Omicron变异株感染者的分析显示,常见的症状依次是:咳嗽(83%)、流涕(78%)、疲劳(74%)、咽痛(72%)和头痛(68%)[18]。新冠病毒感染急性期后出现的持续症状被称为“新冠病毒长期症状”。英国的一项病例对照研究显示,Omicron变异株流行期的感染者中有4.5%的人报告了新冠病毒长期症状,而Delta变异株流行期的感染者中报告新冠病毒长期症状的比例是10.8%,进一步调整多种混杂因素后的分析显示,感染Omicron变异株后发生新冠病毒长期症状的风险比Delta变异株明显降低[19]。
3. 疾病结局:在Omicron变异株流行早期,多项流行病学研究显示,Omicron变异株引起的住院、重症和死亡风险低于先前的变异株。南非前瞻性队列研究显示,在住院患者中,与先前Delta变异株感染者相比,Omicron变异株感染者发生重症的概率显著降低70%(OR=0.3,95%CI:0.2~0.5)[20]。英国前瞻性队列研究显示,与Delta变异株相比,Omicron变异株感染导致的就诊风险降低44%,住院和死亡风险分别降低59%和69%[21]。美国回顾性队列研究显示,相比Delta变异株,Omicron变异株感染者总体入院风险下降41%,其中,入住重症监护室风险下降50%,机械通气风险下降64%,死亡风险下降79%[22]。上述研究均基于国家性的大型人群数据开展,并调整了年龄、性别、疫苗接种和新冠病毒感染史等可能与疾病严重性相关混杂因素。
对Omicron变异株不同亚型的研究显示,不同亚型感染导致的疾病严重程度无显著差异。美国研究显示,与BA.1/BA.1.1亚型相比,BA.2感染导致的疾病严重程度无显著差异[22]。南非研究显示,BA.4/5感染者在入院和发展成重症风险方面与BA.2类似。在病死率方面,研究发现原始株、Beta、Delta、BA.1和BA.4/5变异株的病死率分别为5.3%、6.9%、6.4%、2.5%和1.9%。与BA.1相比,BA.4/5变异株感染者病死率差异无统计学意义(HR=1.12,95%CI:0.93~1.34)[23]。感染新冠病毒后发展为重症危险因素包括男性、老年、肥胖、心血管病和呼吸系统疾病等[24-26]。我国香港地区Omicron变异株流行期间,对2021年12月31日至2022年9月28日的9 943例新冠肺炎死亡病例分析显示,死者中位年龄为86岁,91%患有长期慢性病,69%未接种过任何疫苗或接种过1剂疫苗但未满14 d,28%未接种疫苗加强针[27]。可见,未接种疫苗、老年、合并慢性病为感染Omciron变异株后重症和死亡的重要危险因素。
三、Omicron变异株感染的流行特征1. 优势毒株:2022年9月3日至10月3日,共有超10万2019-nCoV序列信息提交全球禽流感基因共享数据库(Global initiative on sharing all influenza data,GISAID),其中Omicron变异株序列占99.9%[1]。Omicron变异株首次于2021年11月在南非检出,随后取代Delta变异株持续流行演变。2022年2月,BA.2亚型迅速流行全球,成为优势毒株。BA.5亚型于2022年2月首次被南非研究者发现后,造成了感染数量的增加,截至7月已替代BA.2成为全球新的优势毒株。BA.4和BA.5早在5月已成为南非优势毒株,每日增长优势(daily growth advantage)比BA.2分别高0.08 d和0.12 d[28]。2022年9月5-11日(第36周)的GISAID数据显示,BA.5及其后代谱系占Omicron变异株序列的80.8%,BA.4及其后代谱系占7.8%,BA.2及其后代谱系占3.1%[1]。美国CDC新冠病毒监测数据显示,当前美国优势毒株仍为BA.5,但BA.5的检出比例自8月中旬到达顶峰(86.7%)后,在10月16-22日的周报中已经下降至62.2%。与此同时,BA.5亚型新的分支BQ.1、BQ.1.1和BF.7检出比例迅速上升,同期周报数据显示分别占9.4%、7.2%和6.7%,位列美国第三、第四和第五,位列第二位的为BA.4.6(占12.8%)[29]。聚焦国内,2022年2月以来,我国主要流行的毒株为BA.2及其后代谱系;5月,上海市从境外输入病例中首次检出BA.5,7月BA.5引发国内首次本土疫情;9月,呼和浩特市现首例BF.7本土新冠病毒感染者,是BF.7变异株首次在国内引起本土疫情。BF.7是Omicron变异株二代谱系,其在BA.4/5的基础上存在额外突变,免疫逃逸能力更强,其具体特性和重要性有待观察。
2. 传播特征:研究显示,Omicron变异株传染力和传播速度比原始株和其他VOC更强。基本再生数(R0)是描述病毒传染力的指标,R0越大病毒的传染力越强。病毒传染的序列间隔(serial interval)和代际间隔(generation time)会影响其传播速度,其数值越小传播速度越快。新冠病毒原始株、Delta、Omicron变异株的R0分别为2.2[30]、3.2[30]、9.5[31],表明传染力不断增强。我国研究者基于北京市2022年3月报告的78例具有明确传播链的Omicron变异株感染者信息,分析得出Omicron变异株序列间隔时间中位数为3.0 d,明显短于原始株(不同研究中位数介于4~6 d)[32]。英国研究发现Omicron变异株的指数增长率(exponential growth rate)约为0.34(95%CI:0.33~0.35)/d,感染病例的倍增时间为2 d(Delta变异株的倍增时间为5.2 d)[10]。南非研究显示,在Omicron变异株主导的疫情期间,病例的增加速度更快,该地区从首例病例出现到疫情高峰的时间为1个月,而Delta疫情时为2个月[33]。以上研究均提示相对于Delta变异株,Omicron变异株的传播速度更快。我国香港地区基于BA.1和BA.2流行期间的研究则表明了不同亚型传播速度的差异,研究发现BA.1和BA.2的平均序列间隔时间分别为3.30 d和2.72 d,BA.1的平均代际间隔时间为2.36 d[34];既往基于广州市Delta变异株流行期间的数据研究显示Delta变异株的平均代际间隔时间为2.9 d[30]。提示BA.2的传播速度较BA.1快,均明显强于Delta变异株。
3. 传染源:新冠病毒的主要传染源包括确诊患者和无症状感染者。相比其他变异株,Omicron变异株感染后潜伏期明显缩短。针对潜伏期的综述表明,原始株潜伏期为4~8 d,中位潜伏期为5.5 d;Delta变异株中位潜伏期为4 d,Omicron变异株的中位潜伏期为3 d[35]。关于恢复期感染者传染性的证据,研究认为当恢复期的感染者核酸Ct值≥35(Ct值代表人体内病毒含量的多少。Ct值的数值越大,代表病毒含量较少,传染力越弱;反之,传染力越强),样本中未能分离出病毒,没有传染性。《新型冠状病毒肺炎防控方案(第九版)》也明确了以核酸检测Ct值为判断依据进行管理和密接判定的原则[36]。总之,恢复期感染者作为传染源的意义需要根据其Ct值的大小判断。值得注意的是,目前已有来自29个国家的400多种动物感染了新冠病毒,包括宠物、野生动物和家畜等。与原始株和Delta变异株相比,Omicron变异株的宿主范围更广。除了原始株的宿主外,Omicron变异株还能与果子狸、啮齿动物、多种蝙蝠以及一些刺猬的ACE2受体结合[37]。一项对Omicron变异株起源的研究发现,Omicron变异株的祖先大致于2020年中期由人类传到家鼠,在家鼠中经历一年多的进化,于2021年底回传人类[38]。由此可见,Omicron变异株可从人类传播到动物宿主,然后再实现从动物到人类的溢出。人群大流行中,新冠病毒由人类传给动物,受感染动物作为传染源的意义,值得进一步观察。
4. 传播途径:《新型冠状病毒肺炎防控方案(第九版)》指出新冠病毒主要为经呼吸道飞沫和密切接触传播,在相对封闭的环境中可经气溶胶传播,接触被病毒污染的物品后也可能造成感染[36]。
室温下,新冠病毒传染性可在塑料或不锈钢表面保持3~4 d,而在4 ℃的情况下传染性可保持长达14 d。研究表明,新冠病毒在-20 ℃储存后,将比在4 ℃环境下更具稳定性和传染性[39]。既往我国多次出现与冷链物流相关的新冠肺炎病例,提示新冠病毒有可能通过冷链物流传播。与原始株和其他VOC相比,Omicron变异株更具稳定性,室温下可在物体表面存活更久。发表于bioRxiv预印本上的研究表明,在气温为25 ℃,相对湿度为45%~55%的条件下,Omicron变异株在塑料表面可存活193.5 h,在人体皮肤模型表面可以存活21.1 h,对于原始株则分别为56.0和8.6 h[40]。此外,在21~22 ℃实验室环境中,BA.1可在不锈钢、聚丙烯塑胶片以及玻璃等光滑表面上稳定存活超过7 d,在面巾纸、打印纸等多孔、凹凸不平的物体表面上,BA.1可保持30 min的传染性[41]。我国珠海市对Omicron变异株聚集传播案例的流行病学调查显示,进口假牙和牙膜可能是疫情的源头[16];类似地,我国北京市也报告了可能由北美邮寄文件导致的Omicron变异株聚集传播案例[42]。上述研究均提示Omicron变异株及其亚型极有可能通过非冷链污染物体的媒介进行传播。
一项模型研究揭示了Omicron变异株经气溶胶媒介传播的风险[41]。研究表明Omicron变异株感染者有更高的病毒载量,加之Omicron变异株更高的传染性,大大增加了其经气溶胶传播的风险。模拟研究显示,Omicron变异株感染者中有较高概率排出高浓度的病毒,且办公室、餐厅、歌舞厅和公共交通中的大多数情况都难以通过正确佩戴外科口罩达到对Omicron变异株的有效防护。提示气溶胶这一传播媒介在Omicron变异株传播中的重要性,值得关注和警惕。
四、针对Omicron变异株感染的疫苗Omicron变异株具有显著的免疫逃逸和前所未有的传播速度,针对原始株设计的新冠病毒疫苗对Omicron变异株感染引起的新冠肺炎的保护效果及其随时间的变化备受关注。此外,在当前感染人数不断增加的形势下,既往感染与疫苗效果的叠加效果如何也是亟待回答的问题。
1. 疫苗的免疫应答:多项研究证据显示,两剂灭活疫苗基础免疫后,针对Omicron变异株产生的体液免疫和细胞免疫应答水平较低,加强免疫可提高机体免疫应答水平,序贯加强免疫优于同源加强免疫。我国学者研究显示,在完成两剂CoronaVac接种3~5周后,针对Omicron变异株的50%蚀斑减少中和试验(PRNT50)抗体滴度均低于检测下限[43];而两剂BBIBP-CorV接种第14 d时,80%的血清样本对Omicron变异株的中和抗体滴度都低于检测下限[44]。完成第三剂CoronaVac加强免疫后,可诱导广泛而有效地针对Delta和Omicron变异株有效的体液免疫、B细胞和T细胞免疫[45]。相比同源加强免疫,使用mRNA疫苗、重组亚单位疫苗异源加强免疫后,针对Omicron变异株的中和活性更优[43-44, 46]。在灭活疫苗基础免疫后,mRNA疫苗接种明显增强针对Omicron变异株的体液免疫和细胞免疫应答,特异IgG水平是同源加强免疫的8倍,血浆针对Omicron变异株的90%蚀斑减少中和试验(PRNT90)抗体滴度水平是同源加强免疫的4倍,RBD特异性记忆B细胞数量是同源加强免疫的73倍[46]。假病毒中和试验(pVNT)中,重组亚单位疫苗加强产生的针对Omicron变异株的血清中和抗体水平是同源灭活疫苗加强的2倍,对Omicron变异株的中和抗体滴度阳性率达到了100%[44]。此外,研究显示重组蛋白疫苗第三剂与第二剂疫苗接种间隔延长至4~6个月时,与接种间隔1个月组相比,对Omicron变异株的中和抗体滴度高出约30倍,长间隔组对所有Omicron变异株血清阳性率达100%[47]。
2. 疫苗的保护效果及随时间变化:人群流行病学研究表明,现有疫苗基础免疫预防Omicron变异株感染或者发病保护效果较低,但仍可有效防止重症和死亡,接种加强针可提高对Omicron变异株保护效果。英国研究显示,两剂BNT162b2或ChAdOx1-S-(AZD1222)对有症状Omicron变异株感染的保护率低于20%,三剂疫苗接种可将保护效果提升至55%~80%[10]。类似地,南非研究者评估了Omicron变异株流行期间BNT162b2预防新冠肺炎住院的有效性,结果显示Omicron变异株流行期两剂BNT162b2预防住院的有效性为70%[48]。基于中国香港地区BA.2流行期间的研究结果表明,对20~59岁年龄组人群,两剂CoronaVac预防新冠肺炎重症/死亡的保护效果为91.7%(88.7%~94.0%);对≥60岁年龄组老年人,降至69.9%(64.4%~74.6%);三剂CoronaVac对新冠肺炎重症和死亡的保护效果为98.6%(98.0%~99.0%)[49]。
疫苗的免疫效果会随接种时间延长而减弱。荷兰大型人群病例对照研究显示,接种第三剂疫苗3个月后对BA.1和BA.2的保护效果明显下降[50]。美国CDC的数据显示,接种第三剂疫苗5个月后,预防Omicron变异株的有效性仅为19%[51]。美国食品与药品管理局通过了在新冠病毒加强疫苗中添加Omicron变异株成分的建议,并修改了两种mRNA疫苗即BNT162b2和mRNA-1273的紧急使用授权,批准针对BA.4/5与原始株的双价疫苗。经过批准的双价疫苗既包括原始株的mRNA成分(提供预防新冠肺炎的广泛保护),也包含BA.4和BA.5变异株共有的mRNA成分(提供预防Omicron变异株的更好保护)[51]。两款双价疫苗均为单一加强接种,在完成基础免疫或单价疫苗加强后2个月即可接种。单价mRNA疫苗不再被批准用于≥12岁个体的加强接种。
3. 疫苗与既往感染叠加的保护效果:既往感染新冠病毒可在一定程度上诱导机体产生针对Omicron变异株的免疫保护,联合疫苗接种可对Omicron变异株感染提供更有力的保护[50]。葡萄牙一项研究显示,既往感染原始株、Alpha、Delta、BA.1或BA.2变异株均可对BA.5感染提供一定的保护,分别为51.6%、54.8%、61.3%和75.3%[52]。Delta变异株流行期间开展的一项队列研究显示,接种BNT162b2后120 d内,既往新冠病毒感染者与未感染者突破性感染的累积发病率分别为0.15%和0.83%,既往感染可将突破性感染(指接种新冠病毒疫苗后发生的感染)风险降低72%[53]。一项血清学试验则表明,既往新冠病毒感染者接种1剂BNT162b2疫苗3~5周后,产生的针对Omicron变异株的PRNT50抗体滴度为130.0,高于未接种疫苗的既往感染者(77.8)[43]。荷兰一项病例对照显示,与任一种情况相比,既往感染叠加疫苗接种可以提供针对BA.1或BA.2变异株更高的保护效果[50]。既往免疫与疫苗接种叠加效果更好的原因可能与免疫次数有关,研究显示个体免疫的次数(包括自然感染和疫苗接种)越多,机体引发的中和抗体应答水平及广谱程度(包括针对Omicron变异株的中和抗体反应)越高[54],提供的保护效果越高[55]。
五、应对Omicron变异株流行的防控建议与原始株和其他VOC不同,Omicron变异株免疫逃逸能力更强,感染后重症和死亡发生风险较低。未完成基础免疫或加强免疫、老年人以及合并慢性病等因素是感染Omicron后死亡的重要危险因素。考虑到我国人口自然感染免疫水平低、人口基数大、人口老龄化等多种因素,应研发保护效果更好、更持久的疫苗,加大新冠病毒疫苗基础和序贯加强免疫的覆盖,提高人群免疫水平;同时关注重点人群,减少重症和死亡。在疫苗接种率未达到较高水平(比如加强接种覆盖率90%)、抗新冠病毒药物尚不能普及的情况下,仍需要维持一定强度的公共卫生防控措施[56]。Omicron变异株传播方式主要还是通过飞沫传播,因此,仍应坚持规范佩戴口罩、保持社交距离、勤洗手、多通风的疫情常态化防控措施,同时需关注经污染物体和气溶胶媒介传播的风险。
利益冲突 所有作者声明无利益冲突
作者贡献声明 廖春晓:数据收集/整理、论文撰写与修改;王波、吕筠: 论文审阅、修改建议;李立明: 酝酿和设计实验、论文审阅、修改建议,经费支持
| [1] |
World Health Orgainzation. Weekly epidemiological update on COVID-19-5-october 2022[EB/OL]. (2022-10-05)[2022-10-05]. https://www.who.int/publications/m/item/weekly-epidemiological-update-on-covid-19---5-october-2022.
|
| [2] |
World Health Orgainzation. Classification of Omicron (B. 1.1. 529): SARS-CoV-2 Variant of Concern[EB/OL]. (2021-12-01)[2022-09-21]. https://www.who.int/news/item/26-11-2021-classification-of-omicron-(b.1.1.529)-sars-cov-2-variant-of-concern.
|
| [3] |
Wang Q, Guo Y, Iketani S, et al. Antibody evasion by SARS-CoV-2 Omicron subvariants BA. 2.12. 1, BA. 4 and BA. 5[J]. Nature, 2022, 608(7923): 603-608. DOI:10.1038/s41586-022-05053-w |
| [4] |
Giovanetti M, Benedetti F, Campisi G, et al. Evolution patterns of SARS-CoV-2: Snapshot on its genome variants[J]. Biochem Biophys Res Commun, 2021, 538: 88-91. DOI:10.1016/j.bbrc.2020.10.102 |
| [5] |
Hu BJ, Chan JF, Liu H, et al. Spike mutations contributing to the altered entry preference of SARS-CoV-2 Omicron BA. 1 and BA. 2[J]. Emerg Microbes Infect, 2022, 11(1): 2275-2287. DOI:10.1080/22221751.2022.2117098 |
| [6] |
Kimura I, Yamasoba D, Tamura T, et al. Virological characteristics of the novel SARS-CoV-2 Omicron variants including BA. 2.12. 1, BA. 4 and BA. 5[J]. bioRxiv, 2022.
|
| [7] |
Uraki R, Kiso M, Iida S, et al. Characterization and antiviral susceptibility of SARS-CoV-2 Omicron BA. 2[J]. Nature, 2022, 607(7917): 119-127. DOI:10.1038/s41586-022-04856-1 |
| [8] |
Ai J, Wang X, He X, et al. Antibody evasion of SARS-CoV-2 Omicron BA. 1, BA. 1.1, BA. 2, and BA. 3 sub-lineages[J]. Cell Host Microbe, 2022, 30(8): 1077-1083.e4. DOI:10.1016/j.chom.2022.05.001 |
| [9] |
Khan K, Karim F, Ganga Y, et al. Omicron BA. 4/BA. 5 escape neutralizing immunity elicited by BA. 1 infection[J]. Nat Commun, 2022, 13(1): 4686. DOI:10.1038/s41467-022-32396-9 |
| [10] |
WHO Collaborating Centre For Infectious Disease Modelling. Report 49: Growth, population distribution and immune escape of Omicron in England[EB/OL]. (2021-12-20)[2022-10-05]. https://www.imperial.ac.uk/mrc-global-infectious-disease-analysis/covid-19/report-49-omicron.
|
| [11] |
COVID-19 Cumulative Infection Collaborators. Estimating global, regional, and national daily and cumulative infections with SARS-CoV-2 through Nov 14, 2021: a statistical analysis[J]. Lancet, 2022, 399(10344): 2351-2380. DOI:10.1016/S0140-6736(22)00484-6 |
| [12] |
Jones JM, Opsomer JD, Stone M, et al. Updated US Infection- and Vaccine-Induced SARS-CoV-2 Seroprevalence Estimates Based on Blood Donations, July 2020-December 2021[J]. JAMA, 2022, 328(3): 298-301. DOI:10.1001/jama.2022.9745 |
| [13] |
Espenhain L, Funk T, Overvad M, et al. Epidemiological characterisation of the first 785 SARS-CoV-2 Omicron variant cases in Denmark, December 2021[J]. Euro Surveill, 2021, 26(50): 2101146. DOI:10.2807/1560-7917.ES.2021.26.50.2101146 |
| [14] |
刘碧瑶, 王臻, 戚小华, 等. 奥密克戎变异株流行期浙江省≥60岁新型冠状病毒肺炎本土感染者特征分析[J/OL]. 中国公共卫生, 2022. DOI: 10.11847/zgggws1139326. Liu BY, Wang Z, Qi XH, et al. Characteristics of local COVID-19 cases aged ≥ 60 years in Zhejiang province during epidemic period dominantly due to SARS-CoV-2 Omicron variant infection[J/OL]. Chin J Public Health, 2022. DOI: 10.11847/zgggws1139326. |
| [15] |
褚衍茹, 张栋梁, 陈奕, 等. 基于新冠病毒Delta及Omicron变异株流行特征变化对疫情防控措施优化分析[J/OL]. 中国公共卫生, 2022. DOI: 10.11847/zgggws1139538. Chu YR, Zhang DL, Chen Y, et al. Optimization of COVID-19 prevention and control measures based on prevalence characteristics of SARS-CoV-2 Delta and Omicron variants: an analysis on surveillance data of Ningbo city[J/OL]. Chin J Public Health, 2022. DOI: 10.11847/zgggws1139538. |
| [16] |
Ruan F, Zhang X, Xiao S, et al. An Outbreak of the SARS-CoV-2 Omicron Variant BA. 1-Zhuhai City, Guangdong Province, China, January 13, 2022[J]. China CDC Wkly, 2022, 4(30): 669-671. DOI:10.46234/ccdcw2022.032 |
| [17] |
Menni C, Valdes AM, Polidori L, et al. Symptom prevalence, duration, and risk of hospital admission in individuals infected with SARS-CoV-2 during periods of omicron and delta variant dominance: a prospective observational study from the ZOE COVID Study[J]. Lancet, 2022, 399(10335): 1618-1624. DOI:10.1016/S0140-6736(22)00327-0 |
| [18] |
Brandal LT, Macdonald E, Veneti L, et al. Outbreak caused by the SARS-CoV-2 Omicron variant in Norway, November to December 2021[J]. Euro Surveill, 2021, 26(50): 2101147. DOI:10.2807/1560-7917.ES.2021.26.50.2101147 |
| [19] |
Antonelli M, Pujol JC, Spector TD, et al. Risk of long COVID associated with delta versus omicron variants of SARS-CoV-2[J]. Lancet, 2022, 399(10343): 2263-2264. DOI:10.1016/S0140-6736(22)00941-2 |
| [20] |
Wolter N, Jassat W, Walaza S, et al. Early assessment of the clinical severity of the SARS-CoV-2 omicron variant in South Africa: a data linkage study[J]. Lancet, 2022, 399(10323): 437-446. DOI:10.1016/S0140-6736(22)00017-4 |
| [21] |
Nyberg T, Ferguson NM, Nash SG, et al. Comparative analysis of the risks of hospitalisation and death associated with SARS-CoV-2 omicron (B. 1.1. 529) and delta (B. 1.617. 2) variants in England: a cohort study[J]. Lancet, 2022, 399(10332): 1303-1312. DOI:10.1016/S0140-6736(22)00462-7 |
| [22] |
Lewnard JA, Hong VX, Patel MM, et al. Clinical outcomes associated with SARS-CoV-2 Omicron (B. 1.1. 529) variant and BA. 1/BA. 1.1 or BA. 2 subvariant infection in southern California[J]. Nat Med, 2022, 28(9): 1933-1943. DOI:10.1038/s41591-022-01887-z |
| [23] |
Davies M, Morden E, Rosseau P, et al. Outcomes of laboratory-confirmed SARS-CoV-2 infection during resurgence driven by Omicron lineages BA. 4 and BA. 5 compared with previous waves in the Western Cape Province, South Africa[J]. medRxiv, 2022, 2022-2026. DOI:10.1101/2022.06.28.22276983 |
| [24] |
Atkins JL, Masoli J, Delgado J, et al. Preexisting Comorbidities Predicting COVID-19 and Mortality in the UK Biobank Community Cohort[J]. J Gerontol A Biol Sci Med Sci, 2020, 75(11): 2224-2230. DOI:10.1093/gerona/glaa183 |
| [25] |
Cummings MJ, Baldwin MR, Abrams D, et al. Epidemiology, clinical course, and outcomes of critically ill adults with COVID-19 in New York City: a prospective cohort study[J]. Lancet, 2020, 395(10239): 1763-1770. DOI:10.1016/S0140-6736(20)31189-2 |
| [26] |
Zhou F, Yu T, Du R, et al. Clinical course and risk factors for mortality of adult inpatients with COVID-19 in Wuhan, China: a retrospective cohort study[J]. Lancet, 2020, 395(10229): 1054-1062. DOI:10.1016/S0140-6736(20)30566-3 |
| [27] |
Centre for Health Protection of the Department of Health. Provisional Data Analysis on COVID-19 Reported Death Cases[EB/OL]. (2022-09-29)[2022-10-05]. https://www.covidvaccine.gov.hk/pdf/death_analysis.pdf.
|
| [28] |
Tegally H, Moir M, Everatt J, et al. Emergence of SARS-CoV-2 Omicron lineages BA. 4 and BA. 5 in South Africa[J]. Nat Med, 2022, 28(9): 1785-1790. DOI:10.1038/s41591-022-01911-2 |
| [29] |
Centers for disease control and prevention. COVID data trackern[EB/OL]. (2022-10-22)[2022-10-22]. https://covid.cdc.gov/covid-data-tracker/#variant-proportions.
|
| [30] |
Zhang M, Xiao J, Deng A, et al. Transmission Dynamics of an Outbreak of the COVID-19 Delta Variant B. 1.617. 2-Guangdong Province, China, May-June 2021[J]. China CDC Wkly, 2021, 3(27): 584-586. DOI:10.46234/ccdcw2021.148 |
| [31] |
Liu Y, Rocklöv J. The effective reproductive number of the Omicron variant of SARS-CoV-2 is several times relative to Delta[J/OL]. J Travel Med, 2022, 29(3): taac037. DOI: 10.1093/jtm/taac037.
|
| [32] |
杜婧, 王佳敏, 王晶, 等. 北京市新型冠状病毒Omicron变异株的传播力研究[J]. 中华流行病学杂志, 2022, 43(9): 1364-1369. Du J, Wang JM, Wang J, et al. Study of transmissibility of 2019-nCoV Omicron variant in Beijing[J]. Chin J Epidemiol, 2022, 43(9): 1364-1369. DOI:10.3760/cma.j.cn112338-20220410-00274 |
| [33] |
Madhi SA, Kwatra G, Myers JE, et al. Population Immunity and Covid-19 Severity with Omicron Variant in South Africa[J]. N Engl J Med, 2022, 386(14): 1314-1326. DOI:10.1056/NEJMoa2119658 |
| [34] |
Mefsin Y, Chen D, Bond HS, et al. Epidemiology of infections with SARS-CoV-2 Omicron BA. 2 variant in Hong Kong, January-March 2022[J]. Emerg Infect Dis, 2022, 28(9): 1856-1858. DOI:10.3201/eid2809.220613 |
| [35] |
吴俣, 刘民. 不同新型冠状病毒变异株感染的COVID-19潜伏期研究[J]. 中国全科医学, 2022, 25(11): 1309-1313. Wu Y, Liu M. The Incubation Period of COVID-19 Caused by Different SARS-CoV-2 Variants[J]. Chinese General Practice, 2022, 25(11): 1309-1313. DOI:10.12114/j.issn.1007-9572.2022.0078 |
| [36] |
疾病预防控制局. 关于印发新型冠状病毒肺炎防控方案(第九版)的通知[EB/OL]. (2022-06-28)[2022-10-05]. http://www.nhc.gov.cn/jkj/s3577/202206/de224e7784fe4007b7189c1f1c9d5e85.shtml.
|
| [37] |
Li L, Han P, Huang B, et al. Broader-species receptor binding and structural bases of Omicron SARS-CoV-2 to both mouse and palm-civet ACE2s[J]. Cell Discov, 2022, 8(1): 65. DOI:10.1038/s41421-022-00431-0 |
| [38] |
Wei C, Shan KJ, Wang W, et al. Evidence for a mouse origin of the SARS-CoV-2 Omicron variant[J]. J Genet Genomics, 2021, 48(12): 1111-1121. DOI:10.1016/j.jgg.2021.12.003 |
| [39] |
Li YY, Liu HX, Xia W, et al. Cold chain logistics: a possible mode of SARS-CoV-2 transmission?[J]. BMJ, 2021, 375: e66129. DOI:10.1136/bmj-2021-066129 |
| [40] |
Hirose R, Itoh Y, Ikegaya H, et al. Differences in environmental stability among SARS-CoV-2 variants of concern: Omicron has higher stability[J]. bioRxiv, 2022.
|
| [41] |
Riediker M, Briceno-Ayala L, Ichihara G, et al. Higher viral load and infectivity increase risk of aerosol transmission for Delta and Omicron variants of SARS-CoV-2[J]. Swiss Med Wkly, 2022, 152: w30133. DOI:10.4414/smw.2022.w30133 |
| [42] |
Zhang D, Wu S, Ren Z, et al. A Local Cluster of Omicron Variant COVID-19 Likely Caused by Internationally Mailed Document - Beijing Municipality, China, January 2022[J]. China CDC Wkly, 2022, 4(14): 302-304. DOI:10.46234/ccdcw2022.031 |
| [43] |
Cheng S, Mok C, Leung Y, et al. Neutralizing antibodies against the SARS-CoV-2 Omicron variant following homologous and heterologous CoronaVac or BNT162b2 vaccination[J]. Nat Med, 2022, 28(3): 486-489. DOI:10.1038/s41591-022-01704-7 |
| [44] |
Ai J, Zhang H, Zhang Y, et al. Omicron variant showed lower neutralizing sensitivity than other SARS-CoV-2 variants to immune sera elicited by vaccines after boost[J]. Emerg Microbes Infect, 2022, 11(1): 337-343. DOI:10.1080/22221751.2021.2022440 |
| [45] |
Chen Y, Chen L, Yin S, et al. The Third dose of CoronVac vaccination induces broad and potent adaptive immune responses that recognize SARS-CoV-2 Delta and Omicron variants[J]. Emerg Microbes Infect, 2022, 11(1): 1524-1536. DOI:10.1080/22221751.2022.2081614 |
| [46] |
Zuo F, Abolhassani H, Du L, et al. Heterologous immunization with inactivated vaccine followed by mRNA-booster elicits strong immunity against SARS-CoV-2 Omicron variant[J]. Nat Commun, 2022, 13(1): 2670. DOI:10.1038/s41467-022-30340-5 |
| [47] |
Zhao X, Zhang R, Qiao S, et al. Omicron SARS-CoV-2 Neutralization from Inactivated and ZF2001 Vaccines[J]. N Engl J Med, 2022, 387(3): 277-280. DOI:10.1056/NEJMc2206900 |
| [48] |
Collie S, Champion J, Moultrie H, et al. Effectiveness of BNT162b2 Vaccine against Omicron Variant in South Africa[J]. N Engl J Med, 2022, 386(5): 494-496. DOI:10.1056/NEJMc2119270 |
| [49] |
Mcmenamin ME, Nealon J, Lin Y, et al. Vaccine effectiveness of one, two, and three doses of BNT162b2 and CoronaVac against COVID-19 in Hong Kong: a population-based observational study[J]. Lancet Infect Dis, 2022, 22(10): 1435-1443. DOI:10.1016/S1473-3099(22)00345-0 |
| [50] |
Andeweg SP, de Gier B, Eggink D, et al. Protection of COVID-19 vaccination and previous infection against Omicron BA. 1, BA. 2 and Delta SARS-CoV-2 infections[J]. Nat Commun, 2022, 13(1): 4738. DOI:10.1038/s41467-022-31838-8 |
| [51] |
Administration USFD. Coronavirus (COVID-19) Update: FDA Authorizes Moderna, Pfizer-BioNTech Bivalent COVID-19 Vaccines for Use as a BoosterDose[Z]. 2002.
|
| [52] |
Malato J, Ribeiro RM, Leite PP, et al. Risk of BA. 5 Infection among Persons Exposed to Previous SARS-CoV-2 Variants[J]. N Engl J Med, 2022, 387(10): 953-954. DOI:10.1056/NEJMc2209479 |
| [53] |
Abu-Raddad LJ, Chemaitelly H, Ayoub HH, et al. Association of Prior SARS-CoV-2 Infection With Risk of Breakthrough Infection Following mRNA Vaccination in Qatar[J]. JAMA, 2021, 326(19): 1930-1939. DOI:10.1001/jama.2021.19623 |
| [54] |
Walls AC, Sprouse KR, Bowen JE, et al. SARS-CoV-2 breakthrough infections elicit potent, broad, and durable neutralizing antibody responses[J]. Cell, 2022, 185(5): 872-880. DOI:10.1016/j.cell.2022.01.011 |
| [55] |
Khoury DS, Cromer D, Reynaldi A, et al. Neutralizing antibody levels are highly predictive of immune protection from symptomatic SARS-CoV-2 infection[J]. Nat Med, 2021, 27(7): 1205-1211. DOI:10.1038/s41591-021-01377-8 |
| [56] |
Cai J, Deng X, Yang J, et al. Modeling transmission of SARS-CoV-2 Omicron in China[J]. Nat Med, 2022, 28(7): 1468-1475. DOI:10.1038/s41591-022-01855-7 |
 2022, Vol. 43
2022, Vol. 43